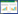扩展功能
文章信息
- 郑永钦, 郑正, 陈燕玲, 黄洪霞, 许美容
- ZHENG Yong-Qin, ZHENG Zheng, CHEN Yan-Ling, HUANG Hong-Xia, XU Mei-Rong
- 柑橘黄龙病菌内参基因的筛选与评估
- Screening and evaluation of reference genes for Candidatus Liberibacter asiaticus
- 微生物学通报, 2019, 46(11): 2985-2995
- Microbiology China, 2019, 46(11): 2985-2995
- DOI: 10.13344/j.microbiol.china.180947
-
文章历史
- 收稿日期: 2018-11-23
- 接受日期: 2019-04-02
- 网络首发日期: 2019-04-19
柑橘黄龙病(Citrus Huanglongbing,HLB)是柑橘生产上最具毁灭性的病害之一,目前普遍存在于东南亚、非洲南部以及美洲地区的柑橘主产区[1],严重制约了世界柑橘产业的发展。在我国,该病害最早在广东潮汕地区发现,并逐渐蔓延至国内其它柑橘种植区[2]。感染黄龙病的柑橘植株表现出叶片斑驳、黄化、缺素状等不同症状[3],寿命变短且严重减产[4]。目前普遍认为黄龙病是由限于韧皮部筛管细胞的革兰氏阴性细菌引起,该细菌属于α-变形菌纲(Proteobacteriacea)的候选韧皮部杆菌属(“Candidatus Liberibacter spp.”)[5],根据热敏性和地区可分为耐热型亚洲种(“Candidatus Liberibacter asiaticus”,CLas)、在巴西发生的美洲种(“Candidatus Liberibacter americanus”,CLam)和在南非发生的热敏型非洲种(“Candidatus Liberibacter africanus”,CLaf)[6],在我国仅检测到亚洲种病原[7-9]。
柑橘黄龙病菌由亚洲柑橘木虱(Diaphorina citri)和非洲柑橘木虱(Trioza erytreae)传播[9],研究表明柑橘黄龙病菌中与转录调节、运输系统、代谢途径、分泌系统和抗逆性相关的基因在昆虫寄主与植物寄主中的表达存在差异[10],其中过氧化物酶、Sec分泌系统效应子(Sec-delivered effectors,SDEs)编码基因在植物寄主中表达上调[11-13],为病原菌检测及致病机理研究奠定基础。由于韧皮部杆菌是难培养菌,至今未能实现人工培养,因此对该病原基因水平上的研究较少。不同寄主植物感染黄龙病后的发病进程和病害症状存在差异[3],然而目前未有黄龙病菌基因在不同植物寄主上及不同侵染时期表达差异的研究。此外,用于研究黄龙病菌的内参基因仅有16S rRNA[10]和gyrA[14],未有系统地筛选及评估黄龙病菌内参基因的研究报道。通过实时荧光定量PCR (Real-time quantitative PCR,RT-qPCR)方法对mRNA表达量进行相对定量是基因表达研究的常用方法[15],相对定量分析中需要内参基因对表达量数据进行标准化以减少实验误差[16],常用的内参基因包括18S rRNA、β-Actin和GAPDH[17],然而RT-qPCR最低标准(Minimum information for publication of real-time quantitative PCR experiment,MIQE)表明没有适用于所有实验条件的内参基因[18]。因此,在研究基因表达差异之前,筛选特定物种及试验条件下表达稳定的内参基因十分重要[19]。近年,首个RT-qPCR内参基因知识库(http://icg.big.ac.cn)收录了209个物种(包含200个真核生物和9个原核生物)已验证的内参基因[20],通过Gene ontology (GO)富集分析表明细菌中功能类别属于DNA复制、转录、运输的基因中存在较多已验证的内参基因,同时统计表明转录产物为DNA旋转酶、RNA聚合酶、细胞分裂蛋白、前蛋白转位酶的基因作为内参基因的可能性较大[21]。
本研究为改变柑橘黄龙病菌内参基因单一的现状,根据已报道的柑橘黄龙病菌CLas、CLam及Claf的全基因组序列,按照功能类别[21]选择了23个候选内参基因,并设计了这些基因针对CLas A4菌株[22]的特异性引物。利用RT-qPCR技术的相对定量方法分析不同候选内参基因在柑橘黄龙病菌CLas A4不同侵染时期及不同种寄主上的差异表达情况。采用Ct值标准差[23]、geNorm[24]、NormFinder[25]及RefFinder[26]方法对各基因表达稳定性进行评价,并利用内参基因研究CLas诱导植株淀粉积累及叶片黄化的致病基因LasΔ5313[27]在不同植物寄主及不同侵染时期的表达水平。本研究通过选择合适的内参基因并进行评估,为今后黄龙病菌分子检测、基因表达和致病机理研究奠定基础,同时为其他韧皮部杆菌属菌种选择内参基因提供参考。
1 材料与方法 1.1 材料 1.1.1 病原材料采用病芽嫁接法将CLas接种到4种柑橘属植株上:十月橘(Citrus reticulata Blanco cv. Shiyue Ju)、柚(Citrus maxima)、年橘(Citrus reticulata Blanco cv. Nian Ju)及柠檬[Citrus limon (L.) Burm. F.],每个种3个生物学重复。病芽材料来自本实验室同一带黄龙病的植株,柑橘材料于防虫网室内自然条件下培养,常规水、肥、药剂管理。在嫁接后3、6、9、12及18个月从每个植株上分别随机采集带有典型黄龙病症状的叶片作为待检样品。阴性对照样品来自4种健康柑橘植株叶片。剪取每个样品的一部分叶片用液氮速冻后,于−80 ℃保存备用;另一部分于4 ℃保存备用。
1.1.2 主要试剂和仪器E.Z.N.A.® High Performance (HP) Plant DNA Midi Kit和E.Z.N.A.® Plant RNA Kit,Omega Bio-Tek公司;TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix,TransGen Biotech公司;Bestar® Sybr Green qPCR Master Mix,DBI® Bioscience公司。NanoDropTM One微量紫外-可见光分光光度计,Thermo Fisher Scientific公司;CFX ConnectTM Real-Time PCR Detection System,Bio-Rad公司。
1.2 DNA提取及“Candidatus Liberibacter asiaticus”的Real-time quantitative PCR检测剪取4 ℃保存的叶片中脉0.1 g,提取植物组织DNA。用1%的琼脂糖凝胶电泳验证所提取DNA质量,经检测合格的DNA于−20 ℃中保存备用。每份DNA样品中CLas含量的检测采用基于16S rRNA基因序列引物HLBas/HLBr[28]的RT-qPCR方法。参照阳性对照及阴性对照的Ct值,检测结果Ct < 32的样品则确定为CLas阳性,Ct > 36的样品为CLas阴性。
1.3 总RNA的提取和cDNA第一链的合成分别取每个样品的叶中脉80–100 mg,在液氮中研磨成粉末,提取各样品的植物组织总RNA。采用1%琼脂糖凝胶电泳检测RNA的完整性,并测定浓度和纯度。根据所测浓度将所有RNA样品的浓度均一化至25 ng/μL。反转录合成第一链cDNA并去除DNA污染,反转录产物于−20 ℃保存。
1.4 引物设计及合成在NCBI的Genome Database中搜索“Candidatus Liberibacter asiaticus”,选择A4菌株(CP010804)的蛋白质信息。根据常用内参基因的功能注释[21],选择其中的管家基因作为候选内参基因(表 1),其中包括已使用但未验证的内参基因gyrA[14]。根据选出的候选内参基因序列,在Primer3 (http://primer3.ut.ee/)中设计引物。引物设计选择Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)结果中特异于CLas的序列,产物长度设置为80–100 bp,熔解温度设置为58.0−64.0 ℃[29],其余均为默认设置。每对引物通过NCBI Primer- BLAST (www.ncbi.nlm.nih.gov/tools/primer-blast/)检验特异性,“Blast expect (E) value”设置为“1 000”,“Blast word size”设置为“16”。引物由生工生物工程(上海)股份有限公司合成,引物序列及相关信息如表 1所示。表 1中同时列出基于16S rRNA基因设计的CLas特异引物HLBas/HLBr[28]。
| 基因 Gene symbol |
功能类别 Functional category |
引物序列 Primers sequence (5′→3′) |
序列号 Accession numbers |
| 16S rRNA | Ribosome | TCGAGCGCGTATGCAATACGCT ACCTTTTTCTACGGGATAACGC | CD16_03520 |
| gyrA | DNA replication | CACGAAAAGCAACAAAAGCC AGCGTCAATATGGTTGCTTTGA | CD16_RS00270 |
| recA | DNA replication | ACAAAAGCACACGTCCCTCCG CATCGTTGAAATTTATGGACCA | CD16_RS00315 |
| rpoA | Transcription | CACCACGCAACGAAGACATT ATAGCAGAACCTCTTCCCCG | CD16_RS00530 |
| rpoB1 | Transcription | ACTCTGGACCCGCAACAATA GGTAAACAGGGGCGCTTTAG | CD16_RS00065 |
| rpoB2 | Transcription | TCCTTCCACCGAACGAATCA AGTTCAGGGGCTGCTATTGT | CD16_RS00070 |
| rpoD | Transcription | ACGGACGAATTACTTGCACT GTGGTTGACGGCGATGATTT | CD16_RS00795 |
| rpoO | Transcription | ACAAGGCATCTATCCCAGGG AGTACCACTTGCAATCTCGC | CD16_RS04090 |
| rho | Transcription | TCCGTACAAGGGGAAGTGAT CTACAAGACACTTAGCTTTCGC | CD16_RS01945 |
| gap | Energy metabolism | GTCAAGACGCGATGATGTGA ACCTTCCATGTACCGAATCGT | CD16_RS02710 |
| ftsA | Cell division | TCATCATCGACAAGAGTGGCT GCAATTAATCGGGCGCATCT | CD16_RS05250 |
| ftsW | Cell division | GGTTGTGCAATGAGTAACGC GAACAAATTCGCCATCCAGAA | CD16_RS04885 |
| ftsZ | Cell division | CTTCCACCGGTAATTTTGGGG AGATAGCTCTCTGACAACGCA | CD16_RS05245 |
| zapE | Cell division | TGGCTTTGGAATTTAAGGGGC CCTTGACCAACATCTCCGTG | CD16_RS04635 |
| cdp1 | Cell division | TTAACTCCGAAAGAAACCTCTCT AGTGGGTGATTATGGTTTGAAAG | CD16_RS02640 |
| cdp2 | Cell division | ACGTTACCAATGATTCTGACCT CCATTGGAGGACATACACGG | CD16_RS05255 |
| secY | Transport | GTCCCACTTAATGACACAGGC GGCGCTGGATATCTCGTTTG | CD16_RS00550 |
| secG | Transport | CCAAAGCGATCGAAGTTGCA TACGTCAGTTCGCAGTACCG | CD16_RS00350 |
| secA | Transport | TTAGCAGCCGTTCTTCCTGT GTGTTGGAATCTCTGCGAGC | CD16_RS00990 |
| yajC | Transport | ACAATTCCTGCTGCTGTGAC GCTTATTCGGCCACAACGTC | CD16_RS04975 |
| gmk1 | Phosphorylation/Nucleotide metabolism | ACCACCATCGCTAAACAGGT ATTGCTTTTCATCGACGCGG | CD16_RS00015 |
| gmk2 | Phosphorylation/Nucleotide metabolism | ATTTTGCCTCCGACAATGCA AATCGTAATTGCACCTTTTCCTT | CD16_RS03955 |
| gmk3 | Phosphorylation/Nucleotide metabolism | TGATTGGGGCATCAGGAGTT TGTAACTCCCACAGGCATGA | CD16_RS05340 |
| adk | Phosphorylation/Nucleotide metabolism | CTCTGCACGCAACATATCCC GGATCTGGAAAAGGAACGCA | CD16_RS00545 |
采用SYBR Green染料法进行实时荧光定量PCR。PCR反应体系(20 μL):SYBR Green qPCR Master Mix 10 μL,1×10−5 mol/L正向引物0.5 μL,1×10−5 mol/L反向引物0.5 μL,cDNA模板(25 ng/μL) 1 μL,ddH2O 8 μL。PCR反应条件:95 ℃ 2 min;95 ℃ 10 s,60 ℃ 30 s,72 ℃ 30 s,40个循环;并在每个72 ℃步骤结束时捕获荧光信号。检测CLas时采用DNA模板1 μL,验证引物特异性时在原程序后添加熔解曲线:70−95 ℃,每隔0.5 ℃停留10 s,检测荧光强度变化。使用Bio-Rad CFX Manager 2.1软件进行数据分析,基线和阈值设置为软件默认。
1.6 数据处理与分析将cDNA样品依照顺序稀释6个梯度,作为RT-qPCR的模板。依次用设计的候选内参基因引物进行扩增,采用Bio-Rad CFX Manager 2.1软件绘制各基因的标准曲线,从而得到引物的斜率(k)、扩增效率(E)、决定系数(R2)等系数。
由于所提取的RNA为携带病原核酸的植物组织总RNA,且每个样品中黄龙病菌RNA的含量不同。因此对黄龙病菌RNA含量进行相对定量时需采用参照基因16S rRNA基因校正表达量数据[10],因此16S rRNA不作为候选内参基因。根据Ct值,通过Excel软件计算相对表达量:Q=(1+E)ΔCt (ΔCt=某内参基因在样品中最低Ct值–某内参基因在各样品中Ct值)[30],最低Ct值样品的相对表达量即为1。
各基因的转录水平分析采用Duncan’s multiple range test (DMRT)法多重比较法。候选内参基因的表达稳定性分析采用Statistical Product and Service Solutions (SPSS)、geNorm、NormFinder及RefFinder软件。其中导入geNorm、NormFinder的数据均为各样品Q值,导入SPSS、RefFinder的数据为各样品Ct值。基于软件分析结果综合评价各候选内参基因,筛选合适的内参基因。目的基因LasΔ5313的相对表达量分析采用2−ΔΔCt法[31]。
2 结果与分析 2.1 样品检测、引物特异性验证及扩增效率健康的柑橘植株在嫁接带CLas芽后,随着时间增加,其中的CLas浓度呈逐渐增长的趋势(表 2),但该规律在每一棵植株上表现不完全一致。因此,根据RT-qPCR结果,筛选出Ct值小于32的样品作为CLas阳性样品;而将健康植株叶片作为CLas阴性对照样品。
| 样品 Sample |
健康 Healthy |
嫁接3个月 Grafted for 3 months |
嫁接6个月 Grafted for 6 months |
嫁接9个月 Grafted for 9 months |
嫁接12个月 Grafted for 12 months |
嫁接18个月 Grafted for 18 months |
| 平均Ct值 Mean Ct |
37.08±0.88 | 22.83±1.60 | 23.06±4.65 | 22.84±1.49 | 24.42±0.90 | 21.62±0.60 |
由于CLas不能纯培养,所提取的核酸背景复杂,包含寄主植物及内生菌的核酸。为初步确定候选内参基因引物的特异性,采用RT-qPCR方法验证CLas阴性及阳性的不同种样品的cDNA。结果显示,有16个候选内参基因在4种柑橘属叶片的CLas阴性样品中无扩增片段,在CLas阳性样品中可扩增出目的片段,特异性良好。而基因rpoA、rpoB2、rho、cdp1、cdp2、secG、adk的引物扩增出目的产物外的基因片段,其原因与核酸背景复杂、RT-qPCR退火温度与时间设置等有关。为进一步确定引物特异性,分析所有引物的熔解曲线:除yajC、gmk3外,14个候选内参基因的熔解曲线都显示单一信号峰(图 1),说明各基因引物均扩增出单一片段。对14个基因的RT-qPCR产物经1%琼脂糖凝胶电泳进行检测,结果显示条带单一且与预期片段大小一致,无非特异性扩增和引物二聚体现象,因此引物具有良好特异性(图 2)。16S rRNA基因引物HLBas/HLBr特异性已验证[28]。
|
| 图 1 柑橘黄龙病菌候选内参基因的RT-qPCR熔解曲线 Figure 1 The melting curve of the real-time PCR products of "Candidatus Liberibacter asiaticus" candidate reference genes Note: A: gyrA (Tm: 79.00); B: recA (Tm: 78.00); C: rpoB1 (Tm: 79.67); D: rpoD (Tm: 80.00); E: rpoO (Tm: 82.00); F: gap (Tm: 78.00); G: ftsA (Tm: 80.50); H: ftsW (Tm: 78.00); I: ftsZ (Tm: 76.50); J: zapE (Tm: 77.00); K: secY (Tm: 80.00); L: secA (Tm: 79.83); M: gmk1 (Tm: 83.00); N: gmk2 (Tm: 77.50). |
|
|

|
| 图 2 候选内参基因RT-qPCR产物的琼脂糖凝胶电泳 Figure 2 Agarose gel electrophoresis result of RT-qPCR products of the candidate reference genes Note: 1: gyrA; 2: recA; 3: rpoB1; 4: rpoD; 5: rpoO; 6: gap; 7: ftsZ; 8: zapE; 9: ftsW; 10: ftsA; 11: secY; 12: secA; 13: gmk1; 14: gmk2. |
|
|
梯度稀释嫁接病芽后18个月的同一植株的cDNA模板以明确各内参基因的扩增效率(表 3)。结果显示,各基因线性决定系数(R2)的变化范围为0.954–0.999,扩增效率范围为85.7%−115.8%,可进行后续RT-qPCR实验。
| 基因 Gene symbol |
扩增长度 Amplicon length (bp) |
斜率 Slope |
相关系数 Correlation (R 2) |
扩增效率 PCR efficiency (%) |
| gyrA | 80 | -3.312 | 0.999 | 100.4 |
| recA | 100 | -3.721 | 0.996 | 85.7 |
| rpoB1 | 85 | -3.597 | 0.999 | 89.7 |
| rpoD | 83 | -3.074 | 0.990 | 111.5 |
| rpoO | 93 | -3.596 | 0.994 | 89.4 |
| gap | 98 | -3.513 | 0.996 | 92.6 |
| ftsA | 83 | -3.633 | 0.999 | 88.5 |
| ftsW | 86 | -3.485 | 0.999 | 93.6 |
| ftsZ | 80 | -3.237 | 0.992 | 103.7 |
| zapE | 80 | -3.334 | 0.954 | 99.5 |
| secY | 83 | -3.422 | 0.999 | 96.0 |
| secA | 98 | -2.997 | 0.999 | 115.8 |
| gmk1 | 94 | -3.378 | 0.981 | 97.7 |
| gmk2 | 92 | -3.652 | 0.983 | 87.9 |
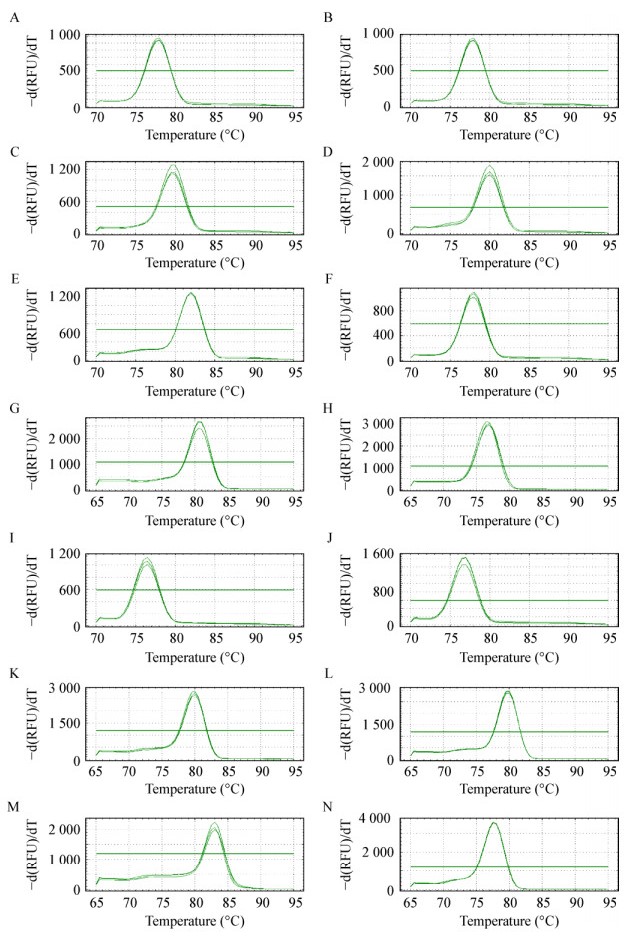
根据每个基因在所有样品中的Ct值平均值进行比较,利用SPSS进行DMRT法多重比较(图 3)。结果显示,各基因Ct值范围为17.00–22.35,16S rRNA基因的表达丰度显著高于候选内参基因(P=0.027 < 0.05)。此外,候选内参基因中gmk2表达量最高(17.74–22.30),recA表达量最低(19.82–24.88),而14个候选内参基因表达水平无显著差异(P=0.078 > 0.05),Ct值范围为20.02–22.35。secA的Ct变化范围最小(18.28–22.02),gmk1的Ct值变化范围最大(17.86–23.28),每个基因在样品中表达均存在差异,说明需要对内参基因进行筛选。
|
| 图 3 候选内参基因的表达分析(SPSS) Figure 3 Expression analysis of candidate reference genes (SPSS) 注:同行相同小写字母者,表示差异不显著(Duncan’s法,P > 0.05). Note: The same lower case letter indicate that the difference isn't significant (Duncan's method, P > 0.05). |
|
|
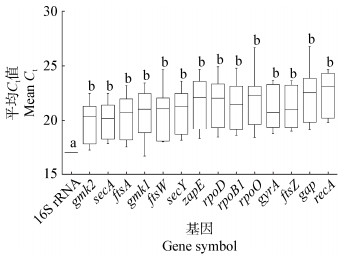
为确定候选内参基因的表达稳定性,采用SPSS、geNorm、NormFinder及RefFinder对RT-qPCR数据进行分析并综合评价。利用geNorm软件分析候选内参基因在不同样品中的表达稳定性(M),M值越小,稳定性越高,以M=1.5为取舍值。根据geNorm软件分析结果,不同候选内参基因表达稳定性从高到低顺序为gyrA (0.349)= ftsZ (0.349) > zapE (0.395) > rpoD (0.431) > secA (0.483) > rpoB1 (0.523) > gap (0.558) > recA (0.586) > ftsA (0.624) > secY (0.658) > ftsW (0.682) > gmk2 (0.698) > rpoO (0.720) > gmk1 (0.789) (图 4A),14个候选内参基因均较稳定。NormFinder软件以稳定因子表达稳定性,其分析结果与geNorm相似,但NormFinder软件无取舍值,稳定性从高到低顺序为ftsZ > rpoB1 > gap > ftsA > gyrA > rpoD > gmk2 > secA > zapE > secY > ftsW > rpoO > recA > gmk1 (图 4B)。利用SPSS统计各内参基因对所有样品扩增值的标准差,标准差越低则表达稳定性越强,稳定性顺序为secA > ftsZ > gyrA > secY > ftsA > gmk2 > rpoB1 > ftsW > rpoD > zapE > recA > gap > rpoO > gmk1 (图 4C)。由各软件的分析结果可看出排序存在差异,因此利用RefFinder在线工具(http://150.216.56.64/referencegene.php)结合geNorm、NormFinder、标准差分析方法的结果对内参基因进行综合排名(图 4D),稳定性综合排序:ftsZ > gyrA > rpoB1 > ftsA > secA > gap > zapE > gmk2 > rpoD > secY > rpoO > ftsW > gmk1 > recA。由稳定性综合排序结果可看出,ftsZ表达最稳定,其次是gyrA。根据geNorm软件计算的内参基因之间的配对变异值Vn/n+1,分析出研究基因表达时引入内参基因的最适数目(n),由图 5可看出,配对变异值均小于0.15,因此选择2对或以上内参基因均可行。
|
| 图 4 候选内参基因的稳定性分析 Figure 4 The stability analysis of candidate reference genes Note: A: Average expressing stability M (geNorm); B: Stability value (NormFinder); C: Standard deviation (SPSS); D: Comprehensive gene stability (RefFinder). |
|
|

|
| 图 5 用于标准化的内参基因最适数目判定(geNorm) Figure 5 Determination of the optional number of control genes for normalization (geNorm) Note: The value of the abscissa indicates the number of reference genes. |
|
|
为进一步证明内参基因的可靠性,根据配对变异值Vn/n+1,选择gyrA+ftsZ的内参基因组合,以16S rRNA基因为参照,比较分析柑橘黄龙病菌重要致病基因LasΔ5313在感染黄龙病的植株叶片中的表达模式(图 6)。结果表明16S rRNA基因作为内参与gyrA+ftsZ作为内参所得的LasΔ5313的表达模式相同,同一侵染时期CLas在年橘、十月橘寄主中LasΔ5313的表达水平高于在柚寄主中的表达水平,但无显著差异(A:P1=0.139 > 0.05;B:P2=0.142 > 0.05);CLas在柚寄主侵染后期LasΔ5313的表达水平显著高于侵染前期(A:P3=0.039 < 0.05;B:P4=0.005 < 0.05)。对比16S rRNA基因作为内参与gyrA+ftsZ作为内参的表达结果,可看出以gyrA+ftsZ作为内参样品相对表达量的标准差较小,且表达差异性更显著(P4 < P3)。

|
| 图 6 基于不同内参基因的柑橘黄龙病菌LasΔ5313基因表达分析 Figure 6 Expression analysis of LasΔ5313 gene in "Candidatus Liberibacter asiaticus" based on different reference genes Note: Y-6: Citrus maxima of 6-month-after inoculation; N-6: Citrus reticulata Blanco cv. Nian Ju of 6-month-after inoculation; S-6: Citrus reticulata Blanco cv. Shiyue Ju of 6-month-after inoculation; Y-12: Citrus maxima of 12-month-after inoculation. |
|
|
在2001年已有利用RT-qPCR筛选内参基因的研究[32],近年来关于细菌内参基因的报道也陆续增加,包括SO2胁迫下胚牙乳杆菌(Lactobacillus plantarum)内参基因rpoB[33]、肺炎杆菌(Klebsiella pneumoniae)的内参基因recA[34]、梨火疫病菌(Erwinia amlovora)的内参基因proC、recA及ffh[35]等。相比动物与植物,细菌内参基因的研究相对较少[20]。目前未有韧皮部杆菌属内参基因筛选的相关报道,研究中使用的内参基因包括16S rRNA[10]、gyrA[14]。本研究中,16S rRNA基因作为内参基因,gyrA作为其中1个候选内参基因与其它候选内参基因进行对比,试验结果表明gyrA在不同植物寄主及不同侵染时期的试验条件下稳定性排第2位,可作为CLas内参基因,而ftsZ表达比gyrA更稳定。由于geNorm配对变异值结果表明需要选择2对或以上内参基因,因此本研究将gyrA+ftsZ作为内参与16S rRNA基因作为内参的分析结果进行对比,所得目的基因LasΔ5313表达模式相同,证明gyrA+ftsZ的可靠性。同时,本研究得出柑橘黄龙病菌的管家基因稳定性排序:ftsZ > gyrA > rpoB1 > ftsA > secA > gap > zapE > gmk2 > rpoD > secY > rpoO > ftsW > gmk1 > recA。
目前常用的稳定性评价软件包括geNorm、NormFinder及BestKeeper。其中BestKeeper实质为比较标准差,且一次性最多评价10个基因[23],因而本试验采用SPSS软件标准差比较法代替。本研究共比较了14个候选内参基因在CLas不同侵染时期及在不同植物寄主上的表达稳定性。以上3种软件的分析结果存在一定差异,因此基于RefFinder综合排序结果进行内参基因选择,避免单个软件分析的片面性,使筛选结果更可靠。
多数内参基因筛选试验选择传统内参基因作为候选内参基因[36-38],而Rocha等[21]表明可根据基因功能类别选择内参基因。本研究认为在全基因组测序完成的基础上,可根据基因的功能注释选择适合特定物种的特定内参基因。目前,柑橘黄龙病菌的全基因组测序已经完成,包括中国地区的菌株gxpsy[39]、A4[22]、YCPsy[40]。本研究利用所在实验室发表的中国CLas A4菌株基因组选择候选内参基因进行稳定性验证,首次对黄龙病的病原内参基因进行系统地研究。本研究发现各功能类别的基因均有效果较好和较差的内参基因,并初步发现细胞分裂和DNA复制功能的管家基因在黄龙病菌中的表达稳定性高于其它功能类别的基因,为后续的其它试验条件下的同类研究提供了参考。同时,本研究进一步证明不同物种适用的内参基因不同,例如其它物种较稳定的内参基因如rpoD[41]、recA[34-35]在黄龙病菌中表达稳定性不高。
综上,本研究筛选并验证柑橘黄龙病菌ftsZ+gyrA的内参基因组合,并且系统评估了14个内参基因的稳定性,发现细胞分裂和DNA复制功能的管家基因较为稳定,为后续内参基因选择及基因表达调控研究奠定基础,并为病原菌的检测及致病机理研究提供指导。
| [1] |
Xu MR, Dai ZH, Kong WW, et al. Citrus Huanglongbing research based on molecular biology techniques[J]. Journal of Fruit Science, 2015, 32(2): 322-334. (in Chinese) 许美容, 戴泽翰, 孔维文, 等. 基于分子技术的柑橘黄龙病研究进展[J]. 果树学报, 2015, 32(2): 322-334. |
| [2] |
Lin KX. Observationgs on yellow shoot of Citrus[J]. Acta Phytopathologica Sinica, 1956, 2(1): 1-11. (in Chinese) 林孔湘. 柑桔黄梢(黄龙)病研究Ⅰ.病情调查[J]. 植物病理学报, 1956, 2(1): 1-11. |
| [3] |
Folimonova SY, Robertson CJ, Garnsey SM, et al. Examination of the responses of different genotypes of citrus to Huanglongbing (citrus greening) under different conditions[J]. Phytopathology, 2009, 99(12): 1346-1354. DOI:10.1094/PHYTO-99-12-1346 |
| [4] |
Bové JM. Huanglongbing: a destructive, newly-emerging, century-old disease of citrus[J]. Journal of Plant Pathology, 2006, 88(1): 7-37. |
| [5] |
Teixeira DDC, Danet JL, Eveillard S, et al. Citrus Huanglongbing in S o Paulo State, Brazil: PCR detection of the 'Candidatus' Liberibacter species associated with the disease[J]. Molecular and Cellular Probes, 2005, 19(3): 173-179. DOI:10.1016/j.mcp.2004.11.002 |
| [6] |
Lopes SA, Frare GF, Bertolini E, et al. Liberibacters associated with Citrus Huanglongbing in Brazil: 'Candidatus Liberibacter asiaticus' is heat tolerant, 'Ca. L. americanus' is heat sensitive[J]. Plant Disease, 2009, 93(3): 257-262. DOI:10.1094/PDIS-93-3-0257 |
| [7] |
Deng XL, Tang WW. The studies on detection of Citrus Huanglongbing pathogen by polymerase chain reaction[J]. Journal of South China Agricultural University, 1996, 17(3): 119-120. (in Chinese) 邓晓玲, 唐伟文. 应用PCR技术检测柑桔黄龙病病原的研究[J]. 华南农业大学学报, 1996, 17(3): 119-120. |
| [8] |
Tian YN, Ke S, Ke C. Detection and quantitation of Citrus Huanglongbing pathogen by polymerase chain reaction[J]. Acta Phytopathologica Sinica, 1996, 26(3): 243-250. (in Chinese) 田亚南, 柯穗, 柯冲. 应用多聚酶链式反应(PCR)技术检测和定量分析柑桔黄龙病病原[J]. 植物病理学报, 1996, 26(3): 243-250. |
| [9] |
Wang N, Pierson EA, Setubal JC, et al. The Candidatus Liberibacter-host interface: insights into pathogenesis mechanisms and disease control[J]. Annual Review of Phytopathology, 2017, 55: 451-482. DOI:10.1146/annurev-phyto-080516-035513 |
| [10] |
Yan Q, Sreedharan A, Wei SP, et al. Global gene expression changes in Candidatus Liberibacter asiaticus during the transmission in distinct hosts between plant and insect[J]. Molecular Plant Pathology, 2013, 14(4): 391-404. DOI:10.1111/mpp.12015 |
| [11] |
Jain M, Fleites LA, Gabriel DW. Prophage-encoded peroxidase in 'Candidatus Liberibacter asiaticus' is a secreted effector that suppresses plant defenses[J]. Molecular Plant-Microbe Interactions, 2015, 28(12): 1330-1337. DOI:10.1094/MPMI-07-15-0145-R |
| [12] |
Prasad S, Xu J, Zhang YZ, et al. SEC-translocon dependent extracytoplasmic proteins of Candidatus Liberibacter asiaticus[J]. Froniers in Microbiology, 2016, 7: 1989. |
| [13] |
Clark K, Franco JY, Schwizer S, et al. An effector from the Huanglongbing-associated pathogen targets citrus proteases[J]. Nature Communications, 2018, 9: 1718. DOI:10.1038/s41467-018-04140-9 |
| [14] |
Pagliaccia D, Shi JX, Pang ZQ, et al. A pathogen secreted protein as a detection marker for Citrus Huanglongbing[J]. Frontiers in Microbiology, 2017, 8: 2041. DOI:10.3389/fmicb.2017.02041 |
| [15] |
Thellin O, ElMoualij B, Heinen E, et al. A decade of improvements in quantification of gene expression and internal standard selection[J]. Biotechnology Advances, 2009, 27(4): 323-333. DOI:10.1016/j.biotechadv.2009.01.010 |
| [16] |
McMillan M, Pereg L. Evaluation of reference genes for gene expression analysis using quantitative RT-PCR in Azospirillum brasilense[J]. PLoS One, 2014, 9(5): e981625. |
| [17] |
Sang J, Han XJ, Liu MY, et al. Selection and validation of reference genes for real-time quantitative PCR in Hyperaccumulating Ecotype of Sedum alfredii under different heavy metals stresses[J]. PLoS One, 2013, 8(13): e82927. |
| [18] |
Huggett JF, Foy CA, Benes V, et al. The digital MIQE guidelines: minimum information for publication of quantitative digital PCR experiments[J]. Clinical Chemistry, 2013, 59(6): 892-902. DOI:10.1373/clinchem.2013.206375 |
| [19] |
Bennett J, Hondred D, Register JC. Keeping qRT-PCR rigorous and biologically relevant[J]. Plant Cell Reports, 2015, 34(1): 1-3. DOI:10.1007/s00299-014-1692-6 |
| [20] |
Sang J, Wang ZN, Li M, et al. ICG: a wiki-driven knowledgebase of internal control genes for RT-qPCR normalization[J]. Nucleic Acids Research, 2018, 46(D1): D121-D126. DOI:10.1093/nar/gkx875 |
| [21] |
Rocha DJP, Santos CS, Pacheco LGC. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis[J]. Antonie van Leeuwenhoek, 2015, 108(3): 685-693. DOI:10.1007/s10482-015-0524-1 |
| [22] |
Zheng Z, Deng XL, Chen JC. A whole genome sequence of 'Candidatus Liberibacter asiaticus' from Guangdong, China, where HLB was first described[J]. The American Phytopathological Society, 2014, 104(S3): 137. |
| [23] |
Pfaffl MW, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [24] |
Vandesompele J, Preter KD, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): research0034. DOI:10.1186/gb-2002-3-7-reports0034 |
| [25] |
Andersen CL, Jensen JL, rntoft TF. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [26] |
Xie FL, Sun GL, Stiller JW, et al. Genome-wide functional analysis of the cotton transcriptome by creating an integrated EST database[J]. PLoS One, 2011, 6(11): e26980. DOI:10.1371/journal.pone.0026980 |
| [27] |
Pitino M, Allen V, Duan YP. LasΔ5315 effector induces extreme starch accumulation and chlorosis as Ca. Liberibacter asiaticus infection in Nicotiana benthamiana[J]. Frontiers in Plant Science, 2018, 9: 113. DOI:10.3389/fpls.2018.00113 |
| [28] |
Li WB, Hartung JS, Levy L. Quantitative real-time PCR for detection and identification of Candidatus Liberibacter species associated with Citrus Huanglongbing[J]. Journal of Microbiological Methods, 2006, 66(1): 104-115. DOI:10.1016/j.mimet.2005.10.018 |
| [29] |
Lu K, Li T, He J, et al. qPrimerDB: a thermodynamics-based gene-specific qPCR primer database for 147 organisms[J]. Nucleic Acids Research, 2018, 46(D1). |
| [30] |
Wu WK, Liu CQ, Zhou ZG, et al. The selection of reference genes in Chlamydomonas reinhardtii P. A. dangeard by real-time quantitative PCR[J]. Plant Physiology Journal, 2009, 45(7): 667-672. (in Chinese) 吴文凯, 刘成前, 周志刚, 等. 用于莱茵衣藻荧光定量PCR分析的内参基因选择[J]. 植物生理学通讯, 2009, 45(7): 667-672. |
| [31] |
Pitino M, Armstrong CM, Duan YP. Rapid screening for citrus canker resistance employing pathogen-associated molecular pattern-triggered immunity responses[J]. Horticulture Research, 2015, 2: 15042. DOI:10.1038/hortres.2015.42 |
| [32] |
Hamalainen HK, Tubman JC, Vikman S, et al. Identification and validation of endogenous reference genes for expression profiling of T helper cell differentiation by quantitative real-time RT-PCR[J]. Analytical Biochemistry, 2001, 299(1): 63-70. DOI:10.1006/abio.2001.5369 |
| [33] |
Lin XZ, He ZG, Li WX, et al. Validation of reference genes for real-time quantitative polymerase chain reaction analysis in Lactobacillus plantarum R23 under sulfur dioxide stress conditions[J]. Australian Journal of Grape and Wine Research, 2018, 24(3): 390-395. DOI:10.1111/ajgw.12331 |
| [34] |
Inácio GA, Stuchi LP, Gonçalves SNM, et al. Selection and validation of reference genes for gene expression studies in Klebsiella pneumoniae using reverse transcription quantitative real-time PCR[J]. Scientific Reports, 2018, 8(1): 9001. DOI:10.1038/s41598-018-27420-2 |
| [35] |
Kałużna M, Kuras A, Puławska J. Validation of reference genes for the normalization of the RT-qPCR gene expression of virulence genes of Erwinia amylovora in apple shoots[J]. Scientific Reports, 2017, 7: 2034. DOI:10.1038/s41598-017-02078-4 |
| [36] |
Zhao WJ, Xu J, Bao QH, et al. Selection of reference genes for real-time quantitative PCR[J]. Microbiology China, 2010, 37(12): 1825-1829. (in Chinese) 赵文静, 徐洁, 包秋华, 等. 实时荧光定量PCR中内参基因的选择[J]. 微生物学通报, 2010, 37(12): 1825-1829. |
| [37] |
Liu XT, Wang SL, Xue JQ, et al. Selection of reference genes for quantitative real-time PCR in different tissue and organ of barbadoslily[J]. Acta Horticulturae Sinica, 2018, 45(5): 919-930. (in Chinese) 刘晓婷, 王顺利, 薛璟祺, 等. 朱顶红实时荧光定量PCR中不同组织器官内参基因的筛选[J]. 园艺学报, 2018, 45(5): 919-930. |
| [38] |
Pan LX, Wang N, Wang AJ, et al. Selection of reference genes for quantitative real-time PCR in Tilletia horrida[J]. Acta Phytopathologica Sinica, 2018, 48(5): 640-647. (in Chinese) 盘林秀, 王娜, 王爱军, 等. 稻粒黑粉病菌实时荧光定量PCR内参基因筛选[J]. 植物病理学报, 2018, 48(5): 640-647. |
| [39] |
Lin H, Han CS, Liu BH, et al. Complete genome sequence of a Chinese strain of "Candidatus Liberibacter asiaticus"[J]. Genome Announcements, 2013, 1(2): e00184-13. DOI:10.1128/genomeA.00184-13 |
| [40] |
Wu F, Zheng Z, Deng X, et al. Draft genome sequence of "Candidatus Liberibacter asiaticus" from Diaphorina citri in Guangdong, China[J]. Genome Announcements, 2015, 3(6): e01316-15. DOI:10.1128/genomeA.01316-15 |
| [41] |
Galisa PS, Silva HAP, Macedo AVM, et al. Identification and validation of reference genes to study the gene expression in Gluconacetobacter diazotrophicus grown in different carbon sources using RT-qPCR[J]. Journal of Microbiological Methods, 2012, 91(1): 1-7. DOI:10.1016/j.mimet.2012.07.005 |
 2019, Vol. 46
2019, Vol. 46