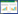扩展功能
文章信息
- 贾化可, 张功友, 陶小买, 吴东明, 隆耀航, 刘红美
- JIA Hua-Ke, ZHANG Gong-You, TAO Xiao-Mai, WU Dong-Ming, LONG Yao-Hang, LIU Hong-Mei
- 短短芽孢杆菌GZDF3中PilZ结构域基因的挖掘和糖基转移酶的原核表达
- Mining gene with PilZ domain and prokaryotic expression of glycosyltransferase of Brevibacillus brevis GZDF3
- 微生物学通报, 2019, 46(11): 2944-2951
- Microbiology China, 2019, 46(11): 2944-2951
- DOI: 10.13344/j.microbiol.china.190215
-
文章历史
- 收稿日期: 2019-03-18
- 接受日期: 2019-05-08
- 网络首发日期: 2019-06-19
2. 贵州医科大学生物与医学工程重点实验室 贵州 贵阳 550025;
3. 贵州省免疫细胞与抗体工程研究中心 贵州 贵阳 550025
2. Guizhou Medical University, Key Laboratory of Biological and Medical Engineering, Guiyang, Guizhou 550025, China;
3. Guizhou Immunology Cell and Antibody Engineering Research Center, Guiyang, Guizhou 550025, China
环二鸟苷酸(Cyclic diguanylate,c-di-GMP)是普遍存在于细菌中的第二信使,参与调节细菌的运动性、生物膜的形成、细胞周期和毒力等生命活动过程[1]。c-di-GMP信号受体类型多样,包括转录调控因子、结构域蛋白、核糖体开关、多核苷酸磷酸化激酶和新发现的蛋白激酶等[2]。c-di-GMP响应性效应蛋白的一个主要结构域是含有C末端的PilZ结构域,与c-di-GMP结合时构象发生变化,导致蛋白与蛋白间的相互作用和变构效应的改变[3-4],两者主要通过PilZ结构域上保守的基序RXXXR和D/NXSXXG结合[5-6]。
PilZ结构域是最早发现可与c-di-GMP结合的结构域,最终从铜绿假单胞菌中鉴定出来并命名为PilZ结构域,通常为β桶状折叠形式,含有两个保守基序[7],广泛分布在细菌基因组中,并且细菌基因组通常编码一个或多个PilZ结构域蛋白[8],PilZ结构域通常作为独立结构域或多结构域蛋白的一部分出现[9]。
PilZ结构域的研究成为近年来细菌研究方面的热点之一。许多PilZ结构域蛋白已在不同细菌中被鉴定,分别具有不同的功能,主要涉及细菌的运动性、多糖的合成、生物膜的形成及细菌毒力的调节[8]。大肠杆菌和沙门氏菌中含有PilZ结构域的蛋白YcgR在高水平c-di-GMP中直接与鞭毛运动元件结合,从而降低细菌的泳动速度,使细菌由浮游状态转变为静止状态,有利于生物膜的形成[10-12]。在枯草芽孢杆菌中有证据表明YcgR的同源物YpfA (现称DgrA)与鞭毛运动马达蛋白MotA相互作用[13]。PilZ结构域蛋白PilZXAC1133和PilB ATP酶形成复合物后,促进T4P复合物的形成,抑制柑橘溃疡病菌的滑动运动[14]。铜绿假单胞杆菌中含PilZ结构域的蛋白FlgZ与高水平的c-di-GMP结合,在多糖产物Pel的协同作用下可抑制菌体的群集运动[15]。伯氏疏螺旋体中含有PilZ结构域的蛋白PlzA和PlzC与c-di-GMP结合后可触发结构重组,从而起到功能转换的作用,而PlzA的缺失会导致异常的运动模式并减弱或消除伯氏疏螺旋体的感染性[4]。绿脓杆菌中包含一个PilZ结构域的Alg44是由操纵子alg编码的内膜蛋白,可与c-di-GMP结合,而Alg44等位基因编码的Alg44蛋白则不能与c-di-GMP结合,也不能产生海藻酸钠,表明海藻酸盐的生物合成需要Alg44与c-di-GMP的结合[16-17]。假单胞菌中的一种鞭毛基因flgZ,编码一种PilZ结构域蛋白,调节假单胞菌的运动状态和生物膜形成[18]。因此,研究PilZ结构域蛋白对于我们从更多方面了解细菌的生命活动规律具有重要的意义。
目前,关于PilZ结构域在短短芽孢杆菌(Brevibacillus brevis) c-di-GMP调控中的研究尚未报道,因此挖掘短短芽孢杆菌基因组中的PilZ结构域,获得纯度较高的含PilZ结构域的蛋白,并对其生物物理性质以及结构的分析,对于进一步确定该蛋白的结构及结合c-di-GMP调节细菌生长状态的机制研究是非常有必要的,同时也能为短短芽孢杆菌的进一步开发利用提供新的研究思路。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒短短芽孢杆菌(Brevibacillus brevis GZDF3)、大肠杆菌[Escherichia coli BL21(DE3)]均保存于贵州医科大学医药生物技术工程研究中心;原核表达质粒pET-28a由贵州医科大学生物与工程学院王兵副教授惠赠。
1.1.2 主要试剂和仪器DNA Marker、T4 DNA连接酶、限制性内切酶Hind Ⅲ和Xho Ⅰ购自宝日医生物技术(北京)有限公司(TaKaRa中国);蛋白质预染Marker购自北京索莱宝科技有限公司;DNA高保真聚合酶购自北京全式金生物技术有限公司;细菌全基因组试剂盒、PCR产物纯化试剂盒和质粒小提试剂盒购自天根生化科技(北京)有限公司;引物的合成和质粒的测序由深圳华大基因完成;Ni-NTA His Bind Resin镍柱购自上海七海复泰生物科技有限公司。
PCR仪,ABI公司;琼脂糖电泳仪、凝胶成像系统,Bio-Rad公司。
1.1.3 主要软件及操作平台操作平台:Windows系统操作平台、Linux系统操作平台。
分析软件:BioEdit (V7.0.9)、HMMER (V3.1b2)、MEGA X。
主要网站:ExPASy (https://www.expasy.org/);NCBI (https://www.ncbi.nlm.nih.gov)。
1.2 GZDF3中含PilZ结构域的蛋白编码基因预测及保守结构域分析 1.2.1 GZDF3中含PilZ结构域的蛋白编码基因预测从Pfam数据库下载PilZ结构域蛋白质家族模型并进行格式化,用华大基因公司测序、SOAPdenove (V2.04)组装、Genemark注释后的GZDF3全基因组序列[19]作为输入序列,在该蛋白质家族模型数据库中通过HMMScan进行搜索比对,确定GZDF3中含有PilZ结构域的蛋白编码基因。
1.2.2 基因保守结构域分析登录NCBI (https://www.ncbi.nlm.nih.gov/)网站,通过Protein BLAST比对分析各候选基因的编码产物;在保守结构域数据库(Conserved domain database,CDD)中通过CD-Search分析该蛋白氨基酸序列保守结构域。
1.3 蛋白基本性质分析登录蛋白质专家分析系统(ExPASy,https://www.expasy.org/),通过ProtParam分析该序列的等电点(pI)、相对分子质量(Molecular weight,MW)、亲疏水性、跨膜区等基本性质,通过SOPMA分析该蛋白的二级结构特征;通过SignalP-5.0 Server预测该蛋白质的信号肽。
1.4 系统发育树构建及高级结构分析通过NCBI下载其他菌株含有PilZ结构域的蛋白质序列Brevibacillus brevis NBRC 110488 (登录号为WP 069846830.1)、Brevibacillus brevis X23 (登录号为WP 017250036.1)、Brevibacillus brevis DZQ 7 (登录号为WP 048031333.1)、Brevibacillus brevis NBRC 100599 (登录号为WP 012684654.1)、Brevibacillus brevis ATCC 35690 (登录号为WP 087342889.1)、Brevibacillus brevis NCTC 3000 (登录号为WP_106654172.1),选用Arabidopsis thaliana (L.) Heynh (登录号为AEE34679.1)作外群,使用软件MEGA X进行多序列比对,使用邻接法(Neighbor-Joining,NJ),Bootstrap值为1 000,构建系统发育树,分析不同菌株中含PilZ结构域蛋白的进化关系;通过SWISS-MODEL分析蛋白的结构。
1.5 该编码基因的克隆及原核表达载体的构建用细菌全基因组提取试剂盒提取过夜培养的短短芽孢杆菌的总DNA,并以总DNA为模板,以引物5′-CCCAAGCTTAGAACGCAGATG-3′ (下划线为Hind Ⅲ酶切位点)和5′-CCGCTCGAGGAACC AATTAAC-3′ (下划线为Xho Ⅰ酶切位点)扩增目标基因。PCR反应体系(30 μL):10×Buffer 3 μL,dNTPs (10 mmol/L) 2 μL,模板DNA (约200 ng) 1 μL,上、下游引物(10 μmol/L)各1 μL,高保真酶(5 U/μL) 0.25 μL,双蒸水21.75 μL。PCR反应条件:95 ℃ 5 min;94 ℃ 1 min,55 ℃ 50 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。用DNA产物纯化回收试剂盒回收PCR产物,并用限制性内切酶Hind Ⅲ和Xho Ⅰ进行双酶切,纯化后连接到pET-28a表达载体上,转化Escherichia coli BL21(DE3)感受态细胞,将菌体涂布于含10 μg/mL Kanr的LB平板,37 ℃培养过夜,挑取单菌落做菌落PCR,选择阳性结果的克隆抽提质粒并进行双酶切验证,取双酶切验证正确的阳性克隆送至华大基因测序,进一步确定插入的目的基因序列是否正确。
1.6 重组蛋白的诱导表达及纯化 1.6.1 重组蛋白的诱导表达将培养过夜的细菌按1%接种量分别接种至30 mL含10 μg/mL Kanr的LB液体培养基,于37 ℃、200 r/min培养至OD600为0.5-0.6时,加入终浓度为0.5 mmol/L的乳糖溶液,30 ℃、200 r/min下分别诱导4、8、20 h,然后4 ℃、6 000 r/min离心10 min收集菌体和菌液上清备用,加入等体积的PBS重悬菌体,4 ℃、6 000 r/min离心10 min收集菌体,重复此步骤1-2次,加入预冷的PBS重悬菌体后进行超声破碎(300 W,工作5 s,间隔5 s,共30 min),破碎后的菌液在4 ℃、8 000 r/min离心15 min,收集破碎后上清。12% SDS-PAGE电泳,考马斯亮蓝染色,确定最佳表达时间。
1.6.2 重组蛋白的分离纯化将1.6.1获得的最佳表达条件进行重组蛋白的大量表达。在4 ℃、6 000 r/min离心10 min收集诱导表达后的菌体,重悬于30 mL的PBS中,再次离心收集菌体,重复此步骤1-2次,加入预冷的PBS重悬菌体,在冰浴中超声破碎(300 W,工作5 s,间隔5 s,共30 min),4 ℃、8 000 r/min下离心15 min,收集破碎后上清备用。通过Ni-NTA His Bind Resin镍柱对上清中的目的蛋白进行纯化,将收集的蛋白样品用12% SDS-PAGE电泳,考马斯亮蓝染色,检验蛋白的纯化效果。
2 结果与分析 2.1 含有PilZ结构域的蛋白编码基因预测及保守结构域分析以下载的PilZ结构域模型作为蛋白数据库,用HMMScan软件扫描短短芽孢杆菌GZDF3全基因组,发现GZDF3中含有PilZ结构域的蛋白编码基因一共有5个,分别命名为Gene1423、Gene1723、Gene2571、Gene2956、Gene4836。Protein BLAST分析显示,Gene1423编码产物属YcgR蛋白超家族,与鞭毛开关复合蛋白FliG和FliM相互作用,调节细菌的运动;Gene1723编码I类透明质酸合成酶,属于糖基转移酶超家族2,其功能结构相对保守[20];Gene2571、Gene2956编码产物为假定蛋白,Gene4836编码产物为糖基转移酶;CD-Search结果表明,Gene4836编码的氨基酸序列C端含有1个PilZ保守结构域(登录号为WP_064202322.1)。
2.2 蛋白基本性质分析ProtParam分析显示Gene4836编码产物糖基转移酶的氨基酸长度为206 aa;分子量为24.08 kD;等电点为6.39,为酸性蛋白;在水溶液中不稳定,不稳定指数为53.93;通过ProtScale分析,Score > 0表示疏水,Score < 0表示亲水,亲水性区域大于疏水性区域,总平均亲水性为-0.336,表明该糖基转移酶为亲水性蛋白(图 1A);通过Tmpred分析,在第107-125位氨基酸之间存在一个跨膜区(图 1B),TM-螺旋长度在17-33之间;通过SOPMA分析,糖基转移酶的二级结构中延伸链(Extended strand)占33.98%,α-螺旋(Alpha helix)占17.48%,β-折叠(Beta turn)占8.25%,无规则卷曲(Random coil)占40.29%。
|
| 图 1 糖基转移酶亲水性/疏水性分析和跨膜区预测 Figure 1 Hydrophilicity/hydrophobicity analysis and membrane-spanning regions prediction of glycosyltransferases 注:A:糖基转移酶亲水性/疏水性分析;B:糖基转移酶跨膜区分析. i-> o表示从膜内到膜外,o-> i表示从膜外到膜内. Note: A: Hydrophilicity/hydrophobicity analysis of glycosyltransferase; B: Membrane-spanning regions analysis of glycosyltransferases. i-> o indicates from inside to outside of the membrane and o-> i indicates from outside to inside of the membrane. |
|
|
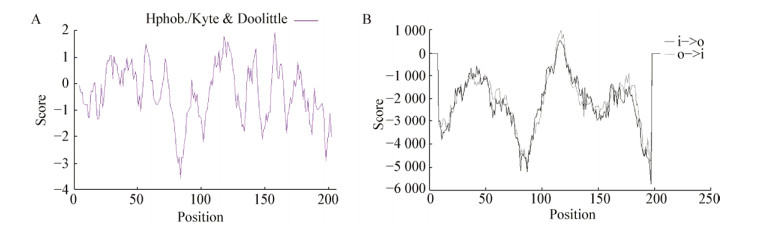
将8条含有PilZ结构域的蛋白质同源编码氨基酸序列和拟南芥的限制性核酸内切酶氨基酸序列进行多序列比对,选用邻接法(Neighbor-Joining,NJ),Bootstrap值为1 000,构建系统发育树如图 2所示,短短芽孢杆菌GZDF3与ATCC35690一致性较高,而其他几株短短芽孢杆菌相似性相对较高;对短短芽孢杆菌GZDF3和ATCC35690菌株中该蛋白的氨基酸序列进行比较分析,发现在28位氨基酸处发生丙氨酸和缬氨酸的相互替换,两者一致性达到99.52% (图 3A);在SWISS-MODEL网站上以该蛋白相似性较高的蛋白5vx6.1.A为模板通过同源建模的方法,最终预测出两种蛋白质的三级结构图(图 3B),可以发现虽然其序列的相似性较高,但在空间结构上存在明显的差异,其中Gene4836蛋白序列首端区域有大约30个氨基酸(包括28位的丙氨酸)未参与建模,而PilZATCC35690只有5个氨基酸未参与建模,从图 3B(1)中可以看出β折叠的数量明显减少,说明28位氨基酸的替换对其空间结构可能具有很重要的影响,图 3B(2)中红色箭头指出位置是PilZATCC35690蛋白序列第28位氨基酸缬氨酸,而该28位氨基酸造成的空间结构差异的最终原因有待于后续实验进行深入的研究。

|
| 图 2 Gene4836的系统进化树 Figure 2 Phylogenetic trees of Gene4836 注:括号内为相应基因序列在GenBank中的登录号;分支点上的数值为1 000次Bootstrap分析所得值. Note: The numbers in parentheses represent the accession numbers in the GenBank for the corresponding gene sequences of those reference strains; Bootstrap values based on 1 000 replications are shown at branch nodes. |
|
|
|
| 图 3 Gene4836和PilZATCC35690的氨基酸序列分析及高级结构预测 Figure 3 Amino acid sequence alignment and advanced structure prediction of Gene4836 and PilZATCC35690 注:A:Gene4836和PilZATCC35690的氨基酸序列分析. B:Gene4836和PilZATCC35690的高级结构预测,(1):蛋白Gene4836;(2):同源蛋白PilZATCC35690. Note: A: Amino acid sequence analysis of Gene4836 and PilZATCC35690. B: Advanced structure prediction of Gene4836 and PilZATCC35690, (1): Protein Gene4836; (2): Homologous protein PilZATCC35690. |
|
|
以短短芽孢杆菌GZDF3总DNA为模板,用PCR扩增Gene4836目的片断,产物经1%的琼脂糖凝胶电泳检测发现扩增产物的大小与理论值(670 bp)一致(图 4),显示获得目的条带,可用于后续实验。对构建的重组表达载体克隆进行大量培养,提取重组质粒,用限制性内切酶Hind Ⅲ和Xho Ⅰ进行双酶切鉴定(图 5),电泳结果显示酶切出一大小为670 bp的插入片段,进一步阳性重组子的测序结果也表明重组质粒构建成功,可用于下一步诱导表达。
|
| 图 4 Gene4836基因的PCR扩增结果 Figure 4 Production of Gene4836 gene amplified by PCR |
|
|
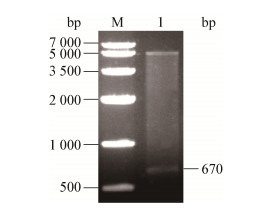
|
| 图 5 重组质粒pET-28a-Gene4836的双酶切结果 Figure 5 Double enzyme digestion results of recombinant plasmid pET-28a-Gene4836 注:M:DL2000 DNA标准条带;1:双酶切结果. Note: M: DL2000 DNA marker; 1: The results of double enzyme digestion. |
|
|
前期预实验表明,重组蛋白在细胞质中以可溶性形式表达;收集乳糖诱导表达4、8、20 h的菌液上清以及每个时间段菌体破碎后的上清备用,利用该融合蛋白的His标签,用Ni-NTA His Bind Resin镍柱进行纯化,取纯化后的样品和备用样品进行SDS-PAGE检测,结果如图 6所示,在25 kD左右处具有明显的条带,与该融合蛋白的理论大小(24.08 kD)相符。
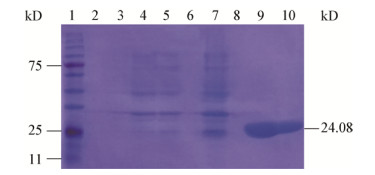
|
| 图 6 SDS-PAGE分析原核表达产物蛋白 Figure 6 SDS-PAGE analysis of prokaryotic expression product protein 注:1:预染的蛋白质标准条带;2:未诱导的菌液上清;3:未诱导的细菌菌体;4:诱导4 h破碎离心后的上清;5:诱导8 h破碎离心后的上清;6:诱导20 h的菌液上清;7:诱导20 h破碎离心后的上清;8:洗脱的杂蛋白;9、10:纯化的目的蛋白. Note: 1: Prestained protein marker; 2: Supernatant of uninduced bacterial fluid; 3: Uninduced bacteria (thallus); 4: The supernatant obtained by centrifugation after induced for 4 h; 5: The supernatant obtained by centrifugation after induced for 8 h; 6: Bacterial supernatant induced for 20 h; 7: The supernatant obtained by centrifugation after induced for 20 h; 8: Other proteins eluted; 9, 10: The purified target protein. |
|
|
短短芽孢杆菌GZDF3是分离于贵州省大方县半夏种植基地根际土壤,对半夏块茎腐烂病病原菌有强抑制作用。短短芽孢杆菌(Brevibacillus brevis)属革兰氏阳性菌,普遍存在于土壤中,具有广泛的生防潜力,可作为生防型菌肥、植物保鲜剂等[21]。PilZ结构域蛋白广泛存在于细菌全基因组中,通过与细菌第二信使c-di-GMP结合调控细菌的运动状态、生物膜形成、细胞分裂和毒力等生命活动过程,在细菌的生长过程中具有重要作用[2]。
本研究以GZDF3全基因组序列为对象,在Pfam数据库中下载PilZ结构域家族模型,使用HMMScan对GZDF3基因组序列进行搜索比对,发现5个含有PilZ结构域的蛋白编码基因,将5个编码基因进行BLAST比对,确定Gene1423编码产物为YcgR蛋白超家族,与鞭毛开关复合蛋白FliG和FliM相互作用,调节细菌的运动;Gene1723编码Ⅰ类透明质酸合成酶,属于糖基转移酶超家族2,其功能结构相对保守[20],通常是一类依赖脂质的完整的膜蛋白;Gene2571、Gene2956编码产物为假定蛋白,Gene4836编码产物为糖基转移酶。糖基转移酶(Gtfs)在生物体内催化活化的糖连接到不同的受体分子,在抗生素生物合成基因簇中已经发现了很多编码糖基转移酶的基因[22],它的功能是在抗生素生物合成的后期使其糖基化,通过糖的位置、类型和数量的改变对抗生素的活性进行调节,这大大增加天然产物的结构多样性[23],将活性糖基从供体转移到受体(小分子化合物),从而改变这些化合物的生物化学性质[24],具有重要的研究意义。
由于Gene1423属于YcgR蛋白超家族,通常参与调节细菌的运动状态,功能相对保守[11];Gene1723为糖基转移酶超家族2中的Ⅰ类透明质酸合成酶,一般为依赖脂质的完整的膜蛋白,在磷脂基团的辅助下可独立完成糖基的转移和加成,其作用机制比较明确[20];而Gene4836为功能未明确的糖基转移酶。为此,我们采用ExPASy中的生物信息学软件进行生物信息学分析,结果该蛋白为酸性亲水性蛋白,在水溶液中不稳定,存在一个明显的跨膜区,推测可能为细菌中的一个膜锚定蛋白,通过与c-di-GMP结合行使调节催化功能。二级结构中含有延伸链(Extended strand)、α-螺旋、β-折叠和无规则卷曲,没有信号肽。系统发育树表明,该糖基转移酶与短短芽孢杆菌ATCC35690中相应的蛋白具有同源性,有可能行使相近的功能。在碳水化合物活性酶数据库(CAZy)中未找到与之相似的蛋白,推测其可能是一种还未分类的糖基转移酶;经UniProt数据库比对分析发现,其C端的PilZ结构域可能与鞭毛运动的开关蛋白FliG和FliM相互作用,进而影响鞭毛的旋转运动。
随后,以GZDF3基因组为模板,以pET-28a为载体构建含有His标签的重组质粒。将该重组质粒转入E. coli BL21(DE3)中,取阳性克隆进行培养,在OD600为0.5、0.5 mmol/L乳糖、30 ℃、200 r/min条件下诱导表达该糖基转移酶,经菌体破碎离心,使用Ni-NTA His Bind Resin镍柱进行纯化,再经SDS-PAGE检验,表明已获得纯度较高的糖基转移酶,为后续的蛋白质的结构与功能的研究奠定基础。由于该糖基转移酶同时含有PilZ结构域,在本研究结果的基础上,接下来有必要通过该基因突变体的构建等方法对其在菌株体内的作用机制做深入研究。
| [1] |
Römling U, Galperin MY, Gomelsky M. Cyclic di-GMP: the first 25 years of a universal bacterial second messenger[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 1-52. |
| [2] |
Yang FH, Tian F, Chen HM, et al. Cyclic di-GMP signal receptor-mediated regulation of bacterial behaviors[J]. Plant Protection, 2017, 43(1): 9-14. (in Chinese) 杨凤环, 田芳, 陈华民, 等. 病原细菌受体介导的c-di-GMP信号传导及其调控机制[J]. 植物保护, 2017, 43(1): 9-14. DOI:10.3969/j.issn.0529-1542.2017.01.002 |
| [3] |
Wirebrand L, Österberg S, López-Sánchez A, et al. PP4397/FlgZ provides the link between PP2258 c-di-GMP signalling and altered motility in Pseudomonas putida[J]. Scientific Reports, 2018, 8(1): 12205. DOI:10.1038/s41598-018-29785-w |
| [4] |
Kostick-Dunn JL, Izac JR, Freedman JC, et al. The Borrelia burgdorferi c-di-GMP binding receptors, PlzA and PlzB, are functionally distinct[J]. Frontiers in Cellular and Infection Microbiology, 2018, 8: 213. DOI:10.3389/fcimb.2018.00213 |
| [5] |
Amikam D, Galperin MY. PilZ domain is part of the bacterial c-di-GMP binding protein[J]. Bioinformatics, 2006, 22(1): 3-6. |
| [6] |
Benach J, Swaminathan SS, Tamayo R, et al. The structural basis of cyclic diguanylate signal transduction by PilZ domains[J]. The EMBO Journal, 2007, 26(24): 5153-5166. DOI:10.1038/sj.emboj.7601918 |
| [7] |
Zhou E, Seminara AB, Kim SK, et al. Thiol-benzo-triazolo- quinazolinone inhibits Alg44 binding to c-di-GMP and reduces alginate production by Pseudomonas aeruginosa[J]. ACS Chemical Biology, 2017, 12(12): 3076-3085. DOI:10.1021/acschembio.7b00826 |
| [8] |
Yang F, Tian F, Chen H, et al. The Xanthomonas oryzae pv. oryzae PilZ domain proteins function differentially in cyclic di-GMP binding and regulation of virulence and motility[J]. Applied and Environmental Microbiology, 2015, 81(12): 4358-4367. |
| [9] |
Sch per S, Steinchen W, Krol E, et al. AraC-like transcriptional activator CuxR binds c-di-GMP by a PilZ-like mechanism to regulate extracellular polysaccharide production[J]. Proceedings of the National Academy of Sciences of the Unites States of America, 2017, 114(24): E4822-E4831. DOI:10.1073/pnas.1702435114 |
| [10] |
Boehm A, Kaiser M, Li H, et al. Second messenger-mediated adjustment of bacterial swimming velocity[J]. Cell, 2010, 141(1): 107-116. DOI:10.1016/j.cell.2010.01.018 |
| [11] |
Paul K, Nieto V, Carlquist WC, et al. The c-di-GMP binding protein YcgR controls flagellar motor direction and speed to affect chemotaxis by a "Backstop Brake" mechanism[J]. Molecular Cell, 2010, 38(1): 128-139. |
| [12] |
Pultz IS, Christen M, Don Kulasekara H, et al. The response threshold of Salmonella PilZ domain proteins is determined by their binding affinities for c-di-GMP[J]. Molecular Microbiology, 2012, 86(6): 1424-1440. DOI:10.1111/mmi.12066 |
| [13] |
Chen Y, Chai YR, Guo JH, et al. Evidence for cyclic di-GMP-mediated signaling in Bacillus subtilis[J]. Journal of Bacteriology, 2012, 194(18): 5080-5090. DOI:10.1128/JB.01092-12 |
| [14] |
Mahdavi J, Royer PJ, Sjölinder HS, et al. Pro-inflammatory cytokines can act as intracellular modulators of commensal bacterial virulence[J]. Open Biology, 2013, 3(10): 130048. DOI:10.1098/rsob.130048 |
| [15] |
Baker AE, Diepold A, Kuchma SL, et al. PilZ domain protein FlgZ mediates cyclic di-GMP-dependent swarming motility control in Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2016, 198(13): 1837-1846. DOI:10.1128/JB.00196-16 |
| [16] |
Merighi M, Lee VT, Hyodo M, et al. The second messenger bis-(3'-5')-cyclic-GMP and its PilZ domain-containing receptor Alg44 are required for alginate biosynthesis in Pseudomonas aeruginosa[J]. Molecular microbiology, 2007, 65(4): 876-895. DOI:10.1111/j.1365-2958.2007.05817.x |
| [17] |
Whitney JC, Whitfield GB, Marmont LS, et al. Dimeric c-di-GMP is required for post-translational regulation of alginate production in Pseudomonas aeruginosa[J]. Journal of Biological Chemistry, 2015, 290(20): 12451-12462. DOI:10.1074/jbc.M115.645051 |
| [18] |
Martínez-Granero F, Navazo A, Barahona E, et al. Identification of flgZ as a flagellar gene encoding a PilZ domain protein that regulates swimming motility and biofilm formation in Pseudomonas[J]. PLoS One, 2014, 9(2): e87608. DOI:10.1371/journal.pone.0087608 |
| [19] |
Li YQ, Ye YN, Tao XM, et al. Analysis of codon bias in whole genome of Brevibacillus brevis GZDF3[J]. Genomics and Applied Biology, 2017, 36(12): 5187-5190. (in Chinese) 李玉权, 叶远浓, 陶小买, 等. 短短芽孢杆菌GZDF3全基因组密码子偏好性分析[J]. 基因组学与应用生物学, 2017, 36(12): 5187-5190. |
| [20] |
Weigel PH. Functional characteristics and catalytic mechanisms of the bacterial hyaluronan synthases[J]. IUBMB Life, 2002, 54(4): 201-211. DOI:10.1080/15216540214931 |
| [21] |
Ma YQ, Wei C, Mao ZC, et al. Effects of bioorganic fertilizers with compound microbes on cucumber and root-knot nematode[J]. Scientia Agricultura Sinica, 2016, 49(15): 2945-2954. (in Chinese) 马玉琴, 魏偲, 茆振川, 等. 生防型菌肥对黄瓜生长及根结线虫病的影响[J]. 中国农业科学, 2016, 49(15): 2945-2954. DOI:10.3864/j.issn.0578-1752.2016.15.009 |
| [22] |
Thoroson JS, Hoster TJ Jr, Jiang JQ, et al. Natures carbohydrate chemists the enzymatic glycosylation of bioactive bacterial metabolites[J]. Current Organic Chemistry, 2001, 5(2): 139-167. DOI:10.2174/1385272013375706 |
| [23] |
Weymouth-Wilson AC. The role of carbohydrates in biologically active natural products[J]. Natural Product Reports, 1997, 14(2): 99. |
| [24] |
Lao J, Oikawa A, Bromley JR, et al. The plant glycosyltransferase clone collection for functional genomics[J]. The Plant Journal, 2014, 79(3): 517-529. DOI:10.1111/tpj.12577 |
 2019, Vol. 46
2019, Vol. 46