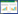扩展功能
文章信息
- 谢琮琮, 李至敏, 王小琴, 雷国风, 张伟, 李志敏
- XIE Cong-Cong, LI Zhi-Min, WANG Xiao-Qin, LEI Guo-Feng, ZHANG Wei, LI Zhi-Min
- 蓝杆藻ATCC51142中琥珀酸半醛脱氢酶的原核表达及生化表征
- Expression and characterization of succinic semialdehyde dehydrogenase from Cyanothece sp. ATCC51142
- 微生物学通报, 2019, 46(11): 2933-2943
- Microbiology China, 2019, 46(11): 2933-2943
- DOI: 10.13344/j.microbiol.china.181034
-
文章历史
- 收稿日期: 2018-12-19
- 接受日期: 2019-02-19
- 网络首发日期: 2019-04-02
2. 江西农业大学理学院 江西 南昌 330045;
3. 江西农业大学 菌物资源保护与利用江西省重点实验室 江西 南昌 330045
2. College of Science, Jiangxi Agricultural University, Nanchang, Jiangxi 330045, China;
3. Jiangxi Key Laboratory for Conservation and Utilization of Fungal Resources, Jiangxi Agricultural University, Nanchang, Jiangxi 330045, China
蓝藻,又称蓝细菌或蓝绿藻,在藻类中是最简单、最原始、最早发现能进行光合释氧的单细胞原核生物[1]。目前已知的蓝藻种类超过2 000种,遍布世界各地[2]。蓝杆藻ATCC51142是一种白天进行光合作用储存糖原,夜间通过固氮作用产氢储存氮源的固氮型蓝细菌[3]。2011年Zhang等报道除少数海洋蓝藻外大多数蓝藻都存在分别编码α-酮戊二酸脱羧酶(α-Ketoglutarate decarboxylase,KGD)和琥珀酸半醛脱氢酶(Succinic semialdehyde dehydrogenase,SSADH)的基因,这2个基因的存在使得蓝藻的三羧酸(Tricarboxylic acid cycle,TCA)循环途径变得完整,打破了长达半个世纪的蓝藻没有完整的TCA循环途径的认知[4-5]。蓝杆藻ATCC51142的基因组序列测序于2008年完成[6]。本课题组通过BLAST序列分析发现,蓝杆藻ATCC51142中编码KGD和SSADH的基因分别是cce4227和cce4228。cce4227基因编码KGD蛋白的生化功能已被证实[7]。
SSADH (EC 1.2.1.79)催化琥珀酸半醛氧化成琥珀酸从而进入TCA循环。因为SSADH是α-酮戊二酸和γ-氨基丁酸代谢旁路中的一个关键酶,所以SSADH的存在丰富了经典的TCA循环途径[8-9]。此外,SSADH在生物体内还发挥重要的作用。例如,在细菌中,SSADH参与调控细胞内的碳氮代谢平衡[10];在真核生物中,SSADH为TCA循环提供碳源[5, 11-12]。植物中的SSADH基因突变或缺失会造成植物发育、表型和对热、光照敏感程度的变化[13-15]。对于人而言,SSADH基因突变或缺失将会导致遗传性神经失调疾病[16-19]。
由于SSADH在生物体内具有重要的生理功能,学术界对SSADH酶学功能的研究也较多[20-22]。SSADH在催化琥珀酸半醛转化成琥珀酸的过程中需要NAD+或NADP+辅因子的参与。最初由于大肠杆菌中存在两种分别由GabD基因和YneI基因编码的SSADH,其分别依赖NADP+和NAD+ (或NADP+)辅因子,因此SSADH又分为GabD和YneI两种类型。辅因子偏好性不同的主要原因是不同类型的SSADH蛋白结构不同。例如,来自酿脓链球菌的SSADH在其活性中心具有较小的Ser158氨基酸残基,可以接纳NADP+辅因子中腺嘌呤核苷的磷酸根,因此其偏好NADP+辅因子[23];然而来自鼠伤寒沙门氏菌的SSADH偏好NAD+辅因子,原因就是其活性中心具有较大的Lys160氨基酸残基,无法容纳磷酸根[22]。本课题组此前研究发现来自鱼腥藻PCC7120的SSADH (all3556基因编码)倾向于使用NADP+辅因子,并且使用NADP+辅因子时其催化活性是使用NAD+辅因子的7倍多[8]。BLAST序列分析发现蓝杆藻ATCC51142中cce4228基因与all3556和a2771基因高度一致,并且蛋白氨基酸序列及建模结构分析发现与鱼腥藻PCC7120和聚球藻PCC7002的SSADH一样,蓝杆藻ATCC51142中cce4228基因编码蛋白在活性中心也具有Ser氨基酸残基。为了证实蓝杆藻ATCC51142中cce4228基因编码蛋白的催化功能并表征其相关生化性质,本研究从蓝杆藻ATCC51142基因组中克隆得到cce4228基因,并将构建的pET-28a-cce4228重组质粒转化至大肠杆菌BL21(DE3)感受态细胞后进行诱导表达,通过Ni-NTA亲和层析分离纯化得到cce4228重组蛋白。生物信息学及酶学研究结果表明cce4228基因编码一个偏好NADP+辅因子的琥珀酸半醛脱氢酶。本研究为进一步探索cce4228蛋白的结构功能关系及催化机制奠定了重要基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒大肠杆菌感受态细胞BL21(DE3)菌株,表达载体pET-28a为本实验室保存。蓝杆藻ATCC51142基因组购于美国模式菌种保藏中心。
1.1.2 主要试剂和仪器限制性内切酶Nde I、Xho I、T4 DNA连接酶、5 000 DNA Marker,北京全式金生物技术有限公司;Protein Marker,Thermo Scientific公司;2×Taq Mix、普通DNA产物纯化试剂盒、琼脂糖凝胶DNA回收试剂盒、质粒提取试剂盒,天根生化科技(北京)有限公司;Ni-NTA树脂,QIAGEN公司;其他主要试剂均为分析纯,北京索莱宝科技有限公司。所用引物由南京金斯瑞生物科技有限公司合成;质粒测序由上海祥音生物技术有限公司完成。PCR仪,Bio-Rad公司;高速冷冻离心机,Beckman公司;细胞破碎仪,宁波新芝生物科技股份有限公司;层析冷柜,上海沪西仪器厂;紫外可见分光光度计,上海仪电科学仪器股份有限公司。
1.2 琥珀酸半醛脱氢酶表达质粒(pET-28a-cce4228)的构建根据蓝杆藻ATCC51142中cce4228基因序列设计特异性引物(表 1),以蓝杆藻ATCC51142基因组为模板进行PCR扩增。PCR反应体系:模板1 μL (约30 ng),上、下游引物(终浓度0.2 μmol/L)各1 μL,2×Taq Mix 25 μL,灭菌水22 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,52 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。PCR扩增产物通过1%琼脂糖凝胶电泳分离纯化后加入Nde I和Xho I限制性内切酶进行双酶切。用同样的方法双酶切pET-28a载体。然后将酶切后纯化回收得到的cce4228基因片段和pET-28a载体线性片段(基因片段与载体片段的摩尔比为6:1)在16 ℃金属浴恒温下用T4 DNA连接酶过夜连接。连接产物5 μL转化大肠杆菌BL21(DE3)感受态细胞(100 μL)后,用含有50 μg/mL卡那霉素的LB固体平板培养基筛选阳性克隆。将经菌落PCR验证正确的阳性克隆单菌落摇菌提取质粒后送检测序。
| 引物名称 Primers name |
引物序列 Sequences (5′→3′) |
| cce4228F-Nde I | TGTAACTCATATGGGTATCGCTACAGTTAACCC |
| cce4228R-Xho I | TATCTCGAGTCACTTGATCCATACGGTTTTTAC |
| 注:引物序列中下划线为相应酶切位点.
Note: The underlined are restriction enzymes cutting sites. | |
将测序正确的单克隆菌落加入10 mL含有卡那霉素(50 μg/mL)的LB液体培养基中,37 ℃、180 r/min培养过夜。然后按1% (体积比)的接种量将过夜培养液加入至800 mL LB液体培养基中(含有50 μg/mL卡那霉素)进行菌体扩大培养。待培养液OD600至0.6时冰水浴后加入IPTG (终浓度为0.2 mmol/L),于25 ℃、180 r/min继续培养24 h。将于4 ℃、6 000 r/min离心20 min所得的菌体用20倍体积的平衡缓冲液(20 mmol/L Tris-HCl,pH 7.5缓冲液)重悬,于冰水浴中超声破碎后于4 ℃、12 000 r/min离心1 h得到菌体上清液,经0.22 μm滤膜过滤后,将上清过Ni-NTA树脂(事先用平衡缓冲液进行平衡处理),然后用含咪唑的平衡缓冲液进行浓度梯度洗脱,分别收集每个浓度的流出液,用SDS-PAGE检测目的蛋白。汇集含有目的蛋白的洗脱液后,于4 ℃下采用透析袋进行浓缩和透析,然后用Bradford方法对目的蛋白进行定量。
1.4 金属离子对琥珀酸半醛脱氢酶催化活性的影响在50 mmol/L Tris-HCl pH 7.5的缓冲液中,分别加入终浓度为2 mmol/L的金属离子(Mg2+,Mn2+,Ca2+,Co2+,Ni2+,Cu2+,Zn2+),测定cce4228蛋白在添加不同金属离子时催化琥珀酸半醛转化成琥珀酸的最大初始反应速度。反应总体积为500 μL,反应体系中含有上述不同的金属离子,0.5 mmol/L NADP二钠盐,0.2 mmol/L琥珀酸半醛和0.135 μmol/L cce4228蛋白。使用紫外分光光度计在25 ℃下连续监测反应混合液在340 nm的吸光值(A340)变化。根据吸光值的变化速率计算反应速度。实验重复3次。
1.5 反应体系pH值对琥珀酸半醛脱氢酶催化活性的影响在不同pH值条件下分别测定cce4228蛋白催化琥珀酸半醛转化成琥珀酸的最大初始反应速度。采用的缓冲液体系如下:50 mmol/L MES pH 5.6,50 mmol/L Bis-Tris pH 6.0、6.5,50 mmol/L HEPES pH 7.0、7.5,50 mmol/L Tris-HCl pH 8.0、8.5,50 mmol/L N-环己基-2-氨基乙磺酸CHES (9.0、9.5,50 mmol/L CAPS) pH 10.0、10.5、11.0。反应总体积为500 μL,在上述不同的反应缓冲液中含有0.2 mmol/L MgCl2,0.5 mmol/L NADP二钠盐,0.2 mmol/L琥珀酸半醛和0.135 μmol/L cce4228蛋白。实验重复3次。
1.6 琥珀酸半醛脱氢酶的活性测定琥珀酸半醛脱氢酶的酶动力学参数在25 ℃下测定。反应总体积为500 μL,反应体系中包含50 mmol/L CHES pH为9.0的缓冲液,0.2 mmol/L MgCl2,0.5 mmol/L NADP二钠盐,不同浓度的琥珀酸半醛和0.067 5 μmol/L cce4228蛋白。测定不同底物浓度(0.005-2 mmol/L)下的初始反应速度,利用KaleidaGraph软件通过米氏方程推导的完全抑制方程V=Vmax/(1+Km/[S]+[S]/Ki),计算出Vmax、Km、Ki和kcat。测定cce4228蛋白与NADP二钠盐的Km时,固定琥珀酸半醛浓度为0.2 mmol/L,Mg2+浓度为0.2 mmol/L,NADP二钠盐的浓度范围为0.005-0.5 mmol/L。测定cce4228蛋白与Mg2+的Km时,固定琥珀酸半醛浓度为0.2 mmol/L,NADP二钠盐浓度为0.5 mmol/L,MgCl2浓度范围为0.01-1 mmol/L。上述反应中cce4228蛋白浓度为0.135 μmol/L。测定在不同浓度的NADP二钠盐或MgCl2时cce4228蛋白催化琥珀酸半醛转化成琥珀酸的最大初始反应速度。实验重复3次。
1.7 琥珀酸半醛脱氢酶的生物信息学分析重组cce4228蛋白的理化性质通过在线网站http://web.expasy.org/protparam/进行预测分析。cce4228蛋白与其他来源的SSADH蛋白用MEGA 6.0软件中ClustalW方法进行多序列比对并构建系统进化树。
1.8 重组cce4228蛋白的三维结构预测重组cce4228蛋白的三级结构通过在线网站http://swissmodel.expasy.org/预测。蛋白质模型结构由Pymol软件根据预测生成的pdb文件制作。
2 结果与分析 2.1 蓝杆藻ATCC51142中cce4228基因的克隆根据蓝杆藻ATCC51142中cce4228基因序列设计分别带有Nde I和Xho I酶切位点的上、下游引物(表 1),以蓝杆藻ATCC51142基因组为模板进行PCR扩增,产物经1%琼脂糖凝胶电泳检测,获得大小约为1.4 kb的特异性产物,该基因片段大小与蓝杆藻ATCC51142中cce4228基因理论碱基数(1 368 bp)相吻合(图 1)。对构建的pET-28a- cce4228重组质粒的测序结果表明:获得的基因序列与蓝杆藻ATCC51142中的cce4228基因序列(GenBank登录号WP_009543699)一致性为100%。因此该重组质粒用于后期进行原核表达实验。
|
| 图 1 蓝杆藻ATCC51142中cce4228基因的PCR扩增 Figure 1 PCR cloning of cce4228 gene 注:M:DNA分子标准;1、2:PCR产物. Note: M: DNA marker; 1, 2: PCR product. |
|
|
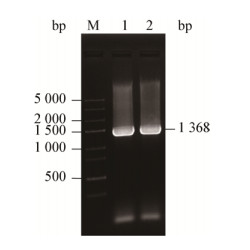
将pET-28a空载体和测序正确的pET-28a- cce4228重组质粒分别转化大肠杆菌BL21(DE3)感受态细胞后进行IPTG诱导表达,结果见图 2。由图 2可见,泳道1在约50 kD处未有大量蛋白表达,然而泳道4在该处有明显的蛋白过表达。蓝杆藻ATCC51142中cce4228基因编码一个49.3 kD的蛋白质,与泳道4中的蛋白质大小接近。此外,泳道5-6表明细胞破碎后大部分重组cce4228蛋白释放到上清液中,而在沉淀中的目的蛋白量相对较少。以上结果表明pET-28a-cce4228重组质粒在大肠杆菌表达体系中能够正确表达,并且重组cce4228具有非常好的溶解性。
|
| 图 2 重组cce4228蛋白在大肠杆菌BL21(DE3)中的表达 Figure 2 Expression of pET-28a plasmid and pET-28a-cce4228 plasmid 注:M:蛋白质标准;1-3:转化pET-28a空载体的大肠杆菌细胞诱导后破碎液,离心后上清液和离心后沉淀悬浮液;4-6:转化pET-28a-cce4228重组质粒的大肠杆菌细胞诱导后破碎液,离心后上清液和离心后沉淀悬浮液. Note: M: Protein marker; 1-3: Cell lysate, supernatant and pellet suspension of pET-28a after induction; 4-6: Cell lysate, supernatant and pellet suspension of pET-28a-cce4228 after induction. |
|
|
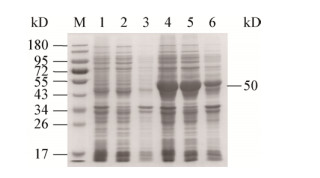
将离心并过滤得到的含有目的蛋白的上清液过平衡缓冲液(20 mmol/L Tris-HCl,pH 7.5)处理好的Ni-NTA树脂,然后用含有20-100 mmol/L咪唑的平衡缓冲液进行洗脱,纯化结果如图 3所示。SDS-PAGE结果表明当咪唑浓度增加到60 mmol/L时,少量重组cce4228蛋白被洗脱,而大部分重组蛋白在咪唑浓度为100 mmol/L时被洗脱。合并图 3中泳道3-8的洗脱液,并对其进行脱盐浓缩,得到纯度大于90%的目的蛋白。用Bradford方法测定浓缩后的重组蛋白的浓度为6.75 g/L (135 μmol/L),目的蛋白的收率为15.6 mg/g菌体。
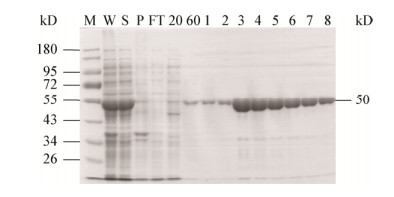
|
| 图 3 重组cce4228蛋白亲和层析纯化组分的电泳分析 Figure 3 SDS-PAGE of cce4228 protein purification 注:M:蛋白质标准;W:细胞破碎液;S:离心后上清液;P:离心后沉淀重悬液;FT:流穿液;20:20 mmol/L咪唑洗脱液;60:60 mmol/L咪唑洗脱液;1-8:100 mmol/L咪唑洗脱液. Note: M: Protein marker; W: pET-28a-cce4228 cell lysate; S: Supernatant; P: Pellet; FT: Flow-through; 20: 20 mmol/L imidazole elution; 60: 60 mmol/L imidazole elution; 1-8: 100 mmol/L imidazole elution. |
|
|
金属离子对重组cce4228蛋白催化琥珀酸半醛氧化成琥珀酸的活性有显著影响。相比较无金属离子添加时,Mg2+、Mn2+、Ca2+离子对cce4228蛋白有激活作用,Mg2+对cce4228蛋白的激活作用最显著;然而Ni2+和Cu2+离子会明显抑制cce4228蛋白的催化活性,Co2+离子会部分抑制cce4228蛋白的催化活性,当添加2 mmol/L Zn2+时cce4228蛋白的催化活性不能被检测到(图 4A)。在标准试验条件下,在不同Mg2+浓度下cce4228蛋白的动力学活性符合米氏方程(图 4B)。测得的最大初始反应速度(Vmax)为0.316±0.005 μmol/(L·s),重组cce4228蛋白与Mg2+的结合常数(Km)为0.006 2±0.000 7 mmol/L,反应所用的cce4228浓度为0.135 μmol/L,因此可以计算得到kcat为2.34 s–1,kcat/Km为3.77×105 L/(mol·s)。
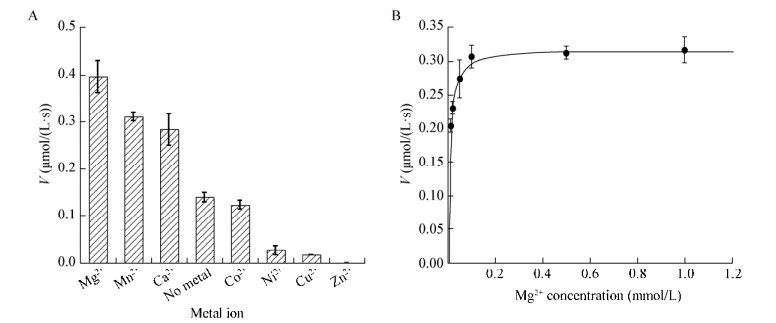
|
| 图 4 金属离子对重组cce4228蛋白催化活性的影响 Figure 4 Effects of metal ions on the catalytic activity of cce4228 protein 注:A:添加不同金属离子时的最大初始反应速度;B:不同Mg2+浓度时的最大初始反应速度.数据以平均值±标准差表示,n=3. Note: A: The maximum initial velocities with addition of different metal ions; B: The maximum initial velocities at different Mg2+ concentrations. Data are expressed as the mean±standard deviation, n=3. |
|
|
反应体系pH值对重组cce4228蛋白催化活性的影响如图 5所示。选定的pH范围为5.6-11.0,在不同pH值缓冲液体系中cce4228蛋白表现出不同的催化活性。当缓冲液体系在50 mmol/L CHES,pH 9.0时,cce4228蛋白的催化活性最高;相比较而言,在碱性条件下cce4228蛋白的催化活性比酸性条件下更高;当反应缓冲液pH低于7.0时,cce4228蛋白的催化活性急剧减弱。
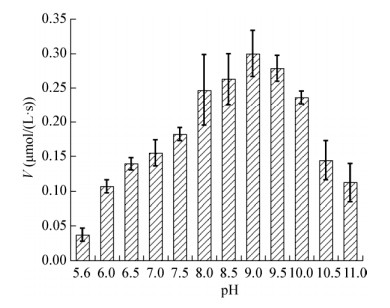
|
| 图 5 反应体系pH值对重组cce4228蛋白催化活性的影响 Figure 5 Effects of pH on the catalytic activity of cce4228 protein |
|
|
在标准试验条件下,当用琥珀酸半醛作为底物,NADP+或NAD+为辅因子时,重组cce4228蛋白的动力学活性符合由米氏方程推导的完全抑制方程:V=Vmax/(1+Km/[S]+[S]/Ki)。详细酶动力学参数如表 2所示。以NADP+ (0.5 mmol/L)作为辅因子时,测得的Vmax为0.26±0.02 μmol/(L·s),cce4228蛋白与底物的Km为0.008±0.003 mmol/L,cce4228蛋白与底物的抑制常数(Ki)为0.8±0.2 mmol/L,反应所用的cce4228蛋白的浓度为0.067 5 μmol/L,因此可以计算得到kcat为3.85 s-1,kcat/Km为4.81×105 L/(mol·s) (图 6A)。当固定琥珀酸半醛浓度为0.2 mmol/L,改变NADP+浓度时,测得Vmax为0.239±0.008 μmol/(L·s),cce4228蛋白与NADP+的Km为0.034±0.002 mmol/L (图 6B),通过计算得到kcat为3.54 s-1,kcat/Km为1.04×105 L/(mol·s)。以NAD+ (10 mmol/L)作为辅因子时,测得的Vmax为0.23±0.01 μmol/(L·s),cce4228蛋白与底物的Km为0.004 1±0.000 5 mmol/L,cce4228蛋白与底物的Ki为0.052±0.004 mmol/L,反应所用的cce4228蛋白的浓度为0.135 μmol/L,因此可以计算得到kcat为1.70 s–1,kcat/Km为4.15×105 L/(mol·s) (图 6C)。当固定琥珀酸半醛浓度为0.02 mmol/L,改变NAD+浓度时,测得Vmax为0.35±0.01 μmol/(L·s),cce4228蛋白与NAD+的Km为1.72±0.17 mmol/L (图 6D),通过计算得到kcat为2.59 s–1,kcat/Km为1.51×103 L/(mol·s)。由此可以得出cce4228蛋白偏好使用NADP+辅因子,为一类NADP+-依赖型的琥珀酸半醛脱氢酶。
| Cofactors | Km (mmol/L) | Ki (mmol/L) | kcat (s-1) | kcat/Km (L/(mol·s)) | Km (mmol/L)* |
| NADP+ | 0.008±0.003 | 0.8±0.2 | 3.85 | 4.81×105 | 0.034±0.002 |
| NAD+ | 0.004 1±0.000 5 | 0.052±0.004 | 1.70 | 4.15×105 | 1.72±0.17 |
| 注:*:辅因子的米氏常数. Note: *: Km for cofactors. | |||||
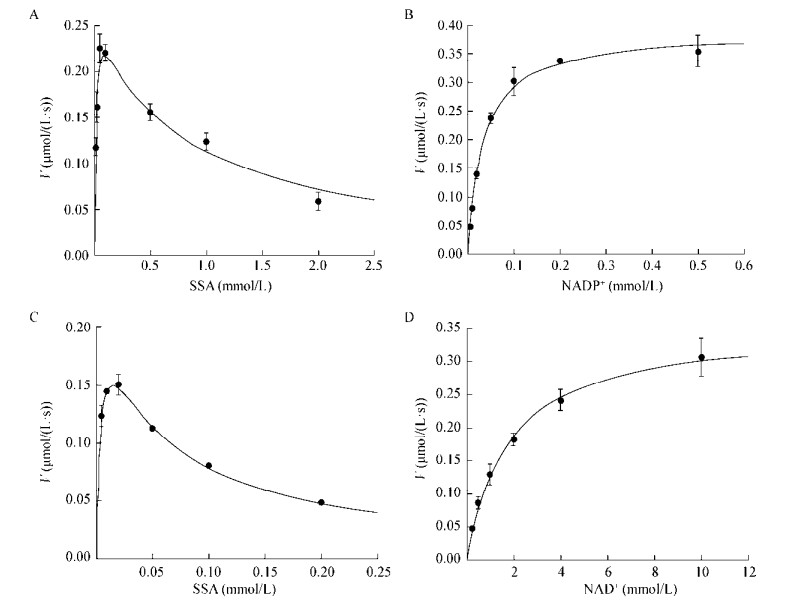
|
| 图 6 重组cce4228蛋白的酶动力学参数 Figure 6 Kinetic profiles of cce4228 protein 注:A:初始反应速度随琥珀酸半醛浓度的变化曲线,固定NADP+的浓度为0.5 mmol/L;B:初始反应速度随NADP+浓度的变化曲线,固定琥珀酸半醛浓度为0.2 mmol/L;C:初始反应速度随琥珀酸半醛浓度的变化曲线,固定NAD+的浓度为10 mmol/L;D:初始反应速度随NAD+浓度的变化曲线,固定琥珀酸半醛浓度为0.02 mmol/L. Note: A: Plot of the initial velocities as function of SSA concentrations, the concentration of NADP+ was fixed at 0.5 mmol/L; B: Plot of the initial velocities as function of NADP+ concentrations, the concentration of SSA was fixed at 0.2 mmol/L; C: Plot of the initial velocities as function of SSA concentrations, the concentration of NAD+ was fixed at 10 mmol/L; D: Plot of the initial velocities as function of NAD+ concentrations, the concentration of SSA was fixed at 0.02 mmol/L. |
|
|
蓝杆藻ATCC51142中的cce4228基因编码的蛋白质含有455个氨基酸残基。重组cce4228蛋白的理论相对分子质量为49 289.69 Da,理论等电点为4.77,摩尔消光系数ε为41 285 L/(mol·cm)。为分析cce4228蛋白与其他来源的SSADH蛋白的进化关系,利用MEGA 6.0软件中的ClustalW方法进行多序列比对,再用Neighbor-Joining (N-J)法进行1 000次Bootstrap构建系统进化树(图 7),其中不同来源的SSADH包括:拟南芥(Arabidopsis thaliana)、人(Human)、大肠杆菌(Escherichia coli GabD和Escherichia coli YneI)、枯草芽孢杆菌(Bacillus subtilis)、酿脓链球菌(Streptococcus pyogenes)、聚球藻(Synechococcus sp. PCC 7002)、鱼腥藻(Anabaena sp. PCC 7120)。图 7结果表明cce4228蛋白与聚球藻和鱼腥藻的SSADH蛋白为同一分支,而与拟南芥、人、大肠杆菌和枯草芽孢杆菌属于不同分支,表明蓝杆藻ATCC51142中的cce4228蛋白与聚球藻和鱼腥藻的SSADH (分别为a2771和all3556蛋白)亲缘关系较近。BLAST序列分析发现,蓝杆藻ATCC51142中cce4228蛋白与鱼腥藻PCC7120中all3556蛋白的氨基酸序列一致性高达73%,与聚球藻PCC7002中a2771蛋白的氨基酸序列一致性达到65%,而聚球藻PCC7002中的a2771和鱼腥藻PCC7120中的all3556蛋白均已被证实为NADP+-依赖型的琥珀酸半醛脱氢酶[4, 21, 24-25]。
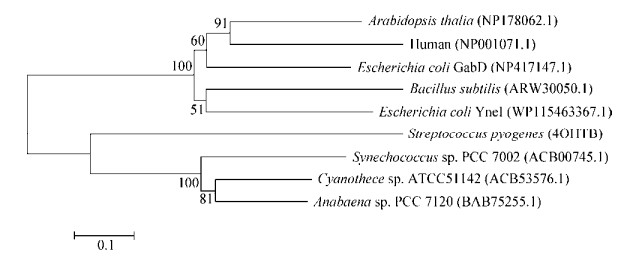
|
| 图 7 蓝杆藻ATCC51142中cce4228蛋白与其他来源的SSADH蛋白系统进化分析 Figure 7 Phylogenetic analysis of cce4228 with other SSADHs from different sources 注:0.1表示距离标尺;线表示分支长度(支长距离越近,差异越小,反之差异越大);数字表示自展值(> 50%认为可信,< 50%隐去). Note: 0.1 represents the distance ruler; Line represents branch length (The closer the branch length is, the smaller the difference, and the greater the difference); Number represents self-expanding value (> 50% believes credible, < 50% hides). |
|
|
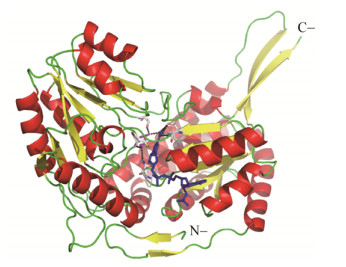
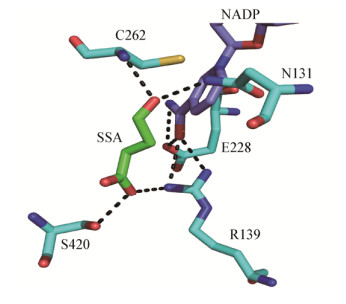
以聚球藻PCC7002中a2771蛋白晶体结构(PDB:3vz3)为模板对cce4228蛋白的三维结构进行建模(图 8)。由图 8可以发现cce4228蛋白的催化中心位于晶体结构的中心区域,NADP+辅因子采取一个伸展的构象,其酰胺基末端深入活性中心区域。cce4228蛋白催化中心的结构示意图如图 9所示。底物琥珀酸半醛的醛基氧原子与Cys262和Asn131的侧链均有氢键作用(距离均 < 0.3 nm,图中未显示),琥珀酸半醛的羧酸根氧原子与Ser420的侧链有氢键作用。此外,NADP+辅因子与Glu228和Arg139的侧链的氢键作用对稳定NADP+的伸展构象起着重要作用(图 9)。
|
| 图 8 重组cce4228蛋白模拟结构卡通示意图 Figure 8 Modeled cartoon structure of cce4228 注:关键氨基酸残基、琥珀酸半醛底物及NADP+辅因子均为棍棒结构,其颜色分别为浅灰色(碳原子)、紫色及蓝色. α-螺旋、β-折叠及无规则卷曲分别为红色、黄色和绿色. N-指蛋白质氮端,C-指蛋白质碳端.黑色虚线为催化中心的氢键. Note: Key residues, SSA substrate and NADP+ cofactor are showed as sticks, represented by light gray (carbon atom), magenta and blue, respectively. The α-helix, β-strands and loops are depicted as red, yellow and green, respectively. N-indicates the N-terminal and C-indicates the C-terminal of cce4228. The hydrogen bonds are shown as black dotted lines. |
|
|
|
| 图 9 重组cce4228蛋白催化中心示意图 Figure 9 Stick view of catalytic center structure of cce4228 注:SSA指琥珀酸半醛(碳原子为绿色),NADP为烟酰胺腺嘌呤二核苷酸磷酸辅因子(紫色碳链),关键氨基酸残基(碳原子为浅蓝色),数字为其在氨基酸序列中的位置.黑色虚线为催化中心的氢键. Note: SSA indicates succinic semialdehyde (green carbon), NADP indicates the nicotinamide adenine dinucleotide phosphate cofactor (purple carbon chain) and the numbers next to the key residues (cyano carbon) are the positions in the amino acids sequence of cce4228. The hydrogen bonds are showed as black dotted lines. |
|
|
由于在蓝藻中没有检测到α-酮戊二酸脱氢酶基因,长期以来蓝藻被认为不具备完整的TCA循环途径。最近的研究发现蓝藻可以通过α-酮戊二酸和γ-氨基丁酸两条代谢旁路使得TCA循环变得完整[26]。琥珀酸半醛是这两条代谢旁路的共同中间代谢物,而SSADH的催化底物正是琥珀酸半醛,催化产物是琥珀酸。因此SSADH在生物体内不但可以降低具有细胞毒性的琥珀酸半醛浓度,还可以生成为TCA循环提供碳源的琥珀酸。
SSADH属于醛脱氢酶超家族第五亚族[27-28]。该家族酶需要NADP+或NAD+辅因子参与电子传递。此外,研究发现SSADH存在明显的非竞争性底物抑制现象,其抑制作用结构基础也得到阐明,即高浓度的琥珀酸半醛底物通过占据辅因子(NADP+或NAD+)结合中心从而抑制SSADH活性[29]。酶动力学结果发现cce4228蛋白与NADP+辅因子的结合能力是与NAD+辅因子结合能力的50倍(表 2、图 6B和6D),同时使用饱和浓度的NADP+辅因子时cce4228蛋白的催化速度常数也是使用饱和浓度的NAD+辅因子时的2倍左右(表 2、图 6A和6C)。本研究同样发现cce4228蛋白存在底物抑制现象,但是使用不同的辅因子时底物抑制浓度有所不同。以NADP+为辅因子时琥珀酸半醛浓度超过0.1 mmol/L时才开始对cce4228蛋白活性有抑制作用,然而以NAD+为辅因子时琥珀酸半醛浓度超过0.02 mmol/L时就开始抑制cce4228蛋白催化活性(图 6A和6C)。因为底物抑制是底物通过占据辅因子的结合中心来实现的,而cce4228蛋白与NADP+辅因子的结合能力要大于与NAD+辅因子的结合能力,因此使用NADP+辅因子时需要更高浓度的底物才会抑制cce4228催化活性。此外,与其他来源的SSADH类似,不同的金属离子和反应体系pH对cce4228蛋白的催化活性有较大影响(图 4和图 5)。
生物信息学分析结果表明蓝杆藻ATCC51142的cce4228蛋白与同为蓝藻的聚球藻PCC7002和鱼腥藻PCC7120来源的SSADH亲缘关系较近(图 7)。事实上,本研究发现cce4228蛋白的酶动力学性质也和鱼腥藻PCC7120的SSADH类似[8]。建模结构分析发现cce4228蛋白的活性中心氨基酸残基与来自聚球藻PCC7002的SSADH高度保守[21, 25],这就为进一步深入研究cce4228蛋白的结构功能关系提供了极大便利。此外,与鱼腥藻PCC7120中的SSADH相同,在cce4228蛋白中与NADP+辅因子的磷酸根基团临近的氨基酸残基为Ser157,这一残基是否决定了cce4228蛋白的辅因子偏好性值得进一步研究。
4 结论本研究通过分子生物学手段成功得到pET-28a-cce4228重组质粒并将其转化至大肠杆菌BL21(DE3)中进行原核表达,通过IPTG诱导表达和Ni-NTA亲和层析纯化获得纯度大于90%的重组cce4228蛋白。运用生物信息学手段对cce4228蛋白的理化性质进行分析,cce4228蛋白与其他不同来源的SSADH蛋白的系统进化分析结果表明cce4228蛋白与聚球藻PCC7002和鱼腥藻PCC7120的SSADH为同一分支且亲缘关系较近。酶动力学结果证实蓝杆藻ATCC51142的cce4228蛋白是依赖于NADP+辅因子的GabD类的SSADH。本研究结果为阐明cce4228蛋白作为SSADH的催化反应机制奠定了重要基础。
| [1] |
Falcón LI, Magallón S, Castillo A. Dating the cyanobacterial ancestor of the chloroplast[J]. The ISME Journal, 2010, 4(6): 777-783. DOI:10.1038/ismej.2010.2 |
| [2] |
Stanier RY, Cohen-Bazire G. Phototrophic prokaryotes: the cyanobacteria[J]. Annual Review of Microbiology, 1977, 31: 225-274. DOI:10.1146/annurev.mi.31.100177.001301 |
| [3] |
Alagesan S, Gaudana SB, Sinha A, et al. Metabolic flux analysis of Cyanothece sp. ATCC 51142 under mixotrophic conditions[J]. Photosynthesis Research, 2013, 118(1/2): 191-198. |
| [4] |
Zhang SY, Bryant DA. The tricarboxylic acid cycle in cyanobacteria[J]. Science, 2011, 334(6062): 1551-1553. DOI:10.1126/science.1210858 |
| [5] |
Steinhauser D, Fernie AR, Araújo WL. Unusual cyanobacterial TCA cycles: not broken just different[J]. Trends in Plant Science, 2012, 17(9): 503-509. DOI:10.1016/j.tplants.2012.05.005 |
| [6] |
Welsh EA, Liberton M, Stöckel J, et al. The genome of Cyanothece 51142, a unicellular diazotrophic cyanobacterium important in the marine nitrogen cycle[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(39): 15094-15099. DOI:10.1073/pnas.0805418105 |
| [7] |
Xie CC, Xu WW, Li ZM, et al. Cloning and expression of α-ketoglutarate decarboxylase from Cyanothece sp. ATCC 51142[J]. Jiangsu Agricultural Sciences, 2018, 46(19): 37-39. (in Chinese) 谢琮琮, 许文武, 李至敏, 等. 蓝杆藻ATCC 51142中α-酮戊二酸脱羧酶的克隆与表达[J]. 江苏农业科学, 2018, 46(19): 37-39. |
| [8] |
Wang XQ, Lai CD, Lei GF, et al. Kinetic characterization and structural modeling of an NADP+-dependent succinic semialdehyde dehydrogenase from Anabaena sp. PCC7120[J]. International Journal of Biological Macromolecules, 2018, 108: 615-624. DOI:10.1016/j.ijbiomac.2017.12.059 |
| [9] |
Zhang SY, Qian X, Chang S, et al. Natural and synthetic variants of the tricarboxylic acid cycle in cyanobacteria: introduction of the GABA shunt into Synechococcus sp. PCC 7002[J]. Frontiers in Microbiology, 2016, 7: 1972. |
| [10] |
Schneider BL, Reitzer L. Pathway and enzyme redundancy in putrescine catabolism in Escherichia coli[J]. Journal of Bacteriology, 2012, 194(15): 4080-4088. DOI:10.1128/JB.05063-11 |
| [11] |
Fait A, Fromm H, Walter D, et al. Highway or byway: the metabolic role of the GABA shunt in plants[J]. Trends in Plant Science, 2008, 13(1): 14-19. |
| [12] |
Kim KJ, Pearl PL, Jensen K, et al. Succinic semialdehyde dehydrogenase: biochemical-molecular-clinical disease mechanisms, redox regulation, and functional significance[J]. Antioxidants & Redox Signaling, 2011, 15(3): 691-718. |
| [13] |
Ludewig F, Hüser A, Fromm H, et al. Mutants of GABA transaminase (POP2) suppress the severe phenotype of succinic semialdehyde dehydrogenase (ssadh) mutants in arabidopsis[J]. PLoS One, 2008, 3(10): e3383. DOI:10.1371/journal.pone.0003383 |
| [14] |
Toyokura K, Watanabe K, Oiwaka A, et al. Succinic semialdehyde dehydrogenase is involved in the robust patterning of Arabidopsis leaves along the adaxial-abaxial axis[J]. Plant and Cell Physiology, 2011, 52(8): 1340-1353. DOI:10.1093/pcp/pcr079 |
| [15] |
Wang LH, Yang ZW, Zhu L, et al. Cloning and prokaryotic expression of sweet sorghum succinic semialdehyde dehydrogenase SbSSADH[J]. Biotechnology Bulletin, 2015, 31(7): 83-90. (in Chinese) 王龙海, 杨泽伟, 朱莉, 等. 甜高粱琥珀酸半醛脱氢酶SbSSADH基因克隆及原核表达[J]. 生物技术通报, 2015, 31(7): 83-90. |
| [16] |
Kim YG, Lee S, Kwon OS, et al. Redox-switch modulation of human SSADH by dynamic catalytic loop[J]. The EMBO Journal, 2009, 28(7): 959-968. DOI:10.1038/emboj.2009.40 |
| [17] |
Parviz M, Vogel K, Gibson KM, et al. Disorders of GABA metabolism: SSADH and GABA-transaminase deficiencies[J]. Journal of Pediatric Epilepsy, 2014, 3(4): 217-227. |
| [18] |
Lin CY, Weng WC, Lee WT. A novel mutation of ALDH5A1 gene associated with succinic semialdehyde dehydrogenase deficiency[J]. Journal of Child Neurology, 2014, 30(4): 486-489. |
| [19] |
Niemi AK, Brown C, Moore T, et al. Evidence of redox imbalance in a patient with succinic semialdehyde dehydrogenase deficiency[J]. Molecular Genetics and Metabolism Reports, 2014, 1: 129-132. DOI:10.1016/j.ymgmr.2014.02.005 |
| [20] |
Park SA, Park YS, Lee KS. Kinetic characterization and molecular modeling of NAD(P)+-dependent succinic semialdehyde dehydrogenase from Bacillus subtilis as an ortholog YneI[J]. Journal of Microbiology and Biotechnology, 2014, 24(7): 954-958. DOI:10.4014/jmb.1402.02054 |
| [21] |
Park J, Rhee S. Structural basis for a cofactor-dependent oxidation protection and catalysis of cyanobacterial succinic semialdehyde dehydrogenase[J]. Journal of Biological Chemistry, 2013, 288(22): 15760-15770. DOI:10.1074/jbc.M113.460428 |
| [22] |
Zheng HY, Beliavsky A, Tchigvintsev A, et al. Structure and activity of the NAD(P)+-dependent succinate semialdehyde dehydrogenase YneI from Salmonella typhimurium[J]. Proteins, 2013, 81(6): 1031-1041. DOI:10.1002/prot.24227 |
| [23] |
Jang EH, Park SA, Chi YM, et al. Kinetic and structural characterization for cofactor preference of succinic semialdehyde dehydrogenase from Streptococcus pyogenes[J]. Molecules and Cells, 2014, 37(10): 719-726. DOI:10.14348/molcells.2014.0162 |
| [24] |
Wang XQ, Li ZM, Lei GF, et al. Preliminary characterization and structural analysis of succinic semialdehyde dehydrogenase from Anabaena sp. PCC7120[J]. Life Science Research, 2017, 21(4): 318-324. (in Chinese) 王小琴, 李至敏, 雷国风, 等. 鱼腥藻PCC7120中琥珀酸半醛脱氢酶的初步表征及结构分析[J]. 生命科学研究, 2017, 21(4): 318-324. |
| [25] |
Yuan ZN, Yin B, Wei DZ, et al. Structural basis for cofactor and substrate selection by cyanobacterium succinic semialdehyde dehydrogenase[J]. Journal of Structural Biology, 2013, 182(2): 125-135. DOI:10.1016/j.jsb.2013.03.001 |
| [26] |
Xiong W, Brune D, Vermaas WFJ. The γ-aminobutyric acid shunt contributes to closing the tricarboxylic acid cycle in Synechocystis sp. PCC 6803[J]. Molecular Microbiology, 2014, 93(4): 786-796. DOI:10.1111/mmi.12699 |
| [27] |
Perozich J, Nicholas H, Wang BC, et al. Relationships within the aldehyde dehydrogenase extended family[J]. Protein Science, 1999, 8(1): 137-146. |
| [28] |
Kirch HH, Bartels D, Wei YL, et al. The ALDH gene superfamily of Arabidopsis[J]. Trends in Plant Science, 2004, 9(8): 371-377. DOI:10.1016/j.tplants.2004.06.004 |
| [29] |
Jang EH, Park SA, Chi YM, et al. Structural insight into the substrate inhibition mechanism of NADP+-dependent succinic semialdehyde dehydrogenase from Streptococcus pyogenes[J]. Biochemical and Biophysical Research Communications, 2015, 461(3): 487-493. DOI:10.1016/j.bbrc.2015.04.047 |
 2019, Vol. 46
2019, Vol. 46