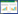扩展功能
文章信息
- 刘珊珊, 刘元元, 余彬彬, 成凤凤, 田文辉, 祝建波, 王爱英
- LIU Shan-Shan, LIU Yuan-Yuan, YU Bin-Bin, CHENG Feng-Feng, TIAN Wen-Hui, ZHU Jian-Bo, WANG Ai-Ying
- 新疆巴音布鲁克草原白蘑蘑菇圈土壤真菌多样性分析
- Soil fungi diversity analysis of Tricholoma mongolicum mushroom ring in Bayinbuluke grassland, Xinjiang
- 微生物学通报, 2019, 46(11): 2909-2918
- Microbiology China, 2019, 46(11): 2909-2918
- DOI: 10.13344/j.microbiol.china.181044
-
文章历史
- 收稿日期: 2018-12-20
- 接受日期: 2019-03-28
- 网络首发日期: 2019-04-23
白蘑(Tricholoma mongolicum Imai)俗称蒙古口蘑、白口蘑、钉子菇等,隶属于担子菌门(Basidiomycota)伞菌目(Agaricales)白蘑科(Tricholomaceae)白蘑属(Tricholoma)[1],主要分布在我国内蒙古、黑龙江、辽宁、吉林、河北等省(区)的草原地区,在新疆主要分布于巴音布鲁克草原,每年七八月为其盛产期,形成大小不一的蘑菇圈,蘑菇圈的形状多数为圆形,也有少数呈现半圆或者弧形[1]。白蘑肉质鲜美,不仅富含蛋白质和多种维生素,而且富含钙、磷、铁等矿物质[2],同时具有医疗保健作用[1]。白蘑至今无法实现人工栽培,加上人们掠夺式的采食、草原环境的破坏,这一珍贵野生食用菌濒临灭绝。
1807年Wollaston首次报道蘑菇圈是由真菌引起的[3],目前已经发现超过60余种担子菌会形成蘑菇圈[4]。蘑菇圈上蘑菇子实体呈圈状或带状分布,其圈上植物浓绿并且明显高于两侧植物[5],蘑菇圈每年向外生长,目前关于蘑菇圈的生长特性研究较少。赵吉等对蒙古口蘑形成的蘑菇圈各环带植物生长状况、土壤特征、微生物类群的分布进行研究,发现蘑菇圈上土壤养分含量显著增加,好气性细菌和芽孢细菌数量在枯草环的数量明显高于绿草环、圈内、圈心和圈外[6]。王芳等对蒙古口蘑蘑菇圈土壤真菌进行分离培养,发现蘑菇圈外真菌种类较圈上丰富[7]。蘑菇圈是由土壤-微生物-植物构成的完整系统,其中的微生物部分的研究目前主要是通过分离培养的方法,这使得不可培养微生物被忽略,限制了对蘑菇圈微生物群落构成及其潜在功能的了解。
高通量测序技术是目前广泛应用的新一代测序技术[8],在分析微生物的群落结构方面具有独特的优势,通过从环境样本中直接获取总DNA并构建文库,对微生物遗传物质中保守区序列的可变区域进行测定和比对,分析土壤中微生物物种组成和群落结构的多样性。高通量测序技术具有高效、信息全面、丰富等特点,为研究微生物生态提供了有利手段[9]。由于白蘑分布地理位置的特点,目前对白蘑的研究较少,应用高通量测序技术研究白蘑蘑菇圈土壤微生物多样性及群落结构还鲜见报道。杨岳等利用高通量测序技术对蒙古口蘑子实体下土壤真菌类型及群落结构组成进行了研究,确定蒙古口蘑子实体下主要优势真菌类群多为世界广布属,且营腐生或寄生生活,或兼具两种营养方式[10]。土壤微生物群落结构是一个十分复杂的环境,其与蘑菇圈的形成以及生长之间的关系有待深入研究。本文采用高通量测序的方法对新疆巴音布鲁克草原上白蘑蘑菇圈的圈上及两侧土壤真菌物种组成和群落构成特点进行分析,初步探索土壤真菌微生物类群对白蘑蘑菇圈形成以及蘑菇圈生态演化过程的影响,以期为白蘑的驯化栽培提供理论依据。
1 材料与方法 1.1 土壤样品采集巴音布鲁克草原属天山半湿润高山气候区,海拔2 400 m以上,气候寒冷、干旱,无霜期短,年平均气温为-4.5 ℃,年平均降水量为276.9 mm[11]。
2017年8月中下旬,在新疆巴音布鲁克草原采集两个相聚100 m左右的白蘑蘑菇圈样地,标记为XB.1和XB.2,每个样地分别选择3个蘑菇圈,在每个蘑菇圈的圈外、圈上、圈内采用五点取样法(采样位置如图 1所示),每个位置去除表面腐殖土层后,挖取0-20 cm剖面土壤样品保存于灭菌的密封袋中,并将5个点的土样混合均匀,做好标记,置于冰袋中冷藏保存。将混合好的土样分为两份,一份土样在-80 ℃下保存,供土壤微生物种群结构多样性分析;另一份土壤风干后供理化性质测定。样品分组及编号为:XB1.W(1-3)、XB1.N(1-3)、XB1.S(1-3)分别表示第1个样地蘑菇圈外、圈内、圈上,1-3表示3个重复;同样XB2.W(1-3)、XB2.N(1-3)、XB2.S(1-3)表示第2个样地。
|
| 图 1 白蘑的蘑菇圈照片(A)及土壤样品的采集示意图(B) Figure 1 Photograph of fairy circle (A) and collection of soil samples (B) |
|
|
PowerSoil® DNA Isolation Kit,Mo BioLaboratories公司;Phusion® High-Fidelity PCR Master Mix with GC Buffer,New England Biolabs公司;TruSeq® DNA PCR-Free Sample Preparation Kit、HiSeq2500仪器,Illumina公司。
1.3 土壤样品微生物总DNA提取及高通量测序将土壤样品碾碎过筛,用去腐殖质的试剂盒提取土壤总DNA并检测浓度,以稀释后的基因组DNA为模板,使用带Barcode的真菌特异引物ITS5-1737F (5′-GGAAGTAAAAGTCGTAACAAG G-3′)和ITS2-2043R (5′-GCTGCGTTCTTCATCGA TGC-3′)以及ITS3-202F (5′-GCATCGATGAAGAA CGCAGC-3′)和ITS4-2409R (5′-TCCTCCGCTTAT TGATATGC-3′)对真菌的ITS1区域和ITS2区域进行PCR扩增。PCR反应体系:2×Phusion Master Mix 15 μL,正、反向引物(2 μmol/L)各3 μL,基因组DNA (1 ng/μL) 7 μL,ddH2O 2 μL。PCR反应条件:94 ℃ 1 min;94 ℃ 20 s,56 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。扩增后PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测;根据PCR产物浓度对样品进行等量混和,完全混匀后同样使用2%的琼脂糖凝胶电泳检测PCR产物,对目的条带使用胶回收试剂盒回收产物。使用TruSeq® DNA PCR-Free Sample Preparation Kit进行文库构建,构建好的文库检验合格后,使用HiSeq 2500进行上机测序。
1.4 生物信息学分析测序获取原始数据(Raw data),首先对双端的Raw data数据进行质量过滤,得到质控后的有效数据(Clean data)[12]。利用FLASH等软件,根据双端(Paired-end,PE) Reads之间的Overlap关系对每对PE reads进行拼接,将成对的Reads拼成一条序列,获得原始拼接序列(Raw conting)。利用Mothur对拼接后的序列去除嵌合体及短序列进行质量控制及过滤,得到有效拼接片段(Clean tags)。然后基于有效数据,利用QIIME 1.7.0,采用Uclust的方法,以序列97%相似性为标准,进行OTU聚类和物种分类分析。
1.5 真菌多样性分析α-多样性通过Chao1指数、Simpson指数、Shannon指数等6个指数分析样品中的物种多样性,所有指数均使用QIIME 1.7.0进行计算分析。Chao1指数用来表征微生物群落丰富度,数值越大,说明群中含有的OTU数目越多,群落的丰富度越大。Simpson指数、Shannon指数用来评估微生物群落多样性,Shannon指数值越大,说明群落多样性越高,Simpson指数则与其他指数呈负相关关系[12]。Good_coverage指数用来表征样品文库的覆盖率。
β-多样性分析用于评估样品间物种组成的变化,是评估生物多样性的重要组成部分[13]。基于加权和未加权的Unifrc距离算法的柱坐标分析(Principal co-ordinates analysis,PCoA)分析比较样本间的相似性,通过非加权组平均法(Unweighted pair-group method with arithmetic means,UPGMA)对样品进行聚类分析。
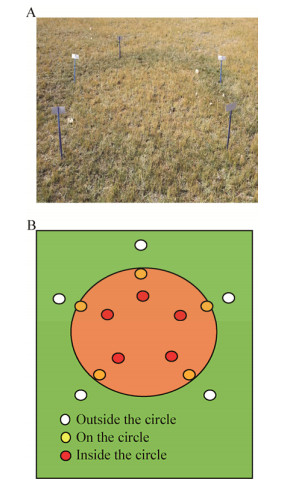
2 结果与分析 2.1 白蘑蘑菇圈土壤样品的测序结果根据测序的结果,滤除低质量和短序列读数后,18个样品总共得到50 193条优质序列;以97%的相似性对其进行聚类,总共得到809个OTU,其中蘑菇圈上的土壤样品中检测到的有效序列占比最高为87.01%,其次为蘑菇圈外81.74%和蘑菇圈内83.77%。图 2显示了每个分类学类别中真菌的分布,在蘑菇圈及其两侧的土壤中,82.60%的序列属于门和纲水平、目、科、属分类等级中所有的序列分别为:77.54%、66.75%、42.32%。
|
| 图 2 有效序列在每个分类等级的数量分布(门、纲、目、科、属、种) Figure 2 Distribution of the number of tags on each classification level (phylum, class, order, family, genus, species) 注:XB1、XB2分别表示白蘑蘑菇圈样地1、2,W、N、S表示圈外、圈内、圈上. Note: XB1 and XB2 respectively represent two mushroom circle plots, and W, N, S represent outside the mushroom circle, inside the circle, and on the circle. |
|
|
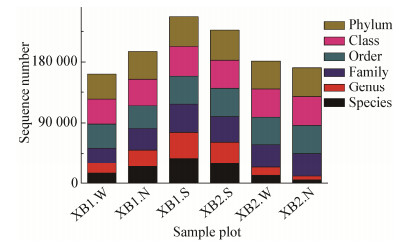
测序共获得了50 193条有效序列,对其进行注释得到809个OTU,分为5个门、26个纲、79个目、166个科、232个属。通过对样品进行种群组成分析,可以反映其在不同分类学水平上的群落结构,有利于分析样品之间的差异,从而分析其群落结构组成的特点。图 3A和3B分别显示纲(Class)水平和属(Genus)水平的群落结构分类比较结果。经过对测序结果分析可得:注释得到的26个纲中,相对丰度较高的前10个纲种类为伞菌纲(Agaricomycetes)、粪壳菌纲(Sordariomycetes),其次是散囊菌纲(Eurotiomycetes)、座囊菌纲(Dothideomycetes)、未定接合菌纲(Incertae_sedis_ Zygomycota)、盘菌纲(Pezizomycetes)、未定子囊菌纲(Incertae_sedis_Ascomycota)、古菌根菌纲(Archaeorhizomycetes)、锤舌菌纲(Leotiomycetes)以及球囊菌纲(Glomeromycetes) (图 3A)。
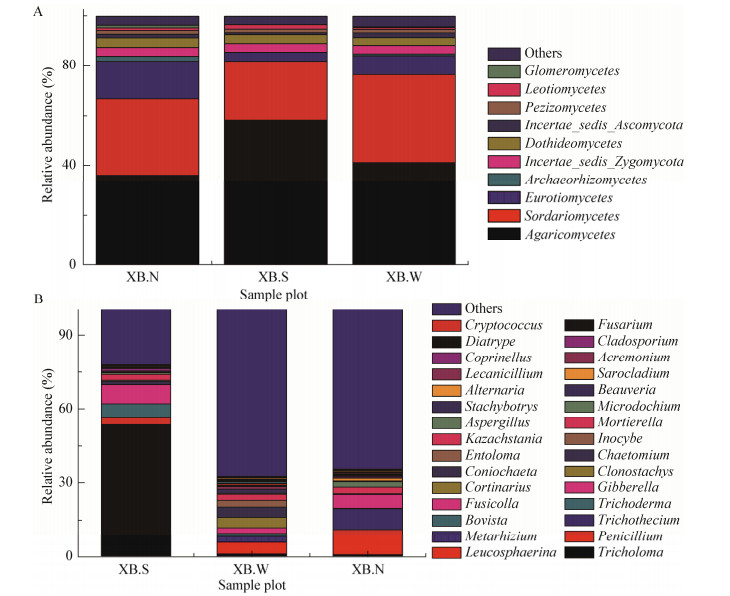
|
| 图 3 白蘑蘑菇圈土壤真菌群落在纲(A)、属(B)分类水平的相对丰度 Figure 3 Communities of fungi of T. mongolicum mushroom circle in the classification standards class (A), genus (B) of abundance 注:XB.W、XB.N、XB.S分别表示蘑菇圈外、圈内、圈上. Note: XB.W, XB.N, XB.S, represent soil samples outside the circle, inside the circle and on the circle. |
|
|
从属的分类水平分析,相对丰度较高的为口蘑属(Tricholoma)、青霉属(Penicillium)、单端孢属(Trichothecium)、木霉属(Trichoderma)、赤霉属(Gibberella)、螺旋聚孢霉属(Clonostachys)、毛壳属(Chaetomium)、丝盖伞属(Inocybe)、被孢霉属(Mortierella)、小羊蹄菌属(Microdochium) (图 3B)。蘑菇圈及其两侧土壤真菌的共有真菌属为青霉属、赤霉属和被孢霉属。圈上口蘑属和赤霉属的相对丰度分别达到53.5%和7.66%,两者在蘑菇圈上表现出非常明显的优势,说明蘑菇圈上真菌优势属的组成较为单一,口蘑属占据主导地位。青霉属在蘑菇圈外及蘑菇圈内相对丰度均为最高,但在蘑菇圈上相对丰度仅为蘑菇圈内的1/4。总体来说蘑菇圈上的真菌种类少于圈外和圈内,圈内与圈上共有属多于圈外与圈上共有属,此外,圈上真菌的种类及丰富度均低于圈外和圈内。毛壳属(Chaetomium)、锥毛壳菌属(Chaetomium)、枝孢属(Cladosporium)、赤霉属(Gibberella)、粉红粘帚霉(Clonostachys rosea)在蘑菇圈上相对丰度均高于圈外和圈内,其中锥毛壳属的相对丰度在圈上高于两侧8倍以上,推测它们可能与口蘑的生长具有正相关的关系。圈外相对丰度较高的真菌属有螺旋聚孢霉属(Clonostachys)、丝盖伞属(Inocybe)、被孢霉属(Mortierella)、单端孢属(Trichothecium),圈内相对丰度较高真菌种类有镰刀霉属(Fusarium)、木霉属(Trichoderma)、被孢霉属(Mortierella)、小羊蹄菌属(Microdochium)、Sarocladium、链格孢属(Alternaria)。
2.2.2 蘑菇圈不同位置土壤真菌群落组成变化蘑菇圈不同位置的土样中真菌菌群分布具有较明显的差异(表 1),蘑菇圈上伞菌纲(Agaricomycetes)显著高于圈外及圈内,伞菌纲群落在蘑菇圈上相对丰度达到59.69%,可以说明蘑菇圈上口蘑是占据绝对优势的种群,伞菌纲群落在蘑菇圈上的分布特点为圈上 > 圈外 > 圈内,由此可以推测蘑菇圈的生长是一个动态的向外扩展过程。散囊菌纲(Eurotiomycetes)和粪壳菌纲(Sordariomycetes)在圈上的相对丰度显著低于圈外和圈内,其中粪壳菌纲(Sordariomycetes)的分布特点为圈内 > 圈外 > 圈上,可以推测粪壳菌纲在蘑菇圈上的生长受抑制,锤舌菌纲(Leotiomycetes)在蘑菇圈上的相对丰度显著高于圈外和圈内,其他种群相对丰度在蘑菇圈及其两侧的分布差异并不显著。
| 纲水平 Class level |
XB.S | XB.N | XB.W |
| Agaricomycetes | 59.69±0.15a | 36.21±12.16b | 41.25±5.64b |
| Sordariomycetes | 22.60±0.57b | 30.8±5.18b | 35.41±2.61a |
| Eurotiomycetes | 2.80±0.48c | 15.18±2.20a | 7.66±1.50b |
| Archaeorhizomycetes | 0.02±0.01a | 1.9±3.26a | 0.74±0.95a |
| Leotiomycetes | 1.83±0.06a | 1.06±0.09b | 0.70±0.11c |
| Dothideomycetes | 3.63±0.09a | 3.86±0.78a | 3.14±0.57a |
| Incertae_sedis_Ascomycota | 1.81±0.04a | 1.93±0.39a | 1.57±0.28a |
| Pezizomycetes | 1.40±0.02a | 1.43±0.73a | 1.33±0.35a |
| Incertae_sedis_Zygomycota | 3.72±0.50a | 3.59±1.17a | 3.40±0.60a |
| Glomeromycetes | 0.06±0.00a | 1.06±0.97a | 0.33±0.35a |
| 注:XB.N、XB.S、XB.W分别表示蘑菇圈圈内、圈上、圈外土壤样品.不同小写字母表示0.05 (P < 0.05)水平下的差异分析,横向比较. Note: XB.N, XB.S, XB.W, represent soil samples inside the circle, on the circle and outside the circle. Different small letters mean significant differences at the 0.05 level (P < 0.05). | |||
α-多样性分析可以反映微生物分布的丰富度与均匀度。由表 2数据可以看出,物种数量范围为533-968,蘑菇圈上物种数量最低,并且显著低于圈外。Chao1指数和Ace指数范围分别为596-1 112和611-1 155,其中Chao1指数和Ace指数均以蘑菇圈上最低,且显著低于蘑菇圈外,蘑菇圈上和蘑菇圈内Chao1指数和Ace指数差异不显著。表征微生物群落多样性的Shannon指数范围为4.2-6.5,圈上最低,并且显著低于圈外和圈内。在蘑菇圈不同位置多样性指数分布特点为:圈外 > 圈内 > 圈上,说明蘑菇圈上土壤真菌物种数量较少,且群落多样性较差。通过对蘑菇圈不同位置多样性指数的分析,推测口蘑的生长可能与特定的真菌有关,可以影响周围真菌的定殖,形成特定的微生物群落结构。
| 土壤样品 Soil sample |
多样性指数Diversity index | |||
| 物种数量 Observed_species |
Shannon指数 Shannon index |
Chao1指数 Chao1 index |
Ace指数 Ace index |
|
| XB.N | 588±38.54b | 5.3±0.49b | 643.9±45.5b | 651.6±41.1b |
| XB.S | 533±43.02b | 4.2±0.16c | 596.1±46.7b | 611.4±52.1b |
| XB.W | 968±94.59a | 6.5±0.25a | 1 112.2±163.8a | 1 155.2±194.2a |
| 注:XB.N、XB.S、XB.W分别表示蘑菇圈圈内、圈上、圈外土壤样品.不同小写字母表示0.05 (P < 0.05)水平下的差异分析. Note: XB.N, XB.S, XB.W, represent soil samples inside the circle, on the circle and outside the circle. Different small letters mean significant differences at the 0.05 level (P < 0.05). | ||||
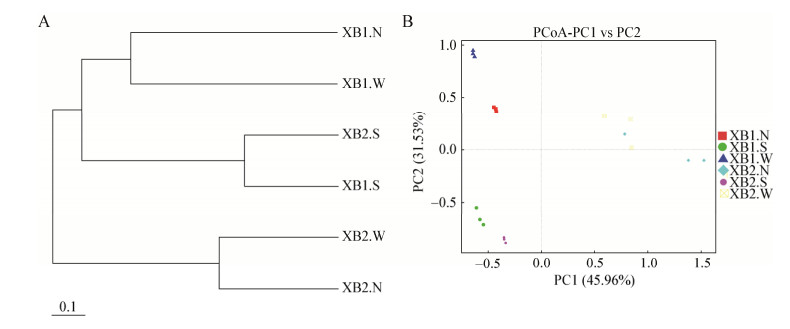
通过Bray-curtis距离,基于OTU水平对蘑菇圈及其两侧土壤样品进行UPGMA (图 4A)和主坐标分析(图 4B),两者的结果基本相同。使用加权的UniFrac距离矩阵进行分析(图 4A),蘑菇圈真菌群落主要分为3个主要群:XB1.W和XB1.N聚为一个群,XB1.S和XB2.S聚为一个群,说明在蘑菇圈上占主导的真菌群落结构相同,XB2.N和XB2.W独自聚为一个群,且圈外与圈上的亲缘关系较近。基于加权的UniFrac PCoA,通过物种组成计算得到距离矩阵,横、纵坐标表示OTU水平真菌群落结构的变异分别为45.96%和31.53%,由图 4B可以看出,在PC2维度上可以将蘑菇圈上的样品与蘑菇圈外及圈内样品分开,即PC2为造成圈上真菌群落与其他位置真菌群落产生差异的主成分。在PC1维度上,XB.1W和XB.1N的距离较近,表明在样地1的圈外和圈内两处土壤真菌的群落结构较为相似,并且样地2的蘑菇圈样品除圈上以外都与样地1样品距离较远,也就是说由于采样地的不同,其微生物群落之间存在差异。
|
| 图 4 白蘑蘑菇圈真菌群落结构的UPGMA聚类分析(A)和PCoA分析(B) Figure 4 UPGMA clustering analysis (A) and principal co-ordinates analysis (B) of the fungal community structure of T. mongolicum mushroom circle 注:A:蘑菇圈样地1;B:蘑菇圈样地2. W、N、S表示圈外、圈内、圈上. Note: A: Mushroom circle plots 1; B: Mushroom circle plots 2. W, N, S represent outside the mushroom circle, inside the circle, and on the circle. |
|
|
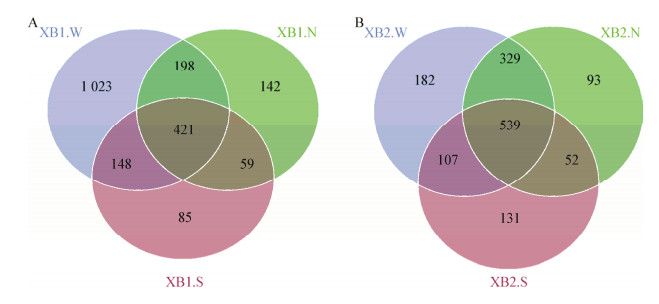
通过构建韦恩图对土壤样品中真菌物种组成进行分析(图 5A、B),蘑菇圈样地1 (图 5A)的土壤样本中(XB1.W、XB1.N、XB1.S)分别检测到1 790、820、713个OTU,其中样本共有的OTU为421个,各自特有的OTU为1 023个(占XB1.W总OTU的57.15%)、142个(占XB1.N总OTU的17.31%)、85个(占XB1.S总OTU的11.92%)。然而在样地2 (图 5B)的土壤样本中(XB2.W、XB2.N、XB2.S)分别检测到1 157、1 013、829个OTU,其中样本共有的OTU为539个,各自特有的OTU为182个(占XB2.W总OTU的15.73%)、93个(占XB2.N总OTU的9.18%)、121个(占XB2.S总OTU的14.60%)。对两个样地蘑菇圈上特有OTU进行分析,对蘑菇圈上特有OTU再取交集,得到9条蘑菇圈上特有OTU,分别为Xylodon nothofagi、Agaricales、Phaeococcomyces、Ochrocladosporium adansoniae、Coniochaeta ligniaria、未知子囊菌、Tomentella amyloapiculata、Dothideomycetes、Incertea_sedis_Helotiales,蘑菇圈上特有的OTU所代表的微生物极有可能与白蘑及蘑菇圈的发生有关。
|
| 图 5 白蘑蘑菇圈样地土壤样品真菌OTU数量韦恩图 Figure 5 Venn profile of OTU number in different soil samples in the mushroom circle of T. mongolicum 注:A:蘑菇圈样地1;B:蘑菇圈样地2. W、N、S表示圈外、圈内、圈上. Note: A: Mushroom circle plots 1; B: Mushroom circle plots 2. W, N, S represent outside the mushroom circle, inside the circle, and on the circle. |
|
|
草原蘑菇圈是十分值得关注的生态景观,我国学者于20世纪80年代开始对蘑菇圈进行相关研究[14]。研究主要分为几个方面:蘑菇圈土壤养分状况、蘑菇圈对植被及土壤的影响,蘑菇圈上细菌、真菌的分布等方面[15-18]。本研究利用新一代高通量测序的技术方法,分析白蘑蘑菇圈及其两侧土壤真菌组成及群落结构,结果表明白蘑蘑菇圈上与圈外和圈内真菌群落结构存在较大差异;圈上真菌多样性及特异性物种均低于圈外和圈内,这与王芳等[7]研究结果一致。蘑菇圈上的真菌分布特点是以白蘑为主的真菌群落结构,分析认为蘑菇圈对除白蘑以外的其他土壤真菌具有一定程度的抑制作用,蘑菇圈的形成与圈上土壤真菌的多样性及丰富度无正相关关系,可能与特异真菌物种相关。叶雷等通过高通量测序的方法对松茸菌柄细菌群落结构分析后发现,变形菌门对松茸的生长可能具有积极贡献[19]。Oh等对松茸产生的蘑菇圈与非蘑菇圈地区土壤的细菌多样性进行了研究,发现松茸圈上细菌多样性较低,在圈上形成以松茸为主的优势菌群,其他微生物受到抑制[20]。Antony-Babu等发现松露的形成与特定的微生物有关[21]。
本研究发现蘑菇圈上的真菌类群除口蘑属外,毛壳属(Chaetomium)、粉红粘帚霉(Clonostachys rosea)、锥毛壳属(Coniochaeta)丰度显著高于圈外及圈内,毛壳属(Chaetomium)、粉红粘帚霉(Clonostachys rosea)被用作防治植物病害的生防菌[22-23],毛壳菌可以有效降解纤维素和有机物,并对土壤中的其他微生物产生拮抗作用[22],此外,蘑菇圈上9种特异真菌多为腐生菌或者植物的内生菌,推测它们极有可能与白蘑的生长及蘑菇圈的形成相关,通过分解土壤有机质为白蘑及圈上植物的生长提供营养物质,同时又可以抑制其他真菌的生长,从而形成土壤-微生物-植物互惠互利的生态环境[6]。杨岳等对蒙古口蘑蘑菇圈子实体下土壤真菌群落结构分析发现,优势属中除口蘑为担子菌门外,其余都为子囊菌门,推测这些优势真菌如镰刀菌属、木霉属等在蒙古口蘑形成子实体时,可通过土壤中的有机物质或分解植物为蒙古口蘑提供出菇所必需的营养物质[10],本研究结果与其存在一定程度的相似性。张功等发现粪链球菌可以促进白口蘑菌丝的生长[24],赵吉等发现两种细菌——浸麻芽孢杆菌(Bacillus macerans)和德氏食酸菌(Acidovorax delafieldii)与蒙古口蘑子实体的发生关系密切[18]。
目前对于白蘑蘑菇圈不断向外生长的研究,被普遍接受的是营养学说[14]。宋超等认为蘑菇圈向外扩展是由于中心区蘑菇圈产生菌将土壤养分消耗殆尽,致使某些营养缺乏,因而菌丝向相反方向生长,由此形成不断向外生长的蘑菇圈环带[5]。本文通过对白蘑蘑菇圈及其两侧真菌种类分析之后发现,在蘑菇圈内相对丰度较高的真菌有镰刀霉属、被孢霉属、小羊蹄菌属、Sarocladium、链格孢属,枝孢属(Cladosporium)、青霉属(Trichoderma),大部分属于腐生菌或动植物病原菌[25-27],认为这也是造成蘑菇圈向外扩展的原因之一,即由于前一年圈上白蘑生长,为腐生菌及病原类真菌提供营养物质,使其大量繁殖,造成真菌群落失衡[20],白蘑生长的微生态环境遭到破坏,圈上植物生长受到抑制,无法给内生菌提供赖以生存的环境,因此菌丝向外生长。
蘑菇圈是草原上一种常见的生态现象,对草原植被的演变发挥重要的作用,本文对白蘑蘑菇圈及其两侧真菌物种组成和群落结构进行了深入分析,探讨了蘑菇圈产生菌白蘑与圈上其他真菌类群之间的关系,从微生物群落结构方面分析了蘑菇圈向外扩展的原因,为解析白蘑蘑菇圈生长发育及其子实体的发生提供理论依据。
| [1] |
Wu EQ, Tuli GE. Research adavancement on Tricholoma mongolicum[J]. Edible Fungi of China, 2007, 26(4): 3-5. (in Chinese) 吴恩奇, 图力古尔. 蒙古口蘑研究进展[J]. 中国食用菌, 2007, 26(4): 3-5. DOI:10.3969/j.issn.1003-8310.2007.04.001 |
| [2] |
Xie YJ, Sun GQ, Pang J, et al. Research advancement on wild edible mushroom Tricholoma mongolicum[J]. Edible and Medicinal Mushrooms, 2017, 25(1): 28-33. (in Chinese) 解亚杰, 孙国琴, 庞杰, 等. 草原野生食用菌蒙古口蘑的研究进展[J]. 食药用菌, 2017, 25(1): 28-33. |
| [3] |
Wollaston WH. On fairy-rings[J]. Philosophical Transactions of the Royal Society of London, 1807, 97: 133-138. DOI:10.1098/rstl.1807.0008 |
| [4] |
Kirk P Jr, Cannon P, Minter D, et al. Dictionary of the Fungi[M]. 10th ed. Wallingford: CABI, 2008: 507-508
|
| [5] |
Song C, Bau T. Fairy ring formation mchanism and its ecological significance[J]. Edible Fungi of China, 2007, 26(6): 9-13. (in Chinese) 宋超, 图力古尔. 蘑菇圈形成机理及其生态学意义[J]. 中国食用菌, 2007, 26(6): 9-13. DOI:10.3969/j.issn.1003-8310.2007.06.002 |
| [6] |
Zhao J, Shao YQ, Bao QH. Soil-vegetation system surround the fairy ring in steppe[J]. Chinese Journal of Ecology, 2003, 22(5): 43-46. (in Chinese) 赵吉, 邵玉琴, 包青海. 草原蘑菇圈的土壤-植物系统研究[J]. 生态学杂志, 2003, 22(5): 43-46. DOI:10.3321/j.issn:1000-4890.2003.05.010 |
| [7] |
Wang F, Lu T, Bau T. The diversity of soil fungi on Leucocalocybe mongolicum fairy ring[J]. Journal of Fungal Research, 2015, 13(2): 115-121. (in Chinese) 王芳, 鲁铁, 图力古尔. 蒙古白丽蘑蘑菇圈土壤真菌多样性[J]. 菌物研究, 2015, 13(2): 115-121. |
| [8] |
Niu SQ, Long Y, Li HY, et al. Microbial diversity in saline alkali soil from Hexi corridor analyzed by Illumina MiSeq high-throughput sequencing system[J]. Microbiology China, 2017, 44(9): 2067-2078. (in Chinese) 牛世全, 龙洋, 李海云, 等. 应用Illumina MiSeq高通量测序技术分析河西走廊地区盐碱土壤微生物多样性[J]. 微生物学通报, 2017, 44(9): 2067-2078. |
| [9] |
Zhou QW, Cao RJ, He HK, et al. Research progress of Baijiu-making microbes based on modern molecular biological technology[J]. Liquor-Making Science & Technology, 2017(6): 95-102. (in Chinese) 周庆伍, 曹润洁, 何宏魁, 等. 现代分子生物学技术对白酒酿造微生物的研究进展[J]. 酿酒科技, 2017(6): 95-102. |
| [10] |
Yang Y, Wei J. High-throughput sequencing analysis of the fungal diversity in the soil under the fairy ring fruitbodies of Leucocalocybe mongolicum[J]. Molecular Plant Breeding, 2018, 16(10): 3412-3422. (in Chinese) 杨岳, 魏杰. 高通量测序分析蒙古白丽蘑蘑菇圈子实体下土壤真菌多样性[J]. 分子植物育种, 2018, 16(10): 3412-3422. |
| [11] |
Xian J. Introduction of wild white mushroom in Bayinbuluke Area[J]. Xinjiang Agricultural Sciences, 2006, 43(S1): 241-242. (in Chinese) 先杰. 巴音布鲁克野生白蘑[J]. 新疆农业科学, 2006, 43(S1): 241-242. |
| [12] |
Xu YR, Fang ZJ, Lu XP, et al. Effects of starane on maize soil bacterial diversity analyzed by high-throughput sequencing technology[J]. Acta Microbiologica Sinica, 2017, 57(7): 985-993. (in Chinese) 许艳蕊, 方志军, 卢晓平, 等. 基于高通量测序技术分析使它隆对玉米土壤细菌多样性的影响[J]. 微生物学报, 2017, 57(7): 985-993. |
| [13] |
Chen SB, Ouyang ZY, Zheng H, et al. Latitudinal gradient in beta diversity of forest communities in America[J]. Acta Ecologica Sinica, 2011, 31(5): 1334-1340. (in Chinese) 陈圣宾, 欧阳志云, 郑华, 等. 美洲森林群落beta多样性的纬度梯度性[J]. 生态学报, 2011, 31(5): 1334-1340. |
| [14] |
Zhao YB, Hu MR, Leng GT, et al. Preliminary study on mushroom circle[J]. Microbiology China, 1985, 12(2): 56-58. (in Chinese) 赵勇斌, 胡美蓉, 冷观悌, 等. 蘑菇圈的初步研究[J]. 微生物学通报, 1985, 12(2): 56-58. |
| [15] |
Shao YQ, Zhao J. Study on dynamic distribution of soil microbial number of the fariy ring in steppe[J]. Grassland of China, 2000, 22(1): 47-50. (in Chinese) 邵玉琴, 赵吉. 草原蘑菇圈中土壤微生物类群数量的动态分布研究[J]. 中国草地, 2000, 22(1): 47-50. |
| [16] |
Zhao J, Sun W, Liu HY, et al. A comparative study on biochemical activities in soil around the fairy rings in grassland[J]. Acta Scientiarum Naturalium Universitatis NeiMongol, 1999, 30(1): 96-100. (in Chinese) 赵吉, 孙维, 柳海鹰, 等. 草原蘑菇圈土壤生物化学活性的比较研究[J]. 内蒙古大学学报:自然科学版, 1999, 30(1): 96-100. |
| [17] |
Shao YQ, Zhao J. A study on soil microbial distribution in the fairy rings of Tricholoma mongolicum[J]. Acta Scientiarum Naturalium Universitatis NeiMongol, 2000, 31(1): 81-83. (in Chinese) 邵玉琴, 赵吉. 蒙古口蘑蘑菇圈土壤微生物类群的分布研究[J]. 内蒙古大学学报:自然科学版, 2000, 31(1): 81-83. |
| [18] |
Zhao J, Shao YQ, Bao QH, et al. A comparative study on distribution of the bacteria in soil around fairy rings of Tricholoma mongolicum[J]. Acta Scientiarum Naturalium Universitatis NeiMongol, 1999, 30(1): 104-105. (in Chinese) 赵吉, 邵玉琴, 包青海, 等. 蒙古口蘑(Tricholoma mongolicum Imai.)蘑菇圈及其两侧的土壤细菌分布的比较研究[J]. 内蒙古大学学报:自然科学版, 1999, 30(1): 104-105. |
| [19] |
Ye L, Fu Y, Li Q, et al. Bacterial community structure of Tricholoma matsutake stipe soil by high-throughput sequencing[J]. Chinese Journal of Applied & Environmental Biology, 2018, 24(3): 583-588. (in Chinese) 叶雷, 付雨, 李强, 等. 高通量测序研究松茸菌柄土壤细菌群落结构[J]. 应用与环境生物学报, 2018, 24(3): 583-588. |
| [20] |
Oh SY, Fong JJ, Park MS, et al. Distinctive feature of microbial communities and bacterial functional profiles in Tricholoma matsutake dominant soil[J]. PLoS One, 2016, 11(12): e0168573. DOI:10.1371/journal.pone.0168573 |
| [21] |
Antony-Babu S, Deveau A, van Nostrand JD, et al. Black truffle-associated bacterial communities during the development and maturation of Tuber melanosporum ascocarps and putative functional roles[J]. Environmental Microbiology, 2014, 16(9): 2831-2847. DOI:10.1111/1462-2920.12294 |
| [22] |
Chi YJ, Yang Q. Biological control of plant diseases with Chaetomium spp. and the problems in its application[J]. System Sciences and Comprehensive Studies in Agriculture, 2002, 18(3): 215-218. (in Chinese) 迟玉杰, 杨谦. 毛壳菌对植物病害的生物防治及存在的问题[J]. 农业系统科学与综合研究, 2002, 18(3): 215-218. DOI:10.3969/j.issn.1001-0068.2002.03.016 |
| [23] |
Wang AX, Zhang LL, Wang X, et al. Study on growth-promoting effects and application patterns of Clonostachys rosea in tomato[J]. Journal of Northeast Agricultural University, 2015, 46(10): 37-44. (in Chinese) 王傲雪, 张莉莉, 王旭, 等. 粉红粘帚菌对番茄促生作用及施用方式研究[J]. 东北农业大学学报, 2015, 46(10): 37-44. |
| [24] |
Zhang G, Bao H. Studis on microbial lineage under T. mongolicum Lmai's mushroom growing ring area[J]. Journal of Inner Mongolia Teachers University (Natural Science Edition), 1990, 20(3): 45-47. (in Chinese) 张功, 包宏. 白口蘑蘑菇圈微生物区系研究初报[J]. 内蒙古师大学报:自然科学, 1990, 20(3): 45-47. |
| [25] |
Hu ZM, Zhang K, Zhou FH, et al. A case of cutaneous hyalohyphomycosis caused by Fusarium solani[J]. Chinese Journal of Mycology, 2009, 4(6): 358-360. (in Chinese) 胡志敏, 张开, 周飞红, 等. 茄病镰刀菌致皮肤透明丝孢霉病1例[J]. 中国真菌学杂志, 2009, 4(6): 358-360. DOI:10.3969/j.issn.1673-3827.2009.06.011 |
| [26] |
Gao F, Wu YH. Progresses in the biocontrol of plant diseases caused by Alternaria[J]. Plant Protection, 2008, 34(3): 1-6. (in Chinese) 高芬, 吴元华. 链格孢属(Alternaria)真菌病害的生物防治研究进展[J]. 植物保护, 2008, 34(3): 1-6. DOI:10.3969/j.issn.0529-1542.2008.03.001 |
| [27] |
Saravanakumar D, Lavanya N, Muthumeena K, et al. Fluorescent pseudomonad mixtures mediate disease resistance in rice plants against sheath rot (Sarocladium oryzae) disease[J]. BioControl, 2009, 54(2): 273-286. DOI:10.1007/s10526-008-9166-9 |
 2019, Vol. 46
2019, Vol. 46