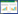扩展功能
文章信息
- 洪义国, 李益本, 吴佳鹏, 陈光仕, 黄天政, 陈帅
- HONG Yi-Guo, LI Yi-Ben, WU Jia-Peng, CHEN Guang-Shi, HUANG Tian-Zheng, CHEN Shuai
- 高通量测序分析多种典型生境中厌氧氨氧化细菌的多样性分布特征
- Diversity analysis of anaerobic ammonium-oxidizing bacteria in typical habitats by high throughput sequencing
- 微生物学通报, 2019, 46(11): 2865-2876
- Microbiology China, 2019, 46(11): 2865-2876
- DOI: 10.13344/j.microbiol.china.180913
-
文章历史
- 收稿日期: 2018-11-15
- 接受日期: 2019-04-18
- 网络首发日期: 2019-06-18
2. 广州大学环境科学与工程学院 广东 广州 510006
2. School of Environmental Science and Engineering, Guangzhou University, Guangzhou, Guangdong 510006, China
在1977年,Broda根据热力学计算分析,推测在自然界中可能存在一种自养型菌,能在厌氧条件下以NO2-作为电子受体氧化NH4+[1]。1995年,Mulder等首次在污水处理系统中发现这种脱氮途径,并命名为厌氧氨氧化(Anaerobic ammonia oxidation,Anammox)[2]。进一步的研究证实Anammox实质上是微生物作用的过程,其电子供体和电子受体分别是NH4+和NO2–[3]。2002年,Dalsgaard等首次在自然环境中发现Anammox过程的存在[4]。此后陆续的研究表明,Anammox在自然生态系统中广泛分布,包括海洋生态系统[5-7]、河口和海湾生态系统[8-9]、淡水生态系统[10-11]、土壤生态系统[12-13]以及一些特殊的生境,如火山口、极地沉积物、高温油库等[14]。Anammox的发现打破了认为反硝化是唯一微生物脱氮途径的传统认识,对理解全球氮循环具有重要的意义,并且在废水脱氮处理中具有广阔的应用前景。
厌氧氨氧化细菌(Anaerobic ammonium-oxidizing bacteria)是厌氧菌,该菌生长缓慢,至今未能通过传统的微生物分离培养方法实现纯培养。1999年,Strous等通过16S rRNA基因进化分析揭示这类微生物属于浮霉菌门(Planctomycetes)[15]。至今发现的厌氧氨氧化细菌有6个属,包括Candidatus Brocadia、Ca. Kuenenia、Ca. Scalindua、Ca. Jettenia、Ca. Anammoxoglobus和Ca. Anammoximicrobium moscowii。目前发现厌氧氨氧化细菌在海洋生态系统中广泛存在且异常活跃,对海洋氮转化的贡献为30%-50%[16];但其种群组成比较单一,Ca. Scalindua属的比例占绝对优势,推测与其具有较强的耐盐性有关[8]。河口和海湾生态系统作为海陆交界地带,淡水和海水的相互作用使得各种营养物质不断变化,形成适合多个种属厌氧氨氧化细菌生存的微环境。研究表明,Ca. Brocadia、Ca. Kuenenia、Ca. Scalindua等多个属的厌氧氨氧化细菌共同存在于这一生境中[9,17]。河流、湖泊等淡水生态系统中的厌氧氨氧化细菌也表现出较高的多样性水平,Ca. Brocadia、Ca. Kuenenia、Ca. Scalindua、Ca. Anammoxoglobus属均被检测到。Schubert等首次在淡水环境中检测到厌氧氨氧化细菌的存在,且大多数为Ca. Brocadia属,其对氮转化的贡献率与海洋环境相似[18]。此外,在湿地、农田土壤、沼泽、永久冻土等不同的土壤生态系统中均检测到厌氧氨氧化细菌的存在[12,19]。然而,目前对于土壤生态系统中Anammox过程的研究还较少,且在一定深度范围内的土壤中发现有厌氧氨氧化细菌的分布,表明了厌氧氨氧化细菌分布的空间异质性。
为了更清晰地展示厌氧氨氧化细菌在不同生境中的分布特征,对已经发表的相关序列进行了搜集整理和系统分析,发现不同生境中具有显著不同的群落结构组成。然而先前的研究都是基于某个生境的独立分析,而且都是基于克隆文库的构建,对于不同生境中厌氧氨氧化菌多样性分布的认识还不够系统和深入,还需要做更进一步的深入分析,为阐释厌氧氨氧化细菌在不同生境中的分布特征提供实验依据。本研究选取了红树林生态系统、海湾和河口生态系统、河流生态系统、土壤生态系统和人工生态系统等多种典型生境的样品,通过对厌氧氨氧化细菌16S rRNA基因的高通量测序,研究多种典型生境下厌氧氨氧化细菌的多样性特征,并结合环境条件分析环境因子对厌氧氨氧化细菌群落结构的影响。本研究将为深入了解不同生境中厌氧氨氧化细菌的多样性分布特征以及与环境因子之间的关系提供理论基础。
1 材料与方法 1.1 样品采集与保存所用样品均为沉积物或者土壤类型,选取了红树林、海湾、河口、河流、土壤和人工6种典型生境共15个样品进行分析。Mros1和Mros2样品采集于广东省惠州市沿海红树林区域;JZB1和JZB2样品采集于胶州湾;PR1、PR2和PR3样品采集于珠江口;DJR1、DJR2和DJR3样品分别采集于东江上游、中游、下游;RS1和RS2的样品采集于湖南省邵阳市的红壤;HC和HN采集于本实验室内构建的人工反应器;WW样品采集于广州市沥滘污水厂污泥。
对现场采集的环境样品混匀后立即置于聚乙烯密封袋中,排尽空气后密封,用冰盒储存带回实验室-80 ℃保存。一部分样品用于理化参数的测定,一部分用于DNA的提取和后续的分子生物学实验。
1.2 主要试剂和仪器PowerSoil® DNA Isolation Kit,Qiagen公司;GoTaq® Green Master Mix,Promega公司;TaKaRa MiniBEST Agarose Gel DNA Extraction Kit,TaKaRa公司;PCR引物由Invitrogen公司合成。PCR仪,ABI公司;NanoDrop核酸定量仪,Thermo公司;凝胶成像系统,Bio-Rad公司。
1.3 理化指标的测定样品混匀后进行离心(5 000 r/min,5 min),抽取离心后的上清液进行盐度和营养盐的测定。样品的盐度采用盐度计直接测定,NO3-的测定采用本实验室建立的小体系连续快速测定水体溶解性硝酸盐浓度的方法[20];NO2-浓度根据《海洋调查规范GB/T 12763.4–2007》规定的重氮-偶氮法来测定;NH4+采用使用小体系连续快速测定水体溶解性铵盐的方法进行[21]。
1.4 样品DNA提取和PCR扩增称取沉积物或者土壤样品约0.3 g,按照土壤DNA提取试剂盒(PowerSoil® DNA Isolation Kit)操作说明书提取样品中的总DNA。将提取到的总DNA溶解在50 μL无核酸酶水中,并用NanoDrop核酸定量仪测定DNA浓度与质量。提取后的DNA放置于-80 ℃保存。
以提取的DNA为模板,采用厌氧氨氧化细菌16S rRNA基因的一对特异性引物A438f (5′-XXX XXXXXGTCRGGAGTTADGAAATG-3′)和A684r (5′-ACCAGAAGTTCCACTCTC-3′)对样品进行PCR扩增[22]。其中XXXXXXXX为8个碱基的标签序列(Barcode),每个样品使用的Barcode序列都是不同的,以便于对后续测定的样品序列进行分拣。该引物特异性好,适合检测不同生境的Anammoxer[22-26]。PCR反应体系(25 μL):2×GoTaq® Green Master Mix 12.5 μL,10 μmol/L的正、反向引物各1 μL,DNA模板1 μL,使用无核酸酶纯水补足至25 μL。PCR反应条件:95 ℃ 3 min;94 ℃ 45 s,49 ℃ 45 s,72 ℃ 45 s,32次循环;72 ℃ 10 min。PCR扩增产物经1.2%琼脂糖凝胶电泳和EB染色后用凝胶成像系统进行验证,并按照样本目标测序深度对验证后的PCR产物进行相应比例混合,按照相同体积PCR产物在凝胶成像系统中的亮度从强到弱依次分为1-4四个等级,分别加入1-4等级的PCR产物为2、4、6和8 μL进行混合。使用胶回收试剂盒对混合后的PCR产物进行切胶纯化,将纯化产物进行高通量测序,测序平台为Illumina HiSeq pe150,委托苏州金唯智生物科技有限公司完成。将得到的Anammoxer 16S rRNA基因高通量测序结果提交至NCBI SRA (登录号为PRJNA506364)。
1.5 高通量测序数据的处理和分析对高通量测序数据进行处理前,需要构建厌氧氨氧化细菌16S rRNA基因参考数据库。收集了近20年来发表的与厌氧氨氧化细菌在不同生境中多样性分布研究的相关文献,根据文献中公布的厌氧氨氧化细菌的16S rRNA基因的序列号(部分序列直接来自数据库),在NCBI基因数据库上下载相关基因序列。总共收集了12 614条序列,这些序列来自全球不同的研究区域和不同类型的生境。首先,从GenBank数据库中下载通过分离或富集培养的厌氧氨氧化细菌16S rRNA基因序列作为种子序列;然后,从FunGene数据库中下载已有的环境样品Anammox基因序列;将获得的所有序列使用Mothur软件进行质控,生成厌氧氨氧化细菌16S rRNA基因参考数据库。
高通量测序的序列经过质控和分拣后,按照Mothur软件(V1.39.5)标准操作流程对序列进行处理和分析。(1)参照构建的厌氧氨氧化细菌16S rRNA基因参考数据库,使用MEGA 7.0软件进行序列对齐。(2)对每个样品的序列长度进行控制,删除过短序列;对序列名称进行简化,并去除Barcode序列。(3)筛选去除冗余序列和嵌合体序列。(4)运用构建好的厌氧氨氧化细菌 16S rRNA基因参考数据库,确定样品序列的归类种属信息;将每一条经质控后的序列与厌氧氨氧化细菌16S rRNA基因参考数据库比对,找出与其最相近且可信度达80%以上的参考序列种属信息。(5)计算序列两两之间的相似度,生成距离矩阵文件。(6)基于生成的距离矩阵,对序列进行操作分类操作单元(Operational taxonomic units,OTU)聚类,相似性阈值为97%。(7)过滤低丰度OTU,去除序列数仅为5的OTU,提取主要OTU的代表序列。(8)基于非稀有OTU在每个样品中的丰度,计算样品中的α多样性指数;基于主坐标分析(Principal co-ordinates analysis,PCoA)进行β多样性分析。
根据稀释曲线可判断样品的测序深度是否满足多样性分析;根据等级丰度曲线可判断样品的物种丰富度和均匀度;统计隶属于每个种属中OTU的序列条数,可得到各样品中厌氧氨氧化细菌在属水平的相对丰度。根据基于OTU的系统发育树和热图,可分析得到不同生境样品中厌氧氨氧化细菌的群落结构和组成。使用Origin、Excel等软件进行厌氧氨氧化细菌的α多样性和β多样性的相关图绘制。在α多样性指标中,利用Ace指数、Chao1指数、Shannon指数、Simpson多样性指数判断群落多样性。在β多样性指标中,利用主坐标分析(PCoA)比较各样点间群落的差异。根据OTU聚类结果以及各个样品的OTU分布状况,结合样品的环境参数,使用CANOCO 5.0软件分析影响厌氧氨氧化细菌群落结构及分布的主要环境因素。
2 结果与分析 2.1 理化指标分析实验测得的15个样品的理化指标数据如表 1所示。结果表明,除了红树林、海湾和河口生态系统存在较高盐度,其他生境样品的盐度都较低。在溶解无机氮(Dissolve inorganic nitrogen,DIN)的含量上,HN样品孔隙水的NO3-浓度比较特殊,高达1 500 μmol/L;而JZB1、JZB2、DJR1、DJR2、DJR3孔隙水的NO3-浓度较高;可以看出:JZB1的NO3-浓度明显低于JZB2,而DJR1、DJR2、DJR3的NO3-浓度的差别也很明显;所有样品除了HC和HN外,NO2-的浓度都比较低;NH4+在不同的样品中差别也较大,在东江、珠江口以及胶州湾的沉积物中含量较高。
| 样品 Sample |
经度 Longitude (°) |
纬度 Latitude (°) |
盐度 Salinity (%) |
铵盐 NH4+ (μmol/L) |
硝酸盐 NO3- (μmol/L) |
亚硝酸盐 NO2- (μmol/L) |
生态系统类型 Ecosystem-type |
| Mros1 | 114.78 | 22.83 | 2.223 | 1.54 | 0.97 | 0.51 | 红树林Mangrove |
| Mros2 | 114.78 | 22.83 | 2.477 | 1.29 | 3.71 | 0.52 | 红树林Mangrove |
| JZB1 | 120.28 | 36.29 | 3.110 | 3.12 | 8.45 | 0.38 | 海湾Bay |
| JZB2 | 120.42 | 36.35 | 3.111 | 12.28 | 30.95 | 0.74 | 海湾Bay |
| PR1 | 113.51 | 23.03 | 0.015 | 29.32 | 92.09 | 4.17 | 河口Estuary |
| PR2 | 113.75 | 22.52 | 0.501 | 4.52 | 10.45 | 0.63 | 河口Estuary |
| PR3 | 113.72 | 21.99 | 3.237 | 2.38 | 12.39 | 1.76 | 河口Estuary |
| DJR1 | 114.72 | 24.05 | - | 2.35 | 14.62 | 0.64 | 河流River |
| DJR2 | 114.60 | 23.16 | - | 15.67 | 68.37 | 0.64 | 河流River |
| DJR3 | 113.99 | 23.09 | - | 34.82 | 146.24 | 1.03 | 河流River |
| RS1 | 116.33 | 27.95 | - | 0.28 | 1.17 | 0.53 | 土壤Soil |
| RS2 | 111.45 | 27.38 | - | 0.58 | 2.09 | 0.58 | 土壤Soil |
| HC | 113.63 | 22.60 | - | 15.23 | 1.23 | 88.72 | 人工系统Artificial system |
| HN | 113.63 | 22.60 | - | 18.92 | 1 500.00 | 6.16 | 人工系统Artificial system |
| WW | 113.35 | 23.04 | - | 1.34 | 2.01 | 0.98 | 人工系统Artificial system |
| 注:-:淡水样品,盐度 < 0.1%. Note: -: Freshwater sample with salinity lower than 0.1%. |
|||||||
对实验全部样品进行α多样性指数统计分析。从每个样品的原始序列库中随机筛选10 000条进行分析。表 2表明,原始序列经过质控后,样品序列数在7 529-9 198之间,样品的OTU数在55-127之间。其中海湾JZB1样品的OTU最多,为127;污泥(WW)的OTU数最少,为55。测序结果覆盖率均在99.7%以上,表明得到的多样性指数有较好的可靠性。结合稀释曲线(图 1A)可以看出,起初随着序列数的增加,样品中OTU迅速增加;而随着序列数的进一步增加,OTU数量增加的趋势逐渐变缓,稀释曲线趋于饱和,说明所用样品的序列数能够充分展示样品中的厌氧氨氧化细菌的多样性,与序列高覆盖率(> 99.7%,见表 2)反映的结果相符。表 2呈现出不同生境样品中明显的多样性变化趋势:红树林生态系统(Mros1、Mros2)、海湾生态系统(JZB1、JZB2)、河口生态系统(PR1、PR2、PR3)这三类生态系统的Ace指数、Chao1指数和Shannon指数都较高,而Simpson多样性指数较低,表明这三类生态系统样品的生物多样性都较高;与之相反的是:红壤(RS1、RS2)和污泥(WW)的Ace指数、Chao1指数和Shannon指数都较低,而Simpson多样性指数较高,表明这两类生态系统的生物多样性都较低。
| Sample | Unique sequences | Coverage | OTUs | Ace | Chao1 | Simpson | Shannon |
| Mros1 | 8 121 | 0.99 | 99 | 140.45 | 125.90 | 0.25 | 2.31 |
| Mros2 | 8 014 | 0.99 | 99 | 111.17 | 110.82 | 0.14 | 2.74 |
| JZB1 | 7 898 | 0.99 | 127 | 144.78 | 145.65 | 0.05 | 3.49 |
| JZB2 | 7 529 | 0.99 | 95 | 125.99 | 107.21 | 0.08 | 2.93 |
| PR1 | 8 485 | 0.99 | 87 | 101.64 | 103.09 | 0.17 | 2.41 |
| PR2 | 7 891 | 0.99 | 126 | 146.75 | 152.58 | 0.08 | 3.16 |
| PR3 | 7 823 | 0.99 | 113 | 122.74 | 125.88 | 0.09 | 3.06 |
| DJR1 | 9 028 | 0.99 | 70 | 81.65 | 84.98 | 0.82 | 0.62 |
| DJR2 | 8 793 | 0.99 | 73 | 134.89 | 107.28 | 0.13 | 2.49 |
| DJR3 | 9 057 | 0.99 | 73 | 93.67 | 87.62 | 0.75 | 0.79 |
| RS1 | 9 198 | 0.99 | 62 | 75.30 | 71.27 | 0.86 | 0.49 |
| RS2 | 9 188 | 0.99 | 67 | 92.51 | 86.37 | 0.88 | 0.41 |
| HC | 8 092 | 0.99 | 66 | 73.42 | 69.67 | 0.19 | 2.20 |
| HN | 8 186 | 0.99 | 58 | 64.57 | 61.99 | 0.18 | 2.27 |
| WW | 9 088 | 0.99 | 55 | 89.64 | 71.12 | 0.88 | 0.42 |
|
| 图 1 厌氧氨氧化细菌16S rRNA基因基于高通量测序分序OTU稀释曲线(A)和等级丰度曲线(B) Figure 1 Rarefaction curves (A) and grade abundance curves (B) of the anammox specific 16S rRNA gene sequences in different habitats |
|
|
结合等级丰度曲线(图 1B)分析,JZB1、PR3、PR2的等级丰度曲线在横轴上的长度都比较长,且比较平滑地下降,表明这3个样品所含物种较丰富、多样性较高。而WW、RS1、RS2的等级丰度曲线在横轴上的长度都比较短,且快速陡然地下降,表明这3个样品所含物种较少,样本中的优势菌群所占比例很高、多样性较低。综合来看,JZB1的生物多样性最高,WW的生物多样性最低。
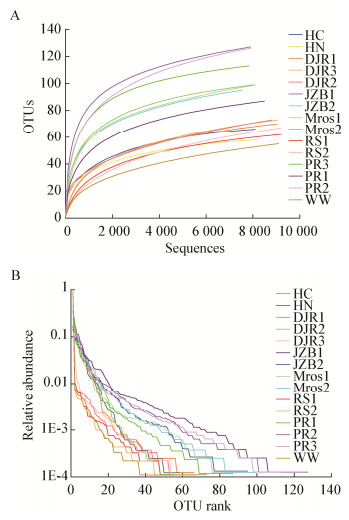
2.3 不同生境中厌氧氨氧化细菌的种属分布特征根据Mothur分析结果绘制每个样品中厌氧氨氧化细菌的种属分布图(图 2)。对单个样品而言,Ca. Scalindua属在Mros1、Mros2、JZB2、PR3中有明显优势,分别达到了64.54%、68.55%、87.18%、59.72%,且在JZB1和PR2也占有一定的比例,分别达到了30.62%、28.66%;Ca. Brocadia属在绝大部分样品(PR1、PR2、DJR1、DJR2、DJR3、RS1、RS2、HC、HN、WW)有明显优势,部分高达90%以上。Ca. Kuenenia属只在部分样品中存在,且比例普遍较低(10%以下);而Ca. Jettenia和Ca. Anammoxoglobus属在样品中的相对丰度非常低。值得注意的是,未明确分类地位的种属(Unclassified)在部分样品中的比例较高,在JZB1中高达64.48%,推测还存在多种未被鉴定的新种属。
|
| 图 2 不同生境中厌氧氨氧化细菌在属水平上的群落结构组成 Figure 2 Composition of anammox bacteria in different habitats in genus level |
|
|
对不同生态系统而言,河流生态系统(DJR1、DJR2、DJR3)、土壤生态系统(RS1、RS2)和污泥(WW)的厌氧氨氧化细菌种属组成类似,Ca. Brocadia属占绝对优势,但多样性水平较低。红树林生态系统(Mros1、Mros2)、海湾生态系统(JZB1、JZB2)和河口生态系统(PR2、PR3)的厌氧氨氧化细菌种属组成相近,种群多样性较高,Ca. Scalindua属是优势种属,另外都能检测到少量的Ca. Kuenenia和Ca. Brocadia属,该结果与先前的研究结果一致[27]。
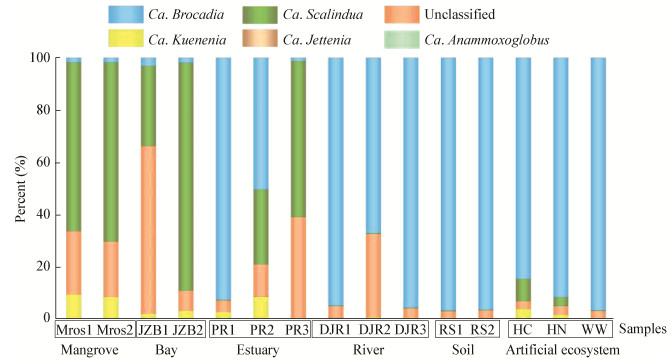
2.4 基于OTU的厌氧氨氧化细菌系统发育和热图分析基于OTU聚类结果,挑选序列数大于总序列数1%的OTU代表序列,共提取了10条代表序列,TOP10 OTU的序列总数占总序列数的97%以上。将提取到的代表序列与已知Anammoxer的基因序列进行比较,将序列对齐后,用MEGA 7.0以邻接(Neighbor-Joining)法构建系统发育树(图 3)。结果表明:选取的15个样品的厌氧氨氧化细菌主要OTU分为3大簇。其中,OTU1 (50.94%)、OTU3 (11.00%)、OTU4 (7.81%)、OTU5 (5.68%)归类为Ca. Brocadia属;OTU2 (11.13%)、OTU6 (2.93%)、OTU8 (2.63%)、OTU9 (2.60%)、OTU10 (2.54%)归类为Ca. Scalindua属;而OTU7 (2.74%)为Unclassified种属(未明确分类地位的菌属)。主要OTU均为Ca. Brocadia属。
|
| 图 3 基于16S rRNA基因高通量测序分析多种生境中厌氧氨氧化细菌主要OTU的系统进化 Figure 3 Phylogenetic analysis of dominant OTU of anammox specific 16S rRNA gene based on the high-throughput sequencing Note: This tree was constructed with a Neighbor-Joining method based on Jukes-Cantor-corrected DNA distances and midpoint rooted. The scale bar represents 2% estimated sequence divergence. The numbers at the nodes are percentages that indicate the levels of bootstrap support based on 1 000 resampled data sets. |
|
|
通过热图展示了厌氧氨氧化细菌的群落结构在OTU水平的分布(图 4),图 4中每个方块代表对应的OTU,方块的颜色代表该OTU的相对丰度水平(颜色从浅蓝到深蓝代表序列丰度由低到高)。可以看出,OTU1主要分布在WW、RS1、RS2、DJR1、DJR3,OTU2主要分布在Mros1,PR2、PR3中10个OTU都有所分布。

|
| 图 4 不同生境中厌氧氨氧化细菌主要OTU在不同生境中的热图分析 Figure 4 Heatmap analysis of dominant OTU of anammox bacteria in different habitat |
|
|
基于OTU聚类结果,采用Mothur软件中的Thetayc算法计算各样品之间的差异,从而对厌氧氨氧化细菌的群落排序,选取解释度最大的两轴作主坐标分析图(Principal co-ordinates analysis,PCoA) (图 5),分析不同生境中厌氧氨氧化细菌的聚类关系。由图 5可知,P1主成分对排序结果的解释度为38.9%,P2主成分对排序结果的解释度为30.1%,P1和P2主成分累计对排序结果的解释度为69.0%。P1和P2主成分共同将15个样品分成3个簇:PR1、PR2、HC、HN这4个样品为一簇,说明PR1、PR2、HC、HN的群落结构比较类似;JZB1、JZB2、Mros1、Mros2、PR3这5个样品为一簇,且距离都比较接近,说明这几个样品的群落结构都比较类似;而DJR1、DJR3、RS1、RS2、WW这5个样品在PCoA图上几乎看不出距离,说明这几个样品的群落结构非常类似。总体而言,簇与簇之间的距离都比较大,说明每个簇内样品的群落结构与其他簇之间都有较大差异。值得注意的是,DJR2与其他样品的距离都比较远,说明DJR2的群落结构与其他样品差异都比较大,推测可能是因为DJR2所在的区域厌氧氨氧化细菌生存的微环境与其他样品间的差异较大。

|
| 图 5 不同生境中厌氧氨氧化细菌16S rRNA基因序列的PCoA分析 Figure 5 Principal co-ordinates analysis (PCoA) for each habitat based on the anammox specific 16S rRNA gene sequences |
|
|
对比样品和它们代表的生境在PCoA图上的位置,可以发现红树林生态系统、海湾生态系统和河口生态系统的群落结构比较类似,但与河流生态系统的群落结构差异较大。结合红树林(Mros1、Mros2)、胶州湾(JZB1、JZB2)和东江(DJR1、DJR2、DJR3)的盐度和可溶解无机氮(Dissolve inorganic nitrogen,DIN) (表 1),推测可能是因为河口和海湾生态系统作为淡水和海水相互作用最强烈的区域,形成的厌氧氨氧化细菌生存的微环境类似,因此这两类生态系统的群落结构差异不大;而DJR1、DJR2、DJR3属于淡水河流生态系统,其环境条件与河口、海湾生态系统区别较大,导致厌氧氨氧化细菌生存的微环境不同,因此群落结构差异较大。
2.6 环境因子对厌氧氨氧化细菌群落结构的影响分析根据OTU聚类结果以及各个样品的OTU分布状况,结合样品的环境参数1使用CANOCO 5.0软件进行冗余分析(Redundancy analysis,RDA),结果如图 6所示。RDA分析产生4个排序轴,图中显示2个,即排序轴RDA1和RDA2。排序轴RDA1和RDA2的特征值分别为0.584和0.185,方差变量解释分别为46.93%和29.78%,累计解释了厌氧氨氧化细菌群落结构累积方差变量的76.71%,说明此次RDA排序结果是可靠的。采用前向选择法(Forward selection)对全部环境变量进行选择,结果表明在盐度、NO2-和NO3-三种环境参数中,只有盐度对厌氧氨氧化细菌起到显著性影响(P=0.002 < 0.05,NO2-和NO3-的P值分别是0.198和0.322)。盐度的方差变量贡献率为86.3% (F=18.3),对群落结构变异信息的解释度为58.4%,这表明盐度是影响本研究中样品厌氧氨氧化细菌群落结构分布的主要因素。不同生态系统环境因素的差异是影响厌氧氨氧化细菌群落结构和分布的重要原因。近年来对环境中Anammox的研究聚焦于环境因子与群落结构、多样性水平、空间分布及丰度差异的关系。2007年,Schmid等对黑海、日本海等11个盐度较高的海域(盐度为17–35 PSU)进行研究,发现这些海域的厌氧氨氧化细菌均属于Ca. Scalindua属[27]。随后,Dale等证实了在海洋环境中,随盐度的递增厌氧氨氧化细菌的优势种群从Ca. Brocadia转变为Ca. Scalindua,而且在高盐环境中仅有Ca. Scalindua存在[8]。2011年,Hirsch等的研究表明盐度是多种生态系统中厌氧氨氧化细菌群落结构和分布特征存在差异的首要原因[28]。大量研究结果表明厌氧氨氧化细菌存在盐度耐受性差异,证实了盐度是导致厌氧氨氧化细菌群落结构和分布特征差异性的重要因素。

|
| 图 6 不同生境以及主要OTU与环境因子的RDA分析 Figure 6 RDA analysis for the relationship between the habitats or dominant OTU of anammox bacteria and environmental factors |
|
|
目前,大量研究结果表明厌氧氨氧化细菌存在盐度耐受性差异。其中,Ca. Scalindua属的盐度耐受性最高,而Ca. Brocadia属最低。在本研究中,从河流生态系统到红树林生态系统,随着盐度的增加,厌氧氨氧化细菌的优势种属由Ca. Brocadia属转变到Ca. Scalindua属,进一步证实了盐度是导致厌氧氨氧化细菌种属组成和分布存在差异的重要因素。除了盐度和氮素营养盐外,温度、溶解氧、深度、pH等也是影响厌氧氨氧化细菌分布特征的环境因子。由于环境因子是同时存在的,因此难以说明单独一种环境因子对厌氧氨氧化细菌的影响,仍需进一步研究厌氧氨氧化细菌应对不同环境因子的适应机制。
3 讨论与结论对于厌氧氨氧化细菌在不同生境的多样性群落结构,先前的研究基本上都是基于单一生境进行的,例如海洋、河口、淡水、在湿地、农田土壤、沼泽、永久冻土等不同的土壤生境中均检测到厌氧氨氧化细菌的存在[4-14,19],而且认识到不同的生境中有不同的多样性分布特征。然而,先前的研究存在问题:一是没有对多种生境进行系统的分析,导致厌氧氨氧化细菌生态分布的信息不系统;二是先前的研究基本是基于克隆文库的构建,得到的基因序列数非常有限,对于样品的厌氧氨氧化细菌多样性分析不够深入。本研究选取多个典型生境的样品作为研究对象,采用高通量测序技术对样品中厌氧氨氧化细菌的多样性进行了深度展示,并用统一的方法对多个生境的样品进行了一致性分析,更客观地揭示了厌氧氨氧化细菌在自然环境的分布特征和多样性特征。
本研究主要采用特异性引物A438f和A684r所产生的250 bp的16S rRNA基因序列,对不同生境中厌氧氨氧化细菌的群落结构进行了分析。首先,先前的研究已经证实了A438f/A684r引物对目前已知厌氧氨氧化菌的类群都有很好的兼并性,为多样性的分析提供了重要基础[25,29]。此外,用这对引物扩增得到的250 bp的产物,为高通量测序也带来了方便,使用高通量测序深度解析了环境中氧氨氧化细菌的多样性组成。同时,我们也注意到,在细菌一个细胞中16S rRNA基因的多拷贝问题是否影响多样性的分析也是值得思考的重要问题。从目前几个典型的被鉴定的厌氧氨氧化菌株之间的相似性来看,在属水平上的16S rRNA基因的相似性会超过3%[28];而且绝大多数原核微生物基因组中多拷贝的16S rRNA基因序列通常是相同或是有非常高相似度(大于99%)[29]。因此,16S rRNA基因的多拷贝应当不会影响氧氨氧化细菌的多样性分析。
本研究得到的主要结论为:(1)红树林、海湾和河口生态系统的样品多样性水平较高,而污泥和土壤生态系统的多样性水平较低。其中,海湾生态系统(JZB1)的多样性水平最高,污泥的多样性水平最低。(2)系统发育分析结果表明,本研究选取的15个样品的厌氧氨氧化细菌可以分为Ca. Brocadia、Ca. Scalindua和未明确分类地位的菌属(Unclassified)这三大簇。主要OTU均属于Ca. Brocadia属。(3)污泥、河流和土壤生态系统种属组成类似;红树林、海湾和河口生态系统种属组成类似。从河流生态系统到红树林生态系统,随着盐度的增加,厌氧氨氧化细菌的优势种属由Ca. Brocadia属转变到Ca. Scalindua属。(4)河口和海湾生态系统的群落结构相似,与河流生态系统差异较大;土壤与河流生态系统的群落结构非常相似。RDA结果表明,盐度是影响厌氧氨氧化细菌群落结构和分布的主要因素。总之,厌氧氨氧化细菌具有多样性的分布特征,在不同生境中有不同的群落结构组成,这可能是长期适应不同环境演化的结果。
| [1] |
Broda E. Two kinds of lithotrophs missing in nature[J]. Zeitschrift für Allgemeine Mikrobiologie, 1977, 17(6): 491-493. DOI:10.1002/jobm.3630170611 |
| [2] |
Mulder A, van de Graaf AA, Robertson LA, et al. Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor[J]. FEMS Microbiology Ecology, 1995, 16(3): 177-183. DOI:10.1111/j.1574-6941.1995.tb00281.x |
| [3] |
van de Graaf AA, de Bruijn P, Robertson LA, et al. Autotrophic growth of anaerobic ammonium-oxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology, 1996, 142(8): 2187-2196. DOI:10.1099/13500872-142-8-2187 |
| [4] |
Dalsgaard T, Thamdrup B. Factors controlling anaerobic ammonium oxidation with nitrite in marine sediments[J]. Applied and Environmental Microbiology, 2002, 68(8): 3802-3808. DOI:10.1128/AEM.68.8.3802-3808.2002 |
| [5] |
Dalsgaard T, Canfield DE, Petersen J, et al. N2 production by the anammox reaction in the anoxic water column of Golfo Dulce, Costa Rica[J]. Nature, 2003, 422(6932): 606-608. DOI:10.1038/nature01526 |
| [6] |
Kuypers MMM, Sliekers AO, Lavik G, et al. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J]. Nature, 2003, 422(6932): 608-611. DOI:10.1038/nature01472 |
| [7] |
Hong YG, Li M, Cao HL, et al. Residence of habitat-specific anammox bacteria in the Deep-Sea subsurface sediments of the South China Sea: analyses of marker gene abundance with physical chemical parameters[J]. Microbial Ecology, 2011, 62(1): 36-47. DOI:10.1007/s00248-011-9849-0 |
| [8] |
Dale OR, Tobias CR, Song B. Biogeographical distribution of diverse anaerobic ammonium oxidizing (anammox) bacteria in Cape Fear River Estuary[J]. Environmental Microbiology, 2009, 11(5): 1194-1207. DOI:10.1111/j.1462-2920.2008.01850.x |
| [9] |
Hou LJ, Zheng YL, Liu M, et al. Anaerobic ammonium oxidation (anammox) bacterial diversity, abundance, and activity in marsh sediments of the Yangtze Estuary[J]. Journal of Geophysical Research: Biogeosciences, 2013, 118(3): 1237-1246. DOI:10.1002/jgrg.20108 |
| [10] |
Hamersley MR, Woebken D, Boehrer B, et al. Water column anammox and denitrification in a temperate permanently stratified lake (Lake Rassnitzer, Germany)[J]. Systematic and Applied Microbiology, 2009, 32(8): 571-582. DOI:10.1016/j.syapm.2009.07.009 |
| [11] |
Yoshinaga I, Amano T, Yamagishi T, et al. Distribution and diversity of anaerobic ammonium oxidation (anammox) bacteria in the sediment of a eutrophic freshwater lake, Lake Kitaura, Japan[J]. Microbes and Environments, 2011, 26(3): 189-197. DOI:10.1264/jsme2.ME10184 |
| [12] |
Shen LD, Liu S, Lou LP, et al. Broad distribution of diverse anaerobic ammonium-oxidizing bacteria in Chinese agricultural soils[J]. Applied and Environmental Microbiology, 2013, 79(19): 6167-6172. DOI:10.1128/AEM.00884-13 |
| [13] |
Yang XR, Li H, Nie SA, et al. Potential contribution of anammox to nitrogen loss from paddy soils in Southern China[J]. Applied and Environmental Microbiology, 2015, 81(3): 938-947. DOI:10.1128/AEM.02664-14 |
| [14] |
Wang TT. Community structure and diversity of anammox bacteria in cow manure compost[D]. Harbin: Master's Thesis of Northeast Agricultural University, 2016 (in Chinese) 王婷婷.牛粪堆肥中厌氧氨氧化菌的群落结构和多样性研究[D].哈尔滨: 东北农业大学硕士学位论文, 2016 |
| [15] |
Strous M, Fuerst JA, Kramer EHM, et al. Missing lithotroph identified as new planctomycete[J]. Nature, 1999, 400(6743): 446-449. DOI:10.1038/22749 |
| [16] |
Devol AH. Nitrogen cycle: Solution to a marine mystery[J]. Nature, 2003, 422(6932): 575-576. DOI:10.1038/422575a |
| [17] |
Wang SY, Zhu GB, Peng YZ, et al. Anammox bacterial abundance, activity, and contribution in riparian sediments of the Pearl River estuary[J]. Environmental Science & Technology, 2012, 46(16): 8834-8842. |
| [18] |
Schubert CJ, Durisch-kaiser E, Wehrli B, et al. Anaerobic ammonium oxidation in a tropical freshwater system (Lake Tanganyika)[J]. Environmental Microbiology, 2006, 8(10): 1857-1863. DOI:10.1111/j.1462-2920.2006.01074.x |
| [19] |
Xu LY, Zhao X, Zhuang LJ, et al. Community structure analysis of anaerobic ammonium oxidation bacteria in wetland sediments based on hydrazine synthase β-subunit[J]. Acta Scientiae Circumstantiae, 2017, 37(12): 4636-4645. (in Chinese) 徐栎亚, 赵雪, 庄林杰, 等. 基于hzsB功能基因研究典型湿地沉积物中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 2017, 37(12): 4636-4645. |
| [20] |
Wu JP, Hong YG, Guan FJ, et al. A rapid and high-throughput microplate spectrophotometric method for field measurement of nitrate in seawater and freshwater[J]. Scientific Reports, 2015, 6: 20165. |
| [21] |
Guan FJ, Hong YG, Wu JP, et al. A fast sodium hypobromite oxidation method for the sequential determination of ammonia nitrogen in small volume[J]. Ecological Science, 2017, 36(2): 42-48. (in Chinese) 关凤杰, 洪义国, 吴佳鹏, 等. 发展一种小体系连续快速测定海洋水体溶解性铵盐的方法[J]. 生态科学, 2017, 36(2): 42-48. |
| [22] |
Han P, Huang YT, Lin JG, et al. A comparison of two 16S rRNA gene-based PCR primer sets in unraveling anammox bacteria from different environmental samples[J]. Applied Microbiology and Biotechnology, 2013, 97(24): 10521-10529. DOI:10.1007/s00253-013-5305-z |
| [23] |
Wu JP, Hong YG, He X, et al. Anaerobic ammonium oxidation in acidic red soils[J]. Frontiers in Microbiology, 2018, 9: 2142. DOI:10.3389/fmicb.2018.02142 |
| [24] |
Humbert S, Zopfi J, Tarnawski SE. Abundance of anammox bacteria in different wetland soils[J]. Environent Microbiopgyl Report, 2012, 4(5): 484-490. DOI:10.1111/j.1758-2229.2012.00347.x |
| [25] |
Han P, Gu JD. More refined diversity of anammox bacteria recovered and distribution in different ecosystems[J]. Applied Microbiology and Biotechnology, 2013, 97(8): 3653-3663. DOI:10.1007/s00253-013-4756-6 |
| [26] |
Wu JP, Hong YG, Chang XY, et al. Unexpectedly high diversity of anammox bacteria detected in deep-sea surface sediments of the South China Sea[J]. FEMS Microbiology Ecology, 2019, 95(3): fiz013. |
| [27] |
Schmid MC, Risgaard-Petersen N, van de Vossenberg J, et al. Anaerobic ammonium-oxidizing bacteria in marine environments: widespread occurrence but low diversity[J]. Environmental Microbiology, 2007, 9(6): 1476-1484. DOI:10.1111/j.1462-2920.2007.01266.x |
| [28] |
Hirsch MD, Long ZT, Song B. Anammox bacterial diversity in various aquatic ecosystems based on the detection of hydrazine oxidase genes (hzoA/hzoB)[J]. Microbial Ecology, 2011, 61(2): 264-276. DOI:10.1007/s00248-010-9743-1 |
| [29] |
Yan YW, Zhang DM. Multiplicity and heterogeneity of 16S rRNA genes in prokaryotes — a mini review[J]. Journal of Biology, 2013, 30(4): 63-66. (in Chinese) 阎永伟, 张德民. 原核生物16S rRNA基因多重拷贝及其序列异化[J]. 生物学杂志, 2013, 30(4): 63-66. DOI:10.3969/j.issn.2095-1736.2013.04.063 |
 2019, Vol. 46
2019, Vol. 46