扩展功能
文章信息
- 周培富, 赵宇中, 谢建平
- ZHOU Pei-Fu, ZHAO Yu-Zhong, XIE Jian-Ping
- 《微生物学》中“微生物物种多样性”的教学内容和教学方式探讨
- Discussion on the contents of microbial diversity and teaching methods in Microbiology
- 微生物学通报, 2019, 46(10): 2794-2800
- Microbiology China, 2019, 46(10): 2794-2800
- DOI: 10.13344/j.microbiol.china.180852
-
文章历史
- 收稿日期: 2018-10-31
- 接受日期: 2019-01-22
- 网络首发日期: 2019-02-28
2. 西南大学生命科学学院现代生物医药研究所 重庆 400715
2. Institute of Modern Biopharmaceuticals, School of Life Sciences, Southwest University, Chongqing 400715, China
在高校的课程体系中,微生物学历来都是各类高校生命科学学院及医、药、农、林、食品等有关专业本科生的必修专业基础课,为学生提供基础、系统、前沿的微生物学知识和理论,地位十分重要[1]。在大学本科的大多数《微生物学》教材中都有类似的描述:自然环境中超过99%的微生物无法在实验室培养条件下生长,也称为未培养微生物[2]。然而,在介绍该知识点时,学生多会提问:这些超过99%的未培养微生物既然无法培养和观察,那如何知道它们的存在?对此,教师会从不依赖于微生物培养的一些微生物多样性研究方法开始逐步解答。然而,由于教材缺乏相关的背景知识介绍,学生不太容易理解。基于此,我们在分析了微生物多样性相关概念、研究方法并对比国内外主流《微生物学》相关教材的基础上,讨论在本科《微生物学》教材中,结合教材的专业倾向,适当补充“微生物多样性相关知识点及领域研究进展”内容的必要性,以期为推动大学本科微生物学的教学工作提供一种思路。
1 微生物多样性的概念及其主要研究方法 1.1 微生物多样性的概念从广义上讲,微生物多样性可从微生物生命活动层次的角度分为遗传(基因)多样性、生理多样性、物种多样性和生态多样性四个层面[3]。而从狭义上讲,微生物多样性只特指微生物物种多样性。物种多样性包括α多样性和β多样性两类,前者表示局部环境中不同物种种类的丰富程度(Richness)以及物种数量分布的均匀程度(Evenness);后者描述不同局部环境之间物种多样性的差异程度。这里的“局部”是广泛地指代某种状态下的环境,如某一空间地点或某一时间点,实际研究中往往就是指某一样本[4]。结合上述提出的问题,在本文中探讨的主要是狭义的微生物多样性。
1.2 微生物多样性研究的意义微生物可谓无处不在,人类一直生活在微生物的海洋里。因此,对各种环境中微生物多样性的研究具有重要意义,不仅可使人类认清自身所生存和接触的环境中微生物的种类及群落结构,更有利于揭示微生物与人类的关系。微生物多样性的研究可促进微生物新种的发现和完善物种进化树[5]、解密微生物与人类健康或疾病[6],以及与人类生存密切相关的环境[7]、能源[8]、食品[9-10]等之间的关系。比如,越来越多的研究发现人类肠道微生物与人类健康有着重要的关系,肠道微生物已被报道与人体营养吸收[11]、肥胖[12]、糖尿病[13-14]、胃肠道炎症[15]、药物疗效[16]等有关,甚至还与抑郁症[17-18]、自闭症[19]、帕金森病[20-21]等有关。目前,从国内外期刊发表的相关论文数量来看(中文论文用“微生物多样性”作为关键词在CNKI数据库中进行检索;英文论文用“Microbial diversity”作为关键词在NCBI PubMed中进行检索),从2005年到2018年期间共有约6 000篇相关中文论文及近20 000篇英文论文发表,且均呈逐年增加的趋势。可见,该领域已经成为国内外研究的热点领域。
1.3 微生物物种多样性的研究方法研究微生物物种多样性的方法包括传统的微生物平板培养法,分子生物学方法如限制性片段长度多态性(Restriction fragment length polymorphism,RFLP)、随机扩增多态性DNA技术(Random amplification of polymorphic DNA,RAPD)、DNA单链构象多态性(Single-strand conformation polymorphism,SSCP)技术、扩增限制性片段长度多态性(Amplified fragment length polymorphism,AFLP)技术、特异引物的PCR标记技术[如简单重复序列(Simple sequence repeats,SSR)、序列特异性扩增区(Sequence-characterized amplified region,SCAR)、序列标志位点(Sequence tagged sites,STS)、酶切扩增多态性序列(Cleaved amplified polymorphism sequences,CAPS)、简单序列重复间区标记技术(Inter simple sequence repeat,ISSR)]、变性梯度凝胶电泳(Denatured gradient gel electrophoresis,DGGE)、温度梯度凝胶电泳技术(Temperature gradient gel electrophoresis,TGGE)等[22],以及基于高通量测序的微生物多样性分析技术。
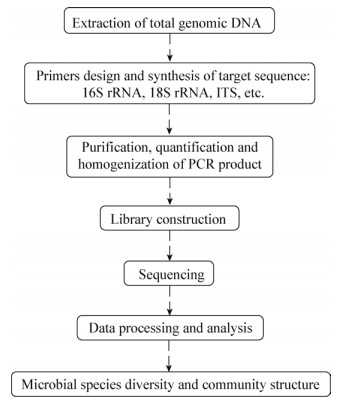
近年来,由于微生物群落总基因组DNA提取方法的改进,以及具有非培养依赖性、分析样品广泛、可对样品中微生物种群进行定性和相对定量分析、商业化程度高、测序技术不断进步且价格日趋便宜等特点,基于高通量测序的微生物多样性分析技术逐渐成为分析特定样品中微生物种类多样性及种群结构的理想手段。微生物高通量测序技术主要包括PCR产物测序和宏基因组学测序技术。PCR产物测序是指通过设计特定的引物,对目的基因片段的PCR扩增产物进行测序;宏基因组学测序是直接对环境样品中微生物群落的遗传物质进行测序和研究,充分发掘未培养和未鉴定微生物的基因多样性与生化反应。如果仅想了解样品中微生物的物种多样性及其群落结构,对其进行PCR产物测序即可。在测序标记基因选择方面,原核生物16S rRNA基因[23]、真核生物18S rRNA[24-25]及ITS序列[26-27]高度保守,是微生物系统分类研究中常用的分子生物标记基因,被广泛应用于微生物物种多样性研究。此外,一些微生物的功能基因因具有类似的保守性,也常被用于特殊微生物的多样性研究,如氨单加氧酶基因amoA[28-29],尿素水解菌的尿素水解酶基因ureC[30],反硝化细菌的nirK、nirS和nosZ基因[31-32]等。通过对这些特定的序列区域进行测序和比对,可以揭示环境微生物的物种组成及其特定生态功能,其简要流程如图 1所示。
|
| 图 1 微生物多样性分析流程 Figure 1 The experimental procedures of microbial diversity analysis |
|
|
教材是体现教学内容和教学要求的知识载体,是进行教学的基本工具,是提高教学质量的重要保证。微生物多样性分析已经是微生物学各领域研究的重要且常用的手段,目前有大量的相关研究报道。然而,众多微生物学相关的教材:高等教育“十三五”规划教材如《微生物学与免疫学》(新世纪第3版,2017,袁嘉丽、刘永琦主编)[33],《微生物学与免疫学》(2016,张雄鹰主编)[34],《微生物学与免疫学》(第8版,2016,沈关心、徐威主编)[35],《食品微生物学》(2017,桑亚新、李秀婷主编)[36],《医学微生物学》(2016,王桂琴、强华主编)[37],《医学微生物学》(第9版,2018,李凡、徐志凯主编)[38],《环境微生物学》(第3版,2019,乐毅全、王士芬主编)[39];高等教育“十二五”规划教材如《医学微生物学》(第3版,2013,张凤民、肖纯凌主编)[40],《医学微生物学》(第3版,2017,戚中田主编)[41],以及周长林老师主编的全国高等医药院校药学类第四轮规划教材《微生物学》(第3版,2015)[42]等均没有涉及微生物多样性相关知识点的介绍,特别是研究方法和原理方面。在何培新主编的普通高等教育“十三五”规划教材《高级微生物学》(2017)[43]中第七章“现代微生物生态学”部分有关于原核生物16S rRNA基因、真核生物18S rRNA基因基本特征介绍,但利用其进行克隆文库测序研究的描述部分相对简略,没有相关实验流程的介绍,因此不足以让学生充分理解和学习。此外,由沈萍和陈向东主编的《微生物学》(第8版,2016)[44]属高等教育“十二五”规划教材,在第十二章“微生物的进化、系统发育与分类鉴定”部分,对原核生物16S rRNA基因、真核生物18S rRNA基因等常用的特征分子标记基因基本信息也做了详细的介绍,并在第八章的微生物“宏基因组及宏基因组学”部分提及了“根据rDNA数据库序列设计引物,通过PCR技术从提纯的宏基因组中扩增细菌rRNA基因,从而获得特定环境中各种细菌的rRNA基因片段,测定序列后,通过系统学分析获得该环境中微生物的遗传多样性和分子生态学信息”,但也没有较具体地介绍相关的实验方法及流程。
从国外主流的微生物学教材来看,由Michael T. Madigan等编著的第15版Brock Biology of Microorganisms (2017)[45]教材中,第19章第6节“PCR methods of microbial community analysis”部分,详细介绍了基于细菌或真菌rRNA基因片段分析特定样品中微生物多样性的方法原理及实验方案;此外,由Eugene L. Madsen等主编的Environmental Microbiology:From Genomes to Biogeochemistry (2015)[46]教材中,第6章第9节“Cloning-based Metagenomic and related methods:procedures and insights”部分也详细地介绍了上述类似的方法原理及实验方案。整体来看,目前国外部分主流教材已经将基于高通量测序的微生物多样性分析方法编入,而在国内大学本科高等教育的《微生物学》教材中,对于微生物中常用的进化遗传分子标记如16S rRNA基因、真核生物18S rRNA基因及ITS序列有零星介绍,但对于如何利用这些分子标记对环境样品进行高通量测序并分析其微生物物种多样性,尚缺乏相对系统的介绍。此外,各专业对教材的选择需要结合本专业特点,使得当前大部分专业学生不能通过教材有效地学习和掌握该知识点。因此,鉴于各领域微生物多样性研究的快速发展及其重要地位,有必要在《微生物学》教材中增加该知识点内容。相关内容主要包括:(1)微生物多样性的概念及主要研究内容(1学时);(2)微生物多样性的主要研究方法技术、原理及数据分析(2学时);(3)结合教材的专业倾向性,重点介绍微生物多样性在特定领域的研究进展及应用实例(1学时)。针对各学科《微生物学》教学学时的差异,在教学学时充裕的情况下,可根据需要给学生补充讲解该部分内容;或者教师可结合专业需求,以专题讲座的方式拓展学生对该知识点的认识。此外,针对目前在部分专业课程学时压缩的同时不断向课程补充新的教学内容这一矛盾,可根据专业特点开展相关教学改革,对原教材中部分相对较陈旧或学生容易自学的内容适当压缩;同时,部分内容可组织学生以兴趣小组汇报讨论的方式学习,调动学生利用课外时间进行学习整理,同时也训练学生在文献查阅、知识点归纳、PPT制作及讲解等方面的综合素质。
3 《微生物学》教材中增加“微生物多样性”相关知识点的意义在《微生物学》及相关教材中增加“微生物多样性”相关知识点,不仅可以让学生系统地学习该研究领域的研究方法及基本知识点,帮助扩展与专业相关的微生物多样性研究前沿,还能更好地促进学生对一些问题的理解。
3.1 回答教材中一些直观的问题首先,可以解答教材中关于“未培养微生物是如何被发现的?”这一直观问题。如果在各领域《微生物学》教材中增加微生物多样性知识点及其研究方法,学生通过对该知识点的学习,能够轻松地理解科学家们具体是采用何种方法来检测和发现未知样品中未培养微生物的存在以及确定它们的种类及种群结构的。其次,可帮助学生理解教材中基于分子生物学的微生物系统分类的核心本质。通过对微生物多样性研究中选取的测序基因如细菌16S rRNA基因、真菌18S rRNA基因及ITS片段等基本信息的介绍,学生将更容易理解教材中微生物系统进化树的绘制依据及物种系统进化关系。最后,可加深学生对微生物多样性和人类健康、环境复杂关系等的理解。作者近几年在药学类本科生《微生物学》教学中,结合专业特点适当补充了微生物多样性相关知识点内容,学生兴趣浓厚,有部分学习小组还自行选择微生物多样性相关的话题做小组PPT汇报交流,一定程度上提升了教学效果。
3.2 帮助学生更好地理解目前大量关于微生物组学的研究微生物组是指一个特定环境或者生态系统中的全部微生物及其遗传信息,包括细胞群体和数量、全部遗传物质(基因组)。微生物组涵盖了微生物群及其全部遗传与生理功能,其内涵包括了微生物与其环境和宿主的相互作用,注重与数、理、化等学科的交叉,被用于解决健康、环境等问题的科技前沿。微生物组学则是以微生物组为对象,研究其结构与功能、内部群体间的相互关系和作用机制,及其与环境或者宿主的相互关系,并最终能够调控微生物群体生长、代谢等,为人类健康和社会可持续发展服务。
微生物组学已经成为一个新的热点研究领域,国际上已经推出了一系列重要的国家级研究计划,例如日本“人体元基因组研究计划”(2005)、加拿大“微生物组研究计划”(2007)、欧盟“人类肠道宏基因组计划”(2008)、美国“人类微生物组计划”(2008)、美国“国家微生物组计划”(2016)和中国“肠道宏基因组计划”(2017)等。从NCBI PubMed中微生物组学领域发表的文章和研究报告统计来看,从2001年到2016年间,文献数量从约100篇增加到超过7 000篇[47]。当前,微生物组学的内容非常前沿,因此对大学本科生特别是非生物相关专业的学生来讲也相对较难理解。在微生物组学研究中,最基础的内容之一也是微生物多样性及微生物群落结构。微生物组涵盖了微生物多样性,微生物多样性是微生物组的重要研究内容之一。如果在《微生物学》教材中增加微生物多样性相关知识点内容及研究方法,可方便感兴趣的学生在理解和掌握微生物多样性及其实验方法的基础上,能进一步学习和了解微生物基因组学等相关内容,拓展知识面。
4 展望微生物学是生物科学中一门重要的专业基础课程,在当前微生物学成果层出不穷的形势下,《微生物学》教材内容在保证经典微生物学的前提下,应当不断地扩充最前沿、最有突破性的微生物多样性科研成果。微生物学多样性相关内容在教材中可独立成章节,也可根据教材的专业倾向性将其基本概念、研究方法和在该领域取得的重要突破和进展等整合在适当章节中,这将极大地促进学生对微生物多样性相关研究前沿的学习和了解。
| [1] |
Chen XD. The China Microbiology teaching reform and personnel training under the trend of open education[J]. Microbiology China, 2018, 45(3): 471-472. (in Chinese) 陈向东. 开放式教育潮流下的我国微生物学教学改革与人才培养[J]. 微生物学通报, 2018, 45(3): 471-472. |
| [2] |
Epstein SS. Uncultivated Microorganisms[M]. New York: Springer, 2009.
|
| [3] |
Zhou JM, Shen RF. Dictionary of Soil Science[M]. Beijing: Science Press, 2013. (in Chinese) 周建民, 沈仁芳. 土壤学大辞典[M]. 北京: 科学出版社, 2013. |
| [4] |
Li JF. Microbial diversity research based on high-throughput sequencing data of 16S rRNA and metagenome[D]. Beijing: Master's Thesis of Tsinghua University, 2015 (in Chinese) 李俊锋.基于16S rRNA和宏基因组高通量测序的微生物多样性研究[D].北京: 清华大学硕士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10003-1016712741.htm |
| [5] |
Castelle CJ, Banfield JF. Major new microbial groups expand diversity and alter our understanding of the tree of life[J]. Cell, 2018, 172(6): 1181-1197. DOI:10.1016/j.cell.2018.02.016 |
| [6] |
Dinan TG, Cryan JF. Gut-brain axis in 2016: Brain-gut-microbiota axis-mood, metabolism and behaviour[J]. Nature Reviews Gastroenterology & Hepatology, 2017, 14(2): 69-70. |
| [7] |
Perveen N, Barot S, Alvarez G, et al. Priming effect and microbial diversity in ecosystem functioning and response to global change: a modeling approach using the SYMPHONY model[J]. Global Change Biology, 2014, 20(4): 1174-1190. DOI:10.1111/gcb.12493 |
| [8] |
Jobard M, Pessiot J, Nouaille R, et al. Microbial diversity supporting dark fermentation of waste[J]. Trends in Biotechnology, 2014, 32(11): 549-550. DOI:10.1016/j.tibtech.2014.09.005 |
| [9] |
Zhang JC, Wang XR, Huo DX, et al. Metagenomic approach reveals microbial diversity and predictive microbial metabolic pathways in Yucha, a traditional Li fermented food[J]. Scientific Reports, 2016, 6: 32524. DOI:10.1038/srep32524 |
| [10] |
Wu RN, Yu ML, Liu XY, et al. Changes in flavour and microbial diversity during natural fermentation of suan-cai, a traditional food made in Northeast China[J]. International Journal of Food Microbiology, 2015, 211: 23-31. DOI:10.1016/j.ijfoodmicro.2015.06.028 |
| [11] |
Glenwright AJ, Pothula KR, Bhamidimarri SP, et al. Structural basis for nutrient acquisition by dominant members of the human gut microbiota[J]. Nature, 2017, 541(7637): 407-411. DOI:10.1038/nature20828 |
| [12] |
Shabana, Shahid SU, Irfan U. The gut microbiota and its potential role in obesity[J]. Future Microbiology, 2018, 13(5): 589-603. DOI:10.2217/fmb-2017-0179 |
| [13] |
Han H, Li YY, Fang J, et al. Gut microbiota and type 1 diabetes[J]. International Journal of Molecular Sciences, 2018, 19(4): 995. DOI:10.3390/ijms19040995 |
| [14] |
Velmurugan G, Ramprasath T, Gilles M, et al. Gut microbiota, endocrine-disrupting chemicals, and the diabetes epidemic[J]. Trends in Endocrinology & Metabolism, 2017, 28(8): 612-625. |
| [15] |
Zhu WH, Winter MG, Byndloss MX, et al. Precision editing of the gut microbiota ameliorates colitis[J]. Nature, 2018, 553(7687): 208-211. DOI:10.1038/nature25172 |
| [16] |
Olson CA, Vuong HE, Yano JM, et al. The gut microbiota mediates the anti-seizure effects of the ketogenic diet[J]. Cell, 2018, 174(2): 497. DOI:10.1016/j.cell.2018.06.051 |
| [17] |
Macedo D, Filho AJMC, Soares de Sousa CN, et al. Antidepressants, antimicrobials or both? Gut microbiota dysbiosis in depression and possible implications of the antimicrobial effects of antidepressant drugs for antidepressant effectiveness[J]. Journal of Affective Disorders, 2017, 208: 22-32. DOI:10.1016/j.jad.2016.09.012 |
| [18] |
Luna RA, Foster JA. Gut brain axis: diet microbiota interactions and implications for modulation of anxiety and depression[J]. Current Opinion in Biotechnology, 2015, 32: 35-41. DOI:10.1016/j.copbio.2014.10.007 |
| [19] |
Li QR, Han Y, Dy ABC, et al. The gut microbiota and autism spectrum disorders[J]. Frontiers in Cellular Neuroscience, 2017, 11: 120. DOI:10.3389/fncel.2017.00120 |
| [20] |
Parashar A, Udayabanu M. Gut microbiota: implications in Parkinson's disease[J]. Parkinsonism & Related Disorders, 2017, 38: 1-7. |
| [21] |
Scheperjans F, Aho V, Pereira PAB, et al. Gut microbiota are related to Parkinson's disease and clinical phenotype[J]. Movement Disorders, 2015, 30(3): 350-358. DOI:10.1002/mds.26069 |
| [22] |
Zhou DB, Jing T, Chen YF, et al. Analysis methods for microbial population diversity in soils and their application prospect in the control of soil-borne diseases[J]. Chinese Journal of Tropical Agriculture, 2018, 38(2): 79-86, 112. (in Chinese) 周登博, 井涛, 陈宇丰, 等. 土壤微生物多样性研究方法及其在土传病害防治中的应用展望[J]. 热带农业科学, 2018, 38(2): 79-86, 112. |
| [23] |
Alegría A, Szczesny P, Mayo B, et al. Biodiversity in Oscypek, a traditional Polish cheese, determined by culture-dependent and -independent approaches[J]. Applied and Environmental Microbiology, 2012, 78(6): 1890-1898. DOI:10.1128/AEM.06081-11 |
| [24] |
Field KG, Olsen GJ, Lane DJ, et al. Molecular phylogeny of the animal kingdom[J]. Science, 1988, 239(4841): 748-753. DOI:10.1126/science.3277277 |
| [25] |
Yarza P, Yilmaz P, Panzer K, et al. A phylogenetic framework for the kingdom Fungi based on 18S rRNA gene sequences[J]. Marine Genomics, 2017, 36: 33-39. DOI:10.1016/j.margen.2017.05.009 |
| [26] |
Martin KJ, Rygiewicz PT. Fungal-specific PCR primers developed for analysis of the ITS region of environmental DNA extracts[J]. BMC Microbiology, 2005, 5: 28. DOI:10.1186/1471-2180-5-28 |
| [27] |
Zhang T, Wei XL, Zhang YQ, et al. Diversity and distribution of lichen-associated fungi in the Ny-Ålesund Region (Svalbard, High Arctic) as revealed by 454 pyrosequencing[J]. Scientific Reports, 2015, 5: 14850. DOI:10.1038/srep14850 |
| [28] |
Alves RJE, Minh BQ, Urich T, et al. Unifying the global phylogeny and environmental distribution of ammonia-oxidising archaea based on amoA genes[J]. Nature Communications, 2018, 9(1): 1517. DOI:10.1038/s41467-018-03861-1 |
| [29] |
Wu RN, Meng H, Wang YF, et al. A more comprehensive community of ammonia-oxidizing archaea (AOA) revealed by genomic DNA and RNA analyses of amoA gene in subtropical acidic forest soils[J]. Microbial Ecology, 2017, 74(4): 910-922. DOI:10.1007/s00248-017-1045-4 |
| [30] |
Jin D, Zhao SG, Zheng N, et al. Differences in ureolytic bacterial composition between the rumen digesta and rumen wall based on ureC gene classification[J]. Frontiers in Microbiology, 2017, 8: 385. |
| [31] |
Yang YD, Hu YG, Wang ZM, et al. Variations of the nirS-, nirK-, and nosZ-denitrifying bacterial communities in a northern Chinese soil as affected by different long-term irrigation regimes[J]. Environmental Science and Pollution Research, 2018, 25(14): 14057-14067. DOI:10.1007/s11356-018-1548-7 |
| [32] |
Zeng W, Zhang J, Wang AQ, et al. Denitrifying phosphorus removal from municipal wastewater and dynamics of "Candidatus Accumulibacter" and denitrifying bacteria based on genes of ppk1, narG, nirS and nirK[J]. Bioresource Technology, 2016, 207: 322-331. DOI:10.1016/j.biortech.2016.02.016 |
| [33] |
Yuan JL, Liu YQ. Microbiology and Immunology[M]. Beijing: China Press of Traditional Chinese Medicine, 2017. (in Chinese) 袁嘉丽, 刘永琦. 微生物学与免疫学[M]. 北京: 中国中医药出版社, 2017. |
| [34] |
Zhang XY. Microbiology and Immunology[M]. Beijing: China Medical Science Press, 2016. (in Chinese) 张雄鹰. 微生物学与免疫学[M]. 北京: 中国医药科技出版社, 2016. |
| [35] |
Shen GX, Xu W. Microbiology and Immunology[M]. 8th ed. Beijing: People's Medical Publishing House, 2016. (in Chinese) 沈关心, 徐威. 微生物学与免疫学[M]. 8版. 北京: 人民卫生出版社, 2016. |
| [36] |
Sang YX, Li XT. Food Microbiology[M]. Beijing: China Light Industry Press, 2017. (in Chinese) 桑亚新, 李秀婷. 食品微生物学[M]. 北京: 中国轻工业出版社, 2017. |
| [37] |
Wang GQ, Qiang H. Medical Microbiology[M]. Beijing: China Medical Science Press, 2016. (in Chinese) 王桂琴, 强华. 医学微生物学[M]. 北京: 中国医药科技出版社, 2016. |
| [38] |
Li F, Xu ZK. Medical Microbiology[M]. 9th ed. Beijing: People's Medical Publishing House, 2018. (in Chinese) 李凡, 徐志凯. 医学微生物学[M]. 9版. 北京: 人民卫生出版社, 2018. |
| [39] |
Le YQ, Wang SF. Environmental Microbiology[M]. Beijing: Chemical Industry Press, 2019. (in Chinese) 乐毅全, 王士芬. 环境微生物学[M]. 北京: 化学工业出版社, 2019. |
| [40] |
Zhang FM, Xiao CL. Medical Microbiology[M]. 3rd ed. Beijing: Peking University Medical Press, 2013. (in Chinese) 张凤民, 肖纯凌. 医学微生物学[M]. 3版. 北京: 北京大学医学出版社, 2013. |
| [41] |
Qi ZT. Medical Microbiology[M]. Beijing: Science Press, 2017. (in Chinese) 戚中田. 医学微生物学[M]. 北京: 科学出版社, 2017. |
| [42] |
Zhou CL. Microbiology[M]. 3rd ed. Beijing: China Medical Science Press, 2015. (in Chinese) 周长林. 微生物学[M]. 3版. 北京: 中国医药科技出版社, 2015. |
| [43] |
He PX. Advanced Microbiology[M]. Beijing: China Light Industry Press, 2017: 230-247. (in Chinese) 何培新. 高级微生物学[M]. 北京: 中国轻工业出版社, 2017: 230-247. |
| [44] |
Shen P, Chen XD. Microbiology[M]. 8th ed. Beijing: Higher Education Press, 2016: 310-348. (in Chinese) 沈萍, 陈向东. 微生物学[M]. 8版. 北京: 高等教育出版社, 2016: 310-348. |
| [45] |
Madigan MT, Bender KS, Bender KS, et al. Brock Biology of Microorganisms[M]. London: Pearson, 2017: 595-599.
|
| [46] |
Madsen EL. Environmental Microbiology: From Genomes to Biogeochemistry[M]. 2nd ed. New Jersey: Wiley-Blackwell, 2015: 280-290.
|
| [47] |
Liu SJ, Shi WY, Zhao GP. China microbiome initiative: opportunity and challenges[J]. Bulletin of the Chinese Academy of Sciences, 2017, 32(3): 241-250. (in Chinese) 刘双江, 施文元, 赵国屏. 中国微生物组计划:机遇与挑战[J]. 中国科学院院刊, 2017, 32(3): 241-250. |
 2019, Vol. 46
2019, Vol. 46