扩展功能
文章信息
- 孟娇, 刘丁玉, 黄灿, 韩北忠, 陈晶瑜
- MENG Jiao, LIU Ding-Yu, HUANG Can, HAN Bei-Zhong, CHEN Jing-Yu
- CRISPR/Cas基因编辑系统在原核微生物细胞工厂构建中的开发与应用
- Development and application of CRISPR/Cas genome editing system in the construction of prokaryotic microbial cell factories
- 微生物学通报, 2019, 46(10): 2730-2742
- Microbiology China, 2019, 46(10): 2730-2742
- DOI: 10.13344/j.microbiol.china.180808
-
文章历史
- 收稿日期: 2018-10-17
- 接受日期: 2018-12-12
- 网络首发日期: 2018-12-29
2. 天津大学化工学院 天津 300350
2. School of Chemical Engineering and Technology, Tianjin University, Tianjin 300350, China
原核微生物是生物圈中重要的组成部分,提供了重要的生物学资源,包括多样性的酶类、丰富功能的代谢网络以及各种分子生物学与合成生物学遗传元件[1]。在能源危机背景下,多样性的原核微生物在以生物炼制替代传统化学合成为目标的工业微生物生产中具有重要价值[2-4]。基因工程是20世纪70年代发展起来的一种新型生物技术,其发展从根本上改变了生物技术的研究和发展模式。利用基因工程技术可以直接、有目的性地在染色体水平上改变微生物的遗传性状。特别是随着DNA重组技术的完善和发展,以基因水平为核心的现代分子定向育种技术越来越受到工业微生物育种学家的关注,通过导入外源DNA,微生物可以表达出自身原来没有的蛋白质。同样,利用基因工程技术也可以使微生物中原来存在量极低但有重要工业生产价值或医药用途的产品得以大量生产[4]。目前,研究者们运用微生物基因组工程育种技术成功实现了多种化工[5-6]、药品[7]、食品[8]等产品的代谢工程生产,并取得了丰硕的成果。
在此过程中,简单、快捷、高效的基因组编辑技术是微生物基因组工程育种的技术基础,通过基因组设计,利用基因组编辑技术在基因组尺度优化代谢网络是微生物代谢工程育种的关键。近几年发展的基因组编辑工具主要包括:核酸酶介导的编辑系统,如基于锌指核酸酶(Zinc-finger nucleases,ZFNs)[9]和转录激活因子样效应因子核酸酶(Transcription activatorlike effector nucleases,TALENs)[10]系统等;多元自动基因组工程(Multiplex automated genome engineering,MAGE)[11];可追踪多元重组工程(Trackable Multiplex Recombineering,TRMR)[12]。其中,核酸酶介导的编辑系统虽具有性能稳定、结构简单等优势,但存在易受基因组上下游序列结构影响、需重复构建融合蛋白、序列甲基化敏感等不足;MAGE和TRMR虽然在基因组编辑技术具有“里程碑式”意义,却有宿主局限性,目前仅限应用于大肠杆菌。
成簇的规律间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)/ CRISPR相关蛋白(CRISPR-associated proteins,Cas)基因编辑系统是从细菌和古细菌的免疫防御研究发展而来,该系统因结构简单、成本低廉、作用高效等优势受到研究者的广泛重视,已经成功应用于多种动物、植物和微生物等的基因组编辑,并取得了显著成果,引发了一场位于多领域的“CRISPR大爆炸”[13-15]。本文将立足于CRISPR/Cas系统的原理,重点概述CRISPR/Cas基因编辑技术在原核微生物中的建立、优化与应用,并探讨该系统所面临的主要问题与未来发展方向。
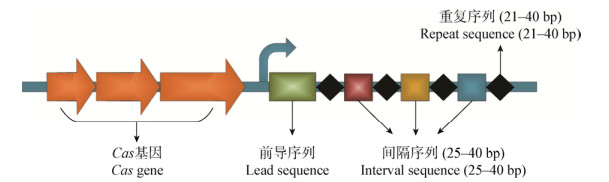
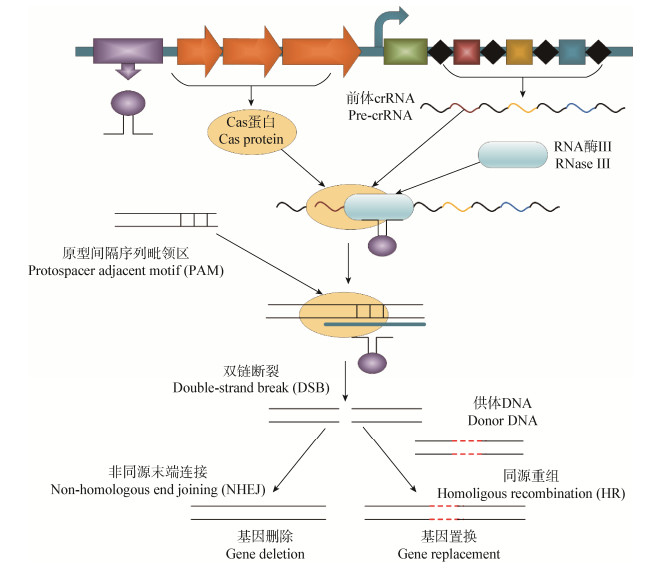
1 CRISPR/Cas系统概述 1.1 CRISPR/Cas系统结构与作用机制CRISPR/Cas系统是细菌和古细菌在与噬菌体竞争中进化出来的一种获得性免疫系统,如图 1所示,主要包括两部分:长度范围为21−40 bp的重复序列(Repeats)与25−40 bp的间隔序列(Spacers)交替排列形成的CRISPR基因座和在CRISPR基因座附近存在的成簇Cas基因[16-18]。其中,CRISPR基因座的Spacers中储存了侵入宿主的核苷酸信息,Cas基因用于编码具有核酸酶、解旋酶、聚合酶等功能的一系列蛋白。外源核苷酸首次侵入宿主细胞后,Cas蛋白(通常为Cas1和Cas2)会从中截取一段原型间隔区(Protospacer)并将其插入到CRISPR基因座中,这段序列在外源核苷酸的二次侵入后会被诱导生成未成熟的CRISPR RNA (Pre-crRNA),随后经过Cas蛋白和核酸酶的加工形成成熟的crRNA,并与系统其它组件结合形成CRISPR核糖核蛋白复合物(CRISPR ribonucleoprotein,crRNP),crRNP中的crRNA含有能与原型间隔区互补配对的间隔序列,从而能够引导crRNP对外源核苷酸进行特异性切割(图 2),最终实现了对外源核苷酸的特异性免疫[19]。
|
| 图 1 CRISPR系统结构 Figure 1 The system structure of CRISPR |
|
|
|
| 图 2 CRISPR/Cas系统介导的基因编辑 Figure 2 Gene editing mediated by CRISPR/Cas system |
|
|
分析CRISPR数据库(http://crispr.i2bc.paris-saclay.fr)结果显示,大约90%的古细菌和40%的细菌中具有CRISPR序列[20]。如此广泛的存在也造就了CRISPR/Cas系统种类的多样性,但无论哪一类型的CRISPR系统,都可以从功能模块化的角度将其划分为适应模块(Adaptation module)、响应模块(Effector module)和附件模块(Accessory module)[18, 21]。其中响应模块包含了与crRNA成熟以及识别和降解外源核苷酸相关的Cas蛋白。响应模块的不同导致其分化种类异常丰富,也构成了目前CRISPR/Cas系统分类的基础,根据响应模块的不同,研究者将目前发现的CRISPR/Cas系统划分为2类6型,共24种亚型[22-23]。如表 1所示,第一大类CRISPR/Cas系统由多亚基效应蛋白复合体发挥免疫功能,包括TypeⅠ、Type Ⅲ和Type Ⅳ;第二大类CRISPR/Cas系统由具有多结构域的单一蛋白组成,包括Type Ⅱ、Type Ⅴ和Type Ⅵ。目前对TypeⅡCRISPR/Cas9系统的研究最为透彻,其主要包含Cas9蛋白、tracrRNA、crRNA和RNase Ⅲ四个元件[24]。Cas9是一个具有两个核酸结构域(RuvC和HNH)的多功能蛋白,不仅参与crRNA的成熟,还可以在tracrRNA:crRNA复合体的引导下切割靶序列DNA,形成一个平末端的双链切口,同时能够识别位于原型间隔区下游的PAM序列(5′-NGG-3′),防止出现自身免疫[25]。
| 分类 Classification |
型别 Type |
亚型 Subtype |
特征蛋白 Characteristic protein |
核酸酶活性类型 Nuclease activity type |
自己/非己序列的识别 Self/non-self sequence identification |
| 1 | Ⅰ | A–F,U | Cas3 | 切割DNA形成SSB Cutting DNA to form SSB |
Protospacer 5′端的PAM PAM at the 5′-end of Protospacer |
| Ⅳ | – | 未知 Unknown |
未知 Unknown |
未知 Unknown |
|
| Ⅲ | A–D | Cas10 | 切割RNA或DNA Cutting RNA or DNA |
crRNA与靶序列附近的碱基错配 Base mismatch between crRNA and target sequence |
|
| 2 | Ⅱ | A–C | Cas9 | 切割DNA形成DSB (平末端) Cutting DNA to form DSB (Blunt end) |
Protospacer 3′端的PAM PAM at the 3′-end of Protospacer |
| Ⅴ | A–E,U | Cas12 | 切割DNA形成DSB (粘性末端) Cutting DNA to form DSB (Sticky end) |
Protospacer 5′端的PAM PAM at the 5′-end of Protospacer |
|
| Ⅵ | A–C | Cas13 | 切割并破坏RNA二级结构 Cutting and destroying RNA secondary structure |
Protospacer 3′端的PFS PFS at the 3′-end of Protospacer |
|
| Note: SSB: Single-stranded break; DSB: Double-stranded break; PAM: Protospacer-adjacent motif; PFS: Protospacer flanking site; crRNA: CRISPR RNA. | |||||
原核微生物CRISPR基因编辑系统的建立通常分为3个阶段:首先需要选择合适的CRISPR/Cas系统类型,不同类型和家族的CRISPR/Cas系统具有不同的特点和应用范围,可适用于不同的原核微生物;随后需要确定CRISPR/Cas基因编辑平台的搭建方案,即系统中各元件的表达方式;最后再根据宿主的遗传背景和特性,对CRISPR/Cas基因编辑系统进行合理的优化以扩大系统的效率和适用性,提高系统的应用质量。
2.1 CRISPR/Cas系统的选择CRISPR/Cas基因编辑系统开发的核心在于基因编辑平台的搭建,而选择合适的CRISPR/Cas系统则是基因编辑平台搭建的基础。根据目前在原核微生物中CRISPR/Cas系统成功应用的案例,可将其分为TypeⅡ型CRISPR/Cas系统与非TypeⅡ型CRISPR/Cas系统。
2.1.1 TypeⅡ型CRISPR/Cas系统在诸多类型CRISPR系统中,源于化脓链球菌(Streptococcus pyogenes)的TypeⅡCRISPR/Cas9被广泛用于原核微生物基因编辑系统的构建。2013年,Jiang等[26]以大肠杆菌为例首次证实了CRISPR/Cas9系统在原核微生物中的适用性,为研究者们进一步在原核微生物中系统地开发CRISPR/Cas9介导的基因编辑技术提供了支持。随后,Jiang等[27]首次在大肠杆菌中建立了完善的CRISPR/Cas9基因编辑循环系统,作者详细介绍了构建CRISPR编辑系统的每一个环节:包括Cas9和gRNA的表达(表达载体的选择,启动子等表达元件的选择)、供体DNA的提供方式以及如何完成迭代操作;最终作者利用此系统最高以接近100%的效率完成单基因敲除和插入,同时尝试进行双位点和三位点同时修饰,分别达到了97%和47%的效率。Jiang等[27]的研究为后续的研究者们在不同原核微生物中开发CRISPR基因编辑系统提供了“研究模板”,该系统中涉及的每个环节都可以延伸到其它菌株,在任何菌株中构建CIRSPR基因编辑系统的过程都是逐个解决这些环节的过程。此后,TypeⅡCRISPR/Cas9系统被应用于多种原核微生物,如:枯草芽孢杆菌[28]、柠檬明串珠菌[29]、蓝藻细菌[30]、链球菌属[31]、链霉菌属[32]、罗伊氏乳杆菌[33]、梭菌属[34]等。
然而随着以Cas9蛋白为基础的CRISPR系统的广泛应用,研究者们也发现了Cas9蛋白存在的局限性,主要体现在其造成的双链断裂对宿主产生的毒性。为了解决这一问题,研究者通过失活Cas9蛋白单个核酸内切酶结构域获得Cas9切口酶(Cas9 nickase,Cas9n),与双链切割活性Cas9相比,Cas9n对宿主细胞生长压力小,单链切口易于修复而且脱靶几率较小,因此更加适用于非同源末端连接(Non-homologous end joining,NHEJ)缺陷的原核微生物[24]。例如在梭状芽孢杆菌(Clostridium cellulolyticum)中,由于宿主自身NHEJ修复机制表达较弱,Cas9蛋白会极大地抑制细胞生长,因此一直无法建立有效的CRISPR/Cas9编辑系统,Xu等[35]在该菌株中采用了CRISPR/Cas9n基因编辑系统成功克服了这一问题,最终单点编辑效率可达95%。值得注意的是,由于Cas9n是对染色体进行单链切割,对于Cas9n可能发生的脱靶效应,被切割的DNA链会利用另外一条未被切割的互补链作为同源重组模板进行精确修复,因此并不会引起不必要的基因突变。
2.1.2 非TypeⅡ型CRISPR/Cas系统在CRISPR/Cas系统中,除了TypeⅡ型Cas9以及它的突变蛋白Cas9n外,另一种Type V-A型Cas蛋白也在原核生物基因编辑系统中发挥着重要应用。这种Cas蛋白最初由张峰课题组[36]于金黄色葡萄球菌中发现,最初被命名为Cpf1,随着近年来研究的深入,将其进一步归类为Cas12a;Cas12a比标准的spCas9更小,且在切割基因组时只需要crRNA参与,操作上比Cas9更简单;此外Cas12a识别与Cas9不同的PAM位点,在目标位点的选择上使CRISPR/Cas系统更具灵活性。
Cas12a的发现最重要的意义在于为CRISPR基因编辑系统多提供了一种选择,在传统的CRISPR/Cas9基因编辑系统构建遇到障碍时便于提供新的途径。这在研究者们构建谷氨酸棒杆菌的CRISPR/Cas编辑系统的研究中体现得尤为明显。来自于不同课题组的研究人员在谷氨酸棒杆菌中建立CRISPR/Cas编辑系统时均发现spCas9对该菌株具有一定的细胞毒性,导致无法获得转化子。Cho等[37]采用传统的研究思路,进一步寻找合适于宿主的Cas9的突变体,最终发现来自天蓝色链霉菌密码子的Cas9对谷氨酸棒杆菌的毒性较低,在结合了单链重组系统后最终在宿主内实现了400 bp的片段敲除,但未继续进行其他的编辑操作。Jiang等[38]在谷氨酸棒杆菌中遇到了同样的问题。与Cho等不同的是,Jiang等直接将CRISPR/Cas9系统替换为CRISPR/Cpf1系统,成功实现了基因敲除、基因插入和位点突变操作,其中位点突变效率可达到100%。从报道的最终结果和工作的全面性上来评价,Jiang等的工作要优于Cho等,也说明了Cas12a比Cas9要更适用于谷氨酸棒杆菌。这两项研究的结果也表明:当遇到不适合spCas9的宿主时,选择非spCas9的CRISPR系统来匹配宿主是一种比调试spCas9更加快捷高效的策略。在Jiang等的研究之后,Yan等[39]先后在大肠杆菌、鼠伤寒耶尔森菌和耻垢分支杆菌中均成功建立了CRISPR/Cas12a编辑系统,证明了Cas12a在原核微生物同样具有相对广泛的适用范围。
除了表达异源CRISPR/Cas系统以外,利用宿主自身内源性CRISPR/Cas系统也是目前的一个重要研究方向,其同样可以起到克服传统Cas9局限性、提高CRISPR系统适用范围的功效。Pyne等[40]在开发巴氏固氮梭菌(Clostridium pasteurianum) CRISPR/Cas9系统时发现正确表达Cas9蛋白的转化子数目极少,因此选择利用宿主内源性TypeⅠCRISPR/Cas系统进行编辑系统开发,作者利用此系统以100%的效率敲除了cpaAIR基因,其转化子数目要比CRISPR/Cas9基因编辑系统提高4倍。值得注意的是,利用内源性CRISPR系统只需要表达crRNA和供体DNA模板两个元件,进一步简化了系统结构,因此在多位点编辑方面显示出广阔的应用前景。
2.2 CRISPR/Cas基因编辑平台的搭建方案选择了CRISPR/Cas系统的类型后,随后的关键在于确定基因编辑平台的搭建方案。目前原核微生物中CRISPR基因编辑系统构建方案有单质粒表达、多质粒表达、染色体整合表达3种。
2.2.1 单质粒表达策略单质粒表达是CRISPR/Cas基因编辑系统构建中最常见的构建策略,即将Cas、gRNA、供体DNA构建在同一表达载体上,该策略在早期的CRISPR/Cas基因编辑系统研究中广泛使用,结合GoldenGate、CPEC等高效构建质粒的分子克隆技术可以极大缩短整个编辑周期。在遗传操作过程中,仅需一轮质粒转化便可完成对基因组的切割和重组修复,再通过质粒消除系统消除质粒,从而可以实现循环编辑。Cobb等[41]在链霉菌属中采用了单质粒表达策略构建CRISPR/Cas9基因编辑系统,该系统操作简单、过程简捷,可应用于3种不同的链霉菌中,片段敲除效率最高可接近100%。Altenbuchner等[28]仿照Cobb等的方法,用同样的策略首次在枯草芽孢杆菌中构建了CRISPR/Cas9基因编辑系统,在基因敲除和单点突变修饰方面均取得了显著成效。此外,单质粒系统在其它一些兼容性质粒较少、转化效率较低的菌株中均有应用,如:谷氨酸棒杆菌[38]、永达尔梭菌[42]、贝氏梭状芽孢杆菌[34]、解纤维梭菌[43]、链霉菌属[32]等。
2.2.2 多质粒表达策略单质粒系统虽然在便捷性上有一定优势,但是其缺点也很明显,最主要的是无法进行更加复杂的基因编辑操作。受制于质粒大小的限制,单质粒载体上可插入的供体DNA大小受限,因此无法用于长片段的插入和多位点的同时编辑,而多质粒表达策略则能够很好地解决这些问题。
同样是在枯草芽孢杆菌中构建CRISPR/Cas9基因编辑系统,So等[44]在之前报道的基础上改用双质粒系统,pHCas9质粒用于表达Cas9蛋白,pB0A质粒则同时提供gRNA和供体DNA,分别转入2个质粒,保证转化效率的同时延长诱导时间,让2个质粒充分表达发挥作用;该系统除了能高效率完成基因敲除和点突变外,还成功在基因组上插入了绿色荧光蛋白基因。同时借助双质粒系统,供体DNA的长度更加自由,作者还研究了不同同源臂长度对编辑效率的影响。双质粒系统是现在CRISPR/Cas编辑系统中最常见的构建策略,在大肠杆菌[45]、枯草芽孢杆菌[44]、谷氨酸棒杆菌[38]、丙酮丁醇梭菌[46]、罗伊氏乳杆菌[33]、蓝藻[46]等多种菌属中均被采纳。例如,Li等[47]在大肠杆菌中构建了一个双质粒CRISPR/Cas9基因编辑系统,该系统单点编辑效率最高可接近100%,同时还设计了质粒消除系统,通过诱导位于Cas9表达质粒上的gRNA表达,使另外一个gRNA表达质粒消除,从而可以进行下一轮编辑,显示出双质粒系统的灵活性。
在大肠杆菌这种分子克隆技术非常成熟且可选质粒较多的菌株中,甚至可以开发三质粒系统。Zhu等[48]在大肠杆菌中分别将Cas9、gRNA、供体DNA用3个质粒表达,由于表达载体各自独立,质粒构建更加方便,在相应载体上可构建多个gRNA或供体DNA;借助菌株本身高效的转化效率,大肠杆菌利用三质粒系统进行了高效率的三位点修饰,进一步体现了多质粒系统的优势。
2.2.3 染色体整合表达策略染色体整合表达策略是将Cas基因和gRNA表达框整合到染色体上表达,但在诸多CRISPR/Cas基因编辑系统构建方案中并不常见,主要原因是其操作流程非常复杂,特别是如果要形成迭代循环系统的话一般需要再引入一套“无痕操作”系统来消除gRNA表达框,这种策略与传统基于负筛选标记的基因编辑策略相比反而更加复杂。然而,染色体整合策略也具有其独特的优势,主要表现在:(1)染色体整合表达能够保证Cas蛋白和gRNA的稳定表达,而质粒表达可能会出现质粒丢失的情况;(2)染色体整合表达比多拷贝质粒对宿主造成更小的生理负担;(3)染色体整合表达策略不需要过多关注于质粒的转化效率对系统编辑效率的影响。一个经典的案例是Westbrook等[49]在枯草芽孢杆菌中构建的CRISPR/Cas9基因编辑盒策略,该作者分别将spCas9和gRNA表达框整合到染色体的lacA和thrC位点,并提供dsDNA作为供体DNA模板以实现对枯草芽孢杆菌的基因组编辑。值得注意的是,该工具盒除了达到高效的单位点编辑效率外,还能够实现基因组上双基因的同时敲除,效率高达85%,目前尚未有在枯草芽孢杆菌中利用质粒表达策略进行多位点编辑的报道。由此可见,染色体整合表达策略在一定程度上能够补充质粒表达策略的不足,对于扩大CRISPR/Cas基因编辑系统在原核微生物中的应用具有重要的意义。
2.3 CRISPR/Cas基因编辑系统的优化 2.3.1 通过引入外源重组系统提高基因编辑效率CRISPR/Cas基因编辑系统的本质是依靠Cas蛋白切割基因组形成缺口,再通过同源重组修复以获得正确基因型,因此提高同源重组概率是提升基因编辑效率的一个有效手段,而其中最有效的方式是引入外源重组系统。其中,最经典的案例是λ-red重组系统的引入。
λ-red重组系统是一种基于λ噬菌体Red重组酶的重组系统,仅需35 bp长的同源臂长度便能够高效介导寡核苷酸链与染色体之间的同源重组,Jiang等[26]在大肠杆菌中构建CRISPR/Cas9基因编辑系统时,发现仅依赖于大肠杆菌内源性同源重组系统,基因编辑效率仅为0.7%,但如果同时表达λ-red重组系统,则能够将编辑效率提升至65%。此后,λ-red重组系统对CRISPR/Cas9基因编辑效率的大幅度提升得到了研究者的广泛关注,并将此优化策略应用到各自的研究中[27, 48, 50-55]。除了λ-red重组系统以外,也有通过引入RecT重组酶提高CRISPR基因编辑效率的报道[33, 37]。
2.3.2 通过改变供体DNA模板提供方式以提高基因编辑效率除了引入外源重组系统以外,供体DNA模板提供方式也是影响重组概率的关键因素。供体DNA模板能够以线性片段或者质粒的形式提供,其中线性片段又可分为线性ssDNA (Single-stranded DNA)和线性dsDNA (Double-stranded DNA),不同形式的模板提供方式对于基因组编辑效率的影响也不同。在λ-red重组系统存在的情况下,以ssDNA的形式提供模板要优于dsDNA,显示出更高的编辑效率,但是因为ssDNA的长度限制,无法提供较长的同源臂,因此无法进行大片段的敲除或者插入。除了线性片段,更多的研究采用质粒形式提供供体DNA,虽然构建质粒会提高实验操作的复杂程度,但是在一些原核微生物中,质粒转化效率相比于线性片段要高很多,同时以质粒形式提供供体DNA模板可以保证片段的稳定存在并延长重组效应的时间,结合对操作流程的改进后,可以在编辑效率和编辑时间上得到明显改进。Zhao等[52]在大肠杆菌中开发CRISPR/Cas9系统时以质粒形式提供供体DNA,供体DNA以游离质粒形式转入宿主后,挑取阳性转化子进行培养,同时诱导Cas9和gRNA表达,培养一段时间后进行涂板筛选。这一过程中不仅Cas9和gRNA表达更充分,而且供体DNA作用时间更长,最终可以在3 d内完成从质粒构建到获得正确基因型菌株的过程。与该菌株中大多数采用线性片段的方法相比,Zhao等开发的流程更加省时和节约成本。
2.3.3 通过降低菌株突变率以提高基因编辑效率提高CRISPR基因编辑效率的另外一个有效策略是降低菌株突变率。宿主靶位点Spacer区域的缺失以及PAM序列的碱基突变是造成假阳性菌出现的主要原因,因此提高基因编辑效率可以通过降低菌株突变率来实现。在野生型大肠杆菌中存在错配修复(Mismatch repair,MMR)系统,该系统能够识别发生错配的碱基并利用正确DNA链作为模板进行精确修复。研究者发现如果失活MMR系统,可以将ssDNA重组效率大幅度提升,但是也会造成基因组的不稳定性,容易发生基因突变等问题。Li等[47]对比了野生型大肠杆菌中MMR系统失活与否对CRISPR/Cas9基因组编辑系统的影响,发现在没有失活MMR系统的菌株中假阳性菌数目比失活了该系统的菌株中减少了90%,而基因编辑效率则提高了475%,从而证实了降低菌株突变率是提高基因编辑效率的一个有效策略。
3 原核微生物细胞工厂构建中CRISPR/Cas基因编辑系统的应用 3.1 在传统代谢工程研究中的应用利用CRISPR/Cas基因编辑系统对宿主细胞进行代谢网络调控是该系统一个重要的应用方向。与传统的基因编辑策略相比,CRISPR/Cas基因编辑技术具有效率高、成本低、应用范围广等优势,因此在短时间内便被应用于多种化学品的代谢工程改造和生产中(表 2)。Heo等[56]将CRISPR/Cas9技术应用于大肠杆菌中丁醇的生产,通过高效精确地修饰gltA基因的5′-UTR区域,调控gltA基因的表达水平,从而改变TCA循环和丁醇合成之间的碳通量分配,获得一系列不同gltA表达水平的丁醇生产菌株,其中最优菌株柠檬酸合成酶活性为出发菌的55%,丁醇产量提高了1.3倍。Meng等[5]在大肠杆菌琥珀酸的生产研究中利用λ-red重组系统并未实现外源pyc基因在大肠杆菌染色体上的整合,然而利用CRISPR/Cas9基因编辑技术,成功将来自谷氨酸棒杆菌大片段的pyc基因整合到大肠杆菌的基因组中,细菌的比生长速率提高了39%,丙酮酸副产物的产量减少了50%,琥珀酸得率提升至1.4 mol/mol葡萄糖,达到理论最大得率的82%。Bassalo等[57]利用CRISPR/Cas9基因编辑系统,在大肠杆菌中进行异丁醇的生产,该研究利用转化耦联重组技术(Transformation-associated recombination,TAR)将5个异丁醇合成相关基因(alsS、ilvC、ilcD、kivD、adhA)进行快速组装,获得了一个长达10 kb的基因簇,随后发挥CRISPR系统在基因编辑方面的优势,将该基因簇一步插入大肠杆菌基因组中,从而实现了异丁醇合成途径在菌株中的一步构建,最后通过筛选获得的最优菌株,其异丁醇产量可达2.2 g/L。Zhu等[48]通过构建的三质粒CRISPR/Cas9系统优化大肠杆菌的木糖利用能力,凭借高效的多位点编辑效率,同时调控木糖利用途径中3个靶点的RBS序列,从得到的菌株中随机挑选200株进行验证,73%的菌株达到三位点的同时突变;通过富集筛选后,最优菌株的木糖利用率与出发菌株相比得到了3倍的提升,为进一步构建高效利用木质纤维素资源生产化学品的细胞工厂奠定了基础。
| 菌种 Strains |
代谢工程应用 Metabolic engineering application |
策略 Strategies |
产量/倍数 Production/Fold change |
参考文献 References |
| 大肠杆菌 Escherichia coli |
β-胡萝卜素 β-Carotene |
迭代编辑 Iterative editing |
2.0 g/L/提高3倍 2.0 g/L/Increase by 3 times |
[47] |
| 木糖利用 Xylose utilization |
木糖利用途径相关基因RBS替换 Replacing the RBS of related genes in the xylose utilization pathway |
–/利用率提高3倍 –/Increase by 3 times at utilization rate |
[48] | |
| 脂肪酸 Fatty acid |
引入外源脂肪酸合成途径 Introduction of exogenous fatty acid synthesis pathway |
–/提高5.3% –/Increase by 5.3% |
[53] | |
| 丁醇Butanol | 修饰gltA基因的5'-UTR区域 Modification of the 5'-UTR region of the gltA gene |
1.03 g/L/提高1.3倍 1.03 g/L/Increase by 1.3 times |
[56] | |
| 异丁醇 Isobutanol |
引入外源异丁醇合成途径 Introduction of exogenous isobutanol synthesis pathway |
2.2 g/L/– | [57] | |
| 异丙醇 Isopropanol |
异丙醇合成途径相关基因RBS替换 Replacing the RBS of related genes in the isopropanol synthesis pathway |
7.1 g/L/– | [58] | |
| 1, 4-丁二醇 1, 4-Butanediol |
结合CRISPR和CRISPRi系统对1, 4-丁二醇合成途径进行改造和调节 Modification and regulation of the 1, 4-butanediol synthesis pathway in combination with CRISPR and CRISPRi systems |
1.8 g/L/– | [59] | |
| 谷氨酸棒杆菌 Corynebacterium glutamicum |
γ-氨基丁酸 γ-Aminobutyric acid |
敲除γ-氨基丁酸合成的竞争途径基因 Deletion of competitive pathway genes for γ-aminobutyric acid synthesis |
27.5 g/L/– | [37] |
| L-脯氨酸 L-Valine |
proB基因密码子饱和突变 Saturated mutation of the proB gene codon |
6.6 g/L/– | [38] | |
| 蓝藻 Cyanobacteria |
琥珀酸 Succinic acid |
琥珀酸合成途径代谢网络改造 Modification of metabolic network of succinic acid synthesis pathway |
435 μg/L/提高11倍 435 μg/L/Increase by 11 times |
[30] |
| 枯草芽孢杆菌 Bacillus subtilis |
环糊精葡萄糖基转移酶 Cyclodextrin glucosyltransferase |
敲除srfC, spoIIAC, nprE, aprE和amyE Deletion of srfC, spoIIAC, nprE, aprE and amyE | 320 U/mL/提高2.5倍 320 U/mL/Increase by 2.5 times |
[60] |
| 丙酮丁醇梭菌 Clostridium acetobutylicum |
异丙醇 Isopropanol |
引入外源异丙醇合成途径 Introduction of exogenous isopropanol synthesis pathway |
4.45 g/L/– | [46] |
| 噬纤维梭菌与拜氏梭菌 Clostridium cellulovorans and Clostridium beijerinckii |
增强溶剂 Enhanced solvent |
结合CRISPR和CRISPRi系统对代谢网络进行改造 Modification of metabolic networks in combination with CRISPR and CRISPRi systems |
22.1 g/L/提高3.2倍 22.1 g/L/Increase by 3.2 times |
[61] |
| 糖丁酸梭状芽胞杆菌 Clostridium saccharoperbutylacetonicum |
丁醇 Butanol |
敲除pta和buk Deletion of pta and buk | 19 g/L/– | [62] |
以上研究均充分发挥了CRISPR/Cas系统在基因编辑方面的优势,在高效精确修饰、大片段整合和多位点编辑方面对代谢工程菌株构建或细胞工厂开发提供了极大帮助。此外,除了对单一代谢途径的调控,CRISPR/Cas系统更大的优势可以体现在对代谢网络的综合调控。例如,Li等[47]应用自主开发的CRISPR/Cas9基因编辑系统进行大肠杆菌高产β-胡萝卜素的生产,综合运用CRISPR/Cas9基因编辑技术,对β-胡萝卜素合成途径、MEP途径和中心碳代谢途径进行逐个优化,通过迭代优化的方法获得最优菌株,再将上下游模块进行不同基因型的组合,从中筛选到最优表型的菌株,最终β-胡萝卜素的产量在5 L发酵罐中达到2.0 g/L;在整个过程中一共测试了33个基因靶点,构建了103株不同靶点组合修饰的菌株,最终菌株中包含15个基因靶点修饰。该研究是CRISPR基因编辑系统在代谢工程中的一个综合应用,从关键基因的调控到单一途径的优化,再扩展到不同途径的协同组合优化,采用传统的基因编辑技术很难高效完成这种大规模的组合修饰。
3.2 高通量技术开发CRISPR/Cas基因编辑系统除了直接进行位点编辑优化代谢途径外,也可以与现有的其他高通量编辑技术和高通量分析方法相结合,进一步开发更加完善的高通量技术。Ronda等[63]通过结合CRISPR/Cas9基因编辑系统与MAGE设计理念开发出一种新型高通量基因编辑系统——CRMAGE,该系统可以大幅度提高基因编辑效率,能够将密码子替换概率从5.4%提升至99.7%,同时该系统也显示出具有多点编辑能力,在单轮循环中能够以几乎100%的效率实现双位点碱基替换。而Garst等[64]开发了一种基于CRISPR的可追踪基因组工程技术(CRISPR-enabled trackable genome engineering,CREATE),该技术最大限度地利用CRISPR/Cas9编辑技术在大肠杆菌中的高效性,在基因组上万个位点中同时引入突变文库,通过富集筛选并借助高通量的深度测序手段在碱基水平上追踪基因型与表型之间的关系;与上一代多元重组工程相比,CREATE技术将编辑数目提高了两个数量级,同时也可以用于研究多基因对表型的叠加影响,显示出巨大的应用潜力;该作者利用CREATE技术准确定位了fis、fadR、rho和fnr四个基因中与乙酸耐受性有联系的突变位点,这些位点的发现有助于对乙酸耐受菌株耐受性机理的进一步解析。
3.3 在工业生产规模微生物发酵中的应用对于原核工业微生物来说,外源DNA片段(质粒或噬菌体)污染会极大地损害菌株的安全性和经济性。传统防治策略像控制感染源、菌株轮换以及基因工程策略能够有效地阻止外源性感染,却对于清除内源性感染方面成效不大[65]。而基于CRISPR/Cas系统构建的防治策略则为清除内源性感染提供了一个新的思路。近期,一个相关的进展是Jakutyte-Giraitiene等[66]在枯草芽孢杆菌表达了源于嗜热链球菌的CRISPR/Cas9系统,并利用此系统将原噬菌体SPP1敲除,同时发现菌株的SPP1抗性水平提高至EOP=1×10−6。而Jang等[29]则成功利用CRISPR/Cas9系统消除了Leuconostoc citreum CB2567中的一个隐蔽性质粒,从而保证了该菌株应用于食品发酵中的安全性。
除了能够用于清除外源DNA片段,CRISPR/Cas基因编辑系统还能被开发成生物抗菌剂,利用系统的细胞毒性来清除杂菌。Gomaa等[67]的研究证实经过合适的crRNA靶位点选择,能够利用CRISPR/Cas系统对混菌中单一菌群进行抑制。然而,若想将此抗菌剂工业化应用还需要解决两个问题:一是如何实现抗菌系统的转染;二是如何设计靶位点实现对复杂环境中微生物组的普遍抑制。
4 结语与展望经过短短几年的发展,基于CRISPR/Cas的基因编辑系统已经在多种原核微生物中得到开发和改进,成为了一种重要的基因编辑工具。与传统的基因编辑方法相比,它具有3个核心优势:精简的系统结构、多位点的编辑能力、高效的编辑效率。这些优势赋予了其在多领域广阔的应用前景,但CRISPR/Cas基因编辑系统在原核微生物中尚未得到大规模的应用,其中一个主要的原因是现有的改进措施还未使其达到最佳优化状态。为了进一步扩大CRISPR/Cas基因编辑系统应用范围,充分发挥系统优势,未来的改进方向主要包括以下几点:
(1) 引入NHEJ修复机制。NHEJ修复机制的引入一方面可以缓解异源Cas效应蛋白对宿主的细胞毒性,从而可以更加容易地实现对染色体的多位点编辑;另一方面也可以进一步简化系统结构,不需要提供供体DNA模板便可进行基因功能失活,结合多位点编辑能力还能够用于高通量基因失活工具的开发,对于阐明基因功能、解析代谢途径等研究具有重要的意义。目前引入NHEJ修复机制可以通过异源表达DNA末端结合蛋白Ku和多功能连接酶LigD来实现[68]。
(2) 开发宿主内源性CRISPR/Cas系统。目前在大约45%的细菌中发现存在自身CRISPR免疫系统,合理利用这部分系统不仅可以突破非模式菌株中可用载体种类不足与现有转化技术效率低下的技术限制,还可以在一定程度上解决表达异源CRISPR/Cas系统存在的细胞毒性以及表达不稳定等问题,对于进一步扩大CRISPR/Cas基因编辑系统的宿主范围具有积极的促进作用。
(3) 开发新型的CRISPR/Cas系统。近年来发现的新型Class 2效应蛋白如C2c1、C2c3等相对于Cas9显示出诸多优势,而相应的系统评估与应用仍处于摸索阶段,未来随着研究的深入以及生物信息学、结构生物学等学科的发展,这些新型CRISPR/Cas系统将会得到开发,从而进一步拓展CRISPR/Cas基因编辑系统的应用范围。
(4) 建立crRNP直接转化系统。在原核微生物中,CRISPR编辑系统各元件均是以DNA的形式在细胞内进行表达,因此每一次基因编辑都需要构建相应的质粒。重复、复杂的质粒构建程序对于需要多个位点编辑的菌株来说并不适用,会成为影响整个基因编辑操作流程的“限速步骤”。在真核生物中,可以将体外组装的crRNP直接转化进入细胞内进行编辑[69],如果相应的方法可以移植到原核微生物中,这将会进一步促进CRISPR/Cas基因编辑系统在原核微生物中的发展与应用。
未来随着各种技术之间的交叉与相互渗透,通过合理的系统构建与优化,CRISPR/Cas基因编辑系统必将克服目前阻碍其发展的种种壁垒,最终成为一种简单、易于操作的基因工程工具,从而进一步促进代谢工程、合成生物学等学科的发展与应用。
| [1] |
Choi KR, Lee SY. CRISPR technologies for bacterial systems: current achievements and future directions[J]. Biotechnology Advances, 2016, 34(7): 1180-1209. DOI:10.1016/j.biotechadv.2016.08.002 |
| [2] |
Peralta-Yahya PP, Zhang FZ, Del Cardayre SB, et al. Microbial engineering for the production of advanced biofuels[J]. Nature, 2012, 488(7411): 320-328. DOI:10.1038/nature11478 |
| [3] |
Keasling JD. Manufacturing molecules through metabolic engineering[J]. Science, 2010, 330(6009): 1355-1358. DOI:10.1126/science.1193990 |
| [4] |
Bailey JE. Toward a science of metabolic engineering[J]. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876 |
| [5] |
Meng J, Wang BY, Liu DY, et al. High-yield anaerobic succinate production by strategically regulating multiple metabolic pathways based on stoichiometric maximum in Escherichia coli[J]. Microbial Cell Factories, 2016, 15: 141. DOI:10.1186/s12934-016-0536-1 |
| [6] |
Chen JY, Zhang L, Chen JC, et al. Biosynthesis and characterization of polyhydroxyalkanoate copolyesters in Ralstonia eutropha PHB− 4 harboring a low-substrate-specificity PHA synthase PhaC2Ps from Pseudomonas stutzeri 1317[J]. Chinese Journal of Chemical Engineering, 2007, 15(3): 391-396. DOI:10.1016/S1004-9541(07)60097-4 |
| [7] |
Yao YF, Wang CS, Qiao JJ, et al. Metabolic engineering of Escherichia coli for production of salvianic acid A via an artificial biosynthetic pathway[J]. Metabolic Engineering, 2013, 19: 79-87. DOI:10.1016/j.ymben.2013.06.001 |
| [8] |
Wu JJ, Du GC, Zhou JW, et al. Metabolic engineering of Escherichia coli for (2S)-pinocembrin production from glucose by a modular metabolic strategy[J]. Metabolic Engineering, 2013, 16: 48-55. DOI:10.1016/j.ymben.2012.11.009 |
| [9] |
Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature, 2005, 435(7042): 646-651. DOI:10.1038/nature03556 |
| [10] |
Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors[J]. Science, 2009, 326(5959): 1509-1512. DOI:10.1126/science.1178811 |
| [11] |
Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement[J]. Science, 2011, 333(6040): 348-353. DOI:10.1126/science.1205822 |
| [12] |
Warner JR, Reeder PJ, Karimpour-fard A, et al. Rapid profiling of a microbial genome using mixtures of barcoded oligonucleotides[J]. Nature Biotechnology, 2010, 28(8): 856-862. DOI:10.1038/nbt.1653 |
| [13] |
Hong KQ, Liu DY, Chen T, et al. Recent advances in CRISPR/Cas9 mediated genome editing in Bacillus subtilis[J]. World Journal of Microbiology and Biotechnology, 2018, 34(10): 153. DOI:10.1007/s11274-018-2537-1 |
| [14] |
Wang HY, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [15] |
Shan QW, Wang YP, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31(8): 686-688. DOI:10.1038/nbt.2650 |
| [16] |
Jansen R, van Embden JDA, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [17] |
Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482(7385): 331-338. DOI:10.1038/nature10886 |
| [18] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [19] |
Carter J, Wiedenheft B. SnapShot: CRISPR-RNA-guided adaptive immune systems[J]. Cell, 2015, 163(1): 260-260.e1. DOI:10.1016/j.cell.2015.09.011 |
| [20] |
Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity[J]. Nature, 2010, 463(7280): 568-571. |
| [21] |
Makarova KS, Wolf YI, Koonin EV. The basic building blocks and evolution of CRISPR-CAS systems[J]. Biochemical Society Transactions, 2013, 41(6): 1392-1400. DOI:10.1042/BST20130038 |
| [22] |
Koonin EV, Makarova KS. Mobile genetic elements and evolution of CRISPR-Cas systems: all the way there and back[J]. Genome Biology and Evolution, 2017, 9(10): 2812-2825. DOI:10.1093/gbe/evx192 |
| [23] |
Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems[J]. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [24] |
Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [25] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [26] |
Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [27] |
Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J]. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [28] |
Altenbuchner J. Editing of the Bacillus subtilis genome by the CRISPR-Cas9 system[J]. Applied and Environmental Microbiology, 2016, 82(17): 5421-5427. DOI:10.1128/AEM.01453-16 |
| [29] |
Jang YJ, Seo SO, Kim SA, et al. Elimination of the cryptic plasmid in Leuconostoc citreum by CRISPR/Cas9 system[J]. Journal of Biotechnology, 2017, 251: 151-155. DOI:10.1016/j.jbiotec.2017.04.018 |
| [30] |
Li H, Shen CR, Huang CH, et al. CRISPR-Cas9 for the genome engineering of cyanobacteria and succinate production[J]. Metabolic Engineering, 2016, 38: 293-302. DOI:10.1016/j.ymben.2016.09.006 |
| [31] |
Selle K, Klaenhammer TR, Barrangou R. CRISPR-based screening of genomic island excision events in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(26): 8076-8081. DOI:10.1073/pnas.1508525112 |
| [32] |
Huang H, Zheng GS, Jiang WH, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces[J]. Acta Biochimica et Biophysica Sinica, 2015, 47(4): 231-243. DOI:10.1093/abbs/gmv007 |
| [33] |
Oh JH, van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J]. Nucleic Acids Research, 2014, 42(17): e131. DOI:10.1093/nar/gku623 |
| [34] |
Wang Y, Zhang ZT, Seo SO, et al. Markerless chromosomal gene deletion in Clostridium beijerinckii using CRISPR/Cas9 system[J]. Journal of Biotechnology, 2015, 200: 1-5. DOI:10.1016/j.jbiotec.2015.02.005 |
| [35] |
Xu T, Li YC, He ZL, et al. Cas9 nickase-assisted RNA repression enables stable and efficient manipulation of essential metabolic genes in Clostridium cellulolyticum[J]. Frontiers in Microbiology, 2017, 8: 1744. DOI:10.3389/fmicb.2017.01744 |
| [36] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [37] |
Cho JS, Choi KR, Prabowo CPS, et al. CRISPR/Cas9-coupled recombineering for metabolic engineering of Corynebacterium glutamicum[J]. Metabolic Engineering, 2017, 42: 157-167. DOI:10.1016/j.ymben.2017.06.010 |
| [38] |
Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J]. Nature Communications, 2017, 8: 15179. DOI:10.1038/ncomms15179 |
| [39] |
Yan MY, Yan HQ, Ren GX, et al. CRISPR-Cas12a-assisted recombineering in bacteria[J]. Applied and Environmental Microbiology, 2017, 83(17): e00947-17. |
| [40] |
Pyne ME, Bruder MR, Moo-Young M, et al. Harnessing heterologous and endogenous CRISPR-Cas machineries for efficient markerless genome editing in Clostridium[J]. Scientific Reports, 2016, 6: 25666. DOI:10.1038/srep25666 |
| [41] |
Cobb RE, Wang YJ, Zhao HM. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J]. ACS Synthetic Biology, 2015, 4(6): 723-728. DOI:10.1021/sb500351f |
| [42] |
Huang H, Chai CS, Li N, et al. CRISPR/Cas9-based efficient genome editing in Clostridium ljungdahlii, an autotrophic gas-fermenting bacterium[J]. ACS Synthetic Biology, 2016, 5(12): 1355-1361. DOI:10.1021/acssynbio.6b00044 |
| [43] |
Xu T, Li YC, Shi Z, et al. Efficient genome editing in Clostridium cellulolyticum via CRISPR-Cas9 nickase[J]. Applied and Environmental Microbiology, 2015, 81(13): 4423-4431. DOI:10.1128/AEM.00873-15 |
| [44] |
So Y, Park SY, Park EH, et al. A highly efficient CRISPR-Cas9-mediated large genomic deletion in Bacillus subtilis[J]. Frontiers in Microbiology, 2017, 8: 1167. DOI:10.3389/fmicb.2017.01167 |
| [45] |
Standage-Beier K, Zhang Q, Wang X. Targeted large-scale deletion of bacterial genomes using CRISPR-nickases[J]. ACS Synthetic Biology, 2015, 4(11): 1217-1225. DOI:10.1021/acssynbio.5b00132 |
| [46] |
Wasels F, Jean-Marie J, Collas F, et al. A two-plasmid inducible CRISPR/Cas9 genome editing tool for Clostridium acetobutylicum[J]. Journal of Microbiological Methods, 2017, 140: 5-11. DOI:10.1016/j.mimet.2017.06.010 |
| [47] |
Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J]. Metabolic Engineering, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006 |
| [48] |
Zhu XN, Zhao DD, Qiu HN, et al. The CRISPR/Cas9-facilitated multiplex pathway optimization (CFPO) technique and its application to improve the Escherichia coli xylose utilization pathway[J]. Metabolic Engineering, 2017, 43: 37-45. DOI:10.1016/j.ymben.2017.08.003 |
| [49] |
Westbrook AW, Moo-Young M, Chou CP. Development of a CRISPR-Cas9 tool kit for comprehensive engineering of Bacillus subtilis[J]. Applied and Environmental Microbiology, 2016, 82(16): 4876-4895. DOI:10.1128/AEM.01159-16 |
| [50] |
Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013, 31(8): 688-691. DOI:10.1038/nbt.2654 |
| [51] |
Zerbini F, Zanella I, Fraccascia D, et al. Large scale validation of an efficient CRISPR/Cas-based multi gene editing protocol in Escherichia coli[J]. Microbial Cell Factories, 2017, 16: 68. DOI:10.1186/s12934-017-0681-1 |
| [52] |
Zhao DD, Yuan SL, Xiong B, et al. Development of a fast and easy method for Escherichia coli genome editing with CRISPR/Cas9[J]. Microbial Cell Factories, 2016, 15(1): 205. DOI:10.1186/s12934-016-0605-5 |
| [53] |
Xia J, Wang L, Zhu JB, et al. Expression of Shewanella frigidimarina fatty acid metabolic genes in E. coli by CRISPR/cas9-coupled lambda Red recombineering[J]. Biotechnology Letters, 2016, 38(1): 117-122. DOI:10.1007/s10529-015-1956-4 |
| [54] |
Reisch CR, Prather KLJ. The no-SCAR (Scarless Cas9 Assisted Recombineering) system for genome editing in Escherichia coli[J]. Scientific Reports, 2015, 5: 15096. DOI:10.1038/srep15096 |
| [55] |
Pyne ME, Moo-Young M, Chung DA, et al. Coupling the CRISPR/Cas9 system with lambda red recombineering enables simplified chromosomal gene replacement in Escherichia coli[J]. Applied and Environmental Microbiology, 2015, 81(15): 5103-5114. DOI:10.1128/AEM.01248-15 |
| [56] |
Heo MJ, Jung HM, Um J, et al. Controlling citrate synthase expression by CRISPR/Cas9 genome editing for n-butanol production in Escherichia coli[J]. ACS Synthetic Biology, 2017, 6(2): 182-189. DOI:10.1021/acssynbio.6b00134 |
| [57] |
Bassalo MC, Garst AD, Halweg-Edwards AL, et al. Rapid and efficient one-step metabolic pathway integration in E. coli[J]. ACS Synthetic Biology, 2016, 5(7): 561-568. DOI:10.1021/acssynbio.5b00187 |
| [58] |
Liang LY, Liu RM, Garst AD, et al. CRISPR enabled trackable genome engineering for isopropanol production in Escherichia coli[J]. Metabolic Engineering, 2017, 41: 1-10. DOI:10.1016/j.ymben.2017.02.009 |
| [59] |
Wu MY, Sung LY, Li H, et al. Combining CRISPR and CRISPRi systems for metabolic engineering of E. coli and 1, 4-BDO biosynthesis[J]. ACS Synthetic Biology, 2017, 6(12): 2350-2361. DOI:10.1021/acssynbio.7b00251 |
| [60] |
Zhang K, Duan XG, Wu J. Multigene disruption in undomesticated Bacillus subtilis ATCC 6051a using the CRISPR/Cas9 system[J]. Scientific Reports, 2016, 6: 27943. DOI:10.1038/srep27943 |
| [61] |
Wen ZQ, Minton NP, Zhang Y, et al. Enhanced solvent production by metabolic engineering of a twin-clostridial consortium[J]. Metabolic Engineering, 2017, 39: 38-48. DOI:10.1016/j.ymben.2016.10.013 |
| [62] |
Wang SH, Dong S, Wang PX, et al. Genome editing in Clostridium saccharoperbutylacetonicum N1-4 with the CRISPR-Cas9 system[J]. Applied and Environmental Microbiology, 2017, 83(10): e00233-17. |
| [63] |
Ronda C, Pedersen LE, Sommer MOA, et al. CRMAGE: CRISPR optimized MAGE recombineering[J]. Scientific Reports, 2016, 6: 19452. DOI:10.1038/srep19452 |
| [64] |
Garst AD, Bassalo MC, Pines G, et al. Genome-wide mapping of mutations at single-nucleotide resolution for protein, metabolic and genome engineering[J]. Nature Biotechnology, 2017, 35(1): 48-55. DOI:10.1038/nbt.3718 |
| [65] |
Shen JT, Xiu ZL. Strategies to prevent bacteriophage infection in industrial fermentation[J]. Chinese Journal of Biotechnology, 2017, 33(12): 1901-1912. (in Chinese) 沈俊涛, 修志龙. 工业微生物的噬菌体感染与防治策略[J]. 生物工程学报, 2017, 33(12): 1901-1912. |
| [66] |
Jakutyte-Giraitiene L, Gasiunas G. Design of a CRISPR-Cas system to increase resistance of Bacillus subtilis to bacteriophage SPP1[J]. Journal of Industrial Microbiology & Biotechnology, 2016, 43(8): 1183-1188. |
| [67] |
Gomaa AA, Klumpe HE, Luo ML, et al. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems[J]. mBio, 2014, 5(1): e00928-13. |
| [68] |
Su TY, Liu FP, Gu PF, et al. A CRISPR-Cas9 assisted non-homologous end-joining strategy for one-step engineering of bacterial genome[J]. Scientific Reports, 2016, 6: 37895. DOI:10.1038/srep37895 |
| [69] |
Zuris JA, Thompson DB, Shu YL, et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo[J]. Nature Biotechnology, 2015, 33(1): 73-80. DOI:10.1038/nbt.3081 |
 2019, Vol. 46
2019, Vol. 46