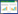扩展功能
文章信息
- 李雨欣, 刘治, 杨金丽, 韩润林
- LI Yu-Xin, LIU Zhi, YANG Jin-Li, HAN Run-Lin
- 铜绿假单胞菌表面(氧化)低密度脂蛋白配体的研究
- Detection of (oxidized) low-density lipoprotein binding ligand on Pseudomonas aeruginosa
- 微生物学通报, 2019, 46(10): 2689-2696
- Microbiology China, 2019, 46(10): 2689-2696
- DOI: 10.13344/j.microbiol.china.180799
-
文章历史
- 收稿日期: 2018-10-16
- 接受日期: 2018-12-13
- 网络首发日期: 2019-01-02
2. 内蒙古农业大学兽医学院 内蒙古 呼和浩特 010018;
3. 内蒙古农业大学血浆脂蛋白免疫学研究中心 内蒙古 呼和浩特 010018
2. College of Veterinary Medicine, Inner Mongolia Agriculture University, Hohhot, Inner Mongolia 010018, China;
3. Research Center of Plasma Lipoprotein Immunology, Inner Mongolia Agriculture University, Hohhot, Inner Mongolia 010018, China
铜绿假单胞菌(Pseudomonas aeruginosa,P. aeruginosa)是一种常见的条件致病菌,广泛分布于医疗器械、动物、植物及人体。约有10%−15%的医源性继发感染是由P. aeruginosa引起的,免疫缺陷、烧伤、患有囊性纤维化及癌症等病人对P. aeruginosa易感[1-2]。P. aeruginosa表达了多种毒力因子,其中最为重要的有脂多糖(Lipopoly-saccharide,LPS)和生物膜结构。生物膜的形成会导致P. aeruginosa由非粘液型菌株转变为粘液型菌株,其耐药性和粘附能力也会随之增强[3]。
RahU蛋白是P. aeruginosa的一种毒性蛋白,由RahU基因编码,分子量大小为14.85 kD,其表达与生物膜的形成、细菌的群体效应相关。RahU蛋白属于杨树菇溶血素(Aegerolysins)蛋白家族,该蛋白家族的蛋白均与LDL/oxLDL特异性结合。有研究证实,RahU蛋白与LDL/oxLDL特异性结合是因为其与LDL/oxLDL的LysoPC相互作用[4]。通过这一发现,我们对P. aeruginosa与LDL/oxLDL的结合进行了研究,发现了二者的特异性相互作用。然而,RahU蛋白在不同菌株的表达差异显著,如果RahU蛋白是P. aeruginosa表面唯一的LDL/oxLDL配体,则P. aeruginosa与LDL/oxLDL的结合可能并不稳定。因此,本研究对RahU蛋白作为P. aeruginosa表面LDL/oxLDL配体的重要性进行了检测。
LDL/oxLDL是与动脉粥样硬化形成密切相关的血浆脂蛋白,其主要功能是参与包括胆固醇在内脂类的运输。除此之外,LDL/oxLDL还可能是一种重要的免疫分子[5],LDL/oxLDL含量的升高可以上调一系列清道夫受体的表达,进而增强吞噬细胞对病原微生物的清除能力,LDL受体(LDL-R)缺失的小鼠抗病原微生物感染的能力显著提高[6]。近年来,有研究发现通过与病原微生物的相互作用,LDL/oxLDL还能发挥调理素功能,促进吞噬细胞对病原微生物的吞噬[7],LDL/oxLDL与病原微生物的相互作用可能是其抗感染免疫作用发挥的前提。
本研究对能与LDL/oxLDL特异性结合的RahU蛋白是否是P. aeruginosa膜表面唯一的LDL/oxLDL配体进行了研究,对LDL/oxLDL与P. aeruginosa相互作用的机制进行了探讨,这可能是对LDL/oxLDL具有抗P. aeruginosa感染能力机制的论证。
1 材料与方法 1.1 供试菌株及培养基P. aeruginosa CMCC10104菌株,中国菌种保藏中心;pEX18Tc、pUCGm载体,武汉汉申生物科技有限公司;E. coli JM109、E. coli SM10菌株,生工生物工程(上海)股份有限公司。LB培养基(g/L):Tryptone 10.0,Yeast extract 5.0,NaCl 5.0,Agar (固体) 10.0。
1.2 主要试剂和仪器细菌基因组DNA提取试剂盒、质粒小提试剂盒购自北京天根生物科技有限公司;LDL购自Alfa asar公司;羊抗人LDL多抗、蛋白酶K及胰蛋白酶购自Sigma公司;驴抗羊IgG-HRP购自R & D Systems;山羊抗鼠IgG-HRP购自北京博奥森生物科技有限公司;Pierce® BCA蛋白测定试剂盒购自Thermo公司;琼脂糖凝胶DNA纯化回收试剂盒购自Promega公司;试验所用限制性内切酶均购自宝生物工程(大连)有限公司;基因测序均由生工生物工程(上海)股份有限公司完成。
基因扩增仪,博日科技有限公司;垂直流超净台、多功能酶标仪,Bio-Tek公司;台式冷冻离心机,Eppendorf公司;轨道型恒温摇床,上海智诚分析仪器制造有限公司;蛋白电泳槽、核酸电泳槽、电脑三恒多用电泳仪,北京六一生物科技有限公司;半干转膜仪,Hoefer公司;湿转仪,Bio-Rad公司;化学发光/荧光凝胶成像系统,Syngene公司。
1.3 RahU基因缺失突变株(ΔRahU菌株)的构建参照Rao等所述,利用等位替换原理构建ΔRahU菌株[8]。LB固体平板过夜活化−80 ℃冻存的P. aeruginosa CMCC10104。次日,挑取单菌落于LB液体培养基,在恒温摇床中37 ℃、200 r/min培养。培养至OD600约为0.8左右,将菌体收集后提取P. aeruginosa基因组DNA。以提取得到的DNA为模板,利用表 1中的引物分别扩增RahUU及RahUD基因片段。PCR反应体系(20 μL):模板0.5 μL,2×PCR master mix 10 μL,20 μmol/L引物各1 μL,ddH2O 7.5 μL。PCR反应条件:94 ℃ 1 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 50 s,30个循环;72 ℃ 5 min。得到的PCR产物用1%的琼脂糖凝胶电泳鉴定。
| Primers name | Primers sequence (5′→3′) | PCR product | Size (bp) |
| RahUU-F | CGGAATTCGCTGGTGGAAAGCGAAGTG | Upstream of RahU gene | 630 |
| RahUU-R | CGGGGTACCGTCGATCTCCGTTAACCATGC | Upstream of RahU gene | 630 |
| RahUD-F | TTCTCCTGACGGTACCTGGA | Downstream of RahU gene | 375 |
| RahUD-R | CCCAAGCTTTGAACACCTCGAACCACCTCA | Downstream of RahU gene | 375 |
| pEX18Tc | GAAGATCTGTTGAATACTCATACTCTTC | Plasmid pEX18Tc | 164 |
| pEX18Tc | GAAGATCTTGTCAGACCAAGTTTACTCAT | Plasmid pEX18Tc | 164 |
于37 ℃用EcoR Ⅰ及Kpn I限制性内切酶对RahUU片段及pEX18Tc质粒分别酶切30 min,酶切后的片段及质粒经凝胶回收、产物纯化后使用T4连接酶连接,连接后的重组质粒命名为pEX18RahUU。pEX18RahUU质粒转化E. coli JM109,筛选阳性克隆并提取质粒。提取得到的pEX18RahUU质粒及RahUD片段分别用Kpn Ⅰ、Hind Ⅲ限制性内切酶在37 ℃酶切30 min,酶切产物经凝胶回收、产物纯化后使用T4连接酶连接,连接后的质粒命名为pEX18RahUUD。pEX18RahUUD质粒转化E. coli JM109,并将阳性克隆培养后提取质粒。将提取得到的pEX18RahUUD质粒及购买的pUCGm质粒用Kpn I限制性内切酶在37 ℃酶切30 min,酶切产物经凝胶回收后,用去磷酸化酶去磷酸化,T4连接酶连接后命名重组质粒为pEXΔRahU::Gm。该质粒中插入了RahU基因上、下游同源臂及830 bp的Gm抗性基因,为等位替换RahU基因的打靶质粒。重组质粒经转化E. coli JM109后筛选阳性克隆,提取pEXΔRahU::Gm质粒转化E. coli SM10菌株。
利用接合转移的方法将E. coli SM10菌株中的穿梭质粒pEXΔRahU::Gm转化P. aeruginosa,利用含Tc (50 µg/mL)、Gm (150 µg/mL)和Sp (25 µg/mL)的LB抗性平板筛选质粒插入后的P. aeruginosa。P. aeruginosa基因组DNA与打靶质粒发生交换的菌株用含5%蔗糖及Gm (150 µg/mL)的LB平板筛选,得到的菌落再分别划线培养于含Gm (150 µg/mL)的LB平板与含Tc (50 µg/mL)的LB平板,能在含Gm (150 µg/mL)的LB平板生长而不能在含Tc (50 µg/mL)的LB平板上生长的菌落为发生双交换的目的菌株ΔRahU菌株。
利用表 2中的引物,以ΔRahU菌株的基因组DNA为模板,通过PCR扩增Gm抗性基因及RahU基因,鉴定ΔRahU菌株中Gm抗性基因的插入和RahU基因的消失。RT-PCR方法分别扩增P. aeruginosa野生型菌株及ΔRahU菌株cDNA中RahU基因,检测基因敲除后RahU基因是否表达。
| Primers name | Primers sequence (5′→3′) | PCR product | Size (bp) |
| RahU-F | GTTAACGGAATTCGACATGGCATACGCAG | RahU gene | 411 |
| RahU-R | GCTCCAGGTACTCGAGGGAGAAGCGGCCG | RahU gene | 411 |
| Gm-F | ATGTTACGCAGCAGCAACGAT | Gm resistance gene | 534 |
| Gm-R | TTAGGTGGCGGTACTTGGGT | Gm resistance gene | 534 |
RahU蛋白的重组表达方法同Rao等[8]的报道,以P. aeruginosa基因组DNA为模板,使用表 2中扩增RahU片段的引物扩增RahU基因,该基因片段与pET28b表达载体连接后转化E. coli Transseta(DE3)菌株。向培养至对数期的E. coli Transseta(DE3)菌液中加1 mmol/L IPTG诱导表达3 h,裂解细菌细胞后使用TALON® Metal亲和柱层析分离目的蛋白,目的蛋白冻存于−20 ℃。15% SDS-PAGE电泳及Western Blot对rRahU蛋白的纯度及特异性进行分析,Pierce® BCA蛋白测定试剂盒测定rRahU蛋白含量(结果未显示)。
购买5−6周龄小鼠采血清,ELISA检测血清中无抗P. aeruginosa抗体。小鼠常规饲养,按照每只小鼠皮下注射75 µg免疫206佐剂乳化后的rRahU蛋白进行初免,同时对照组小鼠每只注射相同体积乳化后PBS (因rRahU由PBS稀释)。14 d后小鼠皮下注射50 µg免疫乳化后rRahU蛋白进行第二次加强免疫,二免14 d后小鼠皮下注射50 µg免疫乳化后rRahU蛋白进行三免。免疫后小鼠眼部采血并分离血清,经Protein G resin纯化得到抗rRahU抗体和抗PBS抗体,纯化得到的抗体经Western-Blot和ELISA检测其特异性(结果未显示)。
1.5 P. aeruginosa野生型菌株及ΔRahU菌株膜蛋白的分离培养P. aeruginosa野生型菌株及ΔRahU菌株各100 mL至对数期,即OD600约为0.9。4 ℃、10 000 r/min离心5 min收集菌体,PBS洗菌3次后,500 µL PBS重悬,−80 ℃冷冻过夜。取出上述冻存菌液室温融化后,分别加2 µL PMSF,以及溶菌酶和DNA酶各1 µL,室温脱色摇床作用20 min后,4 ℃、3 220×g离心15 min。收集上述离心后上清,弃去沉淀。4 ℃、100 000×g超速离心6 h,弃上清,沉淀为菌体膜蛋白。膜蛋白分别用500 µL PBS重悬。
1.6 ELISA检测rRahU蛋白与LDL/oxLDL的相互作用将浓度为20 µg/mL的rRahU或rTuf按照每孔100 µL包被酶标板,同时加阴性对照PBS包被酶标板,室温下孵育1.5 h。之后用TBST每孔200 µL洗板3次,每孔加200 µL 1% BSA-TBST封闭1.5 h。洗板3次后分别按每孔加终浓度为1.0、0.5、0.1 µg/mL的LDL/oxLDL 100 µL (TBST稀释),室温下孵育1.5 h。TBST洗板3次。加入1:10 000稀释的羊抗人LDL,室温下孵育1.5 h后洗板3次。加入1:1 000稀释的驴抗羊-HRP,TBST洗板3次后加显色液TMB底物100 µL,室温孵育5 min。每孔加终止液100 µL,测定450 nm处吸光值。
1.7 ELISA及Western Blot检测P. aeruginosa菌株表面RahU蛋白的表达P. aeruginosa野生型菌株及ΔRahU菌株膜蛋白分别经15% SDS-PAGE电泳分离后,恒压100 V、30 min转膜至0.22 µm NC膜。一抗用小鼠抗rRahU蛋白抗体检测其中的RahU蛋白,小鼠抗PBS抗体作为阴性对照。二抗为山羊抗鼠IgG-HRP,之后加显色液于G:BOX XT4化学发光/荧光凝胶成像系统拍照。
ELISA方法同1.5,用每孔100 µL P. aeruginosa野生型菌株菌液或ΔRahU菌株菌液包被酶标板,随后用抗体检测不同菌株表面RahU蛋白的表达情况,一抗用小鼠抗rRahU蛋白抗体,二抗为山羊抗鼠IgG-HRP。
1.8 ELISA检测RahU蛋白是否是P. aeruginosa CMCC10104菌株表面唯一LDL/oxLDL配体用100 µL每孔P. aeruginosa CMCC10104野生型菌株及该菌株RahU基因缺失突变株ΔRahU菌株菌液包被酶标板,其余方法同1.5。
按照每109 cfu加入蛋白酶K (ProK) 50 µg、胰蛋白酶(Trypsine) 50 µg、ProK和Trypsine各50 µg对ΔRahU菌株进行处理,对照组加入等体积的PBS。37 ℃作用30 min。之后按照每109 cfu加2 mmol/L的PMSF终止反应。得到的菌体经PBS悬浮后每孔100 µL包被酶标板,其余方法同1.5。
2 结果与分析 2.1 ΔRahU菌株的鉴定图 1A为不同引物扩增ΔRahU菌株基因组DNA结果,可以看出Gm抗性基因插入了ΔRahU菌株基因组,而RahU基因在该菌株中消失。
|
| 图 1 PCR及RT-PCR对ΔRahU菌株的鉴定 Figure 1 Identification of ΔRahU strain with PCR and RT-PCR 注:M:DL5000 marker;Gm:用Gm抗性基因引物扩增ΔRahU菌株基因组DNA中插入Gm抗性基因结果;RahU:用RahU基因引物扩增ΔRahU菌株基因组DNA中RahU基因结果;P. aeruginosa RahU+ strain:用RahU基因引物扩增P. aeruginosa野生型菌株mRNA反转录后cDNA中RahU基因结果;ΔRahU strain:用RahU基因引物扩增ΔRahU菌株mRNA反转录后cDNA中RahU基因结果. Note: M: DL5000 marker; Gm: Genome DNA of ΔRahU amplified by primers of Gm; RahU: Genome DNA of ΔRahU amplified by primers of RahU; P. aeruginosa RahU+ strain: cDNA of P. aeruginosa wild strain reverse transcribed mRNA amplified by primers of RahU; ΔRahU strain: cDNA of ΔRahU strain reverse transcribed mRNA amplified by primers of RahU. |
|
|
图 1B为扩增P. aeruginosa野生型菌株及ΔRahU菌株cDNA中RahU基因的结果,可以看出,RahU基因在野生型菌株中表达而在突变株中沉默,证实ΔRahU菌株中RahU基因被敲除。
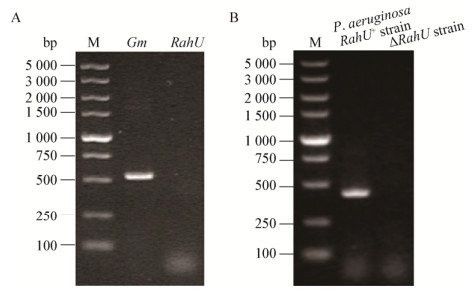
2.2 rRahU蛋白与LDL/oxLDL的相互作用ELISA结果(图 2)显示,与表达rRahU相同方法表达的rTuf与LDL/oxLDL不结合,而rRahU与LDL/oxLDL结合且二者的结合能力与LDL/oxLDL浓度呈正相关,为特异性结合,证实本研究表达的rRahU具有生物学活性。
|
| 图 2 rRahU与LDL/oxLDL的结合 Figure 2 Binding of rRahU to LDL/oxLDL |
|
|
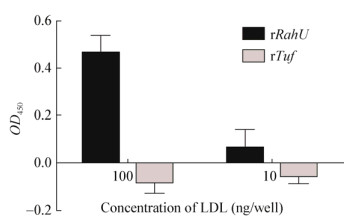
以rRahU蛋白为阳性对照,ΔRahU菌株膜蛋白为阴性对照,Western Blot结果(图 3A)显示小鼠抗rRahU蛋白抗体与P. aeruginosa野生型菌株膜蛋白中的RahU蛋白特异性结合,图 3A为以小鼠抗PBS抗体孵育的阴性对照组,图 3B为以小鼠抗rRahU蛋白抗体孵育的实验组。ELISA结果(图 3C)显示,小鼠抗rRahU蛋白抗体与P. aeruginosa野生型菌株相互作用,而不与ΔRahU菌株相互作用。Western Blot及ELISA试验均以小鼠抗PBS抗体作为阴性对照。证实RahU蛋白在P. aeruginosa CMCC10104菌体表面表达,为其表面的LDL/oxLDL配体之一。
|
| 图 3 ELISA及Western Blot检测P. aeruginosa野生型菌株及ΔRahU菌株表面RahU蛋白的表达 Figure 3 Detection of RahU on P. aeruginosa wild strain and ΔRahU strain membrane with ELISA and Western Blot 注:利用student t检验对数据进行统计学意义分析,*:0.01 < P < 0.05; **:P < 0.01; ***:P < 0.001. Note: A two tailed paired Student's t-test was used to determine statistical significance. *: 0.01 < P < 0.05; **: P < 0.01; ***: P < 0.001. |
|
|
ELISA结果(图 4)表明,RahU基因缺失与否对P. aeruginosa和LDL/oxLDL的结合能力没有显著影响,RahU蛋白不是P. aeruginosa表面唯一的LDL/oxLDL结合位点。
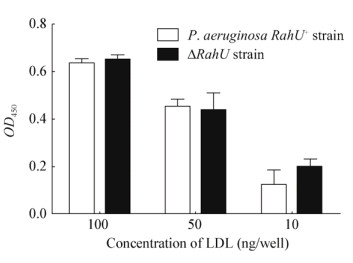
|
| 图 4 P. aeruginosa野生型菌株和ΔRahU菌株与LDL/oxLDL的结合 Figure 4 Binding of P. aeruginosa wild strain and ΔRahU strain to LDL/oxLDL |
|
|
ELISA结果(图 5)表明,不同蛋白酶水解ΔRahU菌株表面蛋白后,ΔRahU菌株与LDL/oxLDL的结合能力无显著差异。该结果说明P. aeruginosa表面与LDL/oxLDL结合的配体可能还有非蛋白组分。
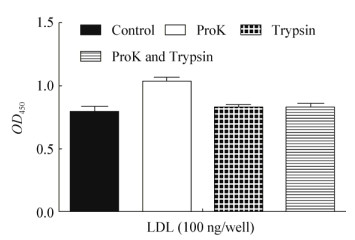
|
|
图 5 不同蛋白酶作用后ΔRahU菌株和LDL/oxLDL的结合
Figure 5 Binding of different protease digested ΔRahU strain to LDL/oxLDL
|
|
|
大量研究证实LDL/oxLDL是一种重要的免疫分子[5],有研究证实LDL/oxLDL可以通过与A群链球菌的Ⅰ型胶原样蛋白相互作用而发挥调理素功能,促进吞噬细胞对该病原微生物的吞噬作用[7]。对LDL/oxLDL与其他病原微生物相互作用的研究可能是对LDL/oxLDL抗感染免疫机制新的补充。同时,有研究证实,很多能与LDL/oxLDL特异性结合的病原微生物感染都与动脉粥样硬化的形成有关[9-11],其中也包括P. aeruginosa[12]。对LDL/oxLDL与病原微生物相互作用的研究可能也是对动脉粥样硬化等疾病形成机理的全新阐述。
本研究中RahU蛋白属于P. aeruginosa的毒力蛋白之一,LDL/oxLDL与该蛋白的相互作用可能会改变P. aeruginosa感染宿主的能力[8]。RahU蛋白在不同的P. aeruginosa菌株中表达差异显著,其非粘液型菌株中该蛋白的表达量是粘液型菌株的4−8倍,同时该蛋白在非粘液型菌株的细胞质及细胞膜上均有表达,而在粘液型菌株中仅在细胞质中表达[4]。本研究证实,RahU蛋白并不是P. aeruginosa细胞膜表面唯一的LDL/oxLDL结合位点。这就说明在不能表达RahU蛋白的P. aeruginosa菌株中,也有其他的LDL/oxLDL配体参与P. aeruginosa与LDL/oxLDL的相互作用。P. aeruginosa与LDL/oxLDL的特异性结合可能与菌株的不同无关,通过对酶处理后ΔRahU菌株和LDL/oxLDL的结合无差异可以看出,P. aeruginosa表面的LDL/oxLDL配体可能不仅有蛋白组分,同时LDL/oxLDL与该病原微生物的相互作用是非常稳定的。
P. aeruginosa含有6.3×107个碱基对,共有5 570个开放式阅读框。在这些开放式阅读框中,编码外膜蛋白的基因就有150个之多[13],因此虽然RahU蛋白是P. aeruginosa表面的LDL/oxLDL受体之一,P. aeruginosa表面也可能还有与LDL/oxLDL相互作用的膜蛋白。有研究称,某些革兰氏阴性菌表面的LPS成分可以与LDL/oxLDL特异性结合[14],而LPS同样是P. aeruginosa的菌表面组分之一,因此P. aeruginosa表面的LPS结构可能也是LDL/oxLDL配体之一。除此而外,P. aeruginosa表面还有生物膜结构,生物膜的成分包括蛋白、脂类及核酸等[15]。研究表明,脂类、蛋白以及核酸都有可能与LDL/oxLDL相互作用[5, 16],因此P. aeruginosa表面的LDL/oxLDL配体可能也与生物膜有关。以上结论还有待于进一步的研究证实。
4 结论本研究证实P. aeruginosa表达的RahU蛋白是P. aeruginosa表面的LDL/oxLDL配体之一,然而P. aeruginosa表面还有其他成分的LDL/oxLDL配体。P. aeruginosa与LDL/oxLDL的结合由多种机制导致且非常稳定。
| [1] |
Singh PK, Schaefer AL, Parsek MR, et al. Quorum-sensing signals indicate that cystic fibrosis lungs are infected with bacterial biofilms[J]. Nature, 2000, 407(6805): 762-764. DOI:10.1038/35037627 |
| [2] |
Rossolini GM, Mantengoli E. Treatment and control of severe infections caused by multiresistant Pseudomonas aeruginosa[J]. Clinical Microbiology and Infection, 2005, 11(S4): 17-32. |
| [3] |
Cayci YT, Coban AY, Gunaydin M. Investigation of plasmid-mediated quinolone resistance in Pseudomonas aeruginosa clinical isolates[J]. Indian Journal of Medical Microbiology, 2014, 32(3): 285-289. DOI:10.4103/0255-0857.136567 |
| [4] |
Rao J, DiGiandomenico A, Unger J, et al. A novel oxidized low-density lipoprotein-binding protein from Pseudomonas aeruginosa[J]. Microbiology, 2008, 154(2): 654-665. DOI:10.1099/mic.0.2007/011429-0 |
| [5] |
Han RL. Plasma lipoproteins are important components of the immune system[J]. Microbiology and Immunology, 2010, 54(4): 246-253. DOI:10.1111/j.1348-0421.2010.00203.x |
| [6] |
Khovidhunkit W, Kim MS, Memon RA, et al. Effects of infection and inflammation on lipid and lipoprotein metabolism: mechanisms and consequences to the host[J]. Journal of Lipid Research, 2004, 45(7): 1169-1196. DOI:10.1194/jlr.R300019-JLR200 |
| [7] |
Zhou LL, Liu L, Yang JL, et al. LDL acts as an opsonin enhancing the phagocytosis of group A Streptococcus by monocyte and whole human blood[J]. Medical Microbiology and Immunology, 2016, 205(2): 155-162. DOI:10.1007/s00430-015-0436-8 |
| [8] |
Rao J, DiGiandomenico A, Artamonov M, et al. Host derived inflammatory phospholipids regulate rahU (PA0122) gene, protein, and biofilm formation in Pseudomonas aeruginosa[J]. Cellular Immunology, 2011, 270(2): 95-102. DOI:10.1016/j.cellimm.2011.04.011 |
| [9] |
Guevara Jr JG, Prashad N, Ermolinsky B, et al. Apo B100 similarities to viral proteins suggest basis for LDL-DNA binding and transfection capacity[J]. Journal of Lipid Research, 2010, 51(7): 1704-1718. DOI:10.1194/jlr.M003277 |
| [10] |
Boddi M, Abbate R, Chellini B, et al. HCV infection facilitates asymptomatic carotid atherosclerosis: preliminary report of HCV RNA localization in human carotid plaques[J]. Digestive and Liver Disease, 2007, 39(S1): S55-S60. |
| [11] |
Magen E, Borkow G, Bentwich Z, et al. Can worms defend our hearts? Chronic helminthic infections may attenuate the development of cardiovascular diseases[J]. Medical Hypotheses, 2005, 64(5): 904-909. DOI:10.1016/j.mehy.2004.09.028 |
| [12] |
Turkay C, Saba R, Pahin N, et al. Effect of chronic Pseudomonas aeruginosa infection on the development of atherosclerosis in a rat model[J]. Clinical Microbiology and Infection, 2004, 10(8): 705-708. DOI:10.1111/j.1469-0691.2004.00920.x |
| [13] |
Stover CK, Pham XQ, Erwin AL, et al. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen[J]. Nature, 2000, 406(6799): 959-964. DOI:10.1038/35023079 |
| [14] |
Kitchens RL, Thompson PA. Impact of sepsis-induced changes in plasma on LPS interactions with monocytes and plasma lipoproteins: roles of soluble CD14, LBP, and acute phase lipoproteins[J]. Journal of Endotoxin Research, 2003, 9(2): 113-118. DOI:10.1177/09680519030090020701 |
| [15] |
Lawrence JR, Swerhone GDW, Leppard GG, et al. Scanning transmission X-ray, laser scanning, and transmission electron microscopy mapping of the exopolymeric matrix of microbial biofilms[J]. Applied and Environmental Microbiology, 2003, 69(9): 5543-5554. DOI:10.1128/AEM.69.9.5543-5554.2003 |
| [16] |
Surewaard BGJ, Reindert N, Spaan AN, et al. Inactivation of staphylococcal phenol soluble modulins by serum lipoprotein particles[J]. PLoS Pathogens, 2012, 8(3): e1002606. DOI:10.1371/journal.ppat.1002606 |
 2019, Vol. 46
2019, Vol. 46