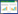扩展功能
文章信息
- 刘洋, 王晓政, 黄婷婷, 周子画, 林双君
- LIU Yang, WANG Xiao-Zheng, HUANG Ting-Ting, ZHOU Zi-Hua, LIN Shuang-Jun
- 链霉菌Streptomyces antibioticus NRRL 8167中Naphthgeranine A生物合成基因簇的分析鉴定
- Analysis and identification of the naphthgeranine A biosynthetic gene cluster from Streptomyces antibioticus NRRL 8167
- 微生物学通报, 2019, 46(10): 2645-2656
- Microbiology China, 2019, 46(10): 2645-2656
- DOI: 10.13344/j.microbiol.china.190274
-
文章历史
- 收稿日期: 2019-04-01
- 接受日期: 2019-05-05
长期以来,天然产物因其结构和生物活性的多样性,始终是药物开发的重要来源。统计数据显示,从1981年到2014年批准上市的药物中约有51%来源于天然产物、天然产物的类似物及其衍生物[1-2]。萘醌类化合物是一类重要的微生物来源天然产物,有着多种多样的生物活性[3]。比如,分离自青霉菌属(Penicillium)的霉酚酸(mycophenolic acid)是肌苷5′-单磷酸盐脱氢酶(inosine 5′-monophosphate dehydrogenase,IMPDH)的抑制剂[4];分离自Aspergillus fumigatus的烟曲霉素(Fumagillin)是治疗微孢子虫病和阿米巴病的重要药物[5]。近年来,得益于分离技术的提高,越来越多来源于微生物的萘醌类化合物被发现,例如Naphterpins[6-8]、Marinones[9-10]、Napyradiomycins[11-12]、Merochlorin[13-14]等。
Naphterpins/Marinones类化合物有着复杂的多环结构[15],通常有4个环,涉及多种取代基,例如羟基、甲基等,有的甚至包含溴原子。其结构中有一个多羟基萘醌环的结构,一个由烷基取代的环醚结构单元和一个被多种基团(例如羟基、烷基和溴原子)取代的环己烯结构。由此可见,这类化合物在结构上有着一定的复杂性。然而截至目前,这类化合物的生物合成途径尚未得到清晰的阐述。其中,naphthgeranine A是最简单的Naphterpin/Marinone类化合物[15],有一个多羟基萘醌环的结构,一个由2个甲基取代的环醚结构和一个甲基取代的环己烯结构。研究表明,Naphthgeranine A具有一定的抗HIV活性[7]。研究Naphthgeranine A可以提高人们对Naphterpins/ Marinones类化合物的认识,为进一步利用这类化合物提供一定的帮助。因此,确定Naphthgeranine A的生物合成基因簇具有重要意义。
本论文研究菌株Streptomyces antibioticus NRRL 8167分离自美国弗吉尼亚州马丁斯维尔(Martinsville,VA)的土壤[16]。通过固体发酵,首次从这个菌株中分离纯化出了Naphthgeranine A (图 1)。结合基因组信息,我们对Naphthgeranine A生物合成基因簇进行了定位,并对这个化合物的生物合成途径进行了推测。这些工作有利于后续对该化合物的生物合成途径进行更加深入细致的解析,拓展微生物天然产物生物合成机制的研究,并为利用组合生物合成及合成生物学创造新的天然产物衍生物奠定基础。
|
| 图 1 Naphthgeranine A的化学结构 Figure 1 The chemical structure of naphthgeranine A |
|
|
研究所用的菌株Streptomyces antibioticus NRRL 8167购自于美国农业研究菌种保藏中心(Agricultural research service culture collection)。
1.1.2 培养基SFM培养基:黄豆饼粉20 g,蒸馏水1 L,1×105 Pa灭菌15 min,用6层纱布过滤收集黄豆饼粉滤汁,加入甘露醇20 g,琼脂粉15 g,pH调至7.5,1×105 Pa灭菌20 min。
1.1.3 主要试剂和仪器甲醇、乙酸乙酯、氯仿等有机试剂,上海国药集团;色谱纯乙腈(Acetonitrile,ACN),Sigma-Aldrich公司;氘代DMSO,Cambridge Isotope Laboratory公司;正相柱层析硅胶,青岛海洋化工有限公司;C18反相柱层析硅胶,YMC公司。
摇床,New Brunswick Scientific公司;旋转蒸发仪,Buchi公司;真空泵,Vacuubrand公司;冷冻干燥机,Labconco公司;高效液相色谱仪(Agilent 1260 infinity HPLC)、高效液相色谱-四级杆飞行时间质谱联用仪,Agilent公司;600 MHz核磁共振波谱仪,Bruker公司。
1.2 方法 1.2.1 菌株的培养及发酵将菌种划线接种于SFM平板培养基,30 °C恒温培养5 d至单菌落表面产生灰色孢子,用无菌棉棒蘸取适量孢子接种于SFM培养基平板上作种子,置于30 C恒温培养箱培养7 d,用无菌棉棒蘸取适量种子转接到SFM培养基平板(9 cm培养皿中含有30 mL固体培养基)上,30 C恒温培养箱培养7 d。共发酵50 L。
1.2.2 次级代谢产物的提取将链霉菌固体发酵物切碎后,用1.5倍体积的乙酸乙酯进行浸泡,超声1 h或搅拌1 h,如此反复浸提3遍将化合物完全提取出来。过滤所得浸提液于40℃进行减压蒸馏,浓缩得黑色粗提物16.2 g。
1.2.3 次级代谢产物的检测方法取少量样品使用HPLC分析,DAD检测器全波长扫描,并选取210、250和290 nm作为检测波长。HPLC以H2O-ACN作为流动相。分析柱为ZORBX SB-C18柱(4.6 mm×250 mm,5 mm,Agilent),流速为0.6 mL/min。洗脱条件为梯度洗脱,乙腈的起始浓度0−2 min为5%;2−15 min乙腈浓度由5%逐渐增加到80%;15−25 min乙腈浓度由80%逐渐增加到100%;26−30 min乙腈浓度持续为100%;30−31 min乙腈浓度由100%逐渐降低到5%;31−35 min乙腈浓度持续为5%,研究中的HPLC检测方法均保持一致。
1.2.4 目标产物的分离纯化及结构鉴定使用正相硅胶柱进行初步分离。取200−300目的硅胶约200 g悬浮于石油醚中,然后装于直径约8 cm的柱中,过夜沉降后待用。粗提物16.2 g用甲醇溶解,然后用100−200目硅胶约30 g进行拌样,采用干法上样。用洗脱体系[石油醚→石油醚:乙酸乙酯(20:1→15:1→10:1→8:1→5:1→2:1→1:1)→乙酸乙酯]逐级洗脱。洗脱体积为2个柱体积。收集洗脱液,使用HPLC进行样品分析,最终合并为FR1–FR8共8个组分。
然后对组分FR4进行反相硅胶柱层析分离,柱子长约20 cm,直径3 cm,采用湿法上样,使用甲醇水混合溶剂作为洗脱体系,甲醇浓度依次为40%→60%→80%→90%→100%。收集洗脱液,使用HPLC进行样品分析检测,确保收集的组分纯度,得到该化合物11 mg。
将纯品组分减压蒸馏蒸干,取少许样品溶解于甲醇中,使用Q-TOF检测,获得高分辨质谱(HPLC条件与产物检测方法一致,电离源为ESI源)。剩余样品溶于0.4 mL氘代DMSO中,以四甲基硅烷(TMS)作为内标进行核磁共振光谱(NMR)分析。检测结束后,样品回收,减压蒸馏去除溶剂,将干燥的样品于−80 °C保存。
1.2.5 基因组DNA的提取及生物合成基因簇分析(1) S. antibioticus NRRL 8167基因组的提取与测序。参照链霉菌操作手册[17]提取菌株S. antibioticus NRRL 8167基因组DNA。纯度检测合格后,由武汉菲沙基因信息有限公司通过PacBio测序平台对该物种的基因组进行测序,测序完成后利用FALCON软件对基因组进行拼接,得到高质量的参考基因组。(2) Naphthgeranine A生物合成基因簇的生物信息学分析。将测序结果上传至AntiSMASH网站(https://antismash.secondarymetabolites.org/#!/start)并进行分析。用NCBI网站(https://www.ncbi.nlm.nih.gov/)上的BLAST软件对菌株基因组核苷酸序列和蛋白质序列进行比对分析。用Frame Plot在线软件(http://biosyn.nih.go.jp/2ndfind/)对DNA序列开放阅读框分析。用BioEdit 7.1.3.0软件中的ClustalW进行多序列比对分析。
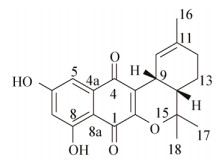
2 结果与分析 2.1 目标化合物的筛选和分离纯化使用HPLC-DAD对发酵产物进行分析(图 2A),发现有一个峰呈现出特殊的紫外吸收光谱(图 2B)。紫外吸收光谱表明化合物有一定的共轭结构,推测其可能属于萘醌类化合物,结构上具有一定的新颖性,于是将这个化合物作为目标化合物进行后续分离。
|
| 图 2 目标化合物的色谱数据 Figure 2 The chromatographic data of candidate compound 注:A:目标化合物(*)在HPLC上的保留时间(检测波长为290 nm);B:目标化合物的紫外吸收图谱. Note: A: The HPLC profile of the metabolites (*) of S. antibioticus NRRL 8167 (detection at 290 nm); B: The UV-vis spectrum of the candidate compound. |
|
|
利用正相硅胶柱层析和反相硅胶柱层析技术对粗提物进行分离纯化,最终分离得到11 mg化合物。
2.2 结构鉴定目标化合物是黄色粉末状固体,可溶于DMSO,微溶于氯仿、甲醇。在正离子模式下,高分辨质谱分析(图 3)得出其准分子离子峰为341.131 6 [M+H]+,推测分子式为C20H20O5,理论分子量为340.131 1。

|
| 图 3 化合物的高分辨质谱图 Figure 3 The HR-MS spectrum of the compound |
|
|
将目标化合物溶解在0.4 mL的氘代DMSO中进行核磁共振(Nuclear magnetic resonance)分析,以四甲基硅烷(TMS)作为内标。1H NMR (图 4A)表明该化合物有3个芳香氢信号δH 6.94 (d,J=2.37 Hz,H-5)、δH 6.49 (d,J=2.28 Hz,H-7)和δH 6.00 (d,J=4.81 Hz,H-10),结合紫外吸收光谱(图 2B)表明该化合物为多取代的萘醌类化合物。1H NMR (图 4A)还显示该化合物有3个甲基信号δH 1.64 (s,H-16)、δH 1.47 (s,H-17)和δH 1.28 (s,H-18)。13C NMR (图 4B)和HSQC显示该化合物有两个羰基信号δC 182.4 (C-1)和δC 183.4 (C-4);7个不饱和季碳信号δC 153.1 (C-2)、δC 123.2 (C-3)、δC 134.8 (C-4a)、δC 165.7 (C-6)、δC 163.9 (C-8)、δC 108.5 (C-8a)和δC 135.7 (C-11);3个不饱和次甲基碳信号δC 107.7 (C-5)、δC 106.7 (C-7)和δC 135.7 (C-11);一个连氧季碳信号δC 80.7 (C-15);两个亚甲基信号δC 20.3 (C-12)和δC 29.6 (C-13);两个次甲基信号δC 31.5 (C-9)和δC 1.81 (C-14);3甲基信号δC 23.8 (C-16)、δC 25.7 (C-17)和δC 25.0 (C-18)。结合1H-1H COSY、HMBC等2D NMR光谱,确定该化合物为Naphthgeranine A,化学结构如图 1所示,核磁数据(表 1)与文献[18]一致。

|
| 图 4 Naphthgeranine A的一维核磁共振谱图 Figure 4 The 1D NMR spectra of naphthgeranine A 注:A:氢谱(600 MHz);B:碳谱(150 MHz). Note: A: 1H NMR (600 MHz); B: 13C NMR (150 MHz). |
|
|
| No. | δH (mult, J/Hz) | δC | 1H-1H COSY | gHMBC |
| 1 | 182.4 | |||
| 2 | 153.1 | |||
| 3 | 123.2 | |||
| 4 | 183.4 | |||
| 4a | 134.8 | |||
| 5 | 6.94 (d, 2.37) | 107.7 | 8a, 4, 7 | |
| 6 | 165.7 | |||
| 7 | 6.49 (d, 2.28) | 106.7 | 5, 8a | |
| 8 | 163.9 | |||
| 8a | 108.5 | |||
| 9 | 3.41 (m) | 31.5 | 10 | 2, 11 |
| 10 | 6.00 (d, 4.86) | 120.7 | 9 | 8, 12 |
| 11 | 135.7 | |||
| 12 | 1.18 (m) | 20.3 | 13 | 14, 16 |
| 13 | 1.96 (m) | 29.6 | 12, 14 | 11, 15 |
| 14 | 1.81 (m) | 39.1 | 13 | 10, 13 |
| 15 | 80.7 | |||
| 16 | 1.64 (s) | 23.8 | 10, 12 | |
| 17 | 1.47 (s) | 25.7 | 14, 18 | |
| 18 | 1.28 (s) | 25.0 | 14, 17 |
S. antibioticus NRRL 8167基因组DNA纯化合格后,委托武汉菲沙基因信息有限公司进行全基因组测序。结果显示:S. antibioticus NRRL 8167基因组含有一条线性染色体,染色体长度为9 379 887 bp,(G+C)mol%含量为71.5%。采用AntiSMASH (V4.0.2)对S. antibioticus NRRL 8167的基因组序列进行分析,发现共含有28个次级代谢物生物合成基因簇,包括3个非核糖体肽合成酶(Nonribosomal peptide synthetase,NRPS)基因簇、2个Ⅰ型聚酮合酶(Type Ⅰ polyketide synthase,PKS)基因簇、1个Ⅱ型聚酮合酶(type Ⅱ polyketide synthase,PKS)基因簇、3个萜类(Terpene)合成基因簇、2个Siderophore合成基因簇、2个Lanthipeptide合成基因簇、1个Butyrolactone合成基因簇、1个Indole合成基因簇、1个Melanin合成基因簇、1个Ectoine合成基因簇、1个Lassopeptide合成基因簇、1个Linardin合成基因簇、1个Bacteriocin合成基因簇、6个杂合基因簇、2个其他类型代谢物合成基因簇。详细信息见表 2。
| 编号 No. |
类型 Type |
大小 Size (kb) |
类似基因簇 Similar known cluster |
相似度 Similarity (%) |
| 1 | Butyrolactone | 9.4 | Lactonamycin | 10 |
| 2 | Others | 41.1 | Akaeolide | 8 |
| 3 | T1pks | 93.9 | Indanomycin | 100 |
| 4 | Nrps | 62.5 | Herboxidiene | 8 |
| 5 | Lanthipeptide-Bacteriocin | 25.5 | Informatipeptin | 100 |
| 6 | Otherks-T1pks | 48.7 | Laspartomycin | 13 |
| 7 | Terpene | 24.5 | Hopene | 92 |
| 8 | Indole | 21.3 | Meridamycin | 5 |
| 9 | Nrps-Siderophore | 60.6 | Scabichelin | 100 |
| 10 | Terpene | 21.7 | Geosmin | 100 |
| 11 | Bacteriocin | 10.0 | ||
| 12 | Siderophore | 11.9 | ||
| 13 | Lanthipeptide | 21.3 | ||
| 14 | Terpene | 17.7 | Albaflavenone | 100 |
| 15 | Siderophore | 11.7 | Desferrioxamine B | 83 |
| 16 | Melanin | 9.3 | Lactonamycin | 8 |
| 17 | Nrps | 57.2 | SCO-2138 | 71 |
| 18 | Ectoine | 10.4 | Ectoine | 100 |
| 19 | Others | 42.6 | ||
| 20 | T2pks | 72.5 | Spore pigment | 83 |
| 21 | Lassopeptide | 22.6 | SRO15-2005 | 40 |
| 22 | T1pks | 40.6 | Maduropeptin | 3 |
| 23 | Terpene-Melanin | 21.2 | Melanin | 71 |
| 24 | Lanthipeptide | 22.6 | Labyrinthopeptin A1, A3 | 60 |
| 25 | Butyrolactone-Nrpsfragment-Otherks | 66.4 | Pyridomycin | 26 |
| 26 | Linaridin | 20.6 | Legonaridin | 33 |
| 27 | Nrps | 48.6 | Mitomycin | 10 |
| 28 | Nrps-T1pks | 57.1 | Antimycin | 100 |
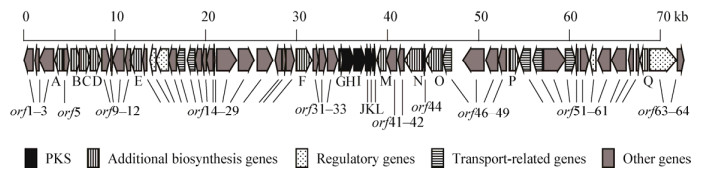
Naphthgeranine A属于萘醌类天然产物,从结构上分析该化合物是由Ⅲ型聚酮合酶与萜类合成酶杂合产生的天然产物。然而,利用AntiSMASH数据库对基因组数据比对分析显示基因组中并不含有这样的基因簇。但是,在该菌株中却包括一个Ⅱ型聚酮合酶基因簇。因此,推测这个基因簇可能负责合成Naphthgeranine A。该基因簇定位于编号20的基因簇,这个生物合成基因簇位于染色体DNA的7 883 630−7 956 142位置,大小为72.5 kb,功能基因的组织排布如图 5所示。同时,对该基因簇的基因进行功能注释,其基因功能见表 3。
|
| 图 5 S. antibioticus NRRL 8167中Naphthgeranine A生物合成基因簇 Figure 5 The biosynthetic gene cluster of naphthgeranine A from S. antibioticus NRRL 8167 |
|
|
| 开放阅读框 Orf |
大小 Size (aa) |
同源蛋白及其大小 Protein homologue, size (aa) |
一致性/相似性 Identity/Similarity (%/%) |
预测的基因功能 Proposed function |
| orf1 | 343 | Sp2771 (3VZ1_A), 457 | 52/69 | Succinate-semialdehyde dehydrogenase |
| orf2 | 144 | PRK10546 (2RRK_A), 140 | 38/53 | CTP pyrophosphohydrolase |
| orf3 | 512 | LigTsib (4EQ5_A), 571 | 34/52 | ATP-dependent DNA ligase |
| NapA | 325 | Hlmi (4NTD_A), 340 | 51/64 | Thioredoxin reductase |
| orf5 | 190 | PlcR (2QFC_A), 293 | 35/59 | Virulence regulator |
| NapB | 323 | Qor (1YB5_A), 351 | 31/52 | Quinone oxidoreductase |
| NapC | 394 | ChsE4-ChsE5 (4X28_A), 400 | 32/45 | Acyl-CoA dehydrogenase |
| NapD | 360 | Chse4-chse5(4X28_D), 373 | 35/46 | Acyl-CoA dehydrogenase |
| orf9 | 286 | TauD (1OIH_A), 301 | 30/48 | Alpha-ketoglutarate-dependent dioxygenase |
| orf10 | 66 | ASD48_36420 (KQX78285.1), 64 | 98/100 | Hypothetical protein |
| orf11 | 409 | NAGPA (CQR65357.1), 413 | 67/78 | Metallophosphoesterase |
| orf12 | 182 | Pat (4JXQ_A), 186 | 38/57 | Acetyltransferase |
| NapE | 400 | Methyltransf_25; Nudix_Hydrolase (AXE84246.1), 390 | 72/81 | Methyltransferase |
| orf14 | 149 | CMD; YciW (2O4D_A), 165 | 39/54 | Carboxymuconolactone decarboxylase |
| orf15 | 222 | LiaR (5HEV_A), 210 | 40/58 | Response regulator protein |
| orf16 | 455 | HisKA_3 (AXE90400.1), 439 | 69/77 | Histidine kinase |
| orf17 | 292 | RbmA (4BE5_A), 262 | 39/50 | Biofilm scaffolding protein |
| orf18 | 309 | tm0544 (1VPL_A), 256 | 32/53 | ABC transporter ATP-binding protein |
| orf19 | 289 | cbiO1 (3GFO_A), 275 | 41/62 | Cobalt import ATP-binding protein |
| orf20 | 252 | CbiQ (5X3X_Q), 244 | 43/57 | Cobalt energy-coupling factor transporter |
| orf21 | 173 | PRK02898 (CBG75386.1), 144 | 71/75 | Cobalt transport protein |
| orf22 | 257 | CbiM (5X3X_M), 222 | 63/75 | Cobalt energy-coupling factor transporter |
| orf23 | 69 | F750_6382 (AGJ58807.1), 73 | 90/92 | Hypothetical protein |
| orf24 | 710 | RsbU (GAQ60155.1), 707 | 74/81 | Phosphoserine phosphatase |
| orf25 | 564 | PucR; HTH_30 (CBG69411.1), 386 | 84/90 | Regulator |
| orf26 | 575 | VanW (SPF06308.1), 579 | 77/86 | Vancomycin B-type resistance protein |
| orf27 | 259 | ASD48_36500 (KQX78308.1), 219 | 99/99 | Hypothetical protein |
| orf28 | 118 | STAS (KPI28265.1), 118 | 95/97 | Sulfate transporter/antisigma-factor antagonist |
| orf29 | 313 | PrsR (EFD71359.1), 309 | 73/81 | Sensor histidine kinase |
| NapF | 572 | Mature chain (Q05355.1), 555 | 78/84 | Polyketide hydroxylase |
| orf31 | 187 | ASD48_23595 (KQX64296.1), 215 | 83/83 | Hypothetical protein |
| orf32 | 248 | BU52_31165 (KES03345.1), 211 | 64/72 | Class F sortase |
| orf33 | 384 | ABM; SchA_CurD; GlnE (AVV43942.1), 385 | 92/94 | TcmI family type Ⅱ polyketide cyclase |
| NapG | 147 | Cupin_2 (SHI48933.1), 147 | 84/93 | Monooxygenase |
| NapH | 418 | KAS_I_II (1TQY_A), 424 | 61/73 | Beta-ketoacyl-acyl carrier protein (ACP) synthase (KAS) |
| NapI | 415 | FabB (1TQY_B), 415 | 46/60 | Polyketide beta-ketoacyl synthase |
| NapJ | 84 | PP-binding (2K0X_A), 86 | 39/55 | Polyketide synthase acyl carrier protein |
| NapK | 159 | TcmN_ARO-CYC_like (3TL1_A), 159 | 68/77 | Polyketide cyclase |
| NapL | 110 | Cyclase_polyket (1TUW_A), 109 | 43/62 | TcmI family type Ⅱ polyketide cyclase |
| NapM | 342 | Calo6 (4Z2Y_A), 359 | 35/51 | Methyltransferase |
| orf41 | 371 | ASD48_23685 (KQX64313.1), 382 | 99/99 | Hypothetical protein |
| orf42 | 219 | ASD48_23680 (KQX64312.1), 219 | 100/100 | Hypothetical protein |
| NapN | 654 | Radical_SAM (AVV47162.1), 651 | 91/93 | RiPP maturation radical SAM protein 1 |
| orf44 | 40 | PRK14985 (2ECP_A), 796 | 67/91 | Maltodextrin phosphorylase |
| NapO | 607 | DAP2 (1VE6_A), 582 | 34/50 | Acylamino-acid-releasing enzyme |
| orf46 | 344 | ASD48_23665 (KQX64310.1), 485 | 99/99 | Hypothetical protein |
| orf47 | 756 | ASD48_23660 (KQX64309.1), 758 | 99/99 | Hypothetical protein |
| orf48 | 427 | Eis (5EBV_A), 422 | 34/46 | Acetyltransferase |
| orf49 | 263 | ASD48_23650 (KQX64307.1), 263 | 100/100 | Hypothetical protein |
| NapP | 377 | UbiH (AQA12410.1), 377 | 76/84 | FAD-dependent oxidoreductase |
| orf51 | 403 | XylH (AWW36118.1), 420 | 83/89 | ABC transporter permease |
| orf52 | 363 | XylF (3M9W_A), 313 | 36/53 | D-xylose binding protein |
| orf53 | 782 | RsbU (TCR54763.1), 838 | 79/84 | Serine phosphatase |
| orf54 | 354 | Chve (3URM_A), 330 | 99/99 | Periplasmic sugar binding protein |
| orf55 | 96 | Tdk (2B8T_A), 223 | 44/60 | Thymidine kinase |
| orf56 | 326 | ASD48_23615 (KQX64300.1), 326 | 100/100 | Hypothetical protein |
| orf57 | 232 | Desr (4LDZ_A), 203 | 32/47 | Transcriptional regulatory protein |
| orf58 | 448 | Peptidase_S13 (2WKE_A), 466 | 37/56 | D-alanyl-d-alanine carboxypeptidase |
| orf59 | 527 | ASD48_23600 (KQX64297.1), 527 | 99/99 | Hypothetical protein |
| orf60 | 187 | ASD48_23595 (KQX64296.1), 215 | 84/84 | Hypothetical protein |
| orf61 | 97 | ASD48_23590 (KQX64295.1), 97 | 98/98 | Hypothetical protein |
| NapQ | 286 | MhpC (3WZL_A), 278 | 34/55 | Zearalenone hydrolase |
| orf63 | 997 | (KQX64293.1), 995 | 99/99 | AfsR family transcriptional regulator |
| orf64 | 239 | Embr (2FEZ_A), 388 | 35/47 | Regulatory protein |
通过对Naphthgeranine A生物合成基因簇各基因的功能注释,结合生物信息学分析,可将其合成途径分为骨架的形成和后修饰两大步骤。
推测NapH、NapI、NapJ、NapK和NapL负责了这个化合物骨架的合成。NapH是KSα (ketosynthase-α),催化Clasien缩合反应。NapI属于KSβ (ketosynthase-β),决定了碳链的长度。结合生物信息学分析和序列比对(图 6)结果表明,NapI 133、137位为G、L,结合分子结构,推测NapI形成一个含有20个碳的碳链[19]。NapK是一个环化酶,与环化酶TcmN一致性高达51.7%,TcmN负责将Decaketidyl-S-ACP环化形成TcmF2[20],因此推测NapK负责Naphthgeranine A中A、B和C环的形成。NapL是一个与TcmI有42.7%一致性的环化酶,TcmI催化TcmF2的D环环化形成了TcmF1[20],因此推测NapL负责了Naphthgeranine A中D环的形成,最终形成了中间体C,至此Naphthgeranine A的骨架就形成了。

|
| 图 6 NapI和其他KSβ的氨基酸序列比对 Figure 6 Alignment of the amino acids sequences of NapI with other KSβ |
|
|
在完成了Naphthgeranine A分子主体骨架碳链组装后,与多肽羟化酶CabE一致性高达55.7%的NapF催化形成了萘醌结构D;之后经过还原形成了E,E在FAD依赖的单加氧酶NapP的作用下,发生Baeyer-Villiger反应形成F;随后F在水解酶NapQ的催化下形成了中间体G,接下来发生亲核取代反应生成H;H在甲基转移酶NapM的催化下形成I,最后通过还原反应最终合成了Naphthgeranine A。详细过程见图 7。

|
| 图 7 推导的Naphthgeranine A的生物合成途径 Figure 7 The proposed biosynthetic pathway of naphthgeranine A |
|
|
本研究从S. antibioticus NRRL 8167中分离到萘醌类天然产物Naphthgeranine A,通过高分辨质谱和核磁共振光谱分析确认了其结构,并基于基因组信息推测出其生物合成基因簇和可能的生物合成途径。以往的研究表明在Naphthgeranine A的类似物Merochlorin和Napyradiomycin的生物合成中分别有两个钒依赖的氯过氧化物酶Mcl24和NapH3,催化了α-羟基酮的重排反应,有推测认为在Naphthgeranine A的生物合成途径中有个关键酶是钒依赖的氯过氧化物酶[15]。基于Naphthgeranine A的化学结构,推测Ⅲ型聚酮合酶负责形成了萘醌环部分的生物合成[15],然而在S. antibioticus NRRL 8167基因组中却没有发现Ⅲ型聚酮合酶,也没有关键酶钒依赖的氯过氧化物酶同源蛋白。因此我们推测S. antibioticus NRRL 8167中通过其他的合成方式来合成Naphthgeranine A。
微生物来源的芳香族聚酮化合物通常是由Ⅱ型聚酮合酶合成的[21]。Ⅱ型聚酮合酶利用酰基载体蛋白活化底物酰基辅酶A,然后在聚酮合酶KS的催化下脱羧缩合形成C−C键[22]。Ⅱ型聚酮合酶仅利用一套重复使用的模块,催化多轮Claisen缩合反应形成-聚酮中间体,再经过环化形成多环的芳香族化合物[20, 23-24]。伴随多种多样的后修饰过程,例如氧化、还原、水解、重排和基团迁移等,最终形成了结构复杂、活性多样的聚酮类化合物[21]。
S. antibioticus NRRL 8167的基因组系列分析显示该基因组中只存在一个Ⅱ型聚酮合酶,基于上述理由推测由该Ⅱ型聚酮合酶合成了Naphthgeranine A,同时在该化合物的生物合成途径中有可能涉及到一些比较新颖的酶催化反应,如氧化重排反应、选择性还原反应等,以上分析表明Naphthgeranine A的生物合成途径与以前推测不同[15]。结合基因组的相关信息,我们对Naphthgeranine A的生物合成途径进行了推测。本文为研究该化合物的体内合成路线奠定了基础,也有利于加深对聚酮类天然产物的认识,但详细的生物合成机理仍然需要后续的研究验证。
| [1] |
Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014[J]. Journal of Natural Products, 2016, 79(3): 629-661. DOI:10.1021/acs.jnatprod.5b01055 |
| [2] |
Newman DJ, Cragg GM. Natural products as sources of new drugs over the 30 years from 1981 to 2010[J]. Journal of Natural Products, 2012, 75(3): 311-335. DOI:10.1021/np200906s |
| [3] |
Matsuda Y, Abe I. Biosynthesis of fungal meroterpenoids[J]. Natural Product Reports, 2015, 33(1): 26-53. |
| [4] |
Sintchak MD, Fleming MA, Futer O, et al. Structure and mechanism of inosine monophosphate dehydrogenase in complex with the immunosuppressant mycophenolic acid[J]. Cell, 1996, 85(6): 921-930. DOI:10.1016/S0092-8674(00)81275-1 |
| [5] |
McCowen MC, Callender ME, Lawlis LF Jr. Fumagillin (H-3), a new antibiotic with amebicidal properties[J]. Science, 1951, 113(2930): 202-203. DOI:10.1126/science.113.2930.202 |
| [6] |
Shin-Ya K, Imai S, Furihata K, et al. Isolation and structural elucidation of an antioxidative agent, naphterpin[J]. The Journal of Antibiotics, 1990, 43(4): 444-447. DOI:10.7164/antibiotics.43.444 |
| [7] |
Wessels P, G hrt A, Zeeck A, et al. Metabolic products of microorganisms. 260: naphthgeranines, new naphthoquinone antibiotics from Streptomyces sp[J]. The Journal of Antibiotics, 1991, 44(9): 1013-1018. DOI:10.7164/antibiotics.44.1013 |
| [8] |
Volkmann C, Jen UH, Zeeck A, et al. Biosynthetic capacities of actinomycetes. 3 naphthgeranine F, a minor congener of the naphthgeranine group produced by Streptomyces violaceus[J]. The Journal of Antibiotics, 1995, 48(6): 522-524[J]. The Journal of Antibiotics, 1995, 48(6): 522-524. DOI:10.7164/antibiotics.48.522 |
| [9] |
Pathirana C, Jensen PR, Fenical W. Marinone and debromomarinone: antibiotic sesquiterpenoid naphthoquinones of a new structure class from a marine bacterium[J]. Tetrahedron Letters, 1992, 33(50): 7663-7666. DOI:10.1016/0040-4039(93)88010-G |
| [10] |
Hardt IH, Jensen PR, Fenical W. Neomarinone, and new cytotoxic marinone derivatives, produced by a marine filamentous bacterium (actinomycetales)[J]. Tetrahedron Letters, 2000, 41(13): 2073-2076. DOI:10.1016/S0040-4039(00)00117-9 |
| [11] |
Takemura S, Hirayama A, Tokunaga J, et al. ChemInform abstract: a concise total synthesis of (±)–A80915G, a member of the napyradiomycin family of antibiotics[J]. ChemInform, 2000, 31(2): 2000. |
| [12] |
Kuniaki T, Yoshiki T, Masazumi K, et al. The first total synthesis of (±)-napyradiomycin A1[J]. Chemistry Letters, 2002, 31(1): 14-15. DOI:10.1246/cl.2002.14 |
| [13] |
Kaysser L, Bernhardt P, Nam SJ, et al. Merochlorins A-D, cyclic meroterpenoid antibiotics biosynthesized in divergent pathways with vanadium-dependent chloroperoxidases[J]. Journal of the American Chemical Society, 2012, 134(29): 11988-11991. DOI:10.1021/ja305665f |
| [14] |
Pepper HP, George JH. Biomimetic total synthesis of (±)-merochlorin A[J]. Angewandte Chemie International Edition, 2013, 52(46): 12170-12173. DOI:10.1002/anie.201307200 |
| [15] |
Murray LA, McKinnie SM, Pepper HP, et al. Total synthesis establishes the biosynthetic pathway to the naphterpin and marinone natural products[J]. Angewandte Chemie International Edition, 2018, 57(34): 11009-11014. DOI:10.1002/anie.201804351 |
| [16] |
Rommel KR, Li CX, Kelly WL. Identification of a tetraene-containing product of the indanomycin biosynthetic pathway[J]. Organic Letters, 2011, 13(10): 2536-2539. DOI:10.1021/ol200570u |
| [17] |
Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces Genetics[M]. Colney: John Innes Foundation, 2000: 125-127.
|
| [18] |
Lu C, Yang C, Xu Z. Three naphthoquinones from Streptomyces sp. XZYN-4[J]. Records of Natural Products, 2016, 10(4): 430-440. |
| [19] |
Tang Y, Tsai SC, Khosla C. Polyketide chain length control by chain length factor[J]. Journal of the American Chemical Society, 2003, 125(42): 12708-12709. DOI:10.1021/ja0378759 |
| [20] |
Zhou H, Li YR, Tang Y. Cyclization of aromatic polyketides from bacteria and fungi[J]. Natural Product Reports, 2010, 27(6): 839-868. DOI:10.1039/b911518h |
| [21] |
Zhang Z, Pan HX, Tang GL. New insights into bacterial type Ⅱ polyketide biosynthesis[J]. F1000Research, 2017(6): 172. |
| [22] |
Weissman KJ. Polyketide biosynthesis: understanding and exploiting modularity[J]. Philosophical Transactions of the Royal Society of London. Series A: Mathematical, Physical and Engineering Sciences, 2004, 362(1825): 2671-2690. DOI:10.1098/rsta.2004.1470 |
| [23] |
Das A, Khosla C. Biosynthesis of aromatic polyketides in bacteria[J]. Accounts of Chemical Research, 2009, 42(5): 631-639. DOI:10.1021/ar8002249 |
| [24] |
Hertweck C. The biosynthetic logic of polyketide diversity[J]. Angewandte Chemie International Edition, 2009, 48(26): 4688-4716. DOI:10.1002/anie.200806121 |
 2019, Vol. 46
2019, Vol. 46