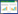扩展功能
文章信息
- 赵祥, 刘红玲, 杨盼, 翟亚萍, 王绍明, 张霞
- ZHAO Xiang, LIU Hong-Ling, YANG Pan, ZHAI Ya-Ping, WANG Shao-Ming, ZHANG Xia
- 滴灌对苜蓿根际土壤细菌多样性和群落结构的影响
- Effects of drip irrigation on bacterial diversity and community structure in rhizosphere soil of alfalfa
- 微生物学通报, 2019, 46(10): 2579-2590
- Microbiology China, 2019, 46(10): 2579-2590
- DOI: 10.13344/j.microbiol.china.180874
-
文章历史
- 收稿日期: 2018-11-05
- 接受日期: 2019-01-25
- 网络首发日期: 2019-02-26
2. 成都师范学院 四川 成都 611130
2. Chengdu Normal University, Chengdu, Sichuan 611130, China
紫花苜蓿(Medicago sativa L.)在我国西北地区大范围种植,号称“牧草之王”。苜蓿作为多年生豆科牧草,具有产量高、适应性强、抗旱耐盐碱、家畜适口性好等特点,是发展节粮型畜牧业的重要基础[1]。随着人工草地种植面积的不断扩大,滴灌作为高效可行的节水灌溉技术得到广泛应用,在节省成本的基础上,克服了山区、丘陵等地形限制,满足作物对水分和养分的需求[2]。滴灌作为典型的节水技术,促进植物生长,同时也改变了农田土壤中微生物群落,为土壤中微生物的群落结构及多样性带来影响。
土壤微生物是农田生态系统中对环境变化极其敏感的生物活性因子,作为土壤与植物的纽带,对农田生态系统的健康状况与植被的生长发育有重要的影响。细菌是土壤微生物中数量最丰富、分布最广泛的类群,参与氮磷循环、土壤矿化和养分吸收等过程[3],对于维持土壤生态系统稳定和保持土壤肥力等方面存在重要作用,且细菌的群落结构和多样性在一定程度上能反映土壤质量的变化趋势[4]。研究土壤细菌群落结构和多样性,对于合理利用农田生态系统的生物学功能,以及实现农田生态系统的可持续发展与农业产量的提高具有重要意义。
随着测序技术的发展,具有高通量、高准确度和低成本优势的第二代高通量测序技术已广泛应用于环境微生物研究的各方面[5-7],与传统测序技术相比,高通量测序技术能更真实全面地反映微生物的群落结构和多样性。目前关于苜蓿根际土壤细菌群落的研究相对较少,为了更真实全面地了解微生物群落特征的变化,本研究基于IonS5TMXL测序平台,利用单端测序(Single-end)方法构建小片段文库进行单端测序,对滴灌和自然降雨苜蓿土壤微生物群落结构和多样性进行分析,旨在揭示滴灌作用对苜蓿土壤细菌多样性及群落分布特征的影响,探讨细菌群落组成与环境因子之间的关系,为滴灌促进植物生长提供微生物方面的科学依据。
1 材料与方法 1.1 主要试剂和仪器Phusion® High-Fidelity PCR Master Mix with GC Buffer,New England Biolabs公司;GeneJETTM Gel Extraction Kit,Thermo Scientific公司;Ion Plus Fragment Library Kit 48 rxns,Thermofisher公司;中性磷酸酶试剂盒,Solarbio®科技有限公司。Ion S5TMXL仪器,Thermofisher公司。
1.2 样品采集与处理调查区位于新疆天山北麓中段,准噶尔盆地南缘的147团11连。该区气候干燥、降雨少、地表蒸发强烈,大面积种植紫花苜蓿、棉花、玉米等农作物。紫花苜蓿处于生长3年的第一茬盛花期,田间平均每10 d进行滴水灌溉,施水量约420 m3/hm2,于灌溉后第6天进行采样。在地埋式滴灌与自然降雨两种人工种植苜蓿田中分别设置3个标准样地,大小为20 m×20 m,各样地之间环境条件相似、距离接近,海拔在358.6 m−360.7 m之间。在每个标准样地内以“S”形五点混合采样法分别收集滴灌苜蓿根际(MR)、非根际(MB)与自然降雨苜蓿根际(NR)、非根际(NB)共4组12个土壤样品。根际土壤取样采用“抖根法”,植物根系从土壤中挖出,抖掉与根系结合松散的土壤,收集与根系紧密结合在4 mm范围内的土壤作为根际土壤,在采集根际土旁选取没有植物生长的位置采集与根际土相同深度的土壤作为非根际土壤样品[8]。土壤于4 ℃保存并尽快带回实验室,一份保存于−80 ℃,供提取土壤DNA进行高通量测序;另一份风干保存,用于基础数据测定。
1.3 土壤理化和酶活性指标测定土壤理化指标的测定参考鲍士旦《土壤农化分析》[9]中的测定方法:全氮(半微量开氏法)、全钾(火焰光度法)、全磷(HClO4-H2SO4法)、碱解氮(碱解扩散法)、有效磷(0.5 mol/L NaHCO3法)、有机质(外加热法)、土壤含水率(铝盒称重法)、pH值(奥豪斯测试笔测定)。土壤中酶活性的测定方法:脲酶(苯酚钠-次氯酸钠比色法)、中性磷酸酶(试剂盒法)。
1.4 细菌16S rRNA基因高通量测序采用CTAB法对样本的基因组DNA进行提取,利用琼脂糖凝胶电泳检测DNA的纯度和浓度,取适量的样品DNA于离心管中,使用无菌水稀释样品至1 ng/μL,每个样品做3个重复。以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物515F (5′-GTGCCA GCMGCCGCGGTAA-3′)和806R (5′-GGACTACHV GGGTWTCTAAT-3′)对16S rRNA基因的V3−V4高变区片段进行PCR扩增。PCR反应体系:2×Phusion Master Mix 15 μL,正、反向引物(2 μmol/L)各3 μL,基因组DNA (1 ng/μL) 7 μL,ddH2O 2 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,50 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。PCR产物使用2%的琼脂糖凝胶进行电泳检测。根据PCR产物浓度进行等量混样,充分混匀后使用1×TAE浓度2%的琼脂糖胶电泳纯化PCR产物,回收目标条带。使用Ion Plus Fragment Library Kit 48 rxns进行文库的构建,经Qubit定量和文库检测合格后,使用Thermofisher的Ion S5TMXL进行上机测序。
1.5 细菌多样性数据分析通过Cutadapt软件[10]对Reads进行过滤,按Barcode拆分出各样品数据,截去Barcode和引物序列并去除嵌合体序列[11],得到最终有效数据。用Uparse软件[12]对序列进行聚类,默认以97%的一致性将序列聚类成为操作分类单元(Operational taxonomic unit,OTU)。根据OTU聚类结果,以SSUrRNA数据库[13]对每个OTU的代表序列做物种注释,获得分类学信息和各样本在各分类水平上的群落组成,用柱状图进行可视化。同时,使用QIIME (V1.9.1)软件进行OTU丰度与α多样性计算,以得到样品内物种丰富度和均匀度信息。
1.6 统计分析使用SPSS 19.0对土壤性质进行单因素方差分析,对土壤性质与细菌多样性进行Pearson相关性分析;利用Canoco 4.5对环境中菌群矩阵进行除趋势对应分析(Detrended correspondence analysis,DCA),发现最大梯度长度小于3,采用冗余分析(Redundancy analysis,RDA)探讨土壤中细菌群落结构和环境因子之间关系。
2 结果与分析 2.1 土壤理化性质和酶活性土壤样品理化性质和酶活性的测定结果如表 1所示,滴灌和自然降雨苜蓿根际土壤中全氮(Total nitrogen,TN)、碱解氮(Alkaline nitrogen,AN)、全钾(Total potassium,TK)、土壤脲酶(Soild urease,S-UE)和土壤中性磷酸酶(Soild neutral phosphatase,S-NP)含量均显著高于非根际土壤;滴灌苜蓿根际土壤含水率(Moisture content,SM)、有机质(Organic matter,OM)、全磷(Total phosphorus,TP)、TK、AN、有效磷(Available phosphorus,AP)、S-UE和S-NP含量均显著高于自然降雨根际土壤,而pH和TN含量却显著低于自然降雨根际土壤。其中S-UE和S-NP活性是评价土壤中氮、磷素生物转化方向与强度的指标,结果表明滴灌作用能显著提高苜蓿生长所必需的养分,且苜蓿根际对土壤中的养分具有一定的聚集吸附效应,发挥了保持肥力的生态功能。
| Samples | pH | SM (%) |
OM (g/kg) |
TN (g/kg) |
TP (g/kg) |
TK (g/kg) |
AN (mg/kg) |
AP (mg/kg) |
S-UE (U/g) |
S-NP (nmol/g) |
| MR | 7.54±0.01c | 12.90±0.78a | 12.95±0.38a | 0.70±0.01b | 0.26±0.01a | 33.96±0.54a | 81.67±9.33a | 36.22±7.19a | 0.44±0.06a | 12 915.31±330a |
| MB | 7.63±0.01b | 14.25±0.33a | 8.89±0.54b | 0.52±0.03c | 0.23±0.00b | 32.31±0.13b | 50.17±2.33b | 24.41±2.51b | 0.12±0.02b | 10 736.43±347b |
| NR | 7.71±0.04a | 11.11±1.78b | 7.82±0.46b | 0.81±0.03a | 0.21±0.00c | 32.49±0.24b | 60.67±2.33b | 20.95±1.67b | 0.12±0.02b | 9 194.99±456c |
| NB | 7.73±0.03a | 13.22±0.43a | 7.90±0.15b | 0.55±0.02c | 0.23±0.00b | 31.29±0.19c | 33.83±4.67c | 24.29±1.55b | 0.08±0.00b | 7 833.83±26d |
| 注:SM:含水率;OM:有机质;TN:全氮;TP:全磷;TK:全钾;AN:碱解氮;AP:有效磷;S-UE:土壤脲酶;S-NP:土壤中性磷酸酶.同列不同小写字母表示不同样品分组之间差异显著(P<0.05). Note: SM: Moisture content; OM: Organic matter; TN: Total nitrogen; TP: Total phosphorus; TK: Total potassium; AN: Alkaline nitrogen; AP: Available phosphorus; S-UE: Soild urease; S-NP: Soild neutral phosphatase. Different lowercase letters in the same column indicated that there was a significant difference among the groups of different samples (P<0.05). |
||||||||||
对4组(12个)土壤样品进行了16S rRNA基因高通量测序,经序列过滤和去除嵌合体序列,最终得到用于后续分析的优质序列共879 675条,平均长度分布在417−418 nt。在97%相似度下将序列聚类为用于物种分类的OTU,统计各样品在不同OTU中的丰度信息(表 2)。4组土壤样品的细菌覆盖率为98.7%−98.9%,说明取样合理,所测结果能够比较真实地反映土壤中细菌群落。稀释曲线反映了样本的测序深度,可用于评估测序数据量的合理性。由图 1所示,曲线趋向平坦,说明测序数据量合理,基本能够反映土壤的细菌群落组成,更多数据量只会产生少量新的物种。
| Samples | Reads | OTUs | Shannon index | Simpson index | Chao1 index | ACE index | Coverage |
| MR | 70 981±9199a | 2 423±120a | 9.478±0.023a | 0.995±0.001a | 2 673±247a | 2 695±224a | 0.989±0.003a |
| MB | 68 591±16372a | 2 312±104a | 9.275±0.044b | 0.996±0.001a | 2 701±210a | 2 679±185a | 0.987±0.005a |
| NR | 78 948±1552a | 2 331±107a | 9.315±0.039b | 0.996±0.001a | 2 654±214a | 2 649±149a | 0.988±0.002a |
| NB | 74 705±9681a | 2 402±52a | 9.118±0.006c | 0.994±0.002a | 2 685±240a | 2 696±176a | 0.988±0.002a |
| 注:同列不同小写字母表示不同样品分组之间差异显著(P<0.05). Note: Different lowercase letters in the same column indicated that there was a significant difference among the groups of different samples (P<0.05). |
|||||||
|
| 图 1 97%相似水平下样品的稀释性曲线 Figure 1 Rarefaction curves of the 97% similarity level 注:MR:滴灌根际土壤;MB:滴灌非根际土壤;NR:自然降雨根际土壤;NB:自然降雨非根际土壤. Note: MR: Drip irrigation rhizosphere soil; MB: Drip irrigation non-rhizosphere soil; NR: Natural rainfall rhizosphere soil; NB: Natural rainfall non-rhizosphere soil. |
|
|
在97%一致性阈值下,使用α多样性指数(Shannon、Simpson、Chao1、ACE)对单个样品复杂性进行统计分析,评估各样品中微生物群落的物种多样性和丰富度的差异。由表 2可知,Shannon指数表明:MR>NR>MB>NB,滴灌苜蓿根际与非根际土壤的细菌多样性均显著高于自然降雨苜蓿的根际与非根际土壤(P<0.05)。此外,Chao1指数显示MB>NB>MR>NR,滴灌苜蓿根际土壤细菌群落丰富度高于自然灌溉根际土壤,但差异不显著(P>0.05)。
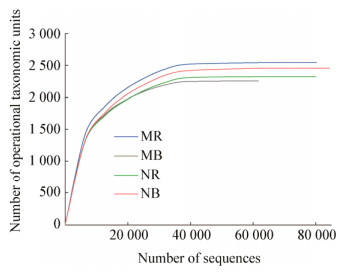
2.4 细菌群落组成与结构利用RDP classifier对各样品OTU从门到属依次进行分类信息分析,挖掘土壤样品在各个水平上的菌群结构。所有供试土壤样品中共检测到细菌46门53纲116目220科469属。选取每个分组在门、属分类水平上最大丰度排名前十的物种,生成物种相对丰度柱形累加图(图 2)。
|
| 图 2 土壤样品细菌类群门(A)和属(B)水平分布图 Figure 2 Phylum-level (A) and genus-level (B) distribution of bacterial in soil samples 注:Unclassified:分类地位不明确的细菌类群;Others:稀有细菌类群. Note: Unclassified: Bacterial sequences that were not identified into anyone in databases; Others: Rare groups. |
|
|
在门分类水平上,共检测到细菌46个门,其中优势门(丰度>1%)数量较少,非优势门数量较多。土壤样品中丰度相对较高的菌门依次为变形菌门(Proteobacteria,25.27%−34.42%)、放线菌门(Actinobacteria,14.70%−26.60%)、拟杆菌门(Bacteroidetes,8.37%−14.42%)、芽单胞菌门(Gemmatimonadetes,8.13%−14.45%)、酸杆菌门(Acidobacteria,6.27%−12.74%)、绿弯菌门(Chloroflexi,5.20%−7.52%)、厚壁菌门(Firmicutes,1.24%−3.42%)、疣微菌门(Verrucomicrobia,0.83%−1.60%)、硝化螺旋菌门(Nitrospirae,0.08%−0.75%),以及部分分类地位不明确的细菌类群(0.65%−4.41%)和其他丰度较低的稀有细菌类群(2.36%−7.11%)。其中,在已分类的优势门中,除放线菌门和厚壁菌门为革兰氏阳性细菌外,其余菌门均是革兰氏阴性细菌。变形菌门作为丰度最大的优势菌门,包含α-变形菌纲(Alphaproteobacteria,11.41%−18.97%)、δ-变形菌纲(Deltaproteobacteria,2.89%−4.50%)以及γ-变形菌纲(Gammaproteobacteria,9.50%−13.79%),α-变形菌纲是主要的变形菌门中的优势菌纲,其主要集中于鞘氨醇单胞菌目(Sphingomonadales,6.87%−22.68%)以及α-变形菌纲内分类不明确的菌群(图 2A)。MR中变形菌门占门水平细菌总数的34.42%,α-变形菌纲、δ-变形菌纲、γ-变形菌纲分别占纲水平细菌总数的18.97%、3.98%和11.38%,NR中变形菌门占门水平细菌总数25.27%,α-变形菌纲、δ-变形菌纲、γ-变形菌纲分别占纲水平细菌总数的12.84%、2.89%和9.50%。
在属分类水平上,共检测到土壤细菌469个属,其中种类繁杂、丰度较低的稀有细菌类群为最主要菌群,分别占80.00%、85.29%、82.28%、85.43%,说明土壤中存在着大量有待发掘的未知微生物资源。已分类的相对丰度较高的6个属依次为鞘氨醇单胞菌属(Sphingomonas,1.00%−4.54%)、节杆菌属(Arthrobacter,0.62%−4.09%)、类诺卡氏菌属(Nocardioides,0.77%−3.03%)、溶杆菌属(Lysobacter,0.27%−2.24%)、马赛菌属(Massilia,0.03%−1.12%)、Haliangium (0.78%−1.79%),同时含有在各个科下属分类地位不明确的菌属(图 2B)。其中节杆菌属和类诺卡氏菌属为革兰氏阳性细菌,而鞘氨醇单胞菌属、溶杆菌属、马赛菌属以及Haliangium均为革兰氏阴性细菌。
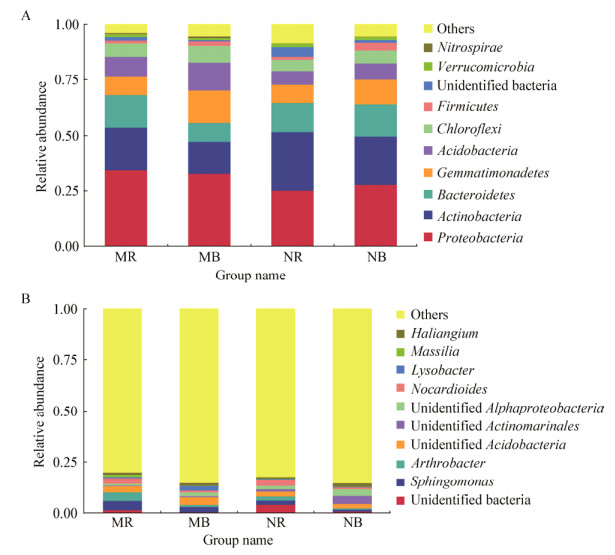
2.5 细菌群落差异性分析进一步探究各土壤样品间微生物群落结构的差异(图 3)。在门水平上,芽单胞菌门、酸杆菌门、绿弯菌门、迷踪菌门(Elusimicrobia)、纤维杆菌门(Fibrobacteres)在滴灌根际土壤中的丰度显著低于非根际土壤,而拟杆菌门的丰度则高于非根际土壤(图 3A)。此外,滴灌与自然降雨苜蓿根际土壤之间的细菌丰度也存在显著差异,滴灌苜蓿根际土壤中变形菌门、酸杆菌门、硝化螺旋菌门、装甲菌门(Armatimonadetes)的丰度显著高于自然降雨根际土壤,而放线菌门和Berkelbacteria的丰度则显著低于自然降雨根际土壤(图 3B)。
|
| 图 3 土壤样品中细菌群落结构在门水平的差异分析 Figure 3 Difference analysis of bacterial community structure at phylum level in soil samples 注:A:滴灌苜蓿根际vs非根际土壤;B:滴灌苜蓿根际土壤vs自然降雨苜蓿根际土壤. Note: A: Drip irrigation rhizosphere vs Drip irrigation non-rhizosphere; B: Drip irrigation rhizosphere vs Natural irrigation rhizosphere. |
|
|
属水平的细菌多样性丰度图(图 4)显示,两种生长模式下苜蓿土壤中菌属的丰度差异较大。MR土壤中的13种菌属:Iamia、鞘氨醇单胞菌属、新鞘脂菌属(Novosphingobium)、Skermanella、Flavisolibacter、蛭弧菌属(Bdellovibrio)、节杆菌属、Devosia、Terrimonas、Adhaeribacter、黄杆菌属(Flavobacterium)、足杆菌属(Pedobacter)和马赛菌属的丰度在MB土壤细菌群落中显著降低,甚至消失;而假单胞菌属(Pseudomonas)、溶杆菌属、Dongia这3种低丰度属在MB中则显著升高。对滴灌与自然降雨苜蓿根际土壤中细菌群落丰度比较时,发现MR土壤中13种菌属的丰度在NR土壤中同样表现出显著降低甚至消失的趋势;而交替赤杆菌属(Altererythrobacter)、芽单胞菌属(Gemmatimonas)、链霉菌属3种低丰度属则显著升高。对土壤样品特有属分析发现,Terrimonas、Adhaeribacter、假单胞菌属、溶杆菌属是滴灌模式下苜蓿土壤中的特有属。

|
| 图 4 土壤样品细菌菌落聚类 Figure 4 Clustering of bacterial colonies in soil samples |
|
|
通过对土壤细菌多样性指数与环境因子之间相关性分析(表 3),Shannon多样性指数与AN、OM、S-UE呈显著正相关,与S-NP呈极显著正相关;丰富度指数(Chao1、ACE)与土壤含水率呈正相关关系,与其他各环境因子间的相关性均未达到显著水平。说明土壤细菌多样性主要受土壤AN、OM、S-UE、S-NP以及土壤含水率的影响。
| Diversity index | TN | TP | TK | AN | AP | OM | pH | SM | S-NP | S-UE |
| Shannon index | 0.342 | 0.517 | 0.163 | 0.676* | 0.520 | 0.604* | −0.467 | 0.026 | 0.747** | 0.667* |
| Simpson index | 0.303 | −0.118 | −0.05 | 0.424 | −0.038 | 0.091 | −0.178 | −0.028 | 0.494 | 0.138 |
| ACE index | −0.103 | 0.022 | −0.147 | −0.134 | −0.175 | 0.007 | −0.036 | 0.641* | 0.077 | −0.029 |
| Chao1 index | −0.133 | −0.012 | −0.254 | −0.145 | −0.256 | −0.049 | 0.012 | 0.632* | 0.062 | −0.077 |
| 注:*:相关达显著水平(P<0.05);**:相关达极显著水平(P<0.01). Note: *: Significant correlation (P<0.05); **: Extremely significant correlation (P<0.01). |
||||||||||
用RDA分析细菌主要群落与土壤环境因子之间的关系,如图 5所示,Gaiella、鞘氨醇单胞菌属、节杆菌属、马赛菌属、足杆菌属、Adhaeribacter、Devosia、Skermanella、黄杆菌属9个属群落丰度与TP、AP、AN、OM、TK、S-NP、S-UE含量显著正相关,而与pH显著负相关;伍斯菌属(Woeseia)和Pelagibius属群落丰度与TP、AP、AN、OM、TK、S-NP、S-UE含量显著负相关,而与pH呈显著正相关关系;此外,土壤含水率与拟杆菌属(Bacteroides)、芽孢杆菌属(Bacillus)、Haliangium、Dongia、Lysobacter呈正相关,而与芽单胞菌属、类诺卡氏属群落丰度呈负相关。

|
| 图 5 细菌群落与环境因子的RDA图 Figure 5 RDA ordination graph for the bacterial community and environmental factors |
|
|
高通量测序技术能够较全面真实地揭示细菌菌落结构特征,本研究在两种生长模式下的土壤中共鉴定出细菌46个门,其中变形菌门、放线菌门、拟杆菌门、芽单胞菌门、酸杆菌门、绿弯菌门在两种施水方式的苜蓿土壤中均是丰度相对较高的菌门(>5%)。变形菌门为最优势细菌类群,且研究区域的农田土壤属于碱性土壤,推测变形菌门是碱性土壤中的主要优势群落,这与Singh等[14]、王鹏等[15]结果相似。同样,土壤中共检测出细菌53个纲,α-变形菌纲和拟杆菌纲在两种灌溉苜蓿土壤中均普遍存在且丰度最高,这些结果说明尽管施水方式不同,但土壤微生物类群具有相似性。
变形菌门、放线菌门是滴灌苜蓿根际土壤中丰度最高优势类群。其中变形菌门在很多土壤细菌群落研究中均为最优势细菌类群[16-19],主要包括α-变形菌纲、δ-变形菌纲以及γ-变形菌纲。目前已有研究报道了16S rRNA基因内部基因组异质性的广泛存在,16S rRNA基因往往同时存在多个拷贝,因此对菌群多样性分析会引起一定程度的高估[20-21],Sun等[22]对16S rRNA基因的拷贝数及基因组内部异质性的研究发现,最优势细菌类群变形菌门的平均拷贝数为3.94±2.62,对该门进一步研究表明,γ-变形菌纲(5.72±2.86)的拷贝数明显高于其他变形菌(少于3个拷贝),因此具有更多16S rRNA基因拷贝的γ-变形菌纲也存在着更高程度的过高估计。在本研究中,α-变形菌纲是最优势细菌纲,考虑到单个基因组内16S rRNA基因多样性的影响,相较于自然降雨,滴灌作用显著提高了苜蓿根际土壤中α-变形菌纲和δ-变形菌纲的丰度。α-变形菌纲中含有大量能与植物共生的固氮细菌[23-24];δ-变形菌纲对农田土壤中氮、磷、硫和有机质循环都有显著的促进作用[24-25]。苜蓿作为富含蛋白质的豆科牧草,根瘤结构以及土壤养分可以显著提高生物量的积累,滴灌作用对此过程存在着正效作用。本研究中,放线菌门是仅次于变形菌门的另一优势类群,而且放线菌在纲、目以及属水平均占据着优势地位。放线菌能产生大量种类繁多的抗生素,表明农田土壤中储存着广泛的抗生素产生菌资源,推测研究区干旱、高温等环境条件有利于放线菌的生长繁殖。此外,有研究表明酸杆菌门多出现在陆地酸性较高的土壤中[26],本研究中滴灌模式下土壤样品中酸杆菌门细菌丰度显著高于自然降雨灌溉土壤,且滴灌模式下苜蓿土壤的pH值显著降低,Sun等[22]的研究发现酸杆菌门的拷贝数仅为1.3±0.4,被高估的可能性较低,表明滴灌作用使得土壤环境存在酸化的倾向,具有缓解土壤盐碱性的可能。
在属水平上除少数常见菌属外,稀有菌属占全部菌属的80.00%−85.43%,反映出土壤中依然存在着大量未被挖掘利用的微生物资源。鞘氨醇单胞菌属为已分类的最优势属,且在滴灌苜蓿根际土壤中的丰度显著高于自然降雨(P<0.05),鞘氨醇单胞菌属已被证明能够分泌过氧化氢酶,提高植物抗逆性,被视为植物益生菌[27]。本研究中,Terrimonas、Adhaeribacter、假单胞菌属、溶杆菌属是滴灌模式下苜蓿土壤中的特有属。其中Terrimonas、Adhaeribacter分别属于拟杆菌门中噬胞菌目和噬纤维菌目,主要促进纤维素的降解;假单胞菌属为变形菌门中假单胞菌目细菌,具有极强分解有机物的能力,在土壤中广泛分布;溶杆菌属属于变形菌门的黄单胞菌目,对土壤中多种病原真菌、细菌和线虫都有突出的拮抗作用[28]。相较于自然降雨灌溉,滴灌技术带来的4种特有属的细菌,提高了对土壤中有机物的分解以及对土壤病害的抵御,对植物的生长发育有不可忽视的作用。对于新疆而言,大部分土地为盐碱土壤,滴灌技术的使用能逐渐改善土壤质量以及改变土壤中微生物群落的结构和组成。
3.2 两种灌溉模式下土壤细菌多样性微生物群落多样性是描述土壤生态特征的关键指标,微生物多样性的升高有助于提高土壤生态系统的稳定性[29]。而细菌是土壤微生物中数量丰富、分布广泛的微生物类群,占土壤微生物总量的70%−90%左右,对土壤养分循环有着重要作用[30]。本研究发现,滴灌苜蓿根际以及非根际土壤的Shannon指数和Chao1指数均高于自然降雨的根际与非根际,且Shannon指数达到显著水平。这说明,与自然降雨相比,滴灌技术的使用增加了土壤中细菌群落多样性及丰度,提高了土壤生态系统的稳定性。此外,土壤微生物多样性也反映了土壤肥力,细菌多样性越高,土壤肥力越丰富[31]。从土壤细菌多样性的研究结果来看,滴灌苜蓿土壤的肥力显著高于自然灌溉土壤,这与土壤养分含量测定的结果相同。
3.3 土壤环境因子对细菌群落结构的影响不同程度的人类活动对于土壤细菌群落结构具有不同的影响。4组土壤样品细菌菌落聚类分析表明,滴灌条件下苜蓿根际土壤中的细菌菌属丰度较高(图 4)。相较于自然灌溉,人工滴灌显著改变了土壤微环境,在土壤性质方面,滴灌苜蓿根际土壤SM、OM、TP、TK、AN、AP、S-UE和S-NP含量均显著高于自然降雨,有研究表明土壤优势细菌类群相对丰度与土壤理化性质有一定的相关性[32-34],这些环境因素极大影响了苜蓿土壤中细菌群落结构。用RDA分析菌属与环境因子的相关性发现,滴灌苜蓿根际土壤内Gaiella、鞘氨醇单胞菌属、节杆菌属、马赛菌属、足杆菌属、Adhaeribacter、Devosia、Skermanella、黄杆菌属共9个细菌属群落丰度与TP、AP、AN、OM、TK、S-NP、S-UE含量显著正相关。目前的研究[35-36]报道节杆菌属、Devosia、Skermanella、Adhaeribacter、黄杆菌属、马赛菌属、鞘氨醇单胞菌属和足杆菌属的细菌,与磷代谢、氮代谢、糖和蛋白质代谢、纤维素降解以及提高植物抗逆性等密切相关,且对土壤养分有较好的适应性。因此滴灌模式下,伴随着土壤中氮、磷等养分含量增加,节杆菌属、Devosia、Skermanella、Adhaeribacter、黄杆菌属、马赛菌属、鞘氨醇单胞菌属和足杆菌属的丰度明显增高。
综上,通过对两种生长方式的苜蓿土壤细菌多样性的比较研究发现,滴灌作为新型的节水灌溉技术在促进植物生长的同时,提高了植物抗逆性以及根际土壤微生物多样性与丰度,并能逐渐改善土壤质量。目前滴灌技术已在我国西北大部分地区广泛使用,而微生物多样性除了与土壤性质有关外,也受到植物根系深度、根际分泌物的种类与数量、植物生长年限以及细菌单个基因组内16S rRNA基因多样性等复杂因素的影响,因此,为了深层次揭示滴灌苜蓿土壤细菌群落多样性特征以及与环境因子的相关性,后续还需进一步深入研究。
| [1] |
Liu YN, Shi YH, Yan XB, et al. Effect of alfalfa hay substituting for part of the concentrate, on the production of cows and economic profit[J]. Acta Prataculturae Sinica, 2013, 22(6): 190-197. (in Chinese) 刘艳娜, 史莹华, 严学兵, 等. 苜蓿青干草替代部分精料对奶牛生产性能及经济效益的影响[J]. 草业学报, 2013, 22(6): 190-197. |
| [2] |
Meng JM, Li WJ. Effects of subsurface drip irrigation on alfalfa growth and seed production[J]. Acta Prataculturae Sinica, 2013, 21(1): 291-295. (in Chinese) 孟季蒙, 李卫军. 地下滴灌对苜蓿的生长发育与种子产量的影响[J]. 草业学报, 2013, 21(1): 291-295. |
| [3] |
Burns KN, Kluepfel DA, Strauss SL, et al. Vineyard soil bacterial diversity and composition revealed by 16S rRNA genes: Differentiation by geographic features[J]. Soil Biology and Biochemistry, 2015, 91: 232-247. DOI:10.1016/j.soilbio.2015.09.002 |
| [4] |
Zhang W, Wei HL, Gao HW, et al. Advances of studies on soil microbial diversity and environmental impact factors[J]. Chinese Journal of Ecology, 2005, 24(1): 48-52. (in Chinese) 张薇, 魏海雷, 高洪文, 等. 土壤微生物多样性及其环境影响因子研究进展[J]. 生态学杂志, 2005, 24(1): 48-52. |
| [5] |
Zhang L, Xu HM, Zhu BL. Association of rhizosphere soil microbiome with the occurrence and development of replant disease-A review[J]. Acta Microbiologica Sinica, 2016, 56(8): 1234-1241. (in Chinese) 张蕾, 徐慧敏, 朱宝利. 根际微生物与植物再植病的发生发展关系[J]. 微生物学报, 2016, 56(8): 1234-1241. |
| [6] |
You J, Wu G, Ren FP, et al. Microbial community dynamics in Baolige oilfield during MEOR treatment, revealed by Illumina MiSeq sequencing[J]. Applied Microbiology and Biotechnology, 2016, 100(3): 1469-1478. DOI:10.1007/s00253-015-7073-4 |
| [7] |
Liu Q, Zhao BX, Li RW, et al. Bacterial community structure in different functional sea areas of Zhoushan archipelago[J]. Acta Microbiologica Sinica, 2018, 58(2): 247-263. (in Chinese) 刘强, 赵本宣, 李汝伟, 等. 舟山群岛不同功能区划海域细菌群落结构分析[J]. 微生物学报, 2018, 58(2): 247-263. |
| [8] |
Grayston SJ, Wang SQ, Campbell CD, et al. Selective influence of plant species on microbial diversity in the rhizosphere[J]. Soil Biology and Biochemistry, 1998, 30(3): 369-378. DOI:10.1016/S0038-0717(97)00124-7 |
| [9] |
Bao SD. Soil and Agricultural Chemistry Analysis[M]. 3rd ed. Beijing: China Agriculture Press, 2000: 1-114. (in Chinese) 鲍士旦. 土壤农化分析[M]. 3版. 北京: 中国农业出版社, 2000: 1-114. |
| [10] |
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1): 10-12. DOI:10.14806/ej.17.1.200 |
| [11] |
Haas BJ, Gevers D, Earl AM, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [12] |
Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [13] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D596. |
| [14] |
Singh D, Shi HL, Adams JM. Bacterial diversity in the mountains of south-west China: climate dominates over soil parameters[J]. Journal of Microbiology, 2013, 51(4): 439-447. DOI:10.1007/s12275-013-2446-9 |
| [15] |
Wang P, Chen B, Zhang H. High throughput sequencing analysis of bacterial communities in soils of a typical Poyang Lake wetland[J]. Acta Ecologica Sinica, 2017, 37(5): 1650-1658. (in Chinese) 王鹏, 陈波, 张华. 基于高通量测序的鄱阳湖典型湿地土壤细菌群落特征分析[J]. 生态学报, 2017, 37(5): 1650-1658. |
| [16] |
Teng QH, Cao H, Cui ZL, et al. PCR-RFLP analysis of bacterial 16S rDNA from a typical garden soil in Taihu region[J]. Biodiversity Science, 2006, 14(4): 345-351. (in Chinese) 滕齐辉, 曹慧, 崔中利, 等. 太湖地区典型菜地土壤微生物16S rDNA的PCR-RFLP分析[J]. 生物多样性, 2006, 14(4): 345-351. DOI:10.3321/j.issn:1005-0094.2006.04.010 |
| [17] |
Nacke H, Thürmer A, Wollherr A, et al. Pyrosequencing-based assessment of bacterial community structure along different management types in German forest and grassland soils[J]. PLoS One, 2011, 6(2): e17000. DOI:10.1371/journal.pone.0017000 |
| [18] |
Zhao AH, Du XJ, Zang J, et al. Soil bacterial diversity in the Baotianman deciduous broad-leaved forest[J]. Biodiversity Science, 2015, 23(5): 649-657. (in Chinese) 赵爱花, 杜晓军, 臧婧, 等. 宝天曼落叶阔叶林土壤细菌多样性[J]. 生物多样性, 2015, 23(5): 649-657. |
| [19] |
Kou WB, Huang ZY, Zhang J, et al. Bacterial community structure and composition in Lake Poyang: a case study in the Songmenshan Region, China[J]. Acta Ecologica Sinica, 2015, 35(23): 7608-7614. (in Chinese) 寇文伯, 黄正云, 张杰, 等. 鄱阳湖湖泊细菌群落组成及结构—以松门山为例[J]. 生态学报, 2015, 35(23): 7608-7614. |
| [20] |
Pei AY, Oberdorf WE, Nossa CW, et al. Diversity of 16S rRNA genes within individual prokaryotic genomes[J]. Applied and Environmental Microbiology, 2010, 76(12): 3886-3897. DOI:10.1128/AEM.02953-09 |
| [21] |
Case RJ, Boucher Y, Dahllöf I, et al. Use of 16S rRNA and rpoB genes as molecular markers for microbial ecology studies[J]. Applied and Environmental Microbiology, 2007, 73(1): 278-288. DOI:10.1128/AEM.01177-06 |
| [22] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 9562-9569. |
| [23] |
Dedysh SN, Ricke P, Liesack W. NifH and NifD phylogenies: an evolutionary basis for understanding nitrogen fixation capabilities of methanotrophic bacteria[J]. Microbiology, 2004, 150: 1301-1313. DOI:10.1099/mic.0.26585-0 |
| [24] |
Madigan MT, Martinko JM, Parker J. Brock's Biology of Microorganisms[M]. 8th ed. Upper Saddle River, NJ: Prentice Hall, 1997.
|
| [25] |
Lv XF, Yu JB, Fu YQ, et al. A Meta-Analysis of the bacterial and archaeal diversity observed in wetland soils[J]. The Scientific World Journal, 2014, 2014: 437684. |
| [26] |
Barns SM, Cain EC, Sommerville L, et al. Acidobacteria phyium sediments in uranium-contaminated subsurface sediments greatly expand the known diversity within the phyium[J]. Applied and Environmental Microbiology, 2007, 73(9): 3113-3116. DOI:10.1128/AEM.02012-06 |
| [27] |
Chen DM, Ke WH, Chen LL, et al. Diversity of bacterial community in rhizosphere soils under effects of continuously planting burley tobacco[J]. Chinese Journal of Applied Ecology, 2010, 21(7): 1751-1758. (in Chinese) 陈冬梅, 柯文辉, 陈兰兰, 等. 连作对白肋烟根际土壤细菌群落多样性的影响[J]. 应用生态学报, 2010, 21(7): 1751-1758. |
| [28] |
Jiang YH, Hu BS, Liu FQ. Selection and identification of antagonistic bacteria against soil-borne plant pathogens[J]. Chinese Journal of Biological Control, 2005, 21(4): 260-264. (in Chinese) 姜英华, 胡白石, 刘凤权. 植物土传病原菌拮抗细菌的筛选与鉴定[J]. 中国生物防治, 2005, 21(4): 260-264. |
| [29] |
Liu BR, Zhang XZ, Hu TH, et al. Soil microbial diversity under typical vegetation zones along an elevation gradient in Helan Mountains[J]. Acta Ecologica Sinica, 2013, 33(22): 7211-7220. (in Chinese) 刘秉儒, 张秀珍, 胡天华, 等. 贺兰山不同海拔典型植被带土壤微生物多样性[J]. 生态学报, 2013, 33(22): 7211-7220. |
| [30] |
Bardgett RD, Freeman C, Ostle NJ. Microbial contributions to climate change through carbon cycle feedbacks[J]. The ISME Journal, 2008, 2(8): 805-814. DOI:10.1038/ismej.2008.58 |
| [31] |
Jiang J, Song MH. Review of the roles of plants and soil microorganisms in regulating ecosystem nutrient cycling[J]. Chinese Journal of Plant Ecology, 2010, 34(8): 979-988. (in Chinese) 蒋婧, 宋明华. 植物与土壤微生物在调控生态系统养分循环中的作用[J]. 植物生态学报, 2010, 34(8): 979-988. DOI:10.3773/j.issn.1005-264x.2010.08.011 |
| [32] |
Zhu D, Zhang L, Wei ZX, et al. Effects of bacterial manure on soil physicochemical properties and microbial community diversity in rhizosphere of highland barley[J]. Acta Pedologica Sinica, 2014, 51(3): 627-635. (in Chinese) 朱丹, 张磊, 韦泽秀, 等. 菌肥对青稞根际土壤理化性质以及微生物群落的影响[J]. 土壤学报, 2014, 51(3): 627-635. |
| [33] |
Lauber CL, Hamady M, Knight R, et al. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J]. Applied and Environmental Microbiology, 2009, 75(15): 5111-5120. DOI:10.1128/AEM.00335-09 |
| [34] |
Rousk J, Bååth E, Brookes PC, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. The ISME Journal, 2010, 4(10): 1340-1351. DOI:10.1038/ismej.2010.58 |
| [35] |
Tang J, Xu XR, Shang CY, et al. Association of bacterial diversity in city area of Nanming river with environmental factors[J]. Acta Microbiologica Sinica, 2015, 55(8): 1050-1059. (in Chinese) 唐婧, 徐小蓉, 商传禹, 等. 南明河城区河段细菌多样性与环境因子的关系[J]. 微生物学报, 2015, 55(8): 1050-1059. |
| [36] |
Liu L, Sun L, Zhang RY, et al. Diversity of IAA-producing endophytic bacteria isolated from the roots of Cymbidium goeringii[J]. Biodiversity Science, 2010(2): 182-187. (in Chinese) 刘琳, 孙磊, 张瑞英, 等. 春兰根中可分泌吲哚乙酸的内生细菌多样性[J]. 生物多样性, 2010(2): 182-187. |
 2019, Vol. 46
2019, Vol. 46