扩展功能
文章信息
- 李曼莉, 王利君, 赵亚超, 蒋昭芳, 周冬生, 童贻刚, 赵宝华
- LI Man-Li, WANG Li-Jun, ZHAO Ya-Chao, JIANG Zhao-Fang, ZHOU Dong-Sheng, TONG Yi-Gang, ZHAO Bao-Hua
- IncFII-FIA-FIB型多重耐药质粒pBTR-CTXM的结构基因组学分析
- Structural genomics of IncFII-FIA-FIB multidrug resistant plasmid pBTR-CTXM
- 微生物学通报, 2019, 46(1): 139-150
- Microbiology China, 2019, 46(1): 139-150
- DOI: 10.13344/j.microbiol.china.180431
-
文章历史
- 收稿日期: 2018-05-31
- 接受日期: 2018-07-27
- 网络首发日期: 2018-08-10
2. 军事科学院军事医学研究院微生物流行病研究所 病原微生物生物安全国家重点实验室 北京 100071;
3. 清华大学附属北京清华长庚医院 清华大学临床医学院 北京 102218
2. State Key Laboratory of Pathogen and Biosecurity, Beijing Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Academy of Military Sciences, Beijing 100071, China;
3. Department of Laboratory Medicine, Beijing Tsinghua Chang Gung Hospital, Tsinghua University, Beijing 102218, China
随着抗菌药物的广泛使用,细菌的多重耐药问题越来越严峻,严重威胁着细菌感染性疾病的治疗。大肠杆菌作为医源性感染的常见条件致病菌之一,可引起尿道感染、新生儿脑膜炎、败血症等疾病[1-2]。近年来,抗菌药物如第三代头孢菌素、喹诺酮类抗生素以及磺胺类药物已成为治疗大肠杆菌感染性疾病的主要选择[1]。在抗菌药物的选择压力下,大肠杆菌的耐药表型也呈现多样化的趋势。而细菌获得耐药性的主要途径——质粒介导的耐药基因的水平传播,不仅造成了耐药基因在同种属细菌内的积聚,还加剧了不同种属间耐药基因的扩散[3]。目前,大肠杆菌中已被发现有多种不相容群的质粒,如IncN、IncK、IncB/O、IncI1和IncF等[3-5]。
IncF型质粒是流行性耐药质粒,大小不一,宿主谱较窄,可以在大肠杆菌内稳定存在并广泛传播[6-7]。IncF质粒同时具备耐药特性和致病特性,通过携带抗生素抗性、毒素、重金属抗性和稀有物质代谢等相关基因,协助宿主抵抗外界不利因素,提高了细菌对周围环境的适应能力[7]。IncF型质粒分为6个亚群(IncFI、IncFII、IncFIII、IncFIV、IncFV和IncFVI),而且复制子类型多样。FII型复制子常与其他不相容群复制子存在于同一个质粒中,形成质粒多复制子结构。IncFII-FIA-FIB型质粒即是IncF型多复制子质粒,包括FII型复制子RepFII、FIA型复制子RepFIA和FIB型复制子RepFIB[8-9]。FII型复制子最早发现于质粒R100 (GenBank登录号NC_002134)中[10],FIA型复制子和FIB型复制子最早发现于F质粒(GenBank登录号AP001918)中[11]。当一个质粒中同时存在FII型复制子与FIA型或/和FIB型复制子时,FII型复制子并不发挥复制的功能[12]。
我们前期已对临床分离得到的一株多重耐药大肠杆菌BTR中IncN1型质粒pNDM-BTR进行了报道[13],阐明了质粒pNDM-BTR的结构特征。在此基础上,本文对大肠杆菌BTR中另一个质粒pBTR-CTXM进行了接合转移能力和介导耐药表型的鉴定、全序结构的精细注释和结构基因组学的分析,旨在进一步阐述大肠杆菌BTR的耐药机制,并加深对IncFII-FIA-FIB型质粒介导耐药基因水平传播机制的理解。
1 材料与方法 1.1 材料 1.1.1 菌株来源大肠杆菌BTR于2013年分离自北京同仁医院一名89岁慢性阻塞性肺疾病女性患者的尿液标本, 经VITEK 2 Compact全自动细菌鉴定及药敏分析仪和16S rRNA基因测序鉴定[14],菌株BTR为大肠杆菌;接合转移实验的受体菌是临床分离株大肠杆菌EC600,由解放军302医院惠赠;电转化实验的大肠杆菌DH5α感受态细胞购自TaKaRa公司。
1.1.2 主要试剂和仪器DNA Marker,北京全式金有限公司;BHI培养基,BD Biosciences公司;SOB培养基,北京鼎国昌盛生物公司;利福平,Sigma公司;High Pure PCR Template Preparation Kit,Roche公司;QIAGEN Plasmid Midi Kit,Qiagen公司;Wizard Genomic DNA Purification Kit,Promega公司;Nextera Mate Pair Sample Preparation Kit,Illumina公司。MiSeq测序仪,Illumina公司;PCR仪,ABI公司;MicroPulser电转化仪,Bio-Rad公司。
1.2 耐药基因筛查利用High Pure PCR Template Preparation Kit提取菌株BTR的基因组DNA,将其作为模板,通过PCR筛查碳青霉烯酶耐药基因[15]、超广谱β-内酰胺酶(Extended spectrum β-lactamase,ESBL)耐药基因[16]、喹诺酮类耐药基因[17]、大环内酯类耐药基因[18]和四环素类耐药基因[19]。PCR反应条件:95 ℃ 10 min;94 ℃ 30 s,相应退火温度30 s,72 ℃ 1 min,30个循环;72 ℃ 7 min。将扩增产物进行1%浓度的琼脂糖凝胶电泳,并利用一代测序验证扩增产物的序列。
1.3 接合转移实验依据pNDM-BTR质粒接合转移的实验方法[13],将菌株BTR (四环素耐药,20 μg/mL)作为供体菌,大肠杆菌EC600 (利福平耐药,2.5 mg/mL)作为受体菌,进行接合转移实验。混合37 ℃、220 r/min培养过夜的菌株BTR和受体菌EC600,5 000 r/min离心5 min后用80 μL的BHI液体培养基重悬,点加在贴于BHI平板的滤膜(1 cm2大小,0.45 μm孔径)上,37 ℃培养12-18 h。再用含2.5 mg/mL利福平和20 μg/mL四环素的BHI液体培养基洗脱菌体,涂布于含有2.5 mg/mL利福平和20 μg/mL四环素的BHI固体培养基上。37 ℃条件下培养24-48 h后,挑取单菌落,通过PCR (引物见表 1)和一代测序检测菌株BTR相应的耐药基因是否存在于接合子(BTR-CTXM-EC600)中。
| 引物 Primers name |
引物序列 Primers sequence (5′→3′) |
产物长度 Amplicon size (bp) |
退火温度 PCR annealing temperature (℃) |
| NDM-1-F | GGTTTGGCGATCTGGTTTTC | 621[15] | 56 |
| NDM-1-R | CGGAATGGCTCATCACGATC | ||
| CTX-M-15-F | AAAAATCACTGCGCCAGTTC | 415[16] | 52 |
| CTX-M-15-R | AGCTTATTCATCGCCACGTT | ||
| qnrS1-F | GCAAGTTCATTGAACAGGGT | 427[17] | 57 |
| qnrS1-R | TCTAAACCGTCGAGTTCGGCG | ||
| tetA(B)-F | CCTTATCATGCCAGTCTTGC | 774[19] | 57 |
| tetA(B)-R | ACTGCCGTTTTTTCGCC |
利用QIAGEN Plasmid Midi Kit提取菌株BTR中的质粒DNA,取大肠杆菌DH5α感受态细胞进行电击转化,之后向电击杯中迅速加入1 mL的SOB液体培养基,重悬细胞后转移到1.5 mL的离心管中。37 ℃、200 r/min复苏培养菌体2 h。将复苏后的菌体涂布到含四环素(20 μg/mL)的SOB固体培养基上,37 ℃培养24 h。挑取单菌落,利用PCR (引物见表 1)和一代测序检测电转子(BTR-CTXM-DH5α)中是否存在菌株BTR相应的耐药基因。
1.5 药敏实验用VITEK 2 Compact全自动细菌鉴定及药敏分析仪测定菌株BTR、接合子BTR-CTXM-EC600、受体菌EC600、电转子BTR-CTXM-DH5α和受体菌DH5α对抗生素的最小抑菌浓度(Minimal inhibitory concentration,MIC)。该试验结果按照美国临床与实验室标准协会(CLSI,2017)标准[20]判读。
1.6 全基因组测序及生物信息学分析利用Wizard Genomic DNA Purification Kit提取菌株BTR的基因组。构建Mate Pair文库,采用二代高通量测序平台进行全基因组测序。过滤低质量数据后,使用Newbler 3.0[21]软件进行序列拼接构建Contigs和Scaffolds,然后利用PCR和Sanger双脱氧链终止法测序技术对Contigs间的缺失部分进行补充,最终得到质粒的全序列。
质粒序列中的开放阅读框用RAST[22]进行预测,并通过BLASTN/BLASTP[23]与RefSeq[24]和Uni Prot-KB/Swiss-Prot[25]等数据库比对分析进行注释。利用ISFinder[26]、INTEGRALL[27]和Tn Number Registry[28]注释移动元件。使用Inkscape V0.48.1软件绘制质粒的结构图。
1.7 序列登录号将pBTR-CTXM全序列提交至GenBank,获得登录号为MF156697。
2 结果与分析 2.1 菌株耐药基因筛查PCR筛查耐药基因结果显示菌株BTR携带碳青霉烯酶基因blaNDM-1,超广谱β-内酰胺类耐药基因blaCTX-M-15、blaTEM,喹诺酮类耐药基因qnrD、qnrS1,大环内酯类耐药基因mph(A)、erm(B),以及四环素类耐药基因tetA(B)等。
2.2 接合转移和电转化实验多次接合转移实验发现耐药基因blaNDM-1和qnrS1与blaCTX-M-15和tetA(B)共转移到接合子BTR-CTXM-EC600中(图 1)。但前期分析已经证实耐药基因blaNDM-1和qnrS1由可自主接合转移的质粒pNDM-BTR携带,而且在接合子BTR-NDM- EC600[13]中PCR扩增blaCTX-M-15和tetA(B)为阴性。为检测耐药基因blaCTX-M-15和tetA(B)是否存于另一个质粒中,进行电转化实验,发现blaCTX-M-15和tetA(B)可转移到电转子BTR-CTXM-DH5α中,而且PCR扩增blaNDM-1和qnrS1为阴性(图 1)。将携带blaCTX-M-15和tetA(B)的质粒命名为pBTR-CTXM。

|
| 图 1 耐药基因blaNDM-1、blaCTX-M-15、qnrS1和tetA(B)的PCR扩增结果 Figure 1 PCR detection of blaNDM-1, blaCTX-M-15, qnrS1, and tetA(B) |
|
|
大肠杆菌BTR及相关菌株的MIC检测结果表明菌株BTR对硝基呋喃类以外的抗生素均耐药,而电转子BTR-CTXM-DH5α对头孢唑啉、头孢呋辛、头孢呋辛酯、头孢曲松、氨苄西林、哌拉西林、氨曲南、四环素和阿奇霉素耐药(表 2)。
| 分类 Category |
抗生素 Antibiotics |
BTR | BTR-CTXM-EC600 | BTR-CTXM-DH5α | EC600 | DH5α |
| 单环β-内酰胺类Monobactam | 氨曲南Aztreonam | ≥64/R | ≥64/R | ≥16/R | ≤1/S | ≤1/S |
| 四环素类Tetracycline | 四环素Tetracycline | ≥32/R | ≥32/R | ≥32/R | ≤1/S | ≤1/S |
| 大环内酯类Macrolide | 阿奇霉素Azithromycin | ≥64/R | ≥64/R | ≥64/R | ≤2/S | ≤2/S |
| 头孢菌素类Cephalosporins | 头孢唑啉Cefazolin | ≥64/R | ≥64/R | ≥64/R | ≤4/S | ≤4/S |
| 头孢呋辛Cefuroxime | ≥64/R | ≥64/R | ≥64/R | 16/I | 4/S | |
| 头孢呋辛酯Cefuroxime axetil | ≥64/R | ≥64/R | ≥64/R | 16/I | 4/S | |
| 头孢曲松Ceftriaxone | ≥64/R | ≥64/R | ≥64/R | ≤1/S | ≤1/S | |
| 头孢吡肟Cefepime | ≥64/R | ≥64/R | 2/I | ≤1/S | ≤1/S | |
| 头孢他啶Ceftazidime | ≥64/R | ≥64/R | 4/S | ≤1/S | ≤1/S | |
| 头孢替坦Cefotetan | ≥64/R | ≥64/R | ≤4/S | ≤4/S | ≤4/S | |
| 青霉素类Penicillins | 氨苄西林Ampicillin | ≥32/R | ≥32/R | ≥32/R | 16/I | ≤2/S |
| 哌拉西林Piperacillin | ≥128/R | ≥128/R | ≥128/R | ≤4/S | ≤4/S | |
| 氨苄西林/舒巴坦Ampicillin/Sulbactam | ≥32/R | ≥32/R | 4/S | 8/S | ≤2/S | |
| 哌拉西林/他唑巴坦Piperacillin/Tazobactam | ≥128/R | ≥128/R | ≤4/S | ≤4/S | ≤4/S | |
| 氨基糖苷类Aminoglycoside | 庆大霉素Gentamicin | ≥16/R | ≥16/R | 8/I | ≤1/S | ≤1/S |
| 碳青霉烯类Carbapenems | 亚胺培南Imipenem | ≥16/R | ≥16/R | ≤1/S | ≤1/S | ≤1/S |
| 美罗培南Meropenem | ≥16/R | 8/R | ≤0.25/S | ≤0.25/S | ≤0.25/S | |
| 叶酸抑制剂类Folate pathway inhibitor | 甲氧苄啶/磺胺甲噁Trimethoprim/Sulfamethoxazole | ≥320/R | ≥320/R | ≤20/S | ≤20/S | ≤20/S |
| 喹诺酮类Fluoroquinolone | 环丙沙星Ciprofloxacin | ≥64/R | 4/R | ≤0.25/S | ≤0.25/S | ≤0.25/S |
| 硝基呋喃类Nitrofuran | 呋喃妥因Nitrofurantoin | ≤16/S | ≤16/S | ≤16/S | ≤16/S | ≤16/S |
| 注:S:敏感;I:中介;R:耐药. Note: S: Susceptible; I: Intermediate; R: Resistant. | ||||||
pBTR-CTXM是全长144 939 bp的环状DNA分子,平均(G+C)mol%含量为51.85%,存在208个编码蛋白的开放阅读框(表 3)。pBTR-CTXM含有3个复制子RepFII、RepFIA和RepFIB,属于IncFII-FIA-FIB型质粒。纳入分析的质粒共3个:第一个完整测序的IncFII-FIA-FIB型质粒pRSB107[9] (GenBank登录号为AJ851089);pBTR-CTXM (本文);与pBTR-CTXM覆盖度、核酸一致性最高(86%+99%)的质粒pRSB225 (GenBank登录号JX127248)[29]。根据基因编码蛋白质的功能差异,这些质粒均可分为骨架区和外源插入区(图 2)。每个质粒的外源插入区均包括一个MDR区、ugp操纵子、iucABCD-iutA基因簇和一些分散的插入序列等(表 3)。
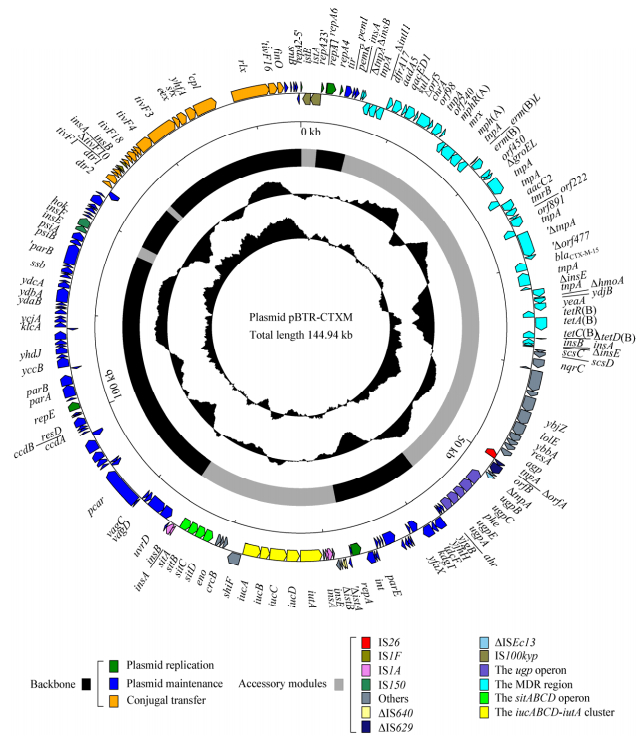
|
| 图 2 质粒pBTR-CTXM全序列示意图 Figure 2 Schematic map of pBTR-CTXM 注:基因按功能用不同颜色的箭头表示;图中最内环表示GC偏移量[(G–C)/(G+C)];次内环表示GC含量;质粒骨架区和外源插入区分别用黑色区和灰色区表示. Note: Genes are denoted by arrows and colored based on gene function classification. The innermost two circles present GC-skew [(G–C)/(G+C)] and GC contents. The backbone and accessory module regions are highlighted in black and gray, respectively. |
|
|
| 特征 Features |
质粒 Plasmids |
||
| pRSB107 | pBTR-CTXM | pRSB225 | |
| 全序长度Total length (bp) | 120 592 | 144 939 | 164 550 |
| 平均(G+C)mol%含量Mean (G+C)mol% content | 53.10 | 51.85 | 52.60 |
| 开放阅读框总数Total number of ORFs | 173 | 208 | 230 |
| 外源插入区Accessory modules | The MDR region | The MDR region | The MDR region |
| The ugp operon | The ugp operon | The ugp operon | |
| The iucABCD-iutA cluster | The iucABCD-iutA cluster | The iucABCD-iutA cluster | |
| IS26 | IS26 | IS26 | |
| IS1A | IS1A | IS1A | |
| ΔIS640 | ΔIS640 | ΔIS640 | |
| ΔISEc13 | ΔISEc13 | ΔISEc13 | |
| ISEc23 | IS1F | ISEc23 | |
| ΔIS1203 | ΔIS629 | ΔIS1203 | |
| IS150 | IS150 | ||
| The sitABCD operon | The sitABCD operon | ||
| IS100kyp | IS1X2 | ||
| ΔISKpn26 | |||
| 注:pBTR-CTXM为本文测序质粒,pRSB107和pRSB225来自于GenBank. Note: pBTR-CTXM was fully sequenced in this work, while pRSB107 and pRSB225 were derived from GenBank. | |||
通过比对pRSB107、pBTR-CTXM和pRSB225的骨架区,发现在 > 58%的覆盖度上存在高于99%的核酸一致性。3个质粒的骨架区主要包括3类复制基因(repA2-6-1-4、repE和repA)、质粒稳定相关基因(ccdAB、resD、parAB和pemIK等)和接合转移相关基因(traIO和finO等) (图 3)。

|
| 图 3 pRSB107、pBTR-CTXM和pRSB225全序线性结构比对图 Figure 3 Linear comparison of pRSB107, pBTR-CTXM and pRSB225 注:基因用箭头表示;基因、移动元件和其他区域按功能用不同颜色表示;阴影部分表示区域核苷酸一致性大于95%. Note: Genes are denoted by arrows. Genes, mobile elements, and other features are colored based on function classification. Shading denotes regions of homology (>95% nucleotide identity). |
|
|
在骨架区中,3个质粒之间的差异主要有:(1) pBTR-CTXM的FII型复制子中repA4基因与其它质粒的该基因一致性仅有91%。由于IS100kyp的插入,pBTR-CTXM的repA2基因被打断成两部分,即repA2-5′和repA2-3′ (图 3A)。(2) parC是parB的结合位点,在pRSB107和pRSB225中为432 bp,而pBTR-CTXM的parC只剩下302 bp (7个43–44 bp串联重复单元:GGGTCTGATTATTAGTCTGGGA CCACGGTCCCACTCGTATCGTC) (图 3B)。(3)与pRSB107相比,pBTR-CTXM和pRSB225在ydbA与dtr2之间插入了一段与质粒稳定相关的序列(图 3C)。(4) 3个质粒的接合转移区长度不同:pRSB107的接合转移区仅含有3个基因(rlx、tivF16和finO),而且不具备自主接合转移的能力[9];pRSB225的接合转移区由dtr1、dtr2、tivF1–tivF16、tivF18、tivF19、eex、sfx、yhfA、cpl、rlx和finO构成,可以进行自主接合转移[29];pBTR-CTXM的接合转移区由于IS1F的插入,导致tivF10基因被截短。与pRSB225相比,pBTR-CTXM缺少部分与Ⅳ型分泌系统相关的基因(tivF2、tivF5–tivF9、tivF11–tivF15和tivF19) (图 3D),可能是其不能自主接合转移的原因。
2.4.3 质粒MDR区分析pRSB107/pRSB225的MDR区(36 817 bp)由复合型转座子Tn6491 (本文新命名)、Tn10残余和IS1R组成。pBTR-CTXM的MDR区(32 439 bp)由新的复合型转座子Tn6492、Tn10残余、Tn2残余、ISEcp1-blaCTX-M-15-Δorf477转座单元和一些插入序列组成(图 4)。

|
| 图 4 pRSB107、pBTR-CTXM和pRSB225质粒MDR区线性比对图 Figure 4 The MDR region of pRSB107, pBTR-CTXM and pRSB225 注:基因用箭头表示;基因、移动元件和其他区域按功能用不同颜色表示;阴影部分表示区域核苷酸一致性大于95%. Note: Genes arc denoted by arrows. Gencs, mobile elements, and other features arc colored based on function classification. Shading regions denote shared DNA regions of homology (>95% nucleotide identity). |
|
|
Tn21是Tn3家族的转座子,结构为IRL (Inverted repeat left,左反向重复序列)-tnpA (Transposase,转座酶)-tnpR (Resolvase,解离酶)-res (Resolution site,重组位点)-tnpM (Modulator protein,调节蛋白)-In2-urf2-merEDACPTR (Mercuric resistance locus,汞耐药位点)-IRR (Inverted repeat right,右反向重复序列),两端分别有5 bp的正向重复序列(Direct repeat,DR)。Tn2670最初在R100中被发现,是由Tn21插入到一个Tn9-like转座子的IS1R与ybjA中间形成的复合型转座子,两端被9 bp的DRs所包围[30]。由于一系列的转座和IS26或IS6100介导的同源重组事件,Tn4352、Tn6029、转座单元IS26-mph(A)-mrx-mphR(A)-IS6100和In191替代了Tn2670中In2的tniA-3′-tniBD1-IS1353-IS1326-orf5-sul1-qacED1-aadA1的位置,形成了Tn6491。Tn4352是一个复合型转座子,以两个方向相同的IS26为基础,携带了aphA1a (Aminoglycoside resistance:氨基糖苷类耐药)基因。复合型转座子Tn6029是由IS26、Tn2和Tn5393c通过同源重组产生,包括sul2 (Sulphonamide resistance:磺胺类耐药)、strAB (Streptomycin resistance:链霉素耐药)和blaTEM-1 (Beta-lactamase resistance:β-内酰胺类耐药)耐药基因,以及3个IS26[31]。IS26-mph(A)-mrx-mphR(A)-IS6100转座单元作为典型的大环内酯转座单元[32],在Tn6491中为完整的结构。Tn402-associated 1型整合子原型的结构为IRi (Inverted repeat at the integrase end,位于整合酶末端的反向重复序列)、5′保守区(5′-Conserved segment,5′CS)的整合酶(Integrase,intI1)和重组位点(attI1)、基因盒阵列,3′保守区[3′-Conserved segment,3′CS:qacED1 (Quaternary ammonium compound resistance:季铵复合物耐药)、sul1、orf5和orf6]、Tn402的tni模块和IRt (Inverted repeat at the tni end,位于tni模块末端的反向重复序列),而且两端被5 bp的DRs所包围。In191是Tn402-associated 1型整合子原型的衍生物,结构为IRi-intI1-dfrA14 (Trimethoprim resistance:甲氧苄啶耐药)-mobC-IRt-IS6100,但与之相比丢失了3′保守区和tni模块。
Tn10是复合型转座子,首尾是方向相反的插入序列IS10,中间携带着tetD(B)、tetC(B)、tetA(B) (Tetracycline resistance:四环素类耐药)、tetR(B)、yeaA、ydjB、hmoA和ydhA,两端由9 bp的DRs包围[33]。在pRSB107和pRSB225中,因为Tn2670的插入,导致Tn10中左手端IS10、ydhA和hmoA-5′的缺失,并且IS1R插入到右手端IS10中,将insE打断成两部分,即insE-5′和insE-3′。在pBTR-CTXM中,Tn10只剩下tetD(B)-5′、tetC(B)、tetA(B)、tetR(B)、yeaA、ydjB和hmoA-3′。
Tn6492由IS26、In54、chrA (Chromate resistance:铬酸盐耐药基因)-orf98转座单元、大环内酯转座单元IS26-mph(A)-mrx-mphR(A)-IS6100、ΔTn6295和Tn6415组成,两端被8 bp的DRs包围。In54属于Tn402-associated 1型整合子,携带dfr17 (Trimethoprim resistance:甲氧苄啶耐药)和aadA5 (Aminoglycoside resistance:氨基糖苷类耐药)两个基因盒。chrA-orf98转座单元可能来源于pCNB1 (GenBank登录号为EF079106)中一个Tn21-like的转座子,经常和IRt-IS6100紧密相连[32]。Tn6295是由首尾为同向的IS26、大环内酯操纵子erm(B)L-erm(B)、orf450、ΔgroEL和ISCR3组成的复合型转座子。Tn6415最早发现于大肠杆菌AR_0119中的质粒Unitig_2 (GenBank登录号为CP021536)中,整体结构为IS26-aacC2 (Aminoglycoside resistance:氨基糖苷类耐药)-tmrB-orf222-orf891-IS26,两端由8 bp的DRs包围。在Tn6492中,In54的整合酶和orf5被截短且丢失了orf6和tni模块,其下游的chrA-orf98转座单元结构完整,而且紧连完整的大环内酯转座单元IS26-mph(A)-mrx-mphR(A)-IS6100。Tn6295与大环内酯转座单元IS26-mph(A)-mrx-mphR(A)-IS6100发生了IS26介导的同源重组事件,而且Tn6295中ISCR3的oriIS和右手端的IS26被下游结构完整的Tn6415截短。
ISEcp1-blaCTX-M-15-Δorf477转座单元是常见介导blaCTX-M-15 (Beta-lactamase resistance:β-内酰胺类耐药)转移的移动元件,两端被5 bp的DRs所包围。ISEcp1是单末端插入序列,可以仅靠单个插入序列识别其自身的反向重复序列(IR:Inverted repeat)以及与IRR-1相似度较高的IRR-2,介导邻近耐药基因的转移[34]。Tn2是Tn3家族转座子,基本组成为IRL-tnpA-res-tnpR-blaTEM-1-IRR,两端被5 bp的DRs所包围[35]。在pBTR-CTXM中,Tn2被ISEcp1-blaCTX-M-15-Δorf477转座单元截短,剩余2 838 bp (只包括IRL和ΔtnpA),而且由于Tn6492的插入,ΔtnpA被截成两部分。
3 讨论与结论质粒pBTR-CTXM通过携带blaCTX-M-15、mph(A)、erm(B)和tetA(B)等耐药基因,介导了大肠杆菌BTR和电转子BTR-CTXM-DH5α对单环β-内酰胺类、部分头孢菌素类、青霉素类(不含酶抑制剂)、大环内酯类和四环素类等抗生素的耐药性。pBTR-CTXM具有IncFII-FIA-FIB型质粒典型的骨架区结构,可能由于接合转移区缺少部分基因,需要在另一个质粒pNDM-BTR的协助下共转移到受体菌EC600中,但可通过电转化进入受体菌大肠杆菌DH5α中,属于可移动质粒。pBTR-CTXM的MDR区是由许多不同移动元件组成的复杂的“马赛克”结构,包含了pBTR-CTXM的全部耐药基因。IS26、In54、chrA-orf98转座单元、IS26-mph(A)-mrx-mphR(A)-IS6100转座单元、ΔTn6295和Tn6415通过复杂的转座和同源重组事件形成了一个新的复合型转座子Tn6492。pRSB107、pBTR-CTXM和pRSB225的骨架区中存在两个插入“热点”,即pemK和ahr之间与orf249和orf165之间(图 3),可整合不同的外源序列,尤其是含有耐药基因的移动元件,从而促进了耐药基因在不同种属细菌间的水平传播。
本文通过对IncFII-FIA-FIB型多重耐药质粒pBTR-CTXM进行结构基因组学分析,进一步明确了大肠杆菌BTR对单环β-内酰胺类、大环内酯类和四环素类等多种抗生素的耐药机制,加深了对IncFII-FIA-FIB型质粒介导的耐药基因水平传播机制的理解,并为感染性疾病的规范化用药和研制新型抗菌药物提供了理论依据。
| [1] |
Mellata M. Human and avian extraintestinal pathogenic Escherichia coli: infections, zoonotic risks, and antibiotic resistance trends[J]. Foodborne Pathogens and Disease, 2013, 10(11): 916-932. DOI:10.1089/fpd.2013.1533 |
| [2] |
Liu CY. Comparative genomics analysis and study on subunit vaccine of porcine extraintestinal pathogenic Escherichia coli[D]. Wuhan: Doctoral Dissertation of Huazhong Agricultural University, 2014 (in Chinese) 刘璨颖.猪源肠外致病性大肠杆菌比较基因组学和亚单位疫苗的研究[D].武汉: 华中农业大学博士学位论文, 2014 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2565916 |
| [3] |
Bengtsson S, Naseer U, Sundsfjord A, et al. Sequence types and plasmid carriage of uropathogenic Escherichia coli devoid of phenotypically detectable resistance[J]. Journal of Antimicrobial Chemotherapy, 2012, 67(1): 69-73. |
| [4] |
Hammerum AM, Larsen J, Andersen VD, et al. Characterization of extended-spectrum β-lactamase (ESBL)-producing Escherichia coli obtained from danish pigs, pig farmers and their families from farms with high or no consumption of third- or fourth-generation cephalosporins[J]. Journal of Antimicrobial Chemotherapy, 2014, 69(10): 2650-2657. |
| [5] |
Duan SS. The dissemination mechanism and genetic characteristics of CTX-M clusters among Avian Escherichia coli[D]. Zhengzhou: Master's Thesis of Henan Agricultural University, 2015 (in Chinese) 段沙沙.禽大肠埃希菌不同CTX-M亚群的散播机制及遗传特性[D].郑州: 河南农业大学硕士学位论文, 2015 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2939129 |
| [6] |
Marcadé G, Deschamps C, Boyd A, et al. Replicon typing of plasmids in Escherichia coli producing extended-spectrum β-lactamases[J]. Journal of Antimicrobial Chemotherapy, 2009, 63(1): 67-71. DOI:10.1093/jac/dkn428 |
| [7] |
Villa L, García-Fernández A, Fortini D, et al. Replicon sequence typing of IncF plasmids carrying virulence and resistance determinants[J]. Journal of Antimicrobial Chemotherapy, 2010, 65(12): 2518-2529. DOI:10.1093/jac/dkq347 |
| [8] |
Osborn AM, da Silva Tatley FM, Steyn LM, et al. Mosaic plasmids and mosaic replicons: evolutionary lessons from the analysis of genetic diversity in IncFII-related replicons[J]. Microbiology, 2000, 146(9): 2267-2275. |
| [9] |
Szczepanowski R, Braun S, Riedel V, et al. The 120 592 bp IncF plasmid pRSB107 isolated from a sewage-treatment plant encodes nine different antibiotic-resistance determinants, two iron-acquisition systems and other putative virulence-associated functions[J]. Microbiology, 2005, 151(4): 1095-1111. DOI:10.1099/mic.0.27773-0 |
| [10] |
Womble DD, Rownd RH. Genetic and physical map of plasmid NR1: comparison with other IncFII antibiotic resistance plasmids[J]. Microbiological Reviews, 1988, 52(4): 433-451. |
| [11] |
Cavalli LL, Lederberg J, Lederberg EM. An infective factor controlling sex compatibility in Bacterium coli[J]. Microbiology, 1953, 8(1): 89-103. DOI:10.1099/00221287-8-1-89 |
| [12] |
Zhan Z, Feng J, Yin Z, et al. Antibiotic-resistant mechanisms of plasmid pA1137 carrying aminoglycoside resistance gene aacC2[J]. Military Medical Sciences, 2017, 41(12): 973-977. (in Chinese) 占喆, 冯娇, 殷喆, 等. 携带氨基糖苷类耐药基因aacC2的质粒pA1137耐药机制研究[J]. 军事医学, 2017, 41(12): 973-977. |
| [13] |
Zhao YC, Wang LJ, Zhang ZY, et al. Structural genomics of pNDM-BTR harboring In191 and Tn6360, and other blaNDM-carrying IncN1 plasmids[J]. Future Microbiology, 2017, 12(14): 1271-1281. DOI:10.2217/fmb-2017-0067 |
| [14] |
Frank JA, Reich CI, Sharma S, et al. Critical evaluation of two primers commonly used for amplification of bacterial 16S rRNA genes[J]. Applied and Environmental Microbiology, 2008, 74(8): 2461-2470. DOI:10.1128/AEM.02272-07 |
| [15] |
Poirel L, Walsh TR, Cuvillier V, et al. Multiplex PCR for detection of acquired carbapenemase genes[J]. Diagnostic Microbiology and Infectious Disease, 2011, 70(1): 119-123. DOI:10.1016/j.diagmicrobio.2010.12.002 |
| [16] |
Dallenne C, Da Costa A, Decré D, et al. Development of a set of multiplex PCR assays for the detection of genes encoding important β-lactamases in Enterobacteriaceae[J]. Journal of Antimicrobial Chemotherapy, 2010, 65(3): 490-495. DOI:10.1093/jac/dkp498 |
| [17] |
Ciesielczuk H, Hornsey M, Choi V, et al. Development and evaluation of a multiplex PCR for eight plasmid-mediated quinolone-resistance determinants[J]. Journal of Medical Microbiology, 2013, 62(12): 1823-1827. |
| [18] |
Phuc Nguyen MC, Woerther PL, Bouvet M, et al. Escherichia coli as reservoir for macrolide resistance genes[J]. Emerging Infectious Diseases, 2009, 15(10): 1648-1650. DOI:10.3201/eid1510.090696 |
| [19] |
Maynard C, Fairbrother JM, Bekal S, et al. Antimicrobial resistance genes in enterotoxigenic Escherichia coli O149:K91 isolates obtained over a 23-year period from pigs[J]. Antimicrobial Agents and Chemotherapy, 2003, 47(10): 3214-3221. DOI:10.1128/AAC.47.10.3214-3221.2003 |
| [20] |
Clinical and Laboratory Standards Institute. M100-S27 Performance standards for antimicrobial susceptibility testing: twenty-seventh informational supplement[Z]. Wayne, PA, USA: CLSI, 2017
|
| [21] |
Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376-380. DOI:10.1038/nature03959 |
| [22] |
Aziz RK, Bartels D, Best AA, et al. The RAST server: rapid annotations using subsystems technology[J]. BMC Genomics, 2008, 9(1): 75. DOI:10.1186/1471-2164-9-75 |
| [23] |
Boratyn GM, Camacho C, Cooper PS, et al. BLAST: a more efficient report with usability improvements[J]. Nucleic Acids Research, 2013, 41: W29-W33. DOI:10.1093/nar/gkt282 |
| [24] |
O'Leary NA, Wright MW, Brister JR, et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation[J]. Nucleic Acids Research, 2016, 44(D1): D733-D745. DOI:10.1093/nar/gkv1189 |
| [25] |
Boutet E, Lieberherr D, Tognolli M, et al. UniProtKB/Swiss-Prot, the manually annotated section of the UniProt KnowledgeBase: how to use the entry view[A]//Plant Bioinformatics. Methods in Molecular Biology[M]. New York: Humana Press, 2016: 23-54
|
| [26] |
Siguier P, Perochon J, Lestrade L, et al. ISfinder: the reference centre for bacterial insertion sequences[J]. Nucleic Acids Research, 2006, 34: D32-D36. DOI:10.1093/nar/gkj014 |
| [27] |
Moura A, Soares M, Pereira C, et al. INTEGRALL: a database and search engine for integrons, integrases and gene cassettes[J]. Bioinformatics, 2009, 25(8): 1096-1098. DOI:10.1093/bioinformatics/btp105 |
| [28] |
Roberts AP, Chandler M, Courvalin P, et al. Revised nomenclature for transposable genetic elements[J]. Plasmid, 2008, 60(3): 167-173. |
| [29] |
Wibberg D, Szczepanowski R, Eikmeyer F, et al. The IncF plasmid pRSB225 isolated from a municipal wastewater treatment plant's on-site preflooder combining antibiotic resistance and putative virulence functions is highly related to virulence plasmids identified in pathogenic E. coli isolates[J]. Plasmid, 2013, 69(2): 127-137. DOI:10.1016/j.plasmid.2012.11.001 |
| [30] |
Partridge SR, Hall RM. Complex multiple antibiotic and mercury resistance region derived from the r-det of NR1 (R100)[J]. Antimicrobial Agents and Chemotherapy, 2004, 48(11): 4250-4255. DOI:10.1128/AAC.48.11.4250-4255.2004 |
| [31] |
Cain AK, Liu XL, Djordjevic SP, et al. Transposons related to Tn1696 in IncHI2 plasmids in multiply antibiotic resistant Salmonella enterica serovar typhimurium from Australian animals[J]. Microbial Drug Resistance, 2010, 16(3): 197-202. DOI:10.1089/mdr.2010.0042 |
| [32] |
Chalmers R, Sewitz S, Lipkow K, et al. Complete nucleotide sequence of Tn10[J]. Journal of Bacteriology, 2000, 182(10): 2970-2972. DOI:10.1128/JB.182.10.2970-2972.2000 |
| [33] |
Partridge SR. Analysis of antibiotic resistance regions in Gram-negative bacteria[J]. FEMS Microbiology Reviews, 2011, 35(5): 820-855. |
| [34] |
Zong ZY, Partridge SR, Iredell JR. ISEcp1-mediated transposition and homologous recombination can explain the context of blaCTX-M-62 linked to qnrB2[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(7): 3039-3042. DOI:10.1128/AAC.00041-10 |
| [35] |
Bailey JK, Pinyon JL, Anantham S, et al. Distribution of the blaTEM gene and blaTEM-containing transposons in commensal Escherichia coli[J]. Journal of Antimicrobial Chemotherapy, 2011, 66(4): 745-751. DOI:10.1093/jac/dkq529 |
 2019, Vol. 46
2019, Vol. 46




