扩展功能
文章信息
- 马燕, 马爽, 尚春香, 格日力
- MA Yan, MA Shuang, SHANG Chun-Xiang, Gerili
- 低氧暴露对大鼠肠道微生物群落的影响
- Effects of hypoxic exposure on rats' gut microbiota
- 微生物学通报, 2019, 46(1): 120-129
- Microbiology China, 2019, 46(1): 120-129
- DOI: 10.13344/j.microbiol.china.180306
-
文章历史
- 收稿日期: 2018-04-16
- 接受日期: 2018-05-22
- 网络首发日期: 2018-06-06
2. 青海省高原医学应用基础重点实验室 青海 西宁 810001
2. Key Laboratory for Application of High Altitude Medicine in Qinghai Province, Xining, Qinghai 810001, China
高原环境是一个特殊而复杂的生态环境系统,具有高寒、低氧、强紫外线辐射等气候特点[1]。高原恶劣的自然环境会对人或动物的生存和繁衍产生诸多影响[2]。以往对高原习服和损伤的研究多集中在呼吸、循环和血液系统,对消化系统的关注较少。高原环境会导致胃肠消化功能障碍,食物摄入、吸收、转运都受到不同程度的影响。低海拔人群快速进入海拔3 000 m以上的高原后除了出现头昏、头痛、心慌、气促、失眠等症状外,消化系统症状发生率较高,而且随着海拔增高,消化系统症状发生率有不同程度的增加,尤其以恶心、呕吐、腹泻、食欲减退、体质量降低等症状最为突出[3]。
肠道微生物作为人或动物的“共生体”,辅助宿主进行营养吸收和能量代谢是其核心功能之一[4]。发酵、甲烷化和硫还原是肠道微生物进行物质代谢的主要方式[5],其以复杂碳水化合物和蛋白质为底物进行发酵,对维持宿主正常生理功能具有重要意义[6]。肠道微生物利用底物发酵后的主要代谢产物短链脂肪酸(Short chain fatty acid,SCFA)参与调节机体营养代谢、能量供应、肠道免疫、炎症反应,甚至对神经系统等诸多方面都有重要影响[7]。
以往就高原低氧对机体肠道微生物影响的研究较少。其中对人体的研究发现平原人暴露于3 505 m的高原地区15 d后,肠道中需氧菌的数量显著减少而兼性厌氧菌的数量明显增加[8]。将大鼠间歇性暴露于模拟海拔4 872.9 m的低氧环境30 d后发现了类似结果[9-10]。将大鼠急性暴露于高原低氧环境后会导致其肠道中双歧杆菌减少,肠道菌群失调,并且随着海拔的升高,肠道菌群的失调率更高[11-12]。然而目前对持续性低氧暴露过程中宿主肠道微生物群落的结构、多样性改变,以及这些变化与宿主习服低氧之间的关系尚未见报道。
本研究拟通过高通量测序技术和生物信息学方法,探讨低氧暴露过程中SD大鼠肠道微生物群落结构、多样性的变化,以及肠道微生物群落的变化与宿主代谢之间可能存在的关系。
1 材料与方法 1.1 实验动物及分组SPF (Specific pathogen free)级SD雄性大鼠40只,体重250-300 g,购于北京维通利华实验动物技术有限公司,合格证号:SCXK(京)2012-10001。按体重大小标号后由随机数字表将大鼠随机分组,分别为低氧暴露1 d组(H1D,5只)、低氧暴露7 d组(H7D,5只)、低氧暴露14 d组(H14D,5只)、低氧暴露21 d组(H21D,5只)、低氧暴露28 d组(H28D,5只)、对照1 d组(C1D,3只)、对照7 d组(C7D,3只)、对照14 d组(C14D,3只)、对照21 d组(C21D,3只)、对照28 d组(C28D,3只)。每个笼具喂养2-3只。H1D、H7D、H14D、H21D、H28D组饲养于青海大学高原医学研究中心低压氧舱(模拟海拔4 500 m),C1D、C7D、C14D、C21D、C28D组饲养于北京维通利华实验动物技术有限公司动物房。
正常饮食(饲料购于北京科奥协力饲料有限公司,产品批号:15073211)。所有器具、饲料均经过灭菌处理,实验期间动物自由采食、进水,并保持垫料干燥,12 h交替照明,温度18-22 ℃。
1.2 主要试剂和仪器PowerFecalTM DNA Isolation Kit,MO Bio Laboratories公司;Phusion® High-Fidelity PCR Master Mix,New England Biolabs公司;GeneJET Gel Extraction Kit,Thermo Scientific公司;TruSeq® DNA PCR-Free Sample Preparation Kit,Illumina公司。
低压氧舱,贵州风雷航空机械有限公司;荧光实时定量仪、凝胶成像系统,Bio-Rad公司;Nano Drop 2000c,Thermo Scientific公司;水平电泳仪,北京六一仪器厂;HiSeq测序平台,Illumina公司。
1.3 样品采集与保存分别在低氧暴露第1天取H1D和C1D组的粪便,在低氧暴露第7天取H7D和C7D组的粪便,低氧暴露第14天取H14D和C14D组的粪便,低氧暴露第21天取H21D和C21D组的粪便,低氧暴露第28天取H28D和C28D组的粪便。粪便采集的具体方法:用75%酒精消毒大鼠肛周皮肤,刺激其排便,使用灭菌镊子从大鼠肛门部取出粪便保存于含磷酸缓冲液(PBS)的无菌EP管,随后迅速投入液氮,保存至-80 ℃备用。
1.4 肠道微生物DNA的提取取0.2 g粪便样品,采用PowerFecalTM DNA Isolation Kit提取样品中的基因组DNA。提取后的DNA经1%琼脂糖凝胶电泳检测其完整性,选取条带清晰、DNA浓度和纯度检测合格的样品送至天津诺禾致源生物信息科技有限公司测序。
1.5 PCR扩增和纯化以稀释后的基因组DNA为模板,使用16S rRNA基因V3-V4区特异性PCR引物(341F:5′-CCTAYGGGRBGCASCAG-3′;806R:5′-XXXXX XGGACTACNNGGGTATCTAAT-3′)进行PCR扩增。30 μL PCR反应体系:Phusion® High-Fidelity PCR Master Mix 15 μL,模板DNA 10 ng,正、反向引物0.2 μmol/L。PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,50 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 5 min。PCR产物4 ℃保存。根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测,采用GeneJET Gel Extraction Kit对目的条带进行回收。
1.6 文库构建和上机测序使用TruSeq® DNA PCR-Free Sample Preparation Kit进行文库构建,检测合格后,使用HiSeq2500 PE250进行上机测序。
1.7 生物信息分析使用QIIME和FLASH软件对测序得到的原始数据进行拼接、过滤,得到有效数据。利用Uparse软件对所有样品的有效数据按97%的相似度进行OTU (Operational taxonomic unit)聚类。筛选OTU中出现频数最高的序列作为OTU的代表序列。对OTU代表序列进行物种注释,用RDP Classifier方法与GreenGene数据库进行物种注释分析(设定阈值为0.8-1.0)。最后对各样品的数据进行均一化处理,以样品中数据量最少的为标准进行均一化处理,后续的α和β多样性分析都是基于均一化处理后的数据。使用QIIME软件和R软件,进行α和β多样性分析,多样性指数的组间差异进行分析(Tukey检验或Wilcox检验)。使用R软件绘制无度量多维标定法(Non-metric multi-dimensional scaling,NMDS)分析图。组间差异性分析使用LEfSe软件分析。LEfSe分析默认设置线性判别分析(Linear discriminant analysis,LDA)的筛选值为4。使用PICRUSt软件对肠道微生物群落的功能进行预测分析。
2 结果与分析 2.1 测序结果测序得到的数据量为3 037 465条序列,平均每个样品为75 937条序列。质控后总体有效数据量及平均有效数据量分别为1 707 496条和42 687条,有效序列的平均长度为416 bp。对所有样品的有效序列进行聚类,以97%的一致性将序列聚类成为OTU。共注释到1 601个OTU,其中平均每个样品743个。
2.2 肠道微生物群落多样性分析 2.2.1 α多样性分析为了研究不同低氧暴露时间组和对照组SD大鼠肠道微生物群落的丰富度和多样性,对各组α多样性指数(表 1)进行了计算和Tukey检验或Wilcox检验。结果显示:Chao1指数在H28D和C28D组间存在显著性差异(Tukey检验);ACE指数在H28D和C28D,H7D和H14D,H7D和H28D组间存在显著性差异(Wilcox检验),说明H28D组微生物群落丰富度显著高于C28D和H7D组,H14D组微生物群落丰富度显著高于H7D组。Simpson指数H14D和C14D组间存在显著差异(Wilcox检验),说明H14D组肠道微生物群落多样性显著低于C14D组;Shannon指数H14D组低于C14D组(Wilcox检验),但差异无显著性。
| Groups | OTUs | α diversity | |||
| Chao1 index | ACE index | Simpson index | Shannon index | ||
| H1D | 3 784 | 751.08±36.67 | 751.69±28.48 | 0.96±0.01 | 6.55±0.39 |
| H7D | 3 489 | 726.39±57.18 | 721.08±41.34 | 0.95±0.03 | 6.38±0.70 |
| H14D | 3 882 | 796.89±54.30 | 797.14±55.45# | 0.93±0.03△ | 6.02±0.52 |
| H21D | 3 744 | 746.43±82.50 | 758.28±72.13 | 0.94±0.05 | 6.35±1.06 |
| H28D | 4 021 | 799.89±60.23* | 796.68±50.28*# | 0.96±0.02 | 6.67±0.44 |
| C1D | 2 252 | 757.40±58.45 | 763.67±53.50 | 0.97±0.01 | 6.89±0.17 |
| C7D | 2 156 | 695.90±73.10 | 714.11±79.59 | 0.96±0.01 | 6.46±0.49 |
| C14D | 2 311 | 810.90±56.30 | 810.93±52.57 | 0.97±0.00 | 6.82±0.06 |
| C21D | 2 304 | 753.22±43.99 | 752.58±43.79 | 0.96±0.01 | 6.66±0.42 |
| C28D | 1 794 | 574.31±75.00 | 585.45±72.06 | 0.96±0.01 | 6.01±0.52 |
| 注:与C28D比,*:P < 0.05;与H7D组比,#:P < 0.05;与C14D比,△:P < 0.05. Note: *: P < 0.05, compared with C28D; #: P < 0.05, compared with H7D; △: P < 0.05, compared with C14D. | |||||
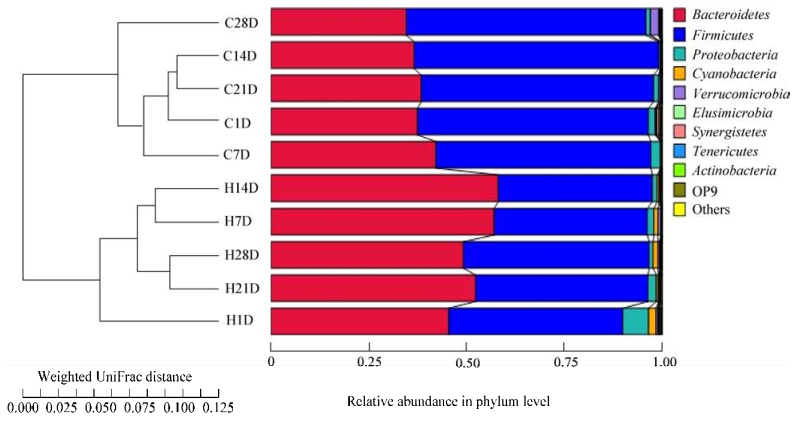
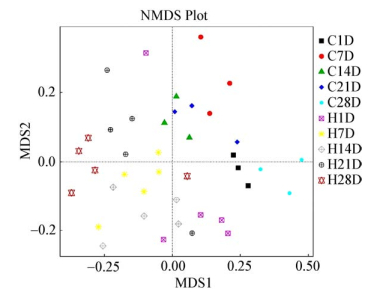
为了研究不同低氧暴露时间组和对照组SD大鼠肠道微生物群落构成的差异,基于加权的UniFrac距离矩阵进行UPGMA聚类分析和NMDS分析。图 1将UPGMA聚类分析的结果与门水平上各样品肠道微生物相对丰度进行了整合展示。不同低氧暴露时间组SD大鼠肠道微生物群落聚为一类,对照组SD大鼠肠道微生物群落聚为一类,其中H7D和H14D组的距离较近,H21D和H28D组的距离较近(图 1、2)。
|
| 图 1 基于加权的UniFrac距离构建的UPGMA聚类树 Figure 1 UPGMA clustering tree based on weighted UniFrac distance and community composition of gut microbiota among groups 注:左侧是UPGMA聚类树结构,标尺是加权的UniFrac距离.右侧是各组在门水平上的物种相对丰度分布图,纵坐标是组名,横坐标是相对丰度,Others表示图中这10个门之外其他所有门的相对丰度之和. Note: UPGMA clustering tree (at left). Scale length represented weighted UniFrac distance. In the stacked bar chart (at right), each bar represents the average relative abundance of each bacterial taxon. The top 10 taxa with high relative abundance in phylum level were illustrated. Others represent taxa beyond the top 10 taxa. |
|
|
|
| 图 2 基于加权的UniFrac的NMDS分析 Figure 2 NMDS analysis based on weighted UniFrac distance 注:每个符号代表了一个样品,符号之间的距离反映了样品群落结构之间的差异. Note: Each symbol represents the gut microbiota of one sample. Distances between symbols on the ordination plot reflect relative dissimilarities in community structures. |
|
|
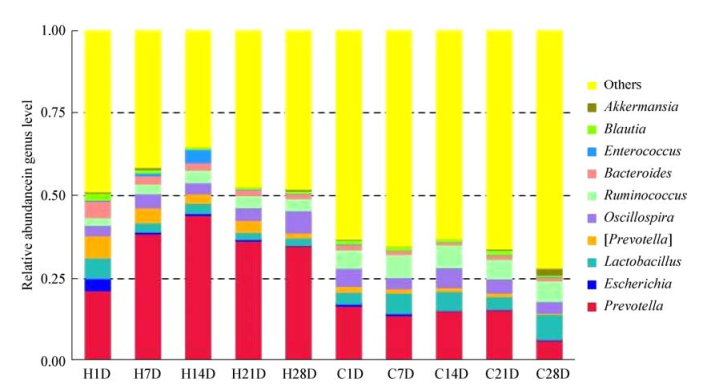
根据物种注释结果,选取每组肠道微生物在分类学门水平和属水平上最大相对丰度排名前10的物种,以柱型图进行展示(图 1、3)。
|
| 图 3 属水平上不同低氧暴露时间组和对照组SD大鼠肠道微生物群落结构相对丰度的比较 Figure 3 Community composition of gut microbiota between hypoxic exposure groups and control groups 注:各组在属水平上的物种相对丰度分布图,横坐标是组名,纵坐标是相对丰度,Others表示图中这10个属之外其他所有属的相对丰度之和. Note: In the stacked bar chart, each bar represents the average relative abundance of each bacterial taxon. The top 10 taxa with high relative abundance in genus level were illustrated. Others represent taxa beyond the top 10 taxa. |
|
|
不同低氧暴露时间组SD大鼠肠道微生物群落中门水平含量大于1%的有4个菌门,分别为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria),平均相对丰度分别为52.36%、42.90%、2.530%、1.02%。
对照组SD大鼠肠道微生物群落中门水平含量大于1%的有3个菌门,分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria),平均相对丰度分别为59.40%、37.81%、1.41%。
不同低氧暴露时间组和对照组SD大鼠肠道微生物群落中厚壁菌门与拟杆菌门的比值(Firmicutes/Bacteroidetes ratio,F/B值)分别为0.98 (H1D)、0.69 (H7D)、0.68 (H14D)、0.84 (H21D)、0.96 (H28D)、1.57 (C1D)、1.30 (C7D)、1.71 (C14D)、1.55 (C21D)、1.77 (C28D)。不同低氧暴露时间组的F/B值低于同时间对照组。
不同低氧暴露时间组SD大鼠肠道微生物群落中属水平含量大于2%的有5个属,分别为:普氏菌属(Prevotella)、颤螺菌属(Oscillospira)、乳酸菌属(Lactobacillus)、胃球菌属(Ruminococcus)、拟杆菌属(Bacteroides),平均相对丰度分别为38.89%、4.32%、3.24%、3.23%、2.61%。
对照组SD大鼠肠道微生物群落中属水平含量大于2%的有4个属,分别为:普氏菌属(Prevotella)、胃球菌属(Ruminococcus)、乳酸菌属(Lactobacillus)、颤螺菌属(Oscillospira),平均相对丰度分别为13.59%、6.25%、5.41%、4.56%。
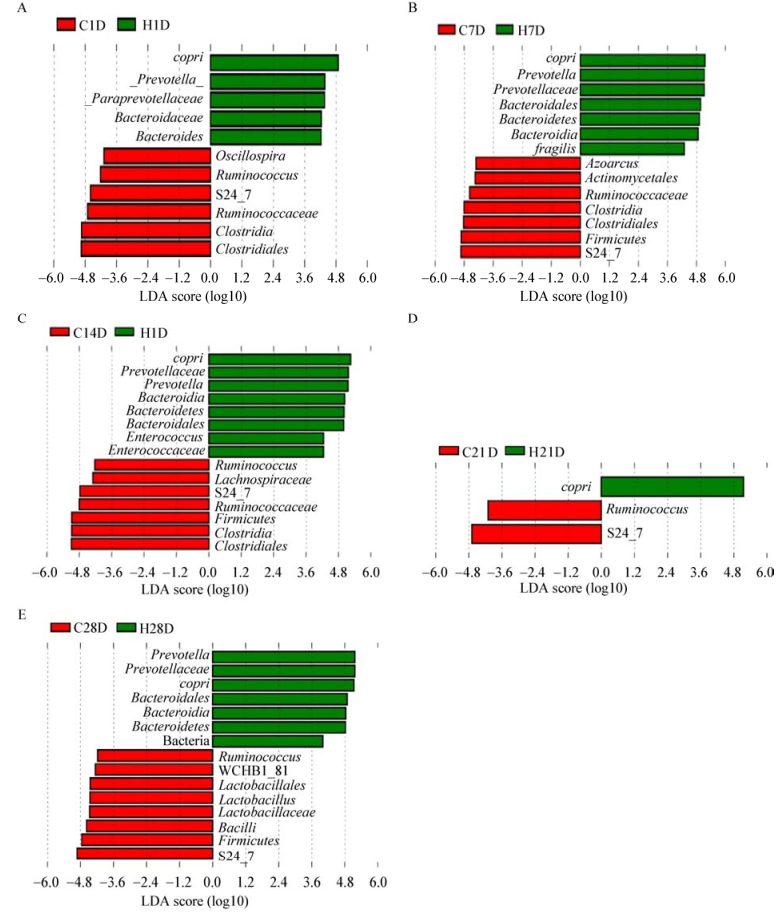
2.3.2 组间差异性肠道微生物群落利用LEfSe分析研究不同低氧暴露时间组和对照组SD大鼠组间差异性肠道微生物群落。结果表明与相应时间对照组相比,低氧暴露组SD大鼠的肠道微生物群落中拟杆菌门(Bacteroidetes)、拟杆菌目(Bacteroidales)、拟杆菌科(Bacteroidaceae)、普氏菌科(Prevotellaceae)、普氏菌属(Prevotella)和普氏菌种(copri)等相对丰度较高且具有统计学意义(图 4);相应时间对照组SD大鼠的肠道微生物群落中,瘤胃球菌科(Ruminococcaceae)、瘤胃球菌属(Ruminococcus)、梭菌纲(Clostridia)和梭菌目(Clostridiales)等相对丰度较高且具有统计学意义(图 4)。
|
| 图 4 不同低氧暴露时间组和对照组SD大鼠肠道微生物群落的LEfSe分析 Figure 4 Analysis of taxonomic relative abundances using LEfSe analysis shows that multiple bacterial taxa are differentially enriched among groups 注:LDA值分布柱状图中展示了不同组中丰度差异显著的物种,柱状图的长度代表差异物种的影响大小(LDA score). Note: Histogram showing significantly enriched bacterial taxa among groups by linear discriminant analysis (LDA) score. |
|
|
为了进一步探讨不同低氧暴露时间组SD大鼠肠道微生物群落的功能,利用PICRUSt软件对肠道微生物群落的功能进行预测分析。根据所有样品在KEGG数据库中的功能注释及丰度信息,选取丰度排名前35的第三层级功能及它们在每个样品中的丰度信息绘制热图(图 5),并从功能差异层面进行聚类。
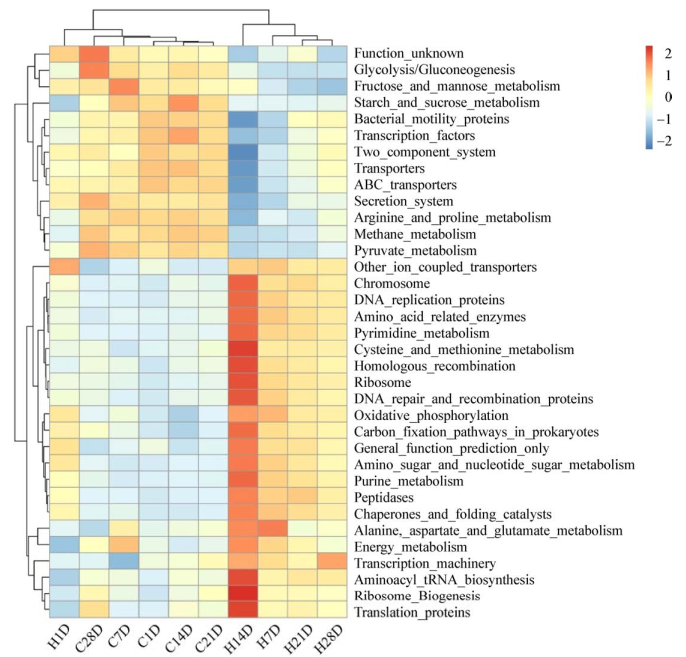
|
| 图 5 低氧不同暴露时间组和对照组SD大鼠肠道微生物群落的功能基因预测热图 Figure 5 Heat map of predicted function of gut micorbiota among hypoxia exposure groups and control groups 注:此聚类热图是基于所有样品在KEGG通路第3层级中的功能相对丰度绘制,图中展示了排名前35的功能聚类. Note: Abundance of top 35 functional genes from the third level of KEGG pathway was shown in the heat map. |
|
|
结果显示,不同低氧暴露时间组SD大鼠肠道微生物群落的功能预测多集中在遗传信息处理和代谢相关通路上,如其他离子偶联转运体、染色体、DNA修复和重组蛋白、氨基酸相关酶、嘧啶代谢、半胱氨酸和蛋氨酸代谢、同源重组、核糖体、氧化磷酸化、原核生物碳固定通路、一般功能预测、氨基糖和核苷酸糖代谢、嘌呤代谢、蛋白酶、分子伴侣和折叠催化剂、丙氨酸和天冬氨酸,及谷氨酸代谢、能量代谢、转录体、氨酰tRNA生物合成、核糖体合成和翻译蛋白。
对照组SD大鼠肠道微生物群落的功能预测为:未知功能、糖酵解和糖异生、果糖和甘露糖代谢、淀粉和蔗糖代谢、转录因子、双组分系统、转运蛋白、ABC转运体、分泌系统、精氨酸和脯氨酸代谢、甲烷代谢和丙酮酸盐代谢。
PICRUst功能预测结果表明,不同低氧暴露时间组和对照组SD大鼠肠道微生物群落的功能存在一定差异。
3 讨论与结论在肠黏膜的绒毛和微绒毛上,微生物通过分泌黏附因子等方式在肠黏膜上定殖[13]。人或动物的肠黏膜处于高灌注状态,因此对缺血、缺氧十分敏感。机体整体或肠道缺血、缺氧的状态会直接损伤肠黏膜[14-15],进一步影响肠道微生物的定殖[8-10]。肠道微生物群落的多样性是在生态和进化压力的双重选择下,由宿主和微生物群落共同进化形成的[5]。本研究通过α多样性分析发现,低氧暴露28 d组SD大鼠肠道微生物群落丰富度显著高于同时间的对照组,这与间歇性低氧处理小鼠6周后其肠道微生物群落的α多样性结果一致[16];低氧暴露14 d组SD大鼠的肠道微生物群落多样性显著低于同时间对照组;低氧暴露14 d组大鼠的微生物群落丰富度显著高于低氧暴露7 d组。通过β多样性分析发现,低氧暴露第1、2周SD大鼠的肠道微生物群落构成相似,低氧暴露第3、4周SD大鼠的肠道微生物群落构成相似。上述结果提示低氧会改变SD大鼠肠道微生物群落的α及β多样性,而且在低氧暴露的过程中SD大鼠肠道微生物群落的结构和丰富度是动态变化的。
除了低氧对机体肠黏膜的损伤造成的肠道微生物群落多样性发生变化外,我们推测低氧未习服物种在度过低氧暴露的急性期后,肠道微生物群落多样性的改变很有可能与机体对营养代谢和能量物质利用的调整有关。本研究发现不同低氧暴露时间组肠道微生物群落的F/B值低于同时间对照组。有研究表明藏族人的F/B值也较低[17]。一般体质量低的个体肠道微生物中厚壁菌门增加而拟杆菌门减少,F/B值低于体质量高的个体。而F/B值的改变可能会导致机体从食物中的能量采集发生变化[18]。碳水化合物是体内氧化代谢的主要产能物质,暴露于低氧环境时机体对碳水化合物的代谢对维持正常的生理功能显得尤为重要。本研究发现低氧暴露组SD大鼠肠道微生物群落中碳水化合物代谢相关菌群(普氏菌和拟杆菌)明显增加。另有研究指出,与汉族相比藏族人肠道微生物中的拟杆菌和普氏菌是优势菌群[17, 19],而高海拔的牦牛和藏系绵羊肠道微生物中的普氏菌也是优势菌群[20]。由此可见,暴露于高原或低氧环境中的人或动物普氏菌是典型的优势菌群。普氏菌属隶属于拟杆菌门,可以发酵碳水化合物和蛋白质底物产生SCFAs,也可以产生醋酸盐和H2,它们定殖在肠黏膜上有利于对营养物质的吸收、肝糖原储存,以及调节宿主免疫和修饰基因表达[21-23]。
本研究通过PICRUst分析发现,低氧暴露组SD大鼠肠道微生物群落的功能预测多在遗传信息处理和代谢通路上,说明低氧对宿主肠道微生物或宿主本身在基因水平上可能造成了损伤。其次,低氧暴露下肠道微生物可能通过加强生糖氨基酸(如半胱氨酸、蛋氨酸、丙氨酸,天冬氨酸和谷氨酸)的代谢参与宿主能量代谢的调整。
综上所述,低氧暴露过程中SD大鼠肠道微生物群落结构和多样性呈动态变化,碳水化合物代谢相关菌群明显增加,可能参与了宿主能量代谢调整,有利于机体对低氧环境的习服。
| [1] |
West JB. Recent advances in high altitude medicine and biology[J]. High Altitude Medicine & Biology, 2015, 16(2): 73. |
| [2] |
West JB. High-altitude medicine[J]. American Journal of Respiratory and Critical Care Medicine, 2012, 186(12): 1229-1237. DOI:10.1164/rccm.201207-1323CI |
| [3] |
Yang ZY, Luo YJ. Research progress of digestive system diseases on highland[J]. People's Military Surgeon, 2017, 60(4): 414-417. (in Chinese) 杨振宇, 罗勇军. 高原地区常见消化系统疾病及研究进展[J]. 人民军医, 2017, 60(4): 414-417. |
| [4] |
Kundu P, Blacher E, Elinav E, et al. Our gut microbiome: the evolving inner self[J]. Cell, 2017, 171(7): 1481-1493. DOI:10.1016/j.cell.2017.11.024 |
| [5] |
Ley RE, Peterson DA, Gordon JI. Ecological and evolutionary forces shaping microbial diversity in the human intestine[J]. Cell, 2006, 124(4): 837-848. DOI:10.1016/j.cell.2006.02.017 |
| [6] |
van Hylckama Vlieg JE, Veiga P, Zhang CH, et al. Impact of microbial transformation of food on health-from fermented foods to fermentation in the gastro-intestinal tract[J]. Current Opinion in Biotechnology, 2011, 22(2): 211-219. DOI:10.1016/j.copbio.2010.12.004 |
| [7] |
Tremaroli V, Bäckhed F. Functional interactions between the gut microbiota and host metabolism[J]. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [8] |
Adak A, Maity C, Ghosh K, et al. Dynamics of predominant microbiota in the human gastrointestinal tract and change in luminal enzymes and immunoglobulin profile during high-altitude adaptation[J]. Folia Microbiologica, 2013, 58(6): 523-528. |
| [9] |
Adak A, Ghosh K, Mondal KC. Modulation of small intestinal homeostasis along with its microflora during acclimatization at simulated hypobaric hypoxia[J]. Indian Journal of Experimental Biology, 2014, 52(11): 1098-1105. |
| [10] |
Adak A, Maity C, Ghosh K, et al. Alteration of predominant gastrointestinal flora and oxidative damage of large intestine under simulated hypobaric hypoxia[J]. Zeitschrift Für Gastroenterologie, 2014, 52(2): 180-186. |
| [11] |
Shan TD, Deng ZY, Zhang FX, et al. Comparative study of molecular biology on acute high altitude and plain intestinal Bifidobacterium in rats[J]. Chinese Journal of Microecology, 2011, 23(3): 197-200. (in Chinese) 单体栋, 邓芝云, 张方信, 等. 急进高原与平原大鼠肠道双歧杆菌的分子生物学实验对比研究[J]. 中国微生态学杂志, 2011, 23(3): 197-200. |
| [12] |
Yang WC, Zhang FX, Wu WM, et al. Changes of intestinal flora microecology in model rats of radical plateau[J]. Chinese Journal of Gastroenterology and Hepatology, 2010, 19(6): 543-545. (in Chinese) 杨文翠, 张方信, 吴文明, 等. 急进高原大鼠肠道微生态变化的探讨[J]. 胃肠病学和肝病学杂志, 2010, 19(6): 543-545. DOI:10.3969/j.issn.1006-5709.2010.06.016 |
| [13] |
Pickard JM, Zeng MY, Caruso R, et al. Gut microbiota: role in pathogen colonization, immune responses, and inflammatory disease[J]. Immunological Reviews, 2017, 279(1): 70-89. DOI:10.1111/imr.12567 |
| [14] |
Taylor CT, Colgan SP. Regulation of immunity and inflammation by hypoxia in immunological niches[J]. Nature Reviews Immunology, 2017, 17(12): 774-785. DOI:10.1038/nri.2017.103 |
| [15] |
Zhou J, Xu Q, Liu JW, et al. Characteristics of pathological injury of intestinal tract in rats under hypobaric hypoxia environment at different high altitude[J]. Progress in Modern Biomedicine, 2017, 17(27): 5238-5241, 5250. (in Chinese) 周静, 许琴, 刘江伟, 等. 不同海拔高原低压缺氧环境下大鼠肠道病理损伤特点[J]. 现代生物医学进展, 2017, 17(27): 5238-5241, 5250. |
| [16] |
Moreno-Indias I, Torres M, Montserrat JM, et al. Intermittent hypoxia alters gut microbiota diversity in a mouse model of sleep apnoea[J]. European Respiratory Journal, 2015, 45(4): 1055-1065. DOI:10.1183/09031936.00184314 |
| [17] |
Lan DL, Ji WH, Lin BS, et al. Correlations between gut microbiota community structures of Tibetans and geography[J]. Scientific Reports, 2017, 7(1): 16982. DOI:10.1038/s41598-017-17194-4 |
| [18] |
Remely M, Haslberger AG. The microbial epigenome in metabolic syndrome[J]. Molecular Aspects of Medicine, 2017, 54: 71-77. DOI:10.1016/j.mam.2016.09.003 |
| [19] |
Li K, Dan Z, Gesang L, et al. Comparative analysis of gut microbiota of native Tibetan and Han populations living at different altitudes[J]. PLoS One, 2016, 11(5): e0155863. DOI:10.1371/journal.pone.0155863 |
| [20] |
Zhang ZG, Xu DM, Li W, et al. Convergent evolution of rumen microbiomes in high-altitude mammals[J]. Current Biology, 2016, 26(14): 1873-1879. DOI:10.1016/j.cub.2016.05.012 |
| [21] |
Zhang HS, DiBaise JK, Zuccolo A, et al. Human gut microbiota in obesity and after gastric bypass[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(7): 2365-2370. |
| [22] |
Kovatcheva-Datchary P, Nilsson A, Akrami R, et al. Dietary fiber-induced improvement in glucose metabolism is associated with increased abundance of Prevotella[J]. Cell Metabolism, 2015, 22(6): 971-982. DOI:10.1016/j.cmet.2015.10.001 |
| [23] |
Ndeh D, Gilbert HJ. Biochemistry of complex glycan depolymerisation by the human gut microbiota[J]. FEMS Microbiology Reviews, 2018, 42(2): 146-164. |
 2019, Vol. 46
2019, Vol. 46




