扩展功能
文章信息
- 刘峰, 冯民权, 王毅博
- LIU Feng, FENG Min-Quan, WANG Yi-Bo
- 汾河入黄口夏季微生物群落结构分析
- Microbial community structure of estuary of the Fenhe River into the Yellow River in summer
- 微生物学通报, 2019, 46(1): 54-64
- Microbiology China, 2019, 46(1): 54-64
- DOI: 10.13344/j.microbiol.china.180252
-
文章历史
- 收稿日期: 2018-03-29
- 接受日期: 2018-06-08
- 网络首发日期: 2018-07-30
河流是地表各物质间迁移转化的载体,在碳、氮、磷等生源要素循环中发挥着重要作用[1]。近年来,大量氮、磷和有机物等污染物排放到河流水体,导致河流水质明显下降,生态环境日益恶化[2-3]。微生物在河流污染物降解和碳、氮、磷等生源要素迁移转化过程中具有不可或缺的作用,同时微生物对外界环境的变化非常敏感[4-7]。因此,河流微生物的多样性可以作为判断流域生态环境健康程度的重要生物指标[8],全面了解河流中微生物群落结构特征及其影响因素,对于保护河流生态环境具有积极意义。
随着分子生物学技术的不断发展,微生物的研究手段已从纯培养技术、各种分子指纹图谱技术[变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)、温度梯度凝胶电泳(Temperature gradient gel electrophoresis,TGGE)、PCR-DGGE、末端限制性片段长度多态性分析(Terminal-restriction fragment length polymorphism,T-RFLP)等]、荧光杂交技术等向高通量测序技术逐渐转化。高通量测序技术可以快速、详细且准确地了解微生物的群落结构[9],已广泛应用于湿地[10]、湖泊[11]、水库[12]、河流[13-14]和海洋[15]等环境中的微生物群落结构研究。基于16S rRNA基因的高通量测序研究表明[16]变形菌门(Proteobacteria)、蓝菌门(Cyanobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和疣微菌门(Verrucomicrobia)等微生物类群在地表水环境中分布广泛。目前,我国已有较多研究者利用分子生物学技术研究了河流微生物群落结构,王鹏等[9]研究发现赣江南昌段细菌优势类群为放线菌门,唐婧等[17]研究发现河流水质发生变化会引起河流细菌群落结构发生改变。然而,河流交汇区微生物群落结构的相关研究却鲜有报道。
汾河是黄河第二大支流,汾河入黄口是典型的河流交汇区,具有独特的生态学研究意义。河流交汇区是水体污染物混合、输移和富集的重要控制点,受到了人们的特别关注,但是关于河流交汇区微生物多样性的研究比较缺乏。目前还未有关于汾河流域微生物群落多样性的相关研究,尚不清楚汾河水系,特别是汾河入黄口处的微生物群落特征。本研究采用高通量测序技术首次分析了汾河入黄口处的微生物群落特征,讨论微生物群落结构与环境因子的关系,这对于更好地了解汾河入黄口微生物多样性和判断汾河入黄口生态环境健康程度,以及对汾河入黄口的环境监测和生态修复具有重要的科学意义。
1 材料与方法 1.1 主要试剂和仪器OMEGA Soil DNA Kit,Omega公司;核酸纯化试剂盒,Beckman公司;HiFi Hot Start Ready Mix高保真酶,KAPA Biosystems公司。PCR仪,Bio-Rad公司;电泳仪、凝胶成像仪,上海天能科技有限公司;台式高速离心机,Eppendorf公司;紫外分光光度计,HACH公司。
1.2 样点布设及水样采集采样区域属于干支流交汇河段,包括入黄口上游黄河段和汾河段、汾河与黄河交汇口、入黄口下游黄河段。如图 1所示,该采样区域共布设9个采样断面,在入黄口上游黄河段布设采样断面H1和H2;考虑交汇口水力条件复杂,在汾河与黄河交汇口设置3个采样断面(H3、H4和H5);入黄口下游黄河段布设采样断面H6和H7;在入黄口上游汾河段布设采样断面F3和F4。2017年8月对汾河入黄口进行样品采集,采集该区域汾河(采样断面F3和F4)、黄河(采样断面H1−H7)中泓垂线水面以下0.5 m处的水体样品,采样断面布设见图 1。现场采集的水样装于灭菌的聚乙烯细口瓶中,并用冰袋冷藏保存。运回到实验室后,用0.22 μm微孔滤膜过滤水样,收集滤膜置于干冰中冷冻保存。
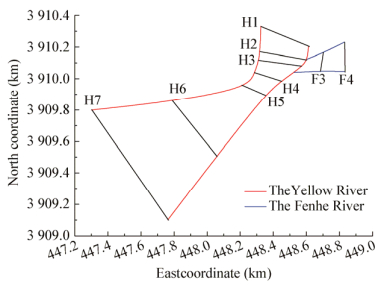
|
| 图 1 采样断面分布图 Figure 1 Map of sampling sections |
|
|
使用便携式多参数分析仪现场测定水样中的溶解氧(Dissolved oxygen,DO)、温度和pH。采用国标法测定水体中化学需氧量(Chemical oxygen demand,COD)、总磷(Total phosphorus,TP)、NH4+-N (氨氮)与总氮(Total nitrogen,TN)。COD按照重铬酸盐法(GB 11914-89)测定;TP按照钼酸铵分光光度法(GB 11893-89)测定;NH4+-N采用纳氏试剂分光光度法(HJ 535-2009)测定;TN采用碱性过硫酸钾消解紫外分光光度法(HJ 636-2012)测定。
1.4 DNA提取与高通量测序用OMEGA Soil DNA Kit提取截留在滤膜上的微生物DNA,上样于1%琼脂糖凝胶孔中,120 V恒压电泳15 min后检测提取的DNA纯度。DNA电泳结果符合质量要求,可以进行后续实验。以提取的DNA为模板,用引物343F (5′-TACGGRAGGCA GCAG-3′)和798R (5′-AGGGTATCTAATCCT-3′)进行PCR扩增微生物16S rRNA基因V3−V4高变区。PCR反应体系(30 μL):2×HiFi Hot Start Ready Mix 15 μL,引物343F和798R (10 μmol/L)各1 μL,DNA模板50 ng,ddH2O补足到30 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 7 min。使用琼脂糖凝胶电泳检测PCR扩增产物的纯度,检测后的产物使用磁珠纯化,纯化后作为二轮PCR扩增的模板,并再次使用琼脂糖凝胶电泳检测产物纯度,检测后使用磁珠纯化,对纯化后的PCR产物进行Qubit定量。按照PCR产物的浓度进行等量混样,在Illumina MiSeq测序平台上测序。
1.5 生物信息学分析由Illumina MiSeq测序所得的原始双端测序数据去杂(去掉3′端或者5′端模糊的碱基或低质量的碱基)并拼接,拼接后的数据进一步去杂。对原始两端序列使用Trimmomatic (V0.35)软件去杂;去杂后的两端数据使用FLASH (V1.2.11)软件进行拼接;使用QIIME (V8.0)软件对拼接后的原始数据进一步去杂;采用UCHIME (V4.2)去除嵌合体得到优质序列;用USEARCH (V2.4.2)软件对所有的优质序列以97%相似度进行OTU (Operational taxonomic unit,可操作分类单元)划分;在划分的OTU中找出丰度最大的序列作为代表序列;与Silva数据库(V123)[18]进行比对后最终得到系统发育树和OTU代表的分类信息,并在各个分类水平上统计每个样品的群落结构组成。
2 结果与分析 2.1 理化因子分析分析结果表明(表 1),汾河水体DO明显低于黄河;汾河和黄河各采样点表层水体水温范围为30.9−32.1 ℃,汾河水温略高于黄河,可能与水体颜色或周围环境有关;汾河与黄河各取样点pH在7.00左右;黄河各样点的COD、TP、NH4+-N与TN等水质指标均低于汾河。汾河水质整体上劣于黄河,汾河水流入黄河使黄河水质变差。
| Sample name | DO (mg/L) | Temperature (℃) | pH | COD (mg/L) | TP (mg/L) | NH4+-N (mg/L) | TN (mg/L) |
| F3 | 4.73 | 32.1 | 6.78 | 76 | 0.60 | 2.50 | 4.10 |
| F4 | 4.28 | 31.2 | 7.12 | 63 | 0.57 | 2.00 | 3.30 |
| H1 | 6.66 | 30.9 | 7.29 | 49 | 0.41 | 1.10 | 2.10 |
| H2 | 6.43 | 31.1 | 7.01 | 39 | 0.48 | 1.02 | 2.00 |
| H3 | 6.62 | 31.3 | 6.98 | 31 | 0.45 | 0.97 | 1.83 |
| H4 | 6.65 | 32.1 | 6.95 | 30 | 0.32 | 0.87 | 2.35 |
| H5 | 5.70 | 32.0 | 7.12 | 28 | 0.37 | 0.72 | 1.23 |
| H6 | 6.72 | 32.0 | 7.20 | 34 | 0.33 | 0.75 | 1.16 |
| H7 | 6.62 | 31.1 | 7.42 | 24 | 0.31 | 0.61 | 1.09 |
9个样点水样通过MiSeq高通量测序共得到高质量16S rRNA基因序列102 977条,以97%的序列相似度对得到的基因序列进行OTU划分,共获得3 478条OTU,单个样点水样的OTU数量为1 620−2 811。汾河入黄口各样点的OTU数量和多样性指数如表 2所示,各样本文库的覆盖率(Coverage)范围都在98.5%以上,表明测序深度可以反映汾河入黄口水体中微生物群落的真实情况。由Chao1丰度指数可知,黄河H5样点微生物丰度最高;汾河F4样点Chao1指数低于2 000,微生物丰度最低。汾河入黄口各样点Shannon多样性指数的变化幅度较大,变化范围在4.91−7.81之间,黄河H5样点Shannon值最大,黄河H6样点Shannon值最小,说明黄河H5样点的微生物多样性最高,汾河与黄河交汇后使黄河样点间微生物多样性差异增大。9个样点的Simpson指数在0.81−0.98之间,其中黄河H6样点Simpson指数值为0.81,与其它样点Simpson指数(0.91−0.98)有差异,说明黄河H6样点水体OTU数的分布与其它样点差异较大。
| Sample name | Number of OTUs | Chao1 index | Coverage | Shannon index | Simpson index |
| F3 | 2 057 | 2 031 | 0.988 6 | 6.59 | 0.93 |
| F4 | 1 620 | 1 990 | 0.988 9 | 7.51 | 0.98 |
| H1 | 2 154 | 2 239 | 0.987 1 | 6.87 | 0.93 |
| H2 | 2 363 | 2 215 | 0.987 3 | 6.66 | 0.94 |
| H3 | 2 426 | 2 385 | 0.985 1 | 6.55 | 0.93 |
| H4 | 2 433 | 2 537 | 0.985 1 | 7.39 | 0.95 |
| H5 | 2 811 | 2 641 | 0.985 2 | 7.81 | 0.97 |
| H6 | 1 882 | 2 004 | 0.986 2 | 4.91 | 0.81 |
| H7 | 2 288 | 2 147 | 0.986 5 | 6.09 | 0.91 |
用等级-多度(Rank-abundance)曲线(图 2)[19]比较了9个取样点微生物的物种丰度和均匀度。由图 2可见,在水平方向上黄河H5样点的曲线宽度最大,其丰富度最大,而汾河F4样点的微生物丰富度最小,汾河微生物丰度低于黄河;黄河H6样点的Rank-abundance曲线最不平滑,表明黄河H6样点物种的均匀度最不均匀,黄河H5样点的曲线最平滑,其物种均匀度最均匀;而各取样点的Rank-abundance曲线形状相似,可近似认为汾河入黄口物种组成的均匀程度基本一致,物种组成均匀。Venn图分析表明(图 3),黄河H5点的OTU数目最多,汾河F4点的OTU数目最少;9个点共有816个相同的OTU,占OTU总数的6.04%。Weighted UniFrac距离和Unweighted UniFrac两个指标可以用来衡量样品间的相异系数,其值代表 2个样品在微生物物种多样性方面的相似性。UniFrac距离数值在0−1之间,距离为0则相似性高,而距离为1则差异较大。根据Weighted UniFrac和Unweighted UniFrac距离绘制Heatmap图(图 4),分析表明汾河和黄河的微生物差异性大于汾河与黄河内各样点间的微生物差异性,汾河与黄河微生物群落组成具有一定的差异。非度量多维尺度分析(Nonmetric multidimensional scaling,NMDS)表明(图 5),黄河H1、H2、H3样点间具有相似的微生物群落结构;汾河F3和F4样点间具有相似的微生物群落结构;而黄河H4、H5、H6和H7间微生物群落结构差异较大。汾河与黄河交汇前河流各采样点具有相似的微生物群落结构,但汾河与黄河各样点微生物群落结构差异明显。汾河与黄河交汇后,汾河水携带大量微生物进入黄河,黄河水质发生变化,导致黄河各采样点微生物群落结构差异较大。

|
| 图 2 Rank-abundance曲线 Figure 2 Rank-abundance curve |
|
|

|
| 图 3 OTU数量的韦恩图 Figure 3 Venn diagram of OTUs number in each sampling site |
|
|
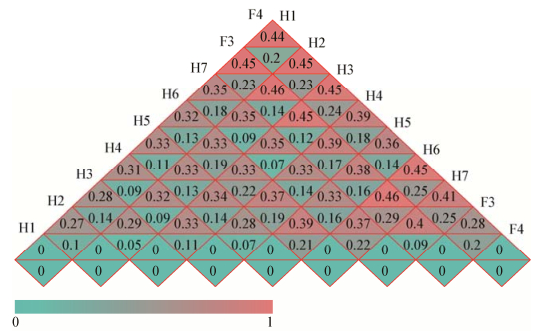
|
| 图 4 Weighted UniFrac和Unweighted UniFrac距离Heatmap图 Figure 4 Weighted and Unweighted UniFrac distances heatmap 注:同一方格中,上下两个值分别代表Unweighted UniFrac距离和Weighted UniFrac距离. Note: In the same square, the upper and lower values represent the unweighted UniFrac distance and the weighted UniFrac distance, respectively. |
|
|
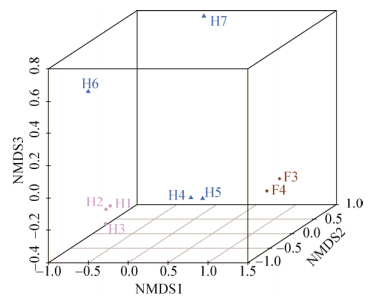
|
| 图 5 NMDS分析图 Figure 5 Analysis diagram of NMDS |
|
|
汾河入黄口水体微生物群落结构在门分类水平上具有较高的多样性(以丰度排序前三十的物种绘制相对丰度图,见图 6),门类数目远超过赣江南昌段等河流[9]。从门分类水平看,相对丰度从高到低依次是变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)、酸杆菌门(Acidobacteria)、绿菌门(Chlorobi)、蓝细菌门(Cyanobacteria)、硝化螺旋菌门(Nitrospirae)、疣微菌门(Verrucomicrobia)、迷踪菌门(Elusimicrobia)、纤维杆菌门(Fibrobacteres)、螺旋体门(Spirochaetae)、衣原体门(Chlamydiae)、TM6、Parcubacteria、梭杆菌门(Fusobacteria)、浮霉菌门(Planctomycetes)、绿弯菌门(Chloroflexi)、柔膜菌门(Tenericutes)、Gracilibacteria、装甲菌门(Armatimonadetes)、TA06、Latescibacteria、Candidate_division_OP3、OC31、WCHB1_60、Saccharibacteria、Aminicenantes、嗜热丝菌门(Caldiserica)。分析各个取样点的微生物群落结构,汾河入黄口菌群主要属于9个细菌门类(平均相对含量大于0.5%,见表 3)。总体上,变形菌门是汾河入黄口的优势菌门,各采样点相对丰度均在40%以上,F4样点高达75.04%。厚壁菌门、拟杆菌门、放线菌门、芽单胞菌门和酸杆菌门是汾河入黄口微生物群落的次优势菌门,5个菌门的平均相对丰度比例分别占总微生物群落的23.67%、9.17%、6.45%、1.35%和1.19%,其它菌门的平均相对丰度均低于1%。变形菌门下的Alphaproteobacteria、Betaproteobacteria、Deltaproteobacteria、Gammaproteobacteria、Epsilonproteobacteria等5个变形菌纲在汾河入黄口各个取样点均被检出,但汾河和黄河的优势变形菌纲存在差异,其中Betaproteobacteria是汾河水体中的优势菌纲,在汾河采样点相对丰度较高,介于25.85%−48.03%之间;Alphaproteobacteria是黄河水体中的优势菌纲,相对丰度介于15.62%−43.66%之间。

|
| 图 6 各样点在门分类水平上的微生物类群比较 Figure 6 Comparison of microorganism groups in each sampling site at phylum level |
|
|
| Phylum | H1 | H2 | H3 | H4 | H5 | H6 | H7 | F3 | F4 |
| Proteobacteria | 58.89 | 46.82 | 45.83 | 57.37 | 60.94 | 61.74 | 41.45 | 43.67 | 75.04 |
| Firmicutes | 12.39 | 30.16 | 35.37 | 19.65 | 16.15 | 8.60 | 42.74 | 42.01 | 5.93 |
| Bacteroidetes | 10.95 | 9.67 | 8.30 | 11.39 | 12.65 | 5.26 | 6.49 | 7.62 | 10.19 |
| Actinobacteria | 10.59 | 6.65 | 4.48 | 5.35 | 3.39 | 17.97 | 4.59 | 2.29 | 2.74 |
| Gemmatimonadetes | 2.11 | 1.61 | 1.77 | 1.55 | 1.29 | 1.02 | 1.14 | 0.75 | 0.94 |
| Acidobacteria | 1.52 | 1.24 | 1.04 | 1.08 | 1.01 | 2.50 | 1.02 | 0.54 | 0.72 |
| Chlorobi | 0.86 | 0.66 | 0.47 | 0.61 | 0.66 | 1.19 | 0.40 | 0.56 | 0.71 |
| Cyanobacteria | 0.55 | 1.17 | 0.48 | 0.37 | 0.60 | 0.56 | 0.57 | 0.31 | 0.78 |
| Nitrospirae | 0.60 | 0.48 | 0.48 | 0.65 | 0.79 | 0.34 | 0.30 | 0.42 | 0.58 |
在属水平上,相对丰度从高到低依次是芽孢杆菌属(Bacillus)、乳球菌属(Lactococcus)、hgcI_clade、硫杆菌属(Thiobacillus)、亚硝氮氧化菌属(Candidatus_Nitrotoga)、MWH_UniP1_aquatic_group、嗜氢菌属(Hydrogenophaga)、CL500_29_marine_ group、OM27_clade、链球菌属(Streptococcus)、Methyloversatilis、陶厄氏菌属(Thauera)、marine_group、肉食杆菌属(Carnobacterium)、12up、肠球菌属(Enterococcus)、Perlucidibaca、Acidibacter、假螺菌属(Pseudospirillum)、Leptothrix、Rheinheimera、BD1_7_clade、Fluviicola、硝化螺旋菌属(Nitrospira)、亚硝酸菌属(Nitrosomonas)、Sulfuritalea、水杆菌属(Aquabacterium)、Polynucleobacter、纤维弧菌属(Cellvibrio)、蛭弧菌属(Bdellovibrio)。对样品和OTU类型进行聚类后按相对丰度绘制热图(图 7),以颜色深浅表示相对丰度数值大小。芽孢杆菌属(Bacillus)、乳球菌属(Lactococcus)和hgcI_clade在各取样点丰度均很高,表明这3个属是汾河入黄口的优势菌属。
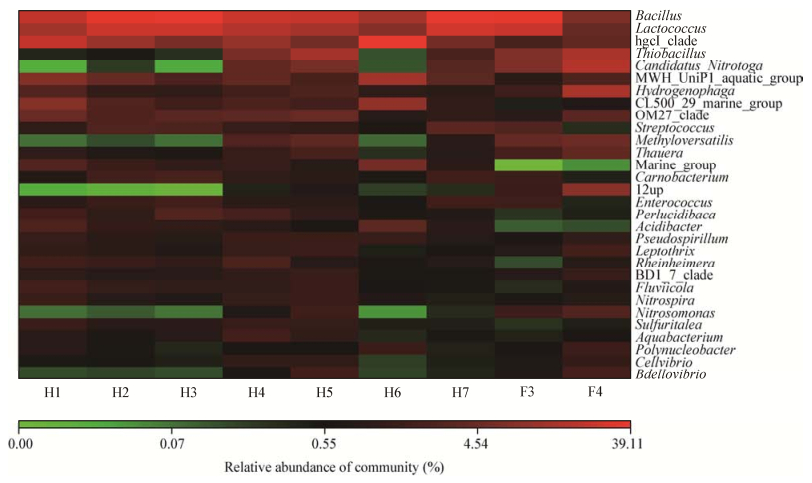
|
| 图 7 各样点优势菌属热图 Figure 7 Heatmap of dominant bacterial genus in each sampling site |
|
|
图 8是水体环境因子与微生物群落之间关系的典范对应分析(Canonical correspondence analysis,CCA),由CCA分析可以直接看出微生物群落组成与环境因子的关系,若图中箭头越长并且与两轴夹角越小,则此环境因子与微生物群落关系就越密切。由图 8可以看出,各环境因子与两轴都有夹角,说明水体环境因子对微生物群落的组成都有一定的影响。汾河入黄口微生物群落与环境因子的CCA分析的显著性P值表明:DO (P=0.003)对微生物群落结构影响的显著性最高;pH (P=0.005)对微生物群落结构的影响也较为显著;微生物群落的组成受其它环境因子如温度(P=0.538)、COD (P=0.017)、TP (P=0.202)、NH4+-N (P=0.009)、TN (P=0.015)的影响显著性较小(P > 0.005),所以pH和DO是影响微生物群落组成的主要环境因子。由图 8中各样点与环境因子的夹角(锐角表示正相关,钝角表示负相关)可以看出:F3、F4、H5样点的微生物群落组成与温度、COD、TP、NH4+-N、TN正相关,与pH、DO负相关;而H1、H2、H3、H6、H7样点微生物群落组成与温度、COD、TP、NH4+-N、TN负相关,与pH、DO正相关。将代表环境因子的箭头延长,样点垂直投射于射线上,沿着箭头方向,环境因子对样点微生物群落的影响程度增大。pH对H7样点微生物群落组成影响程度最大;H2样点微生物群落组成受DO的影响程度最大;TP、NH4+-N、TN、COD对F3样点微生物群落组成影响程度最大。
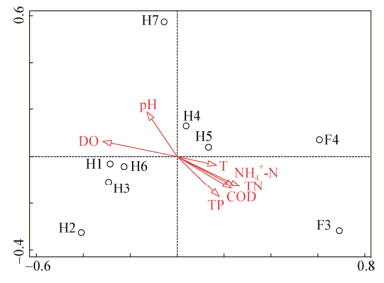
|
| 图 8 微生物群落与环境因子的典范对应分析图 Figure 8 Canonical correspondence analysis (CCA) graph for microorganisms community and environmental factors |
|
|
优势菌属与水质指标间的Spearman相关性分析如表 4所示,hgcI_clade、MWH_UniP1_aquatic_ group、marine_group和CL500_29_marine_group与DO显著正相关,而硫杆菌属、亚硝氮氧化菌属、嗜氢菌属、Methyloversatilis和Thauera与DO显著负相关;部分优势菌属(hgcI_clade、MWH_UniP1_ aquatic_group、Methyloversatilis、marine_group)与温度及pH显著相关;嗜氢菌属与COD、TP、NH4+-N、TN呈正相关;Methyloversatilis和Thauera与TN呈正相关关系,而marine_group与TN呈负相关关系;芽孢杆菌属、乳球菌属、OM27_clade、Carnobacterium和12up与环境因子的相关性都很小。
| Genus | DO | Temperature | pH | COD | TP | NH4+-N | TN |
| Bacillus | −0.18 | 0.17 | −0.39 | −0.17 | 0.05 | −0.03 | −0.02 |
| Lactococcus | −0.12 | 0.07 | −0.26 | −0.30 | −0.08 | −0.17 | −0.15 |
| hgcI_clade | 0.88 | −0.36 | 0.46 | −0.20 | −0.52 | −0.33 | −0.45 |
| Thiobacillus | −0.85 | 0.33 | −0.33 | 0.07 | 0.30 | 0.18 | 0.45 |
| Candidatus_Nitrotoga | −0.82 | 0.38 | −0.23 | 0.13 | 0.28 | 0.15 | 0.35 |
| MWH_UniP1_aquatic_group | 0.77 | −0.52 | 0.70 | −0.15 | −0.53 | −0.32 | −0.47 |
| Hydrogenophaga | −0.73 | 0.16 | −0.35 | 0.55 | 0.63 | 0.65 | 0.78 |
| CL500_29_marine_group | 0.84 | −0.25 | 0.30 | −0.13 | −0.38 | −0.25 | −0.37 |
| OM27_clade | −0.37 | −0.05 | −0.18 | −0.07 | 0.25 | 0.12 | 0.20 |
| Streptococcus | −0.12 | 0.07 | −0.26 | −0.30 | −0.08 | −0.17 | −0.15 |
| Methyloversatilis | −0.84 | 0.53 | −0.49 | 0.17 | 0.37 | 0.25 | 0.50 |
| Thauera | −0.76 | 0.27 | −0.25 | 0.25 | 0.33 | 0.35 | 0.60 |
| Marine_group | 0.83 | −0.52 | 0.59 | −0.22 | −0.47 | −0.37 | −0.60 |
| Carnobacterium | −0.19 | −0.03 | −0.25 | −0.33 | −0.05 | −0.15 | −0.13 |
| 12up | −0.63 | 0.35 | −0.03 | 0.18 | 0.17 | 0.15 | 0.35 |
| 注:表中数值展示Spearman相关系数值,正值代表正相关,负值代表负相关,数值大小代表相关性的高低.
Note: The values of Spearman correlation are shown in the table; Positive values represent positive correlations, negative values represent negative correlations, and numerical values represent high or low correlations. |
|||||||
微生物多样性分析结果受选择的16S rRNA基因扩增区域的影响。Claesson等[20]对16S rRNA基因9个可变区进行了扩增并分析比对到属水平上的准确率,结果表明V3、V4和V5区相对较好,V3/V4区相对其他区域对特异性引物有显著的扩增偏好性。V3−V5区引物应用较为广泛[21],本研究对微生物16S rRNA基因V3−V4高变区进行了测序,分析了汾河入黄口区域的水体中微生物群落多样性。以97%的序列相似性把得到的基因序列划分为1 620−2 811条OTU。汾河入黄口微生物Shannon多样性指数远远高于以16S rRNA克隆文库技术对细菌多样性进行分析的黄河下游河段(Shannon值为2.17−4.40)[22],说明利用高通量测序研究微生物多样性优于16S rRNA克隆文库分析。汾河与黄河的微生物群落结构具有一定的差异性,这与Jackson等[23]的研究结果有一致性,他们发现密西西比河的主干流细菌群落与支流的细菌群落组成显著不同。
本研究发现,汾河入黄口水体中变形菌门、放线菌门和厚壁菌门处于优势地位。汾河入黄口微生物群落组成中的变形菌门、放线菌门和厚壁菌门是典型的淡水菌门,已有众多研究者发现它们在淡水生态系统中广泛分布[24-25]。汾河入黄口水体中拟杆菌门丰度高,这与一些河流[14, 26]、湖泊[27]细菌多样性的研究基本一致;拟杆菌在水体中多呈附着型生长[24],汾河入黄口水体中含有的大量泥沙为拟杆菌提供了良好的生长环境。有研究者发现黄河水体中拟杆菌门和放线菌门的丰度大于厚壁菌门[22],而本研究发现在汾河入黄口厚壁菌门(Firmicutes)的相对丰度大于Bacteroidetes和Actinobacteria。Jeon等[28]发现厚壁菌门是沙尘中的优势细菌类群;而在汾河入黄口微生物中厚壁菌丰度较高,可能由于夏季汛期雨水较多造成大量地表沙尘冲入河流,导致汾河入黄口水体中厚壁菌门相对丰度偏高。汾河采样点水体中硫杆菌属、亚硝氮氧化菌属、嗜氢菌属、Methyloversatilis、陶厄氏菌属、12up、亚硝酸菌属菌群丰度明显高于交汇口及黄河下游河段;而入黄口上游黄河河段水体中丰度较低的亚硝氮氧化菌属、Methyloversatilis、12up、亚硝酸菌属和蛭弧菌属,在与汾河交汇后菌群丰度呈现不同程度的增加。硫杆菌属能利用环境中硫化物获取能量,许多硫杆菌在无机电子供体存在时同化有机物,亚硝氮氧化菌属能参与硝化过程,进而加速氮的有效转化,嗜氢菌属能够脱氮并降解多种有机物[29]。硫杆菌属、亚硝氮氧化菌属和嗜氢菌属在汾河取样点的丰度大于黄河取样点,可能是由于汾河水质较差,这几种微生物所需的营养物充足,有利于微生物的生长。
汾河入黄口水体优势菌属与环境因子的Spearman相关性分析发现,DO会抑制硫杆菌属、亚硝氮氧化菌属、嗜氢菌属的生长,而COD、TP、NH4+-N和TN却在不同程度上提高了它们的丰度,表明硫杆菌属、亚硝氮氧化菌属、嗜氢菌属对河流内氮、磷等有机污染物变化具有较好的适应性;DO对hgcI_clade、MWH_UniP1_aquatic_group丰度的提高较为显著,而TP对它们有抑制作用。汾河入黄口水体中pH变化差异小,而一些微生物对pH表现出敏感性,hgcI_clade、MWH_UniP1_aquatic_ group和marine_group受到pH显著的促进作用,而pH对硫杆菌属、嗜氢菌属和Methyloversatilis有一定的抑制。环境因子的变化会对菌群产生一定的影响,不同菌属受不同环境因子的影响程度不同。
4 结论(1) 本研究采用基于16S rRNA基因的高通量测序技术,首次分析了汾河入黄口区域水体中的微生物群落结构,结果表明该区域微生物群落多样性较高。汾河入黄口优势微生物类群为变形菌门(Proteobacteria),其次为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria);主要优势菌属为芽孢杆菌属(Bacillus)、乳球菌属(Lactococcus)、hgcI_clade。
(2) 汾河入黄口水体中微生物群落结构的相似度较高,9个取样点共有816个相同的OTU。汾河和黄河的微生物差异性大于汾河与黄河内各样点间的微生物差异性,汾河与黄河微生物群落结构具有一定的差异,汾河水的汇入导致交汇区下游黄河微生物结构差异明显。Spearman相关性分析及CCA分析表明不同菌属受水体环境因子的影响程度差异较大,pH和DO是影响汾河入黄口微生物群落组成的主要环境因子。
| [1] |
Cole JJ, Prairie YT, Caraco NF, et al. Plumbing the global carbon cycle: integrating inland waters into the terrestrial carbon budget[J]. Ecosystems, 2007, 10(1): 172-185. DOI:10.1007/s10021-006-9013-8 |
| [2] |
Yan WJ, Mayorga E, Li XY, et al. Increasing anthropogenic nitrogen inputs and riverine DIN exports from the Changjiang River basin under changing human pressures[J]. Global Biogeochemical Cycles, 2010, 24(4): GB0A06. |
| [3] |
Hao LX, Sun RH, Chen LD. Health assessment of river ecosystem in Haihe River basin, China[J]. Environmental Science, 2014, 35(10): 3692-3701. (in Chinese) 郝利霞, 孙然好, 陈利顶. 海河流域河流生态系统健康评价[J]. 环境科学, 2014, 35(10): 3692-3701. |
| [4] |
Dong P, Sun YJ, Wang HQ, et al. Study of micro-ecosystem of the Wenyu River by terminal restriction fragment length polymorphism analysis[J]. China Environmental Science, 2011, 31(4): 631-636. (in Chinese) 董萍, 孙寓姣, 王红旗, 等. 利用T-RFLP技术对温榆河微生物群落结构研究[J]. 中国环境科学, 2011, 31(4): 631-636. |
| [5] |
Cébron A, Garnier J. Nitrobacter and Nitrospira genera as representatives of nitrite-oxidizing bacteria: detection, quantification and growth along the lower Seine River (France)[J]. Water Research, 2005, 39(20): 4979-4992. DOI:10.1016/j.watres.2005.10.006 |
| [6] |
Findlay S. Stream microbial ecology[J]. Journal of the North American Benthological Society, 2010, 29(1): 170-181. DOI:10.1899/09-023.1 |
| [7] |
Jiang JG, Shen YF. Development of the microbial communities in Lake Donghu in relation to water quality[J]. Environmental Monitoring and Assessment, 2007, 127(1/3): 227-236. |
| [8] |
Li ZH. Spatial and seasonal diversity of bacterioplankton communities in Lake Dongping[D]. Tai'an: Master's Thesis of Shandong Agricultural University, 2010 (in Chinese) 李正华.东平湖浮游细菌群落多样性分析[D].泰安: 山东农业大学硕士学位论文, 2010 http://cdmd.cnki.com.cn/Article/CDMD-10434-1011019136.htm |
| [9] |
Wang P, Chen B, Li CQ, et al. Bacterial communities in Nanchang section of the Ganjiang River in wet seaon[J]. China Environmental Science, 2016, 36(8): 2453-2462. (in Chinese) 王鹏, 陈波, 李传琼, 等. 赣江南昌段丰水期细菌群落特征[J]. 中国环境科学, 2016, 36(8): 2453-2462. DOI:10.3969/j.issn.1000-6923.2016.08.027 |
| [10] |
Wei JM, Cui LJ, Li W, et al. Characteristics of bacterial communities in surface-flow constructed wetlands[J]. Environmental Science, 2016, 37(11): 4357-4365. (in Chinese) 魏佳明, 崔丽娟, 李伟, 等. 表流湿地细菌群落结构特征[J]. 环境科学, 2016, 37(11): 4357-4365. |
| [11] |
Zhang YJ, Li K, Zhu HR, et al. Community structure of microorganisms and its seasonal variation in Beihai Lake[J]. Environmental Science, 2017, 38(8): 3319-3329. (in Chinese) 张雅洁, 李珂, 朱浩然, 等. 北海湖微生物群落结构随季节变化特征[J]. 环境科学, 2017, 38(8): 3319-3329. |
| [12] |
Chen ZJ, Ding CY, Zhu JY, et al. Community structure and influencing factors of bacterioplankton during low water periods in Danjiangkou Reservoir[J]. China Environmental Science, 2017, 37(1): 336-344. (in Chinese) 陈兆进, 丁传雨, 朱静亚, 等. 丹江口水库枯水期浮游细菌群落组成及影响因素研究[J]. 中国环境科学, 2017, 37(1): 336-344. |
| [13] |
Staley C, Johnson D, Gould TJ, et al. Frequencies of heavy metal resistance are associated with land cover type in the Upper Mississippi River[J]. Science of the Total Environment, 2015, 511: 461-468. DOI:10.1016/j.scitotenv.2014.12.069 |
| [14] |
Liu R, Wu W, Zhou XD, et al. Bacterioplankton community structure in Weihe River and its relationship with environmental factors[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 934-944. (in Chinese) 刘睿, 吴巍, 周孝德, 等. 渭河浮游细菌群落结构特征及其关键驱动因子[J]. 环境科学学报, 2017, 37(3): 934-944. |
| [15] |
Bai J, Liu XS, Hou R, et al. Community structure and influencing factors of bacterioplankton in the southern South China Sea[J]. China Environmental Science, 2014, 34(11): 2950-2957. (in Chinese) 白洁, 刘小沙, 侯瑞, 等. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学, 2014, 34(11): 2950-2957. |
| [16] |
Zwart G, Crump BC, Kamst-Van Agterveld MP, et al. Typical freshwater bacteria: an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers[J]. Aquatic Microbial Ecology, 2002, 28(2): 141-155. |
| [17] |
Tang J, Xu XR, Shang CY, et al. Association of bacterial diversity in city area of Nanming river with environmental factors[J]. Acta Microbiologica Sinica, 2015, 55(8): 1050-1059. (in Chinese) 唐婧, 徐晓蓉, 商传禹, 等. 南明河城区河段细菌多样性与环境因子的关系[J]. 微生物学报, 2015, 55(8): 1050-1059. |
| [18] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): 590-596. |
| [19] |
Bates ST, Clemente JC, Flores GE, et al. Global biogeography of highly diverse protistan communities in soil[J]. The ISME Journal, 2013, 7(3): 652-659. DOI:10.1038/ismej.2012.147 |
| [20] |
Claesson MJ, Wang Q, O'Sullivan O, et al. Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions[J]. Nucleic Acids Research, 2010, 38(22): e200. DOI:10.1093/nar/gkq873 |
| [21] |
Liu C, Li JB, Rui JP, et al. The applications of the 16S rRNA gene in microbial ecology: current situation and problems[J]. Acta Ecologica Sinica, 2015, 35(9): 2769-2788. (in Chinese) 刘驰, 李家宝, 芮俊鹏, 等. 16S rRNA基因在微生物生态学中的应用[J]. 生态学报, 2015, 35(9): 2769-2788. |
| [22] |
Xia N, Xia XH, Liu T, et al. Characteristics of bacterial community in the water and surface sediment of the Yellow River, China, the largest turbid river in the world[J]. Journal of Soils and Sediments, 2014, 14(11): 1894-1904. DOI:10.1007/s11368-014-0974-5 |
| [23] |
Jackson CR, Millar JJ, Payne JT, et al. Free-Living and particle-associated bacterioplankton in large rivers of the Mississippi River Basin demonstrate biogeographic patterns[J]. Applied and Environmental Microbiology, 2014, 80(23): 7186-7195. DOI:10.1128/AEM.01844-14 |
| [24] |
Newton RJ, Jones SE, Eiler A, et al. A guide to the natural history of freshwater lake bacteria[J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14-49. |
| [25] |
Ren LJ, He D, Xing P, et al. Bacterial diversity and ecological function in lake water bodies[J]. Biodiversity Science, 2013, 21(4): 421-432. (in Chinese) 任丽娟, 何聃, 邢鹏, 等. 湖泊水体细菌多样性及其生态功能研究进展[J]. 生物多样性, 2013, 21(4): 421-432. |
| [26] |
Liu ZH, Huang SB, Sun GP, et al. Phylogenetic diversity, composition and distribution of bacterioplankton community in the Dongjiang River, China[J]. FEMS Microbiology Ecology, 2012, 80(1): 30-44. DOI:10.1111/j.1574-6941.2011.01268.x |
| [27] |
Jones SE, Newton RJ, McMahon KD. Evidence for structuring of bacterial community composition by organic carbon source in temperate lakes[J]. Environmental Microbiology, 2009, 11(9): 2463-2472. DOI:10.1111/emi.2009.11.issue-9 |
| [28] |
Jeon EM, Kim HJ, Jung K, et al. Impact of Asian dust events on airborne bacterial community assessed by molecular analyses[J]. Atmospheric Environment, 2011, 45(25): 4313-4321. DOI:10.1016/j.atmosenv.2010.11.054 |
| [29] |
Wang LH, Lü Z, Hao CB, et al. Degrading bacteria community structure in groundwater of a petroleum-contaminated site[J]. Environmental Science and Technology, 2013, 36(7): 1-8, 146. (in Chinese) 王丽华, 吕铮, 郝春博, 等. 某石油污染场地地下水中降解菌群落结构研究[J]. 环境科学与技术, 2013, 36(7): 1-8, 146. DOI:10.3969/j.issn.1003-6504.2013.07.001 |
 2019, Vol. 46
2019, Vol. 46




