扩展功能
文章信息
- 罗长财, 缪静, 李国莹, 杜瑶, 余晓斌
- LUO Zhang-Cai, MIAO Jing, LI Guo-Ying, DU Yao, YU Xiao-Bin
- N-糖基化对一种新型重组耐高温β-甘露聚糖酶(ReTMan26)稳定性的影响
- Effect of N-glycosylation on the stability of a novel recombinant thermostable β-mannanase (ReTMan26)
- 微生物学通报, 2019, 46(1): 11-19
- Microbiology China, 2019, 46(1): 11-19
- DOI: 10.13344/j.microbiol.china.180192
-
文章历史
- 收稿日期: 2018-03-14
- 接受日期: 2018-05-18
- 网络首发日期: 2018-06-06
2. 鲁东大学生命科学学院 山东 烟台 264025
2. School of Life Sciences, Ludong University, Yantai, Shandong 264025, China
β-甘露聚糖酶(EC3.2.1.78)是一种能催化β-1, 4-甘露糖苷键降解的糖苷水解酶,被广泛用于饲料、食品加工、纺织、造纸、生物能源等领域[1-2]。由于饲料制粒及生物能源生产等环节涉及到高温处理过程,易使酶失活[3-4],因此,提高β-甘露聚糖酶的耐高温性能亟待解决。
为开发一种具有优良耐高温性能的β-甘露聚糖酶,从热泉中筛选出一株嗜热枯草芽胞杆菌(TBS2),该菌产生的β-甘露聚糖酶(BS-Man)在90 ℃处理10 min后酶活仍保留63.6%,但其发酵水平低,难以实现工业化生产[5]。由于毕赤酵母具有表达量高、操作简单、能对蛋白进行翻译后修饰等优点,被广泛用于外源蛋白的表达[6-7]。为此,将该酶基因从TBS2中克隆并在毕赤酵母X33中实现了高效表达,经鉴定该酶属于糖苷水解酶26家族(GH26),是一种新型耐高温β-甘露聚糖酶[8]。检测发现,由毕赤酵母表达的重组耐高温β-甘露聚糖酶(ReTMan26)的耐高温性能比BS-Man有较大幅度的提高。进一步分析发现,ReTMan26序列中存在3个N-糖基化位点(N8、N26及N255)。N-糖基化是毕赤酵母进行翻译后修饰的一种主要形式[9],当表达的蛋白中含有N-糖基化位点时,毕赤酵母就可能对其进行N-糖基化修饰[10-11],因此推测ReTMan26耐高温性能的提高可能与N-糖基化存在一定的关联。
本研究通过构建ReTMan26蛋白质三维结构模型,初步分析了N-糖基化在蛋白结构中的位置及其作用,然后采用天然蛋白去糖基化试剂盒对ReTMan26进行脱N-糖基化处理,并对脱N-糖基化前后的ReTMan26进行了纯化及对比测试,以确定N-糖基化对该酶的pH稳定性、耐热稳定性及抗消化蛋白酶性能的影响,便于今后进行更深入的研究和开发。
1 材料与方法 1.1 材料 1.1.1 菌株(1) 天然菌株TBS2:从水温高于68 ℃的热泉中筛选出的一株产耐高温β-甘露聚糖酶BS-Man的嗜热枯草芽胞杆菌(Thermophilic Bacillus subtilis)[5]。
(2) 重组毕赤酵母Orig-pPICZαA-X33:以TBS2为出发菌株,通过基因克隆、电转化毕赤酵母X33感受态细胞,筛选获得的一株用于生产ReTMan26的重组毕赤酵母[8]。
1.1.2 主要试剂和培养基槐豆胶、天然蛋白去糖基化试剂盒(NDEGLY)购自Sigma-Aldrich公司;酵母粉、胰蛋白胨购自Oxoid公司;其余试剂均为国产分析纯。
TBS2发酵培养基(g/L):魔芋精粉20.0,硫酸铵10.0,硝酸钠5.0,七水硫酸镁2.5,磷酸二氢钾1.5,初始pH 6.5。
YPD培养基(g/L):胰蛋白胨20.0,酵母粉10.0,葡萄糖20.0。
BSM培养基:85%磷酸26.7 mL,硫酸钾18.2 g,七水硫酸镁14.9 g,氢氧化钾4.13 g,二水硫酸钙0.9 g,微量元素(PTM1) 4.4 mL,加水定容至1 L。
微量元素(PTM1,g/L):七水硫酸亚铁65.0,氯化锌20.0,五水硫酸铜6.0,一水硫酸锰3.0,氯化钴0.5,二水钼酸钠0.2,生物素0.2,碘化钠0.08,硼酸0.02,98%浓硫酸5 mL,采用纯净水定容至1 L。其中生物素采用过滤灭菌,待PTM1高温灭菌并冷却至室温后加入。
1.1.3 主要仪器恒温恒湿培养箱、恒温摇床,上海一恒科学仪器有限公司;电泳仪,北京君意东方电泳设备有限公司;紫外-可见光分光光度计,岛津公司;50 L自动液体发酵罐,上海保兴生物设备工程有限公司。
1.2 ReTMan26中N-糖基化位点及其作用分析使用蛋白质在线建模软件SWISS-MODEL (http://swissmodel.expasy.org/)构建ReTMan26的三维结构模型,再通过PyMOL软件进行分析,以初步判断N-糖基化位点在ReTMan26蛋白结构中的位置及可能的作用。
1.3 β-甘露聚糖酶活性检测采用0.1 mol/L pH 5.5的乙酸-乙酸钠缓冲液进行酶液稀释及反应底物(槐豆胶)配制。检测时吸取2.0 mL经37 ℃预热的待测酶液至试管中,加入2.0 mL经37 ℃预热且浓度为6 g/L的底物溶液,振荡混匀,于37 ℃温育30 min。然后加入5.0 mL DNS试剂并振荡混匀,以终止酶解反应。在沸水中加热5 min后用自来水冷却,加水定容至25.0 mL,振荡混匀。以纯净水作为空白对照,于540 nm测定吸光值,然后通过标准曲线计算酶活。
酶活定义为:在37 ℃、pH 5.5的条件下,每分钟从3 mg/mL的甘露聚糖溶液中水解释放1 μmol还原糖所需的酶量为1个酶活单位(U)。
1.4 BS-Man摇瓶发酵及纯化采用嗜热枯草芽孢杆菌TBS2进行摇瓶发酵以获得耐高温β-甘露聚糖酶BS-Man。摇瓶发酵条件为:500 mL摇瓶装100 mL TBS2发酵培养基,初始pH 7.0,温度50 ℃,转速200 r/min,发酵36 h。发酵结束后于8 000×g离心10 min,上清液经10 kD超滤膜浓缩后进行盐析,然后取盐析产物通过Sephadex G-75层析柱(洗脱液为0.1 mol/L pH 5.5的乙酸-乙酸钠缓冲液)进行纯化,纯化效果通过SDS-PAGE进行验证。
1.5 ReTMan26高密度液体发酵挑取重组毕赤酵母Orig-pPICZαA-X33单菌落,接种至YPD培养基中,30 ℃、220 r/min培养24 h制备摇瓶种。50 L发酵罐中装BSM培养基20 L (其中预先加有320 g纯甘油作为碳源),采用17% (质量体积比)的氨水作为氮源并调节pH。高密度液体发酵条件为:接种量10% (体积比),搅拌速率500 r/min,发酵pH 4.6,通气量50 L/min,温度30 ℃。接种培养12 h后,开始流加50% (质量体积比)的甘油,流加速率为:12−16 h,32 g/h;16−20 h,40 g/h;20−24 h,48 g/h。甘油流加结束约15 min后,溶氧大幅升高,即开始甲醇诱导。甲醇的流加速率为:24−30 h,30 mL/h;30−40 h,40 mL/h;40−50 h,50 mL/h;50−60 h,60 mL/h;60−80 h,70 mL/h;80−100 h,80 mL/h;100−192 h,90 mL/h。
1.6 ReTMan26脱N-糖基化处理及纯化参照天然蛋白去糖基化试剂盒使用说明,发酵液经8 000×g离心10 min去除菌体后,取2 mL酶液分别加入20 μL的F1、F2、F3组分及400 μL缓冲液,于37 ℃进行脱N-糖基化处理6 h。脱N-糖基化前后的酶液采用10 kD超滤膜进行超滤浓缩后,再采用Sephadex G-75层析柱(洗脱液为0.1 mol/L pH 5.5的乙酸-乙酸钠缓冲液)进行纯化,纯化产物通过SDS-PAGE验证纯化效果。脱N-糖基化后的ReTMan26标识为ReTMan26-DG。
为确定脱N-糖基化效果,各取10 μL经纯化后的ReTMan26、ReTMan26-DG及BS-Man一起进行SDS-PAGE电泳。
1.7 稳定性对比实验 1.7.1 最适反应pH及pH稳定性采用不同pH值的反应体系:pH 1.5−5.0 (磷酸氢二钠-盐酸缓冲液)、pH 5.0−8.5 (磷酸氢二钠-磷酸二氢钾缓冲液)、pH 8.5−10.0 (甘氨酸-氢氧化钠缓冲液),分别添加0.2 mL经适当稀释的ReTMan26及ReTMan26-DG酶液,在37 ℃、不同pH (1.5−10.0)条件下反应30 min,将活性最高的实验组酶活设定为100%,其余不同反应pH时的酶活与之相比,得到该酶在不同pH时的相对酶活,以确定其最适反应pH。
为测定酶的pH稳定性,采用ReTMan26及ReTMan26-DG于37 ℃、不同pH (1.5−9.0)条件下预处理4 h,然后检测酶活,以未经处理的对照组酶活为100%,各处理组与之相比获得不同pH处理后的剩余相对酶活。
1.7.2 最适反应温度及耐热稳定性在最适pH及相同条件下,分别采用0.2 mL经适当稀释的ReTMan26和ReTMan26-DG酶液在不同温度(20−80 ℃)进行反应,将活性最高的实验组酶活设定为100%,其余反应温度下所测酶活与之相比获得各温度下的相对酶活,以确定其最适反应温度。
为检测ReTMan26及ReTMan26-DG的耐热性能,在相同条件下,各实验组取0.2 mL酶液于不同温度(60−100 ℃)预处理10 min,然后测定酶活。以未经热处理的对照组酶活为100%,各实验组所测酶活与之相比获得该酶经不同温度处理后的剩余相对酶活,从而确定其耐热性能。
为进一步反映N-糖基化对ReTMan26耐热稳定性的影响,分别采用ReTMan26及ReTMan26-DG在不同温度(60−100 ℃)进行不同时间的热处理,并检测剩余酶活,以确定脱N-糖基化前后的ReTMan26在不同温度时的半衰期。
1.7.3 抗消化蛋白酶的稳定性根据单胃动物消化道特点,在体外进行蛋白酶消化模拟实验。各取0.20 mg的ReTMan26及ReTMan26-DG (纯化后的冻干粉)分别加入消化酶液A、B中,于37 ℃下消化2 h。
消化酶液A (胃液)组成:0.1 mol/L磷酸氢二钠-盐酸缓冲液,pH 2.0,胃蛋白酶80 U/mL。
消化酶液B (肠胰液)组成:0.2 mol/L磷酸二氢钾-氢氧化钠缓冲液,pH 6.8,胰蛋白酶150 U/mL。
消化结束后,分别以未经消化酶处理的ReTMan26及ReTMan26-DG酶活性为100%,各处理组的酶活与之相比获得剩余相对酶活。
2 结果与分析 2.1 N-糖基化位点及其作用分析据报道,不同来源的β-甘露聚糖酶在氨基酸序列上会存在一定差异,但在蛋白质结构上通常具有一些相同特征,例如,属于GH5、GH26及GH113家族的β-甘露聚糖酶的催化域,一般都是由8个α-螺旋和8个β-折叠交替排列形成桶状结构,其中排列在一起的8个β-折叠片段形成桶状内部凹槽,与之相连的α-螺旋则组成桶的外沿。另外,氨基酸序列上还存在8个保守氨基酸构成该桶状结构的活性口袋,其组成依次为精氨酸(Arg)、组氨酸(His)、天冬氨酸(Asn)、谷氨酸(Glu)、组氨酸(His)、酪氨酸(Tyr)、谷氨酸(Glu)及色氨酸(Trp),且其中的2个谷氨酸位点分别位于第4及第7个β-折叠的C末端,高度保守,是β-甘露聚糖酶的亲核催化及酸碱催化位点[12-13]。
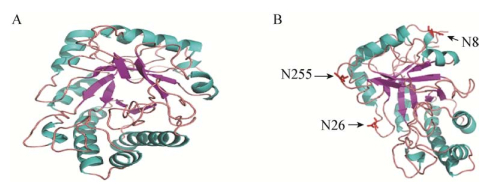
图 1A为ReTMan26的总体三维结构图,从中可以看出,ReTMan26蛋白形成了明显的桶状结构,这与报道的β-甘露聚糖酶结构特征相同。
|
| 图 1 ReTMan26蛋白质三维结构模型(A)及其N-糖基化位点分析(B) Figure 1 Three-dimensional structure model of ReTMan26 (A) and its N-glycosylation sites analysis (B) |
|
|
为更好地展示ReTMan26中的3个N-糖基化位点,图 1B为经过适当旋转后的ReTMan26三维结构图。可以发现,N8、N26及N255这3个N-糖基化位点均处于ReTMan26催化域外围,这些位点的N-糖基化对酶催化性能的影响可能很小。
图 2A为ReTMan26活性口袋的三维结构图,依次由Arg52、His87、Asn90、Glu149、His182、Tyr224、Glu283、Trp284组成,其中位于第4、7个β-折叠C末端的Glu149与Glu283两个谷氨酸位点,是ReTMan26中高度保守的酸碱催化和亲核催化位点,这2个位点对于酶的催化活性及稳定性具有重要作用。图 2B则为3个N-糖基化位点及2个酸碱催化、亲核催化位点(Glu149、Glu283)在ReTMan26三维结构中的分布图,可以发现,N26与Glu149、N255与Glu283在空间上处于相邻状态,推测这2个N-糖基化位点在酸碱、高温或消化蛋白酶等存在时,能在一定程度对Glu149、Glu283或附近其它重要催化域部位提供保护,从而提高ReTMan26的稳定性。

|
| 图 2 ReTMan26活性口袋(A)及3个N-糖基化位点对酶蛋白的稳定性作用分析(B) Figure 2 Active pocket (A) and function analysis of the three N-glycosylation sites to the stability of ReTMan26 (B) |
|
|
重组毕赤酵母Orig-pPICZαA-X33在50 L液体发酵罐中的发酵过程曲线见图 3。

|
| 图 3 重组毕赤酵母Orig-pPICZαA-X33高密度液体发酵 Figure 3 High-cell-dentisity fermentation by recombinant P. pastoris Orig-pPICZαA-X33 |
|
|
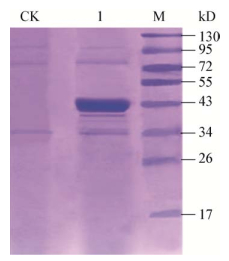
发酵结束后,发酵液中ReTMan26酶活为5 435 U/mL,为了分析发酵液中蛋白分布情况,取发酵结束后的离心上清液进行SDS-PAGE电泳;同时,取空白毕赤酵母X33 (含不带任何外源基因的pPICZαA空白质粒)发酵后的离心上清液作对照,结果见图 4。
|
| 图 4 ReTMan26在毕赤酵母X33中表达后的SDS-PAGE电泳分析 Figure 4 SDS-PAGE analysis of ReTMan26 expressed by P. pastoris X33 注:CK:空白毕赤酵母X33发酵后的离心上清液;1:重组毕赤酵母Orig-pPICZαA-X33发酵192 h后的离心上清液;M:蛋白质Marker. Note: CK: Supernatant of negative P. pastoris X33 after fermentation; 1: Supernatant of recombinant P. pastoris Orig-pPICZαA-X33 after fermentation for 192 h; M: Protein marker. |
|
|
与对照(CK)相比,重组毕赤酵母Orig-pPICZαA- X33发酵192 h后的离心上清液在42 kD附近出现一条粗大、明亮的条带,由于纯化后的ReTMan26分子量也约为42 kD (图 5),说明该条带是ReTMan26蛋白条带。因为ReTMan26的理论分子量为36.132 kD,小于42 kD,说明经毕赤酵母表达的ReTMan26已发生了相应的翻译后修饰。由于ReTMan26中存在3个N-糖基化位点,初步判断ReTMan26极可能形成了N-糖基化。

|
| 图 5 纯化后各种酶的SDS-PAGE电泳分析 Figure 5 SDS-PAGE analysis of different purified enzymes 注:M:蛋白质Marker;1:纯化后的ReTMan26;2:纯化后的ReTMan26-DG;3:纯化后的BS-Man. Note: M: Protein marker; 1: ReTMan26 after purification; 2: ReTMan26-DG after purification; 3: BS-Man after purification. |
|
|
天然蛋白去糖基化试剂盒(NDEGLY)含有多种N-糖基化水解酶组分,可以在不经蛋白变性及温和条件下专一性地切除N-多糖链,这样去除N-糖基化后,酶蛋白仍可保持活性。为了检测去糖基化试剂盒脱N-多糖链及后续纯化的效果,以纯化后的BS-Man为对照,对ReTMan26脱N-糖基化前后经过纯化的产物进行了SDS-PAGE电泳,结果见图 5。
经纯化后的ReTMan26为42 kD左右的单一条带,而去除N-糖基化并纯化后的产物ReTMan26- DG为36 kD左右的单一条带,这与嗜热枯草芽孢杆菌TBS2通过摇瓶发酵产生并纯化后的β-甘露聚糖酶BS-Man条带一致。枯草芽孢杆菌作为一种原核生物,对表达的酶蛋白不能进行翻译后修饰,不会形成糖基化,而且ReTMan26-DG与该酶的理论分子量基本相同,这说明经过去糖基化试剂盒处理后,N-多糖链已去除且纯化效果良好,同时也证明通过毕赤酵母产生的ReTMan26中发生其它糖基化(如O-糖基化)或磷酸化修饰的情况很少或几乎没有。
2.4 稳定性对比实验结果 2.4.1 最适反应pH及pH稳定性纯化后的ReTMan26及ReTMan26-DG最适反应pH及pH稳定性检测结果分别见图 6、7。

|
| 图 6 ReTMan26与ReTMan26-DG的最适反应pH Figure 6 Optimum reaction pH of ReTMan26 and ReTMan26-DG |
|
|

|
| 图 7 ReTMan26与ReTMan26-DG的pH稳定性 Figure 7 pH stability of ReTMan26 and ReTMan26-DG |
|
|
ReTMan26与ReTMan26-DG的最适反应pH均为6.0,这说明N-糖基化对ReTMan26最适反应pH基本没影响。
从图 7可以发现,在pH 1.5−9.0范围内,ReTMan26的pH稳定性比ReTMan26-DG有一定程度的提高,其中在pH 6.0时提高幅度最低,仅约2.2%;在pH 8.5时提高幅度最大,为17.9%,因此N-糖基化可小幅提高ReTMan26的pH稳定性。
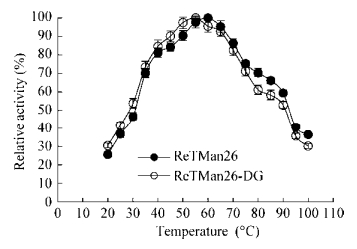
2.4.2 最适反应温度及耐热性ReTMan26的最适反应温度为60 ℃,而ReTMan26-DG最适反应温度为55 ℃,因此ReTMan26的最适反应温度比ReTMan26-DG提高了5 ℃。最适反应温度检测结果见图 8。
|
| 图 8 ReTMan26与ReTMan26-DG最适反应温度 Figure 8 Optimum temperature of ReTMan26 and ReTMan26-DG |
|
|
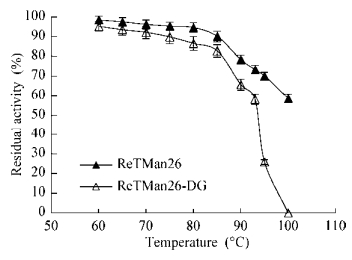
ReTMan26与ReTMan26-DG在60−100 ℃分别处理10 min后的耐热实验结果见图 9。ReTMan26经100 ℃处理10 min后剩余酶活仍达到58.6%,ReTMan26-DG在93 ℃处理10 min后剩余酶活为58.2%,而在100 ℃处理10 min则完全失活,因此N-糖基化可明显提高ReTMan26的耐热性能。
|
| 图 9 ReTMan26与ReTMan26-DG的耐热稳定性 Figure 9 Thermostability of ReTMan26 and ReTMan26-DG |
|
|
ReTMan26与ReTMan26-DG在不同温度的半衰期结果见表 1。在100 ℃时,ReTMan26的半衰期约为12 min,而去除N-糖基化后的ReTMan26-DG仅为0.25 min左右,在其它温度时ReTMan26的半衰期也比ReTMan26-DG有较明显的提高。
| Enzymes | t1/2 (min) | ||||
| 60 ℃ | 70 ℃ | 80 ℃ | 90 ℃ | 100 ℃ | |
| ReTMan26 | 360±15 | 252±10 | 120±3 | 20±1.0 | 12.00±0.50 |
| ReTMan26-DG | 300±12 | 200±5 | 90±3 | 15±0.5 | 0.25±0.01 |
动物消化道内含有较高浓度的消化蛋白酶,因此,ReTMan26作为饲料酶进行应用需具有较好的抗消化性能,以防被动物所分泌的消化蛋白酶水解而失去作用。
ReTMan26经胃蛋白酶及胰蛋白酶各处理2 h后,剩余酶活分别为70.5%及91.2%,与ReTMan26- DG相比分别提高23.7%及25.6% (图 10)。

|
| 图 10 ReTMan26与ReTMan26-DG抗消化蛋白酶的性能 Figure 10 Resistance of ReTMan26 and ReTMan26-DG to digestive proteases |
|
|
针对毕赤酵母Orig-pPICZαA-X33所产的ReTMan26耐高温性能比其基因来源菌株(嗜热枯草芽孢杆菌TBS2)所产的BS-Man有明显提高的现象,本研究通过构建ReTMan26的蛋白质三维结构模型,初步判断其中存在的3个N-糖基化位点经毕赤酵母修饰后,可能会对该酶的稳定性产生一定影响。为此采用天然蛋白去糖基化试剂盒脱除了ReTMan26中的N-多糖链,并对脱除N-糖基化前后的ReTMan26进行了相应的稳定性对比实验。
脱N-糖基化前后的ReTMan26最适反应pH均为6.0,这与基因来源于枯草芽胞杆菌MAFIC-S11并在毕赤酵母中表达的β-甘露聚糖酶相同[14]。该结果说明N-糖基化对ReTMan26的最适反应pH基本上不会产生影响,这是由于酶的最适反应pH一般由其关键催化位点或催化域确定,N-糖基化对其催化特性并不会产生影响。虽然如此,在pH 1.5−9.0范围内,N-糖基化可以小幅提高ReTMan26的pH稳定性,这可能因为在酸碱处理时,处于合适位置的N-多糖链能对关键催化位点或催化域提供一定程度的保护,减少酶蛋白的变性或失活,从而提高其pH稳定性。
ReTMan26的最适反应温度为60 ℃,与来源于枯草芽胞杆菌WL-3的β-甘露聚糖酶[15]及来源于烟色曲霉(A. fumigates)的β-甘露聚糖酶MANA相同[16],而脱除N-糖基化后最适反应温度为55 ℃。通常在一定范围内,反应温度越高,酶的催化活性越强,没有N-多糖链的影响,酶与底物可更好地接触,获得更高的酶促反应速率,所以去除N-糖基化后的酶可表现出更低的最适反应温度。
在耐热性能方面,经100 ℃处理10 min后ReTMan26剩余酶活仍达到58.6%,而去除N-糖基化后,该酶经93 ℃处理10 min后剩余酶活为58.2%,100 ℃处理10 min则完全失活。与之相对应,在100 ℃时,ReTMan26的半衰期约为12 min,去除N-糖基化后的半衰期仅为0.25 min左右。这说明通过毕赤酵母形成的N-糖基化能明显提高ReTMan26的耐热稳定性。类似地,Zou等报道了一种通过毕赤酵母表达的重组β-葡糖苷酸酶(β-glucuronidase) GUS,经65 ℃处理2 h,脱N-多糖链后的GUS完全失活,而含N-糖基化的GUS酶活仍保留80%左右[17]。另外,Han等报道,基因来源于铜绿假单孢菌(Pseudomonas aeruginosa)并在毕赤酵母中表达的重组弹性蛋白酶rPAE,含N-糖基化的rPAE在70 ℃的半衰期为32.2 min,而去除N-多糖链后的rPAE在70 ℃的半衰期仅为23.1 min[18]。以上这些结果佐证了N-糖基化可以提高蛋白质经受各种变性条件(如变性剂、高温等)的稳定性[19]。
在体外模拟实验中,ReTMan26抗胃蛋白酶与胰蛋白酶的消化性能比去除N-糖基化后分别提高了23.7%及25.6%,造成这种现象的原因可能是毕赤酵母在表达外源蛋白时,经翻译后修饰形成的N-多糖链能包裹住蛋白分子中的某些蛋白酶水解位点或催化域中的一些重要位点,如ReTMan26中的酸碱催化及亲核催化位点(Glu149、Glu283)等,从而提高其对消化蛋白酶的抗性。
因此,N-糖基化可提高ReTMan26的pH稳定性、耐热稳定性,及抗胃蛋白酶、胰蛋白酶的消化性能,这对该酶的应用及深入研究具有重要意义。另外,本研究结果也可为今后采用N-糖基化方法提高蛋白质稳定性的定向改造提供参考。
| [1] |
Chauhan PS, Puri N, Sharma P, et al. Mannanases: microbial sources, production, properties and potential biotechnological applications[J]. Applied Microbiology and Biotechnology, 2012, 93(5): 1817-1830. DOI:10.1007/s00253-012-3887-5 |
| [2] |
Choct M, Annison G. The inhibition of nutrient digestion by wheat pentosans[J]. British Journal of Nutrition, 1992, 67(1): 123-132. DOI:10.1079/BJN19920014 |
| [3] |
Turner P, Mamo G, Karlsson EN. Potential and utilization of thermophiles and thermostable enzymes in biorefining[J]. Microbial Cell Factories, 2007, 6: 9. DOI:10.1186/1475-2859-6-9 |
| [4] |
Viikari L, Alapuranen M, Puranen T, et al. Thermostable enzymes in lignocellulose hydrolysis[A]//Advances in Biochemical Engineering/Biotechnology[M]. Vol. 108. Berlin, Heidelberg: Springer, 2007: 121-145
|
| [5] |
Luo ZC, Miao J, Li GY, et al. Screening and identification of a strain producing a highly thermostable β-mannanase[J]. Letters in Biotechnology, 2018, 29(2): 233-237. (in Chinese) 罗长财, 缪静, 李国莹, 等. 一株产耐高温β-甘露聚糖酶菌株的筛选鉴定[J]. 生物技术通讯, 2018, 29(2): 233-237. DOI:10.3969/j.issn.1009-0002.2018.02.015 |
| [6] |
Çelik E, Çalık P. Production of recombinant proteins by yeast cells[J]. Biotechnology Advances, 2012, 30(5): 1108-1118. DOI:10.1016/j.biotechadv.2011.09.011 |
| [7] |
Macauley-Patrick S, Fazenda ML, McNeil B, et al. Heterologous protein production using the Pichia pastoris expression system[J]. Yeast, 2005, 22(4): 249-270. DOI:10.1002/(ISSN)1097-0061 |
| [8] |
Luo ZC, Miao J, Li GY, et al. A recombinant highly thermostable β-mannanase (ReTMan26) from thermophilic Bacillus subtilis (TBS2) expressed in Pichia pastoris and its pH and temperature stability[J]. Applied Biochemistry and Biotechnology, 2017, 182(4): 1259-1275. DOI:10.1007/s12010-017-2397-4 |
| [9] |
Gellissen G. Heterologous protein production in methylotrophic yeasts[J]. Applied Microbiology and Biotechnology, 2000, 54(6): 741-750. DOI:10.1007/s002530000464 |
| [10] |
Damasceno LM, Huang CJ, Batt CA. Protein secretion in Pichia pastoris and advances in protein production[J]. Applied Microbiology and Biotechnology, 2012, 93(1): 31-39. DOI:10.1007/s00253-011-3654-z |
| [11] |
Skropeta D. The effect of individual N-glycans on enzyme activity[J]. Bioorganic & Medicinal Chemistry, 2009, 17(7): 2645-2653. |
| [12] |
Zhao YJ, Xue YF, Ma YH. Recent advances and prospect on structural biology of β-mannanase - a review[J]. Acta Microbiologica Sinica, 2009, 49(9): 1131-1137. (in Chinese) 赵月菊, 薛燕芬, 马延和. β-甘露聚糖酶的结构生物学研究现状和展望[J]. 微生物学报, 2009, 49(9): 1131-1137. DOI:10.3321/j.issn:0001-6209.2009.09.001 |
| [13] |
Hilge M, Gloor SM, Rypniewski W, et al. High-resolution native and complex structures of thermostable β-mannanase from Thermomonospora fusca-substrate specificity in glycosyl hydrolase family 5[J]. Structure, 1998, 6(11): 1433-1444. DOI:10.1016/S0969-2126(98)00142-7 |
| [14] |
Lv JN, Chen YQ, Pei HL, et al. Cloning, expression, and characterization of β-mannanase from Bacillus subtilis MAFIC-S11 in Pichia pastoris[J]. Applied Biochemistry and Biotechnology, 2013, 169(8): 2326-2340. DOI:10.1007/s12010-013-0156-8 |
| [15] |
Yoon KH, Chung S, Lim BL. Characterization of the Bacillus subtilis WL-3 mannanase from a recombinant Escherichia coli[J]. The Journal of Microbiology, 2008, 46(3): 344-349. DOI:10.1007/s12275-008-0045-y |
| [16] |
Puchart V, Vršanská M, Svoboda P, et al. Purification and characterization of two forms of endo-β-1, 4-mannanase from a thermotolerant fungus, Aspergillus fumigatus IMI 385708 (formerly Thermomyces lanuginosus IMI 158749)[J]. Biochimica et Biophysica Acta (BBA) - General Subjects, 2004, 1674(3): 239-250. DOI:10.1016/j.bbagen.2004.06.022 |
| [17] |
Zou SP, Huang S, Kaleem I, et al. N-glycosylation enhances functional and structural stability of recombinant β-glucuronidase expressed in Pichia pastoris[J]. Journal of Biotechnology, 2013, 164(1): 75-81. DOI:10.1016/j.jbiotec.2012.12.015 |
| [18] |
Han MH, Wang XF, Ding HY, et al. The role of N-glycosylation sites in the activity, stability, and expression of the recombinant elastase expressed by Pichia pastoris[J]. Enzyme and Microbial Technology, 2014, 54: 32-37. DOI:10.1016/j.enzmictec.2013.09.014 |
| [19] |
Kwon KS, Yu MH. Effect of glycosylation on the stability of α1-antitrypsin toward urea denaturation and thermal deactivation[J]. Biochimica et Biophysica Acta (BBA)-General Subjects, 1997, 1335(3): 265-272. DOI:10.1016/S0304-4165(96)00143-2 |
 2019, Vol. 46
2019, Vol. 46




