扩展功能
文章信息
- 朱春苗, 蒋群, 张风丽, 李志勇
- ZHU Chun-Miao, JIANG Qun, ZHANG Feng-Li, LI Zhi-Yong
- 海绵共生萎缩芽孢杆菌C89 PPTase bap基因的异源表达
- Heterologous expression of phosphopantetheinyl transferase gene bap from marine sponge-associated Bacillus atrophaeus C89
- 微生物学通报, 2018, 45(9): 2014-2021
- Microbiology China, 2018, 45(9): 2014-2021
- DOI: 10.13344/j.microbiol.china.180296
-
文章历史
- 收稿日期: 2018-04-12
- 接受日期: 2018-05-25
- 网络首发日期(www.cnki.net): 2018-06-28
海绵共生微生物能产生具有潜在药用价值的次级代谢产物,包括非核糖体肽类化合物[1-2]。为了解海绵共生微生物合成非核糖体肽类化合物的潜能,Pimentel-Elardo等[3]对海绵Aplysina aerophoba的宏基因组进行了研究,发现其共生绿弯菌中有一个非核糖体肽合成酶(NRPS)基因簇,并在该基因簇下游发现了一个磷酸泛酰巯基乙胺基转移酶(PPTase)基因lubD。肽酰载体蛋白(PCP)是NRPS的关键功能域,PPTase催化磷酸泛酰巯基乙胺基从辅酶A转移到PCP活性丝氨酸的残基上,将PCP由无活性的脱辅基形态(apo-)转化为有活性的全辅基形态(holo-)[4-5]。PPTase作为激活NRPS中PCP的活性开关,在非核糖体肽类化合物的生物合成中起着不可或缺的作用[5]。根据蛋白质结构,PPTase分为3种类型[4]:Ⅰ型为AcpS型,约120个氨基酸,含P2 (GxD)、P3 [(F/W)(S/T/A)xKE(S/A)xxK]两个保守结构域[6];Ⅱ型为Sfp型,约240个氨基酸,含P1 (PxxP)、P2和P3三个保守结构域[4-7];Ⅲ型PPTase是融合在聚酮合酶(PKS)/脂肪酸合酶(FAS)上的一个结构域。一般而言,NRPS的PCP由Sfp型PPTase催化[7]。
萎缩芽孢杆菌(Bacillus atrophaeus)是重要的工业菌株[8-9]。本实验室从中国南海贪婪倔海绵中分离到B. atrophaeus C89,该菌株产具有抗藻活性[10]的非核糖体肽类化合物Bacillamide C[11]及其类似物Neobacillamide A[12],我们推测B. atrophaeus C89中的nrps基因簇[13]和一个Sfp型PPTase基因参与了Bacillamides的生物合成。Bacillamides在自然条件下生物合成量低,化学合成易造成环境污染,因此nrps基因簇的异源表达是解决其生物合成产量低和解析Bacillamides生物合成途径的重要手段。NRPS的关键功能域PCP需要Sfp型PPTase催化完成翻译后修饰,所以B. atrophaeus C89中Sfp型PPTase对B. atrophaeus C89中Bacillamides的生物合成途径研究意义重大。
Bacillus subtilis 168是芽孢杆菌异源表达常用模式菌株,该菌株中Sfp型PPTase基因sfp突变[6],不能催化NRPS中PCP,因此在B. subtilis 168中表达NRPS时需要引入能够催化该NRPS中PCP的PPTase[14]。Zobel等[15]将来源于真菌的一个NRPS与Sfp在B. subtilis 168中共表达,获得了相应的活性化合物Enniatin。Surfactin是枯草芽孢杆菌合成的非核糖体肽类化合物[7],具有溶血活性[16]。B. subtilis 168有完整的Surfactin操纵元基因srfA[17],但其sfp基因突变不能产生Surfactin[7]。因此在B. subtilis 168中表达Sfp型PPTase时,可通过溶血测试检测Surfactin是否合成,从而判断表达的PPTase能否激活NRPS中PCP[15]。刘丽霞等[18]将B. subtilis Nja-9的sfp基因整合到B. subtilis 168的基因组中,通过溶血测试筛选到产Surfactin的重组菌。因此,在研究B. atrophaeus C89中Sfp型PPTase的功能时,选择B. subtilis 168为异源宿主,以Surfactin为指示剂,通过检测重组菌是否合成Surfactin,验证该PPTase能否激活NRPS中PCP。
本研究首先通过生物信息学分析找到B. atrophaeus C89中Sfp型PPTase Bap,然后将bap基因在B. subtilis 168中异源表达,获得了产Surfactin的重组菌B. subtilis 168-bap,说明Bap在B. subtilis 168中成功表达,并能激活Surfactin合成酶NRPS中所有的PCP (图 1)。
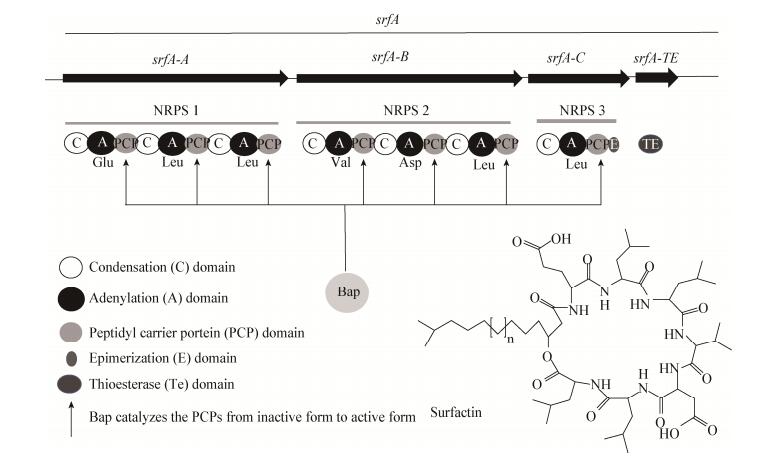
|
| 图 1 重组菌B. subtilis 168-bap中Surfactin的生物合成示意图 Figure 1 Schematic diagram of the surfactin biosynthesis of recombinant B. subtilis 168-bap 注:Surfactin的编码基因srfA-ABC编码合成3个NRPS,srfA-TE编码合成1个硫酯酶,NRPS中的A结构域识别氨基酸作为底物[19],PPTase Bap将辅因子4′磷酸泛酰巯基乙胺基转移到每个PCP结构域上将其激活从而启动Surfactin的合成. Note: The encoding genes srfA-ABC of surfactin encode three NRPSs and the gene srfA-TE encodes a thioesterase. Aminos are recognized by A domains of NRPSs as substrates[19]. PPTase Bap introduces the 4′-Ppan to each PCP domain to activate them so that initiating the synthesis of surfactin. |
|
|
B. atrophaeus C89由本实验室分离保存[13];B. subtilis 168由本实验室保存;枯草芽孢杆菌表达载体pHT01由上海科技大学黄娇芳老师惠赠;克隆载体pEASY-T1 Simple、克隆感受态细胞大肠杆菌Trans1-T1购自北京全式金生物技术有限公司。
1.1.2 培养基枯草芽孢杆菌和大肠杆菌培养用LB (Luria-Bertani)培养基(g/L):酵母粉5.0,蛋白胨10.0,氯化钠10.0,固体培养基中加入1.5%琼脂粉。B. atrophaeus C89培养用人工海水配制的LB培养基,人工海水(g/L):氯化钠26.518,氯化镁2.447,硫酸镁3.305,氯化钙1.141,氯化钾0.725,碳酸氢钠0.202,溴化钠0.083,pH 7.0-7.2。枯草芽孢杆菌电转感受态细胞制备用培养基:生长培养基GM (g/L):酵母粉5.0,蛋白胨10.0,氯化钠10.0,山梨醇90.0,pH 7.2;复苏培养基RM (g/L):酵母粉5.0,蛋白胨10.0,氯化钠10.0,山梨醇90.0,甘露醇70.0;电击缓冲液ETM (g/L):山梨醇90.0,甘露醇92.5,甘油100 mL。溶血测试用血琼脂培养基(g/L):蛋白胨10.0,牛肉膏3.0,氯化钠5.0,脱纤维羊血50 mL,琼脂粉15.0。
1.1.3 主要试剂和仪器氨苄青霉素、氯霉素、异丙基-β-D-硫代半乳糖苷(IPTG),生工生物工程(上海)股份有限公司;Surfactin标准品,西格玛奥德里奇贸易有限公司;细菌基因组DNA快速提取试剂盒,上海捷瑞生物工程有限公司;高保真酶KOD-Plus-Neo,东洋纺(上海)生物科技有限公司;DNA凝胶回收试剂盒,宝生物工程(大连)有限公司;2×EasyTaq PCR Super Mix、质粒小提试剂盒、连接酶,北京全式金生物技术有限公司;限制性内切酶,NEB (北京)有限公司;甲醇、乙腈,上海星可高纯溶剂有限公司。
振荡培养箱,上海旻泉仪器有限公司;电热恒温培养箱,上海新苗医疗器械制造有限公司;超高效液相色谱-四极杆飞行时间质谱联用仪,沃特世公司。
1.2 方法 1.2.1 B. atrophaeus C89中PPTase的生物信息学分析检索B. atrophaeus C89基因组(JQ687535)[13]中的PPTase序列,利用NCBI对检索到的PPTase序列进行BLAST。通过DNAMAN对BLAST结果显示为Sfp型PPTase的Bap序列与2个已知的Sfp型PPTase序列进行比对,这两个编码蛋白分别为来源于真菌Evernia prunastri的EppA (ALS87659.1)[20]和来源于B. subtilis的Sfp (BAA09125.1)[7],检测Bap是否含有Sfp型PPTase的3个保守结构域。
1.2.2 bap基因的克隆和重组表达载体pHT-bap的构建28 ℃、180 r/min培养过夜的B. atrophaeus C89菌液,6 000 r/min离心1 min收集菌体,用细菌基因组DNA快速提取试剂盒提取B. atrophaeus C89基因组DNA。以基因组DNA为模板,根据bap基因(675 bp)设计引物进行PCR扩增:Fbap (5′-GCTCTAGAATGAAGATCTATGGAATTTGTATGG-3′)和Rbap (5′-TCCCCCCGGGCTATAGCAGC TCTTCATACATTTTC-3′),下划线为酶切位点。PCR反应体系(50 μL):模板(100 ng/μL) 1 μL,KOD-Plus-Neo (1 U/μL) 1 μL,dNTPs (2 mmol/L) 5 μL,MgSO4 (25 mmol/L) 3 μL,上、下游引物(10 μmol/L)各1.5 μL,10×PCR buffer 5 μL,添加无菌双蒸水至50 μL。PCR反应条件:94 ℃ 2 min;98 ℃ 10 s,55 ℃ 30 s,68 ℃ 30 s,38个循环;72 ℃ 10 min。切胶回收目的片段,连接到pEASY-T1 Simple载体上,转化大肠杆菌Trans1-T1。挑转化子送至上海美吉生物医药科技有限公司测序,得到重组载体,命名为T1-bap。
重组载体T1-bap用Xba Ⅰ/Xma Ⅰ双酶切后回收连接到用同样的酶处理过的pHT01上,转化大肠杆菌Trans1-T1。挑转化子进行菌落PCR检测,引物为pHbapF (5′-ATTAAAGGAGGAAGGATCCTCT AG-3′)和pHbapR (5′-CCCGGGCTATAGCAGCTCT TCATAC-3′)。PCR反应体系(25 μL):2×EasyTaq PCR Super Mix 12.5 μL,上、下游引物(10 μmol/L)各1 μL,添加无菌双蒸水至25 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。提取阳性菌株质粒进行电泳检测,重组质粒命名为pHT-bap。
1.2.3 重组质粒pHT-bap电转化B. subtilis 168制备B. subtilis 168电转感受态细胞:将B. subtilis 168在LB固体培养基上划线,37 ℃培养过夜。挑单菌落于5 mL LB中,37 ℃、200 r/min培养过夜。将获得的菌液以1:16的比例接种于50 mL GM中,37 ℃、200 r/min培养至OD600为0.85-0.95,冰浴10 min。4 ℃、5 000 r/min离心10 min,收集菌体。用40 mL预冷的ETM洗涤菌体,4 ℃、5 000 r/min离心10 min,去上清,ETM洗涤3次,将洗涤后的菌体重悬于500 μL ETM中,每管分装60 μL,-70 ℃保存。
电转实验中实验组为pHT-bap质粒,对照组为pHT01质粒。向60 μL B. subtilis 168感受态细胞中加入1-6 μL质粒DNA,混匀,转移至预冷的电转杯(1 mm Gap)中,冰浴5 min,置于电转仪中,电击条件:2.0 kV,25 μF,200 Ω,4.5-5.0 ms。电击结束后立即加入1 mL RM,37 ℃、200 r/min培养3 h,涂布于含氯霉素(终浓度5 μg/mL)的LB固体培养基上,37 ℃培养过夜。提取质粒,电泳检测阳性重组菌B. subtilis 168-bap和空载对照菌B. subtilis 168/pHT01。
1.2.4 溶血测试检测重组菌B. subtilis 168-bap合成Surfactin挑取15个重组菌落B. subtilis 168-bap点接于含诱导剂IPTG (终浓度1 mmol/L)和不含诱导剂的血琼脂抗性平板上,37 ℃培养48 h,通过溶血圈检测重组菌能否合成Surfactin。
1.2.5 重组菌B. subtilis 168-bap发酵产物提取及UPLC-Q/TOF MS检测将产生溶血圈的重组菌B. subtilis 168-bap与对照B. subtilis 168/pHT01接种于5 mL LB抗性培养基中,37 ℃、200 r/min培养过夜。将该菌液以1:100比例接种于300 mL LB中,37 ℃、200 r/min培养至OD600为0.8,加入终浓度1 mmol/L IPTG诱导目的蛋白表达,继续培养36 h,酸沉淀法[21]提取发酵产物中的脂肽化合物,将得到的脂肽化合物和Surfactin标准品溶液(1 mg/mL)经0.22 μm有机滤膜过滤,进行UPLC-Q/TOF MS检测。
色谱柱为BEH C18 (100 mm×2.1 mm,1.7 μm),柱温50 ℃。Surfactin标准品进样量为0.2 μL,其他样品进样量为3 μL。流动相A:超纯水,含0.1%甲酸;流动相B:甲醇:乙腈:异丙醇=2:2:1,含0.1%甲酸。梯度洗脱条件为:0-0.5 min 75% B,0.5-7.0 min 75%-95% B,7-9 min 95%-100% B,流速0.35 mL/min。质谱离子扫描模式为正离子模式。
2 结果与分析 2.1 B. atrophaeus C89中PPTase的生物信息学分析通过B. atrophaeus C89全基因组扫描发现,B. atrophaeus C89中有2个PPTase序列。BLAST结果显示,其中长度为224个氨基酸的PPTase序列为Sfp型PPTase,将该基因命名为bap (B. atrophaeus C89 PPTase,序列已提交至GenBank,登录号为MH095983),编码蛋白Bap。氨基酸多序列比对结果显示,Bap含有Sfp型PPTase所特有的P1 (PYAP)、P2 (GAD)和P3 (WSMKESFIK) 3个保守结构域(图 2)。

|
| 图 2 B. atrophaeus C89中Bap的序列比对 Figure 2 Sequence alignment of Bap from B. atrophaeus C89 注:Sfp (BAA09125.1):来源于B. subtilis;EppA (ALS87659.1):来源于E. prunastri;P1、P2、P3表示Sfp型PPTase所特有的3个保守结构域. Note: Sfp (BAA09125.1): From B. subtilis; EppA (ALS87659.1): From E. prunastri; P1, P2, P3 represent the 3 conserved regions of Sfp-type PPTases. |
|
|
获得的重组菌B. subtilis 168-bap在含诱导剂IPTG和不含诱导剂的血琼脂平板上培养过夜后,均长出菌落。继续培养至48 h,含诱导剂的平板上产生溶血圈(图 3A),无诱导剂的平板上不产生溶血圈(图 3B),说明含Bap的重组菌合成了溶血活性化合物Surfactin。

|
| 图 3 重组菌B. subtilis 168-bap溶血测试 Figure 3 Haematolysis test of recombinant B. subtilis 168-bap 注:A:培养基中有诱导剂IPTG;B:培养基中无诱导剂. Note: A: Addition of IPTG to the medium; B: No IPTG in the medium. |
|
|
MS测定重组菌B. subtilis 168-bap和B. subtilis 168/pHT01发酵产物中脂肽化合物的分子量,并与Surfactin标准品的分子量(图 4)对比。结果显示,Surfactin标准品在保留时间5.23 min有一个质谱峰(图 4A),其对应的m/z值(M+H+)为1 036.69 (图 4B)。B. subtilis 168-bap的脂肽化合物在5.23 min有对照菌B. subtilis 168/pHT01没有的质谱峰(图 5A、B),且其m/z值(M+H+)为1 036.69 (图 5C),说明B. subtilis 168-bap合成了Surfactin。
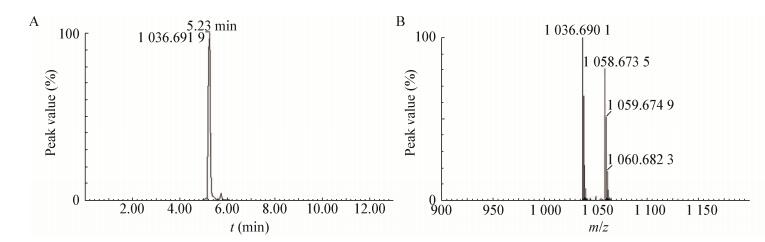
|
| 图 4 Surfactin标准品的MS检测结果 Figure 4 MS analysis of standard surfactin |
|
|

|
| 图 5 重组菌脂肽化合物的MS检测结果 Figure 5 MS analysis of lipopeptide compounds from the recombinant strains 注:A、C:B. subtilis 168-bap脂肽化合物的MS检测结果;B:B. subtilis 168/pHT01脂肽化合物的MS检测结果. Note: A, C: MS analysis of lipopeptide compounds from B. subtilis 168-bap; B: MS analysis of lipopeptide compounds from B. subtilis 168/pHT01. |
|
|
PPTase是非核糖体肽类化合物生物合成途径中重要的调控因子。本研究鉴定了中国南海贪婪倔海绵共生B. atrophaeus C89中Sfp型PPTase Bap,同时构建的重组菌B. subtilis 168-bap能够合成非核糖体肽类化合物Surfactin,证实B. atrophaeus C89的Sfp型PPTase能在B. subtilis 168中异源表达,并能催化宿主中NRPS的PCP从无活性的脱辅基形态转化为有活性的全辅基形态,从而启动非核糖体肽类化合物的生物合成。一般认为产Surfactin的枯草芽孢杆菌都有sfp基因[15],Hsieh等[22]甚至以sfp基因为靶点筛选产Surfactin的枯草芽孢杆菌。B. subtilis 168-bap成功合成Surfactin,一方面说明bap基因可以回补sfp基因的功能,能够激活NRPS的PCP,另一方面说明B. subtilis 168-bap具有表达海洋萎缩芽孢杆菌中NRPS基因簇的潜能。
萎缩芽孢杆菌是一种顽固细菌,难以利用常规分子生物学手段实现外源DNA的遗传转化[23],因此难以直接在萎缩芽孢杆菌上进行遗传操作研究其活性化合物的生物合成途径。异源表达是研究萎缩芽孢杆菌中基因的重要手段[24]。枯草芽孢杆菌被认为是一种安全的微生物,该菌遗传背景清晰,遗传操作较为成熟,是很好的异源宿主[15],因此萎缩芽孢杆菌NRPS基因簇异源表达可考虑以枯草芽孢杆菌为异源宿主。本研究构建的重组菌B. subtilis 168-bap来源于芽孢杆菌异源表达常用模式菌株B. subtilis 168,含有连接了B. atrophaeus C89 Sfp型PPTase基因bap的重组表达载体pHT-bap,并能表达具有Sfp型PPTase生物活性的Bap。因此,可考虑以B. subtilis 168-bap为宿主表达海洋萎缩芽孢杆菌的NRPS基因簇并对其进行遗传操作。本研究为海洋萎缩芽孢杆菌NRPS生物合成基因簇的异源表达奠定了基础。
致谢: 感谢上海科技大学黄娇芳老师惠赠pHT01载体。| [1] |
Faulkner DJ. Marine natural products[J]. Natural Product Reports, 2001, 18(1): 1-49. DOI:10.1039/b006897g |
| [2] |
Sipkema D, Osinga R, Schatton W, et al. Large-scale production of pharmaceuticals by marine sponges: sea, cell, or synthesis?[J]. Biotechnology and Bioengineering, 2005, 90(2): 201-222. DOI:10.1002/(ISSN)1097-0290 |
| [3] |
Pimentel-Elardo SM, Grozdanov L, Proksch S, et al. Diversity of nonribosomal peptide synthetase genes in the microbial metagenomes of marine sponges[J]. Marine Drugs, 2012, 10(6): 1192-1202. |
| [4] |
Beld J, Sonnenschein EC, Vickery CR, et al. The phosphopantetheinyl transferases: catalysis of a post-translational modification crucial for life[J]. Natural Product Reports, 2014, 31(1): 61-108. DOI:10.1039/C3NP70054B |
| [5] |
Yao LD, Wang YY, Fan WM, et al. Substrate specificity of phosphopantetheinyl transferases to carrier proteins in Streptomyces[J]. Microbiology China, 2014, 41(6): 1188-1194. (in Chinese) 姚黎栋, 王月月, 樊伟明, 等. 链霉菌磷酸泛酰巯基乙胺基转移酶对载体蛋白的底物选择性的研究进展[J]. 微生物学通报, 2014, 41(6): 1188-1194. |
| [6] |
Lambalot RH, Gehring AM, Flugel RS, et al. A new enzyme superfamily - the phosphopantetheinyl transferases[J]. Chemistry & Biology, 1996, 3(11): 923-936. |
| [7] |
Quadri LEN, Weinreb PH, Lei M, et al. Characterization of Sfp, a Bacillus subtilis phosphopantetheinyl transferase for peptidyl carrier protein domains in peptide synthetases[J]. Biochemistry, 1998, 37(6): 1585-1595. DOI:10.1021/bi9719861 |
| [8] |
Sella SRBR, Vandenberghe LPS, Soccol CR. Bacillus atrophaeus: main characteristics and biotechnological applications - a review[J]. Critical Reviews in Biotechnology, 2015, 35(4): 533-545. DOI:10.3109/07388551.2014.922915 |
| [9] |
Mwita L, Chan WY, Pretorius T, et al. Gene expression regulation in the plant growth promoting Bacillus atrophaeus UCMB-5137 stimulated by maize root exudates[J]. Gene, 2016, 590(1): 18-28. DOI:10.1016/j.gene.2016.05.045 |
| [10] |
Jeong SY, Ishida K, Ito Y, et al. Bacillamide, a novel algicide from the marine bacterium, Bacillus sp. SY-1, against the harmful dinoflagellate, Cochlodinium polykrikoides[J]. Tetrahedron Letters, 2003, 44(43): 8005-8007. DOI:10.1016/j.tetlet.2003.08.115 |
| [11] |
Yuwen L, Zhang FL, Chen QH, et al. The role of aromatic L-amino acid decarboxylase in bacillamide C biosynthesis by Bacillus atrophaeus C89[J]. Scientific Reports, 2013, 3: 1753. DOI:10.1038/srep01753 |
| [12] |
Yu LL, Li ZY, Peng CS, et al. Neobacillamide A, a novel thiazole-containing alkaloid from the marine bacterium Bacillus vallismortis C89, associated with South China Sea Sponge Dysidea avara[J]. Helvetica Chimica Acta, 2009, 92(3): 607-612. DOI:10.1002/hlca.v92:3 |
| [13] |
Liu F, Sun W, Su F, et al. Draft genome sequence of the sponge-associated strain Bacillus atrophaeus C89, a potential producer of marine drugs[J]. Journal of Bacteriology, 2012, 194(16): 4454. DOI:10.1128/JB.00835-12 |
| [14] |
Tsuge K, Matsui K, Itaya M. Production of the non-ribosomal peptide plipastatin in Bacillus subtilis regulated by three relevant gene blocks assembled in a single movable DNA segment[J]. Journal of Biotechnology, 2007, 129(4): 592-603. DOI:10.1016/j.jbiotec.2007.01.033 |
| [15] |
Zobel S, Kumpfmüller J, Süssmuth RD, et al. Bacillus subtilis as heterologous host for the secretory production of the non-ribosomal cyclodepsipeptide enniatin[J]. Applied Microbiology and Biotechnology, 2015, 99(2): 681-691. DOI:10.1007/s00253-014-6199-0 |
| [16] |
Cosmina P, Rodriguez F, Ferra F, et al. Sequence and analysis of the genetic locus responsible for surfactin synthesis in Bacillus subtilis[J]. Molecular Microbiology, 1993, 8(5): 821-831. DOI:10.1111/mmi.1993.8.issue-5 |
| [17] |
Bernheimer AW, Avigad LS. Nature and properties of a cytolytic agent produced by Bacillus subtilis[J]. Journal of General Microbiology, 1970, 61(3): 361-369. DOI:10.1099/00221287-61-3-361 |
| [18] |
Liu LX, Gao L, Bie XM, et al. Reconstruction of Bacillus subtilis 168 to produce surfactin[J]. Journal of Nanjing Agricultural University, 2014, 37(6): 97-102. (in Chinese) 刘丽霞, 高玲, 别小妹, 等. Bacillus subtilis 168菌株产surfactin改造[J]. 南京农业大学学报, 2014, 37(6): 97-102. |
| [19] |
Eppelmann K, Stachelhaus T, Marahiel MA. Exploitation of the selectivity-conferring code of nonribosomal peptide synthetases for the rational design of novel peptide antibiotics[J]. Biochemistry, 2002, 41(30): 9718-9726. DOI:10.1021/bi0259406 |
| [20] |
Schimming O, Schmitt I, Bode HB. Identification of the Sfp-Type PPTase EppA from the lichenized fungus Evernia prunastri[J]. PLoS One, 2016, 11(1): e0145624. DOI:10.1371/journal.pone.0145624 |
| [21] |
Wang S, Gao SF, Gao XW, et al. Fermentation optimization in lipopeptide productivity of Bacillus subtilis G1[J]. Chinese Journal of Biological Control, 2007, 23(4): 342-347. (in Chinese) 王帅, 高圣风, 高学文, 等. 枯草芽孢杆菌脂肽类抗生素发酵和提取条件[J]. 中国生物防治学报, 2007, 23(4): 342-347. |
| [22] |
Hsieh FC, Li MC, Lin TC, et al. Rapid detection and characterization of surfactin-producing Bacillus subtilis and closely related species based on PCR[J]. Current Microbiology, 2004, 49(3): 186-191. |
| [23] |
Aune TEV, Aachmann FL. Methodologies to increase the transformation efficiencies and the range of bacteria that can be transformed[J]. Applied Microbiology and Biotechnology, 2010, 85(5): 1301-1313. DOI:10.1007/s00253-009-2349-1 |
| [24] |
Saini M, Bindal S, Gupta R. Heterologous expression of γ-glutamyl transpeptidase from Bacillus atrophaeus GS-16 and its application in the synthesis of γ-D-glutamyl-L-tryptophan, a known immunomodulatory peptide[J]. Enzyme and Microbial Technology, 2017, 99: 67-76. DOI:10.1016/j.enzmictec.2017.01.003 |
 2018, Vol. 45
2018, Vol. 45




