扩展功能
文章信息
- 曲雯雯, 吴蕾蕾, 周顺, 史晓翀, 张晓华
- QU Wen-Wen, WU Lei-Lei, ZHOU Shun, SHI Xiao-Chong, ZHANG Xiao-Hua
- 采用随机突变方法对β-琼胶酶YM01-3活性位点的研究
- Exploration on the active sites of β-agarase YM01-3 with random mutation
- 微生物学通报, 2018, 45(9): 2000-2005
- Microbiology China, 2018, 45(9): 2000-2005
- DOI: 10.13344/j.microbiol.china.180449
-
文章历史
- 收稿日期: 2018-06-06
- 接受日期: 2018-07-17
- 网络首发日期(www.cnki.net): 2018-08-15
2. 青岛海洋科学与技术试点国家实验室 海洋生态与环境科学功能实验室 山东 青岛 266071
2. Laboratory for Marine Ecology and Environmental Science, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao, Shandong 266071, China
琼胶酶是一类能够降解琼胶的糖苷水解酶,根据降解底物方式及产生产物不同,可分为α-琼胶酶(E.C.3.2.1.158)和β-琼胶酶(E.C.3.2.1.81)[1]。目前在糖苷水解酶数据库(CAZy)中,β-琼胶酶的数量远多于α-琼胶酶。琼胶酶在制备琼胶寡糖[2]、制备原生质体[3]、回收琼脂糖凝胶中DNA[4]等方面具有潜在的应用价值。
本实验室前期从一株海洋新属新种嗜琼胶卵链菌YM01中克隆表达出β-琼胶酶YM01-3 (GenBank登录号:KF413621)[5],该酶是一种耐高温琼胶酶,最适反应温度达到60 ℃;在最适反应条件下,β-琼胶酶YM01-3的酶活力为1.14×104 U/mg,是目前报道活性较高的琼胶酶,由于其较高的酶活力及温度稳定性,使其具有良好的应用前景。
随机突变(Random mutation)是在实验室环境下模拟蛋白质自然进化的过程,诱导蛋白质的核酸序列在扩增时产生错配。该过程不需要了解蛋白质三维结构、活性位点等信息,是开发新型酶的常见手段。常见方法有易错PCR、化学诱变等[6]。定点突变(Site-directed mutation)是在特定位点处对氨基酸进行突变、缺失或插入,进而分析该处氨基酸突变对蛋白质结构、活性、底物结合能力等的影响[7]。
本研究利用随机突变和定点突变的方法,确定了2个影响β-琼胶酶YM01-3的新位点,为研究β-琼胶酶YM01-3构效关系及其开发利用奠定了基础。
1 材料与方法 1.1 材料 1.1.1 实验材料、主要试剂和仪器pNCMO2载体、短芽孢杆菌感受态细胞,TaKaRa公司;E. coli JM109感受态细胞、质粒提取试剂盒、琼脂糖凝胶DNA回收试剂盒,博迈德公司;限制性内切酶BamH I、EcoR I,Thermo公司。
PCR仪,Applied Biosystems公司;酶标仪,太康公司;蛋白电泳仪,伯乐公司。
1.1.2 培养基MTNm培养基(g/L):葡萄糖10.0,多聚蛋白胨10.0,牛肉粉5.0,酵母膏2.0,FeSO4 0.01,MnSO4 0.01,ZnSO4 0.001,pH 7.0。固体培养基添加琼脂粉15.0 g。
TM培养基(g/L):葡萄糖10.0,多聚蛋白胨10.0,牛肉粉5.0,酵母膏2.0,FeSO4 0.01,MnSO4 0.01,ZnSO4 0.001,MgCl2 4.1,pH 7.0。
1.2 方法 1.2.1 β-琼胶酶基因YM01-3的随机突变根据GenBank中β-琼胶酶YM01-3的序列设计引物,引物由上海铂尚有限公司合成。YM01-3上游引物5′-CACGGATCCATGTATGCAGCAGA-3′ (划线部分为BamH I的酶切位点);YM01-3下游引物5′-CAGGAATTCGTGGTGGTGGTGGT-3′ (划线部分为EcoR I的酶切位点)。以重组β-琼胶酶YM01-3质粒为模板,进行易错PCR扩增。PCR反应体系:Mn2+ 1.6 mmol/L,Mg2+ 5 mmol/L,dNTPs组分是dATP、dGTP 2.5 mmol/L,dCTP、dTTP 20 mmol/L。PCR反应条件:94 ℃ 5 min;98 ℃ 10 s,56 ℃ 15 s,72 ℃ 90 s,30个循环;72 ℃ 10 min;4 ℃保温。PCR产物与载体pNCMO2连接,并将连接产物转化至E. coli JM109感受态细胞。从E. coli JM109感受态细胞中提取重组质粒,参照宝生物NTP转化方法(www.takarabiomed.com.cn/DownLoad/HB200.pdf),将质粒转入短芽孢杆菌(Bacillus brevis)感受态细胞中,将感受态细胞稀释至合适的浓度涂布于MTNm平板上构建突变体文库。
1.2.2 β-琼胶酶YM01-3突变体的初步筛选及鉴定短芽孢杆菌可将β-琼胶酶YM01-3突变酶分泌到胞外,通过观察宿主菌在琼脂平板上透明圈的大小对突变体酶活进行初步高通量筛选[8]。筛选在琼脂平板上没有活性的克隆,用菌液PCR方法对突变体基因与质粒连接情况进行再次验证,排除质粒与片段连接不正确的克隆。10 μL菌液PCR体系:2×Taq PCR Mix 5 μL,引物T7、T7-Ter (10 mmol/L)各0.1 μL,菌液0.5 μL,ddH2O补充体积到10 μL。
1.2.3 β-琼胶酶YM01-3突变体的复筛将筛选出的阳性突变体转接至5 mL TM液体培养基培养8 h,超纯水将突变体稀释,酶标仪检测至OD600=0.3,取突变体粗酶液利用DNS法测定其酶活力[9]。根据酶活力测定结果,从中挑选低酶活的突变体80株,送生工生物工程(上海)股份有限公司进行测序。
1.2.4 酶活关键位点的分析及验证将突变菌株的氨基酸序列与原始氨基酸序列进行比对,发现Y137、D237是在大多突变体中高频出现的突变位点;参照β-琼胶酶YM01-3的三维结构发现Y137、D237均位于催化腔内,因此推测Y137、D237氨基酸可能是影响酶活变化的关键位点。对Y137、D237两个氨基酸位点进行定点突变验证,突变氨基酸选择为性质完全相反原则,方法为PCR介导的定点突变[10]。Y137A上游引物:5′-AT GGAAGCTGCNGGTAGTGATAGGC-3′,Y137A下游引物:5′-TGCATCTAATTCTTGAGTTGAATCA-3′;D237K上游引物:5′-ATTATTATTAARATGGAACAT CAAG-3′,D237K下游引物:5′-ATGTAATGGTTTGT TGATACCTGTG-3′,下划线为突变氨基酸位点。
1.2.5 突变蛋白酶活测定采用DNS法[9]检测定点突变酶Y137A、D237K的酶活力。
2 结果与分析 2.1 建立随机突变文库通过易错PCR获得的PCR产物与载体pNCMO2连接,转入E. coli JM109感受态细胞。为降低突变体的损失,易错PCR产物质粒在E. coli JM109扩增时选用液体培养基培养。从E. coli JM109中提取易错PCR产物质粒,将易错PCR产物质粒转入短芽孢杆菌中构建随机突变文库。
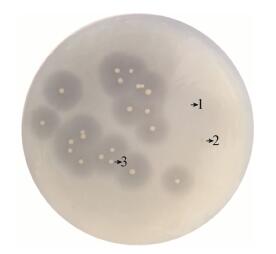
2.2 突变体的初筛通过宿主短芽孢杆菌产生透明圈的大小,利用平板法对随机突变文库中的突变体进行快速筛选。本实验共获得约200个转化平板,构建包含约10 000株突变体的随机突变文库。含突变体的转化平板如图 1所示,菌落1与菌落2完全没有透明圈,推测菌落1与菌落2完全丧失琼胶酶活性;菌落3透明圈直径较小,推测菌落3琼胶酶活性降低。将初筛突变体进行菌液PCR验证其阳性情况,选择菌液PCR结果为阳性的菌株留作下一步筛选,2次筛选得到227株酶活力丧失明显的突变体。
|
| 图 1 易错PCR转化结果 Figure 1 The results of error prone PCR transformation 注:1、2:无透明圈菌落;3:透明圈较小菌落. Note: 1, 2: Colony without transparent circle; 3: Colony with small transparent circle. |
|
|
利用DNS法对获得的227株突变体的琼胶酶酶活进行复筛,从复筛结果中选取酶活最低的80株突变体进行测序。
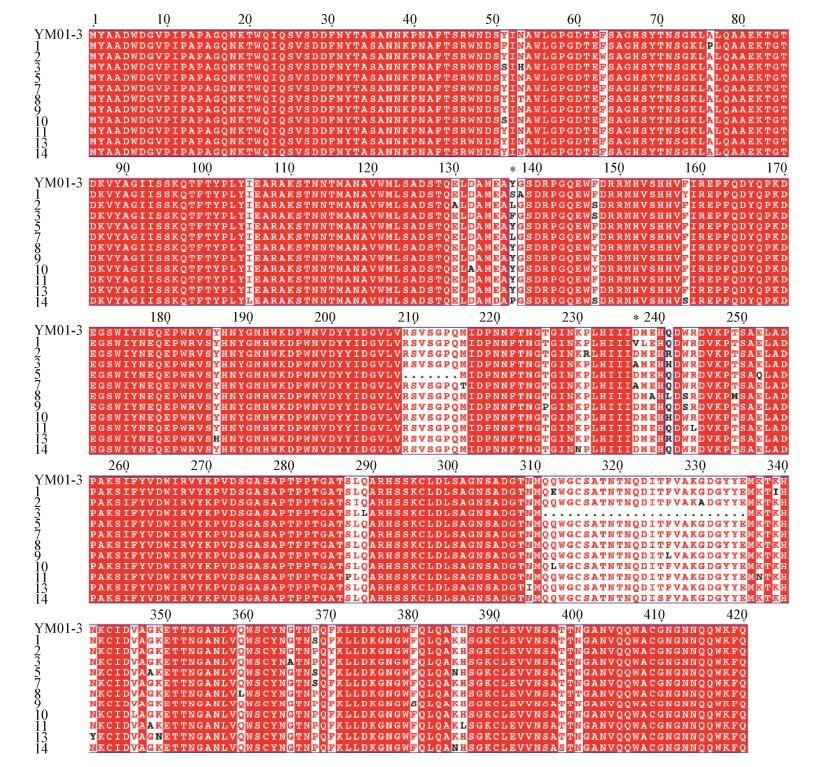
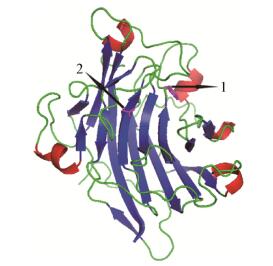
2.4 随机突变菌株测序结果的整理及位点分析将突变体氨基酸序列与β-琼胶酶YM01-3氨基酸序列进行比对(图 2),发现多条序列在Y137、D237处发生突变(突变位点已用*标出)。将β-琼胶酶YM01-3于Swiss-Model网站数据库进行序列比对发现,其与已知三维结构的β-琼胶酶MtAgaA[11]的序列相似度最高,为52.7%。以β-琼胶酶MtAgaA为模型模拟β-琼胶酶YM01-3的三维结构。从β-琼胶酶YM01-3的三维结构可知Y137、D237两个位点均位于催化腔中,如图 3所示,因此推测Y137、D237可能是影响β-琼胶酶YM01-3酶活性丧失的关键位点。
|
| 图 2 β-琼胶酶YM01-3氨基酸序列与突变体多序列比对结果 Figure 2 The alignment of the multiple sequence and β-agarases YM01-3 and mutants |
|
|
|
| 图 3 β-琼胶酶YM01-3结构中突变位点的位置 Figure 3 The sites of mutants in the β-agarase YM01-3 structure 注:1:氨基酸位点Y137;2:氨基酸位点D237. Note: 1: Amino acid site of Y137; 2: Amino acid site of D237. |
|
|
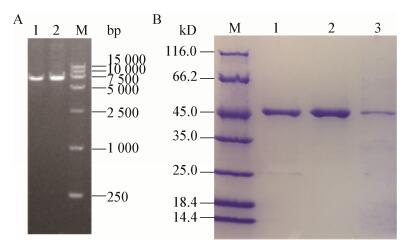
利用PCR介导的定点突变分别对Y137、D237位氨基酸进行突变,将突变PCR产物连接成重组质粒,转入E. coli BL21(DE3)进行表达,并对突变蛋白进行表达及纯化(图 4)。
|
| 图 4 β-琼胶酶基因YM01-3突变体的PCR扩增及纯化蛋白聚丙烯酰胺凝胶电泳 Figure 4 PCR amplification of mutants β-agarase YM01-3 gene and SDS-PAGE analysis of purified protein 注:A:M:15 000 bp DNA marker;1:Y137A突变体基因扩增产物;2:D237K突变体基因扩增产物. B:M:蛋白Marker;1:YM01-3蛋白;2:Y137A突变蛋白;3:D237K突变蛋白. Note: A: M: 15 000 bp DNA marker; 1: PCR production of Y137A mutant gene; 2: PCR production of D237K mutant gene. B: M: Protein marker; 1: Protein of YM01-3; 2: Y137A mutant protein; 3: D237K mutant protein. |
|
|
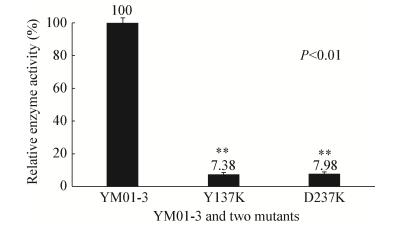
利用DNS法对突变体酶活检测,如图 5所示,突变体Y137A与突变体D237K纯蛋白的酶活力分别为β-琼胶酶YM01-3酶活力的7.38%与7.98%,这2个位点的突变均使原始酶活力显著下降,说明这2个位点是影响β-琼胶酶YM01-3酶活的关键位点。
|
| 图 5 β-琼胶酶YM01-3与突变蛋白活性比较 Figure 5 The comparison of the enzyme activity between β-agarase YM01-3 and mutants |
|
|
目前,绝大多数的研究者都是通过随机诱变的方式来改变酶性能,如Yang等[12]通过随机突变和组合密码子诱变(Combinatorial codon mutagenesis)对人工金属酶(Artificial metalloenzymes)酶活进行提升。本研究是通过随机突变构建突变文库并从中筛选影响酶活力的新位点,这是与目前大多数研究不同的新思路。本研究通过外泌型宿主短芽孢杆菌建立对随机突变文库中突变体的高通量筛选,最终确定2个影响酶活力变化的氨基酸新位点Y137、D237。
将β-琼胶酶YM01-3的氨基酸序列与测序成功的丧失酶活突变体的氨基酸序列进行比对,可了解到多条序列在Y137、D237位氨基酸处发生突变,且这2个位点在β-琼胶酶YM01-3的催化腔内,推测这2个位点是影响β-琼胶酶YM01-3酶活力变化的新位点。Takagi等[11]研究β-琼胶酶MtAgaA (Microbulbifer sp.)发现,色氨酸与底物结合有关。137位酪氨酸,其侧链为对羟基丙酸,与色氨酸侧链苯环相比极性更强,可与底物形成更为稳定的结构。将其突变成丙氨酸,丙氨酸极性低,与底物结合能力差,因此酶活力降低。Allouch等[13]研究β-AgaA_CM (Zobellia sp.)和β-AgaB (Zobellia sp.)的三维结构发现β-AgaA_CM的Glu254和β-AgaB的Glu256突变会使酶活力显著下降,并认为其通过影响蛋白质结构使酶活力降低。237位天冬氨酸侧链与Glu254和Glu256相似也具有羧基,当其由酸性带负电荷的天冬氨酸突变为带正电荷的赖氨酸后,由于电荷的排斥,导致蛋白质结构改变,酶活降低。以上推论还需要通过蛋白质结晶等进一步验证。
| [1] |
Araki T, Hayakaw M, Lu Z, et al. Purification and characterization of agarases from a marine bacterium, Vibrio sp. PO-303[J]. Journal of Marine Biotechnology, 1998, 6(4): 260-265. |
| [2] |
Wu SC, Pan CL. Preparation of algal‐oligosaccharide mixtures by bacterial agarases and their antioxidative properties[J]. Fisheries Science, 2004, 70(6): 1164-1173. DOI:10.1111/fis.2004.70.issue-6 |
| [3] |
Ramos KRM, Valdehuesa KNG, Nisola GM, et al. Identification and characterization of a thermostable endolytic β-agarase Aga2 from a newly isolated marine agarolytic bacteria Cellulophaga omnivescoria W5C[J]. New Biotechnology, 2018, 40: 261-267. DOI:10.1016/j.nbt.2017.09.006 |
| [4] |
Chen XL, Hou YP, Jin M, et al. Expression and characterization of a novel thermostable and pH-stable β-agarase from deep-sea bacterium Flammeovirga sp. OC4[J]. Journal of Agricultural and Food Chemistry, 2016, 64(38): 7251-7258. DOI:10.1021/acs.jafc.6b02998 |
| [5] |
Cui FY, Dong SJ, Shi XC, et al. Overexpression and characterization of a novel thermostable β-Agarase YM01-3, from marine bacterium Catenovulum agarivorans YM01T[J]. Marine Drugs, 2014, 12(5): 2731-2747. DOI:10.3390/md12052731 |
| [6] |
Hida K, Hanes J, Ostermeier M. Directed evolution for drug and nucleic acid delivery[J]. Advanced Drug Delivery Reviews, 2007, 59(15): 1562-1578. DOI:10.1016/j.addr.2007.08.022 |
| [7] |
Irfan M, Gonzalez CF, Raza S, et al. Improvement in thermostability of xylanase from Geobacillus thermodenitrificans C5 by site directed mutagenesis[J]. Enzyme and Microbial Technology, 2018, 111: 38-47. DOI:10.1016/j.enzmictec.2018.01.004 |
| [8] |
Ohta Y, Hatada Y, Nogi Y, et al. Enzymatic properties and nucleotide and amino acid sequences of a thermostable β-agarase from a novel species of deep-sea Microbulbifer[J]. Applied Microbiology and Biotechnology, 2004, 64(4): 505-514. DOI:10.1007/s00253-004-1573-y |
| [9] |
Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [10] |
Ke SH, Madison EL. Rapid and efficient site-directed mutagenesis by single-tube 'megaprimer' PCR method[J]. Nucleic Acids Research, 1997, 25(16): 3371-3372. DOI:10.1093/nar/25.16.3371 |
| [11] |
Takagi E, Hatada Y, Akita M, et al. Crystal structure of the catalytic domain of a GH16 β-agarase from a deep-sea bacterium, Microbulbifer thermotolerans JAMB-A94[J]. Bioscience, Biotechnology, and Biochemistry, 2015, 79(4): 625-632. DOI:10.1080/09168451.2014.988680 |
| [12] |
Yang H, Swartz AM, Park HJ, et al. Evolving artificial metalloenzymes via random mutagenesis[J]. Nature Chemistry, 2018, 10(3): 318-324. DOI:10.1038/nchem.2927 |
| [13] |
Allouch J, Jam M, Helbert W, et al. The three-dimensional structures of two β-agarase[J]. The Journal of Biological Chemistry, 2003, 278(47): 47171-47180. DOI:10.1074/jbc.M308313200 |
 2018, Vol. 45
2018, Vol. 45




