扩展功能
文章信息
- 林国荣, 王春忠, 孙富林, 郑志鹏
- LIN Guo-Rong, WANG Chun-Zhong, SUN Fu-Lin, ZHENG Zhi-Peng
- 海水混养池塘虾蛤肠道与养殖环境的微生物多样性
- Microbial community diversity in seawater polyculture pond of shrimp and clam
- 微生物学通报, 2018, 45(9): 1989-1999
- Microbiology China, 2018, 45(9): 1989-1999
- DOI: 10.13344/j.microbiol.china.171017
-
文章历史
- 收稿日期: 2017-12-01
- 接受日期: 2018-04-23
- 网络首发日期(www.cnki.net): 2018-05-10
2. 福建省莆田市水产科学研究所 福建 莆田 351100;
3. 中国科学院南海海洋研究所 广东 广州 510301;
4. 莆田市天然星农业开发有限公司 福建 莆田 351100
2. Putian Institute of Aquaculture Science of Fujian Province, Putian, Fujian 351100, China;
3. South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou, Guangdong 510 301, China;
4. Putian Tian Ran Xing Agriculture Development Co. Ltd., Putian, Fujian 351100, China
微生物在养殖生态系统的物质循环和能量流动中发挥着巨大作用[1-2],不仅在养殖环境的物质循环中承担着重要角色,而且在维持养殖环境稳定的过程中有重要的作用。同时,微生物与养殖生物的健康生长也有密切的联系,正常的肠道菌群对动物体的维生素合成、生长发育和物质代谢以及免疫防御功能都有重要的作用,是维持动物健康的必要因素。很多的研究表明,动物肠道微生物的组成与肠道的发育、营养状况、免疫反应以及宿主对疾病的抗性相关[1, 3-5]。
虾贝混养是常见海水池塘养殖方式,主要利用贝类滤食特点,摄取水中的浮游植物和虾类残饵、粪便等悬浮有机物,净化底质和水质,达到养殖空间充分利用、产量和经济增加的目的[6]。微生物是池塘养殖系统重要组成部分,不仅影响养殖生物的健康,而且对养殖水域生态系统有机物的代谢和分解能力的良好运转起着关键的作用。混养系统环境中微生物群落的结构[7-8]、水生动物肠道微生物生物学[9]以及单养品种肠道与环境的微生物群落结构影响[10]均有研究。前期通过构建微生物16S rRNA基因和18S rRNA基因克隆文库的方法来研究养殖区进水口水体、养殖池底泥、养殖池水体以及对虾肠道内微生物及真核生物群落组成情况,比较不同样品微生物组成结构的差异,分析微生物从水源到对虾肠道内的演替规律,揭示养殖环境在养殖动物肠道微生物群落结构中所起的重要作用[10-11]。但对混养中多品种动物肠道与环境的微生物群落结构关系尚未见报道。
本实验应用高通量测序技术等方法,研究海水池塘养殖日本囊对虾、硬壳蛤及其环境的微生物群落特征,分析三者之间微生物群落结构的相异性,揭示虾、贝混养模式微生物的组成规律,为池塘养殖虾、贝疾病防治和混养结构的优化提供参考。
1 材料与方法 1.1 采样地点与时间样品于2015年12月采集自福建省莆田市北江垦区对虾养殖区,整个垦区35个池塘,每个池面积均为3.33 hm2,平均水深1 m,泥沙底质,泥׃沙比例为81׃19。8月份投放规格2-3 cm硬壳蛤(Mercenaria mercenaria)苗种100 kg,10月投放平均规格1.0 cm日本囊对虾(Penaeus japonicus)苗种50万尾,每天早晚投相同对虾饲料一次。在1#、6#号池塘前、后、中位置3个点采集水、底泥、蛤和虾(只取一个池)的肠道混合,样品编号分别为水(LZ1、LZ6)、底泥(YN1、YN6)、虾(XD1)、蛤(CD1、CD6)。同时测定水体温度、盐度、pH、溶解氧DO和COD,水样经过0.45 μm滤膜过滤后,测定水体亚硝酸盐、硝酸盐、氨氮及磷酸盐等参数,结果见图 1。底泥样品采用抓斗式采泥器采集,用无菌取样器去掉暴露在外空气部分,采集0-4 cm泥样,放置于15 mL无菌离心管中。水体用有机玻璃采水器采集,先预过滤除去大颗粒物质,再用孔径为0.2 μm的聚碳酸酯膜过滤500 mL,将滤膜用锡箔纸包好保存。每个池塘采集健康虾和蛤各8个,其虾体长4.2±0.6 cm左右,蛤壳长4.9±0.5 cm。样品采集后迅速置于4 ℃,在实验室无菌条件下剖取对虾和蛤肠道,再用无菌的镊子取出肠道内的粪便等杂物,将对虾和蛤肠道分别置于灭菌的1.5 mL离心管中。所有样品保存于-80 ℃以备DNA提取。

|
| 图 1 混养池塘理化参数分析 Figure 1 Physico-chemical parameters in polyculture environment |
|
|
无菌条件下取出滤膜,剪碎后放置于1.5 mL离心管中,水样滤膜的提取参照文献[10]的方法。称取1 g (湿重)底泥和肠道样品,参照土壤核酸提取试剂盒(OMEGA公司)说明步骤提取总DNA。本实验16S rRNA基因测序以V3–V4为目标区域进行引物设计,引物序列为319F (5′-ACTCCTACG GGAGGCAGCAG-3′)和806R (5′-GGACTACHVG GGTWTCTAAT-3′),扩增片段大小大约469 bp。PCR反应体系(25 μL):5×FastPfu缓冲液4 μL,dNTPs (2.5 mmol/L) 2 μL,FastPfu聚合酶(5 U/μL) 0.5 μL,引物(5.0 μmol/L)各1.0 μL,模板DNA 10 ng。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,30个循环;72 ℃ 10 min。在正反引物两端加上不同的Barcode以区分不同的样品,扩增完成后的PCR产物使用Beads纯化之后进行上机测序。
1.3 数据分析对于MiSeq测序获得的双端数据,首先根据Barcode信息进行样品区分,然后根据Overlap关系进行拼接,随后进行Q20、Q30等质控分析,对最终获得Clean数据归一化之后进行OTU (Operational taxonomic units)聚类分析和物种分类学分析。采用CD-HIT工具将序列相似性大于97%的Tags定义为一个OTU聚类。采用QIIME软件对样品序列进行Alpha多样性分析,包括物种数目(Observed species)、Shannon指数(Shannon index)、Chao1指数(Chao1 index)。选择OTU操作单元里的一条代表序列(默认丰度最高)采用RDP classifier软件进行物种分类,分类阈值默认为0.8。利用CANOCO 4.5软件对微生物群落结构进行除趋势对应分析(Detrended correspondence analysis),揭示样品间微生物种类的关系。利用R语言Venn Diagram程序包绘制韦恩图。
2 结果与分析 2.1 高通量测序数据通过Illumina MiSeq测序,总共测定出95 771条序列,在97%相似性水平为标准观测到7 117个物种,各样本结果见表 1。结果显示底泥微生物种类、多样性以及丰富度高于水体和肠道;两池蛤肠道的物种数目和丰富度Chao1指数均高于虾肠道(XD1),但是其多样性指数均略低于对虾肠道。
| 样品Samples | 物种数Species | 香农指数Shannon index | 辛普森指数Simpson index | 丰富度Chao1 index |
| CD1 | 295 | 2.037 | 0.63 | 295 |
| XD1 | 188 | 2.198 | 0.69 | 188 |
| CD6 | 296 | 1.936 | 0.60 | 296 |
| LZ1 | 1 158 | 3.907 | 0.93 | 1 158 |
| LZ6 | 1 442 | 3.558 | 0.91 | 1 442 |
| YN1 | 5 065 | 7.181 | 1.00 | 5 065 |
| YN6 | 3 294 | 6.567 | 0.99 | 3 294 |
UPGMA微生物群落结构聚类分析见图 2。这些样品分为3个类群:2个池塘蛤肠道的微生物群落(CD1,CD6)最为相似,并与虾肠道XD1聚类在一起,三者间相似度大于90%;2个池塘水体LZ1和LZ6聚在一起,微生物群落结构相似度为65%;两池塘底泥样品YN1和YN6聚在一起,相似度60%。说明在同一养殖区2个池塘养殖动物肠道微生物群落结构相似性比环境微生物群落高,同种生物基本相同,虽然试验缺少另一虾肠道样品,但与2个池蛤肠道相似度大于90%,同样推理,两池虾肠道也极为相似。

|
| 图 2 UPGMA微生物群落结构聚类分析 Figure 2 UPGMA cluster analysis of bacterial community among different samples |
|
|
通过对样品微生物种类信息进行注释,比较不同样品所对应的微生物种类在样品中的丰度信息。如图 3A所示,从细菌门纲分类水平分析,厚壁菌门(Firmicutes)、γ-变形杆菌(Gamma Proteobacteria)、α-变形杆菌(Alpha Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidete)和δ-变形杆菌(Delta Proteobacteria)是优势细菌门类。养殖虾与蛤肠道微生物以厚壁细菌(Firmicutes,74.96%)和γ-变形杆菌(Gamma Proteobacteria,22.38%)为主;池塘水体以放线菌门(Actinobacteria,51.32%)、α-变形杆菌(Alpha Proteobacteria,10.16%)、拟杆菌门(Bacteroidete,14.14%)为主;底泥以γ-变形杆菌(Gamma Proteobacteria,20.28%)和δ-变形杆菌(Delta Proteobacteria,20.59%)为主。

|
| 图 3 在门分类(A)和目分类(B)水平样品中细菌种群结构及其相对丰度 Figure 3 Taxonomic diversity of bacterial communities among the samples in phylum level and order level |
|
|
从图 3B目分类水平分析,样品之间微生物种类差异更为明显(ANOVA,P < 0.01)。以相对丰度大于5%计算,动物肠道微生物优势种类为Lactobacillales (73.66%)和Pseudomonadales (20.31%)。池塘水体微生物优势种类为分别为Actinomycetales (50.04%)、Flavobacteriales (11.70%)、Rhodobacterales (5.11%);底泥微生物以Desulfobacterales (17.40%)、Alteromonadales (4.25%)、Thiotrichales (4.28%)为主。
从图 4属分类水平分析,虾蛤肠道微生物以Lactococcus sp. (68.17%)、Pseudomonas sp. (14.24%)、Psychrobacter sp. (3.19%)、Enhydrobacter sp. (2.59%)、Brochothrix sp. (2.50%)、Streptococcus sp. (2.49%)为主。池塘水体微生物主要为Candidatus Aquiluna sp. (21.34%)、Unclassified Actinomycetales (11.17%)、Unclassified Microbacteriaceae (16.99%)、Lishizhenia sp. (4.47%)、Flavobacterium sp. (4.49%)、Unclassified Rhodobacteraceae (6.34%)、Candidatus Pelagibacter sp. (3.04%)。底泥微生物主要为Unclassified Piscirickettsiaceae (3.97%)、Unclassified Marinicellaceae (4.67%)、Unclassified Desulfuromonadaceae (3.18%)、Unclassified Desulfobulbaceae (3.72%)、Unclassified Desulfobacteraceae (4.29%)、Sulfurovum sp. (4.10%)、Unclassified Rhodobacteraceae (2.23%)、Desulfopila sp. (4.20%)。

|
| 图 4 微生物群落在属水平的热图分析 Figure 4 Heatmap showing phylogenetic distribution of bacteria samples 注:图中显示细菌在属分类(y轴)在每个样品(x轴)中的相对丰度;颜色条代表相对丰度大小. Note: The relative percentage of genus classification of bacteria (y axis) within each sample (x axis clustering) was shown. Colour bars represent the relative percentage. |
|
|
为了更好地理解样品微生物类群之间的关系,用除趋势对应分析(DCA)用来表观微生物群落间的差异与相似性(图 5)。排序图表明了细菌在属分类水平上样品间联系。模型的第一和第二特征值为0.946 9和0.222 1。从图 5可以看出,肠道微生物样品(CD1、XD1、CD6)、池塘水体样品(LZ1、LZ6)和池塘底泥样品(YN1、YN6)之间差异大,样品间相距较远,样品间共有的细菌种类极少。虾蛤肠道微生物均以Lactococcus sp.和Pseudomonas sp.等异养细菌为特征微生物种类;池塘水体以Candidatus Aquiluna sp.、Actinomycetales、Microbacteriaceae、Lishizhenia sp.、Flavobacterium sp.和Lewinella sp.等需氧细菌为特征微生物种类,沉积物以Desulfopila sp.、Desulfobacteraceae、Desulfobulbaceae、Desulfuromonadaceae和Desulfococcus sp.等厌氧性硫还原细菌为特征微生物种类。
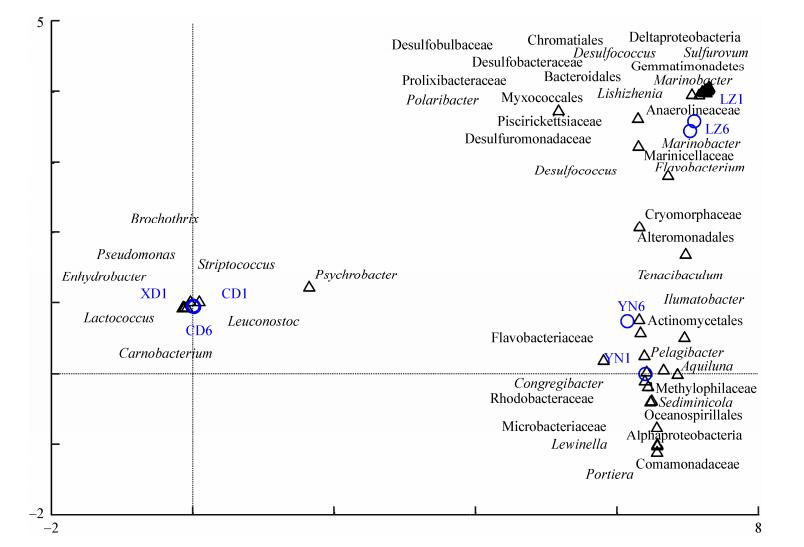
|
| 图 5 样品间微生物群落的对应分析 Figure 5 Correspondence analysis of bacterial communities among samples |
|
|
为了更详细分析环境以及动物肠道微生物的差异,分析了28个优势OTU (丰度 > 2%)的分布特征。图 6表明动物肠道、水体和底泥样品具有显著的微生物差异(ANOVA,P < 0.01),而虾蛤2种动物肠道极为相似;韦恩图分析结果表明(图 7),混养虾蛤肠道微生物具有135个共同的OTU;而蛤肠道微生物同水体以及底泥共有OTU数目分别为69和73;虾肠道微生物同水体以及底泥共同的OTU分别为38和41。对不同样品中所含菌群丰度最高的10个OTU的研究有助于了解样品中所含的主要细菌类型和其生态功能,表 2体现了样品间主要物种丰度和种类变化。经序列比对发现养殖环境及肠道微生物的主要种类组成为厚壁菌门、变形菌门、拟杆菌门、放线菌门和酸杆菌门的细菌。虾蛤肠道具有高丰度的厚壁细菌和γ-变形杆菌如Lactococcus sp.和Pseudomonas sp.等;池塘水体具有较高的需氧细菌如Microbacteriaceae、Candidatus Aquiluna rubra sp.、Rhodobacteraceae、Flavobacteriales等;底泥具有较高丰度的厌氧细菌如Desulfobulbaceae、Desulfobacteraceae、Marinicellaceae、Desulfobulbaceae等。这些微生物群落结构充分体现了不同环境条件微生物特征。
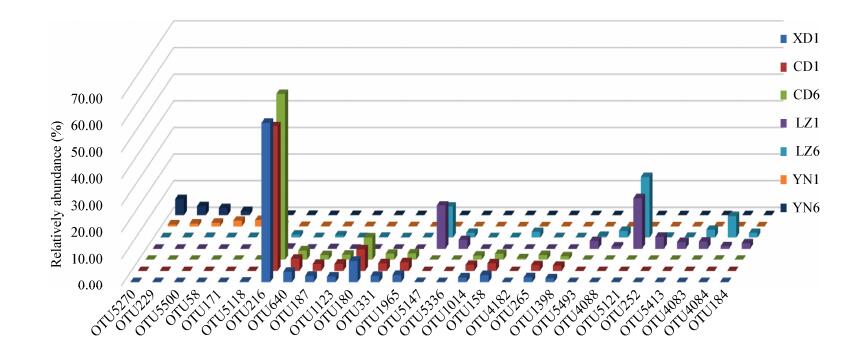
|
| 图 6 样品间优势OTU分布特征(显示相对丰度 > 2%) Figure 6 Rank abundance histograms for the top OTUs (> 2% abundance) |
|
|
| CD1 | XD1 | CD6 | LZ1 | LZ6 | YN1 | YN6 |
| Lactococcus piscium 60.78% | Lactococcus piscium 55.61% | Lactococcus piscium 63.39% | Microbacteriaceae 22.12% | Candidatus Aquiluna rubra 23.02% | Gammaproteobacteria 7.83% | Desulfobulbaceae 6.19% |
| Lactococcus lactis 7.98% | Lactococcus lactis 8.09% | Lactococcus lactis 8.31% | Candidatus Aquiluna rubra 19.64% | Actinomycetales 15.15% | Piscirickettsiaceae 4.91% | Desulfobacteraceae 5.29% |
| Pseudomonas 7.53% | Pseudomonas 9.66% | Pseudomonas 6.68% | Rhodobacteraceae 8.67% | Microbacteriaceae 11.87% | Marinicellaceae 4.52% | Sulfurovum 5.28% |
| Pseudomonas fragi 5.92% | Pseudomonas fragi 7.19% | Pseudomonas fragi 5.35% | Actinomycetales 7.19% | Lishizhenia caseinilytica 8.05% | Desulfuromonadaceae 3.86% | Marinicellaceae 4.83% |
| Psychrobacter 3.08% | Psychrobacter 3.41% | Psychrobacter 2.38% | Lewinella 4.45% | Flavobacterium 5.34% | Desulfobulbaceae 3.56% | Gammaproteobacteria 4.81% |
| Enhydrobacter 2.7% | Enhydrobacter 2.83% | Enhydrobacter 2.24% | Alphaproteobacteria 4.02% | Rhodobacteraceae 4.01% | Desulfobacteraceae 3.3% | Desulfobulbaceae 3.87% |
| Brochothrix 2.34% | Brochothrix 2.85% | Brochothrix 2.31% | Flavobacterium 3.65% | Candidatus Pelagibacter 3.87% | Sulfurovum 2.91% | Piscirickettsiaceae 3.02% |
| Streptococcus 1.91% | Streptococcus 2.43% | Streptococcus 1.96% | Candidatus Portiera 2.36% | Tenacibaculum 2.55% | Rhodobacteraceae 2.61% | Desulfuromonadaceae 2.5% |
| Carnobacterium 1.51% | Carnobacterium 2.06% | Carnobacterium 1.4% | Candidatus Pelagibacter 2.21% | Cryomorphaceae 2.12% | Desulfobulbaceae 2.22% | Desulfococcus 2.35% |
| Leuconostoc 1.04% | Leuconostoc 0.84% | Leuconostoc 0.97% | Comamonadaceae 2.21% | Polaribacter 2.11% | Actinobacteria 1.79% | Rhodobacteraceae 1.84% |
| 注:表中数字为各OTU相对丰度;样品编号CD1、CD6为蛤肠道;XD1为虾肠道;LZ1、LZ6为水样;YN1、YN6为底泥.
Note: The number in the table represent the relative abundance of each OTUs; CD1 and CD6 represent clam’s intestines; XD1 represent shrimp’s intestines; LZ1 and LZ6 represent water samples; YN1 and YN6 represent sediment samples. | ||||||
本文通过Illumina平台的高通量技术测定海水垦区虾蛤混养动物肠道以及养殖水体的微生物群落结构特征,分析了它们的微生物组成、多样性以及遗传进化信息。结果表明,虾蛤肠道微生物以厚壁细菌和γ-变形细菌为主;池塘水体以放线菌、α-变形细菌以及拟杆菌为主,底泥以γ-变形细菌和δ-变形细菌为主;这个同其他研究水生动物肠道及环境微生物相类似[12-13]。从微生物多样性和丰富度显现出底泥 > 水体 > 肠道,这与通过克隆文库的方法结果相似[10-11]。有研究表明水环境中pH、氨氮和亚硝氮等理化性质的改变均能够影响环境中的菌群结构,水产动物肠道菌群与宿主及水环境性质具有密切的联系[14]。本试验同一养殖区不同的养殖池具有相同的养殖模式,2个池塘水体、底泥之间的微生物结构存在一定的差异,而不同池的蛤肠道微生物结构之间具有极高的相似性。蛤肠道微生物群落与虾肠道也具有极高的相似性,其肠道微生物中有约75%的OTU存在于虾肠道微生物中;另外,虾蛤肠道相当数量的微生物OTU都能在养殖环境内找到;以CD1为例,其肠道微生物中有24%的OTU也存在于养殖水体和沉积物中(图 2、图 7),说明水体和肠道微生物组成之间存在紧密的相关关系,养殖水体环境对肠道微生物的组成起到重要的影响作用。说明水产动物肠道菌群与水环境相关,还可能与饲养条件密切相关,这与[15-16]研究饲养条件对肠道微生物组成也有明显的影响相一致。
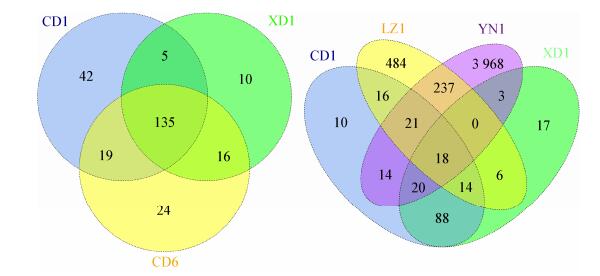
|
| 图 7 肠道与环境样品间共有微生物种类分析 Figure 7 Venn diagram showing the unique and shared OTUs (3% distance level) in the different libraries |
|
|
肠道微生物主要优势菌为乳球菌属Lactococcus sp.和假单胞菌属Pseudomonas sp.,其中Lactococcus sp.占到70%以上。乳酸菌在水产上的应用较多,主要是拌入饲料一起投喂给养殖动物。水产养殖上希望以乳酸菌代替抗生素或饲料添加剂应用于鱼类养殖,提高养殖鱼的免疫和抗病能力。OTU水平比对发现,在99%相似度下动物肠道内70%以上为Lactococcus piscium sp.。据报道Lactococcus piscium sp.具有提高虾质量以及抑制病原菌的作用[17]。另外还发现较高丰度的L. lactis,其作为鱼类健康的益生菌而被广泛报道[18-19]。高丰度的L. lactis在养殖池能产生抗菌肽和蛋白,对某些革兰阳性和阴性菌产生抗菌活性[20],因此在养殖动物出现肠炎等状况下其可以通过抑制特定的微生物而达到调节肠道的作用。另外Pseudomonas sp. (假单胞菌)的一些种类是水产养殖的潜在抗菌药物,可以不通过产生活性物质而抵抗水霉菌的感染,提供了一种新的方式来减轻养殖病害的模式[21];但是对于它们潜在的对养殖动物的保护作用了解仍较为有限。总之,这些肠道益生菌的存在提高了垦区养殖动物对病原菌的抗性。
本试验水体中存在较高丰度的黄杆菌等细菌种群,其优势种类是Sediminicola sp.、Flavobacteriales、Tenacibaculum sp.、Owenweeksia sp.和Winogradskyella sp.。黄杆菌的某些种类也是潜在的致病菌,如黄杆菌属(Flavobacterium sp.)中一些种类对淡水鱼类、海洋鱼类、两栖动物、其他水生动物以及海洋微藻都有致病作用[22],患病鱼体会出现烂鳃、体表溃疡、组织坏死及鳍条腐烂等症状,死亡率可高达100%,全球每年因此病所造成的经济损失极其严重[23]。Tenacibaculum sp.是一种溃疡性疾病称为滑动细菌病,这种病原菌也会对养殖动物产生高死亡率,同时增加其他病原体的易感性[24]。而黄杆菌在本实验中虾和蛤的肠道含量较低,几乎没有测定出,但其对海水生态中的作用和影响亟待进一步研究。
池塘底泥Desulfopil a sp.和Desulfobacteraceae等硫还原细菌(Desulfobacterales和Desulfuromonadales)的含量较高。Desulfobacterales和Desulfuromonadales是严格厌氧硫还原细菌,能还原硫酸盐获得能量。这些硫酸盐还原菌可在缺氧条件下转化硫化氢,对水产动物和环境造成严重的危害。这些结果表明,养殖池容易产生低氧或缺氧,并增加硫还原细菌的丰度。应密切关注水体硫化氢等水质指标,加强养殖管理,采取相关措施提高水体溶解氧,而且要防患黄杆菌这类致病菌感染引发疾病的发生。
4 结论综上所述,本研究采用高通量测序技术研究混养条件虾蛤养殖池细菌群落结构的变化,结果显示微生物生态结构的变化与养殖环境紧密相关。相同混养条件下,不同池塘虾蛤肠道微生物群落结构在水体、底泥微生物结构存在一定差异情况下,其表现出高度相似,说明养殖动物肠道微生物结构受到养殖环境和养殖模式的双重影响。养殖水体和沉积物形成自己特征性细菌类群;水体微生物以需氧细菌为主,主要是放线菌和黄杆菌类群;沉积物以厌氧细菌为主,主要是硫还原细菌。本研究揭示海水混养池塘养殖微生物的特点,为混养条件下微生物的作用机理以及病害的发生原因及预防的研究提供一定的理论参考。
| [1] |
Moriarty DJW. The role of microorganisms in aquaculture ponds[J]. Aquaculture, 1997, 151(1/4): 333-349. |
| [2] |
Abraham TJ, Ghosh S, Nagesh TS, et al. Distribution of bacteria involved in nitrogen and sulphur cycles in shrimp culture systems of West Bengal, India[J]. Aquaculture, 2004, 239(1/4): 275-288. |
| [3] |
Verschuere L, Rombaut G, Sorgeloos P, et al. Probiotic Bacteria as Biological Control Agents in Aquaculture[J]. Microbiology and Molecular Biology Reviews, 2000, 64(4): 655-671. DOI:10.1128/MMBR.64.4.655-671.2000 |
| [4] |
Gatesoupe FJ. The use of probiotics in aquaculture[J]. Aquaculture, 1999, 180(1/2): 147-165. |
| [5] |
Crab R, Avnimelech Y, Defoirdt T, et al. Nitrogen removal techniques in aquaculture for a sustainable production[J]. Aquaculture, 2007, 270(1/4): 1-14. |
| [6] |
Dong GC, Tian XL, Dong SL, et al. An experimental study on energy budget and conversion efficiency in different polyculture modes of shrimp, bivalve and seaweed[J]. Periodical of Ocean University of China, 2007, 37(6): 899-906. (in Chinese) 董贯仓, 田相利, 董双林, 等. 几种虾、贝、藻混养模式能量收支及转化效率的研究[J]. 中国海洋大学学报, 2007, 37(6): 899-906. |
| [7] |
Liu RJ, Tian XL, Dong SL, et al. The functional diversity of flora in polyculture ecosystem of swimming crab, shrimp and short-necked clam[J]. Fisheries Science, 2014, 33(9): 535-544. (in Chinese) 刘瑞娟, 田相利, 董双林, 等. 蟹虾贝混养池塘生态系统微生物群落功能多样性研究[J]. 水产科学, 2014, 33(9): 535-544. DOI:10.3969/j.issn.1003-1111.2014.09.001 |
| [8] |
Liu RJ. Preliminary study on variation of microbe quantity and community composition in polyculture ecosystem of crab, shrimp and clam[D]. Qingdao: Master's Thesis of Ocean University of China, 2014 (in Chinese) 刘瑞娟.蟹虾贝混养系统细菌群落结构与功能多样性初步研究[D].青岛: 中国海洋大学硕士学位论文, 2014 |
| [9] |
Zhang ML, Du ZY. Review and perspective: Function of intestinal microbiota in aquatic animals[J]. Journal of East China Normal University (Natural Science), 2016(1): 1-8. (in Chinese) 张美玲, 杜震宇. 水生动物肠道微生物研究进展[J]. 华东师范大学学报:自然科学版, 2016(1): 1-8. |
| [10] |
Wang CZ, Lin GR, Yan T, et al. Microbial community in the shrimp (Penaeus penicillatus) intestine and its culture environment[J]. Journal of Fisheries of China, 2014, 38(5): 706-712. (in Chinese) 王春忠, 林国荣, 严涛, 等. 长毛对虾海水养殖环境以及虾肠道微生物群落结构研究[J]. 水产学报, 2014, 38(5): 706-712. |
| [11] |
Wang CZ, Lin GR, Yan T, et al. The cellular community in the intestine of the shrimp Penaeus penicillatus and its culture environments[J]. Fisheries Science, 2014, 80(5): 1001-1007. DOI:10.1007/s12562-014-0765-3 |
| [12] |
Oxley APA, Shipton W, Owens L, et al. Bacterial flora from the gut of the wild and cultured banana prawn, Penaeus merguiensis[J]. Journal of Applied Microbiology, 2002, 93(2): 214-223. DOI:10.1046/j.1365-2672.2002.01673.x |
| [13] |
Roeselers G, Mittge EK, Stephens WZ, et al. Evidence for a core gut microbiota in the zebrafish[J]. ISME Journal Multidisciplinary Journal of Microbial Ecology, 2011, 5(10): 1595-1608. |
| [14] |
Zhang JS, Duan YF, Zhang ZZ, et al. Research progress of intestinal microbial flora in shrimp[J]. South China Fisheries Science, 2015, 11(6): 114-119. (in Chinese) 张家松, 段亚飞, 张真真, 等. 对虾肠道微生物菌群的研究进展[J]. 南方水产科学, 2015, 11(6): 114-119. DOI:10.3969/j.issn.2095-0780.2015.06.016 |
| [15] |
Wu SG, Wang GT, Angert ER, et al. Composition, diversity, and origin of the bacterial community in grass carp intestine[J]. PLoS One, 2012, 7(2): e30440. DOI:10.1371/journal.pone.0030440 |
| [16] |
Carda-Diéguez M, Mira A, Fouz B. Pyrosequencing survey of intestinal microbiota diversity in cultured sea bass (Dicentrarchus labrax) fed functional diets[J]. FEMS Microbiology Ecology, 2014, 87(2): 451-459. DOI:10.1111/1574-6941.12236 |
| [17] |
Fall PA, Leroi F, Cardinal M, et al. Inhibition of Brochothrix thermosphacta and sensory improvement of tropical peeled cooked shrimp by Lactococcus piscium CNCM I-4031[J]. Letters in Applied Microbiology, 2010, 50(4): 357-361. DOI:10.1111/lam.2010.50.issue-4 |
| [18] |
Balcázar JL, Decamp O, Vendrell D, et al. Health and nutritional properties of probiotics in fish and shellfish[J]. Microbial Ecology in Health and Disease, 2006, 18(2): 65-70. DOI:10.1080/08910600600799497 |
| [19] |
Ringø E, Gatesoupe FJ. Lactic acid bacteria in fish: a review[J]. Aquaculture, 1998, 160(3/4): 177-203. |
| [20] |
Twomey D, Ross RP, Ryan M, et al. Lantibiotics produced by lactic acid bacteria: structure, function and applications[J]. Antonie van Leeuwenhoek, 2002, 82(1/4): 165-185. DOI:10.1023/A:1020660321724 |
| [21] |
Liu YY, Rzeszutek E, van der Voort M, et al. Diversity of aquatic Pseudomonas species and their activity against the fish pathogenic oomycete saprolegnia[J]. PLoS One, 2015, 10(8): e0136241. DOI:10.1371/journal.pone.0136241 |
| [22] |
Bemardet JF, Family I, In W. Whitman(ed.) Bergey's Manual of Systematic Bacteriology[M]. 2nd ed. The Williams & Wilkins Co., Baltimore, 2011: 106-111
|
| [23] |
Verma DK, Rathore G. Molecular characterization of Flavobacterium columnare isolated from a natural outbreak of columnaris disease in farmed fish, Catla catla from India[J]. The Journal of General and Applied Microbiology, 2013, 59(6): 417-424. DOI:10.2323/jgam.59.417 |
| [24] |
Avendaño-Herrera R, Toranzo AE, Magariños B. Tenacibaculosis infection in marine fish caused by Tenacibaculum maritimum: a review[J]. Diseases of Aquatic Organisms, 2006, 71(3): 255-266. |
 2018, Vol. 45
2018, Vol. 45




