扩展功能
文章信息
- 程梦丽, 李晓峰, 秦成峰
- CHENG Meng-Li, LI Xiao-Feng, QIN Cheng-Feng
- 寨卡病毒蛋白结构与功能研究进展
- Research progress on the structure and function of Zika virus proteins
- 微生物学通报, 2018, 45(8): 1787-1793
- Microbiology China, 2018, 45(8): 1787-1793
- DOI: 10.13344/j.microbiol.china.180307
-
文章历史
- 收稿日期: 2018-04-16
- 接受日期: 2018-06-15
- 网络首发日期(www.cnki.net): 2018-07-05
2. 军事科学院军事医学研究院微生物流行病研究所 病原微生物生物安全国家重点实验室 北京 100071
2. State Key Laboratory of Pathogen and Biosecurity, Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Beijing 100071, China
寨卡病毒(Zika virus,ZIKV)属黄病毒科黄病毒属,是一种通过蚊虫传播的虫媒病毒,该属成员包括登革病毒(Dengue virus,DENV)、西尼罗病毒(West Nile virus,WNV)、黄热病毒(Yellow fever virus,YFV)及乙型脑炎病毒(Japanese encephalitis virus,JEV)等一系列重要的人类病原体[1]。寨卡病毒最早于1947年从乌干达热带丛林中分离,并因而得名,在其被发现的60多年里只有零散的少数病例报道,而且临床表现非常轻微,可引起患者发热、咽喉痛、结膜充血、面部发红、四肢和躯干的大面积斑状丘疹[2]。2015年寨卡病毒感染在南美洲传播并迅速蔓延至70多个国家和地区,且首次发现寨卡病毒可引起胎儿小头症、宫内发育迟缓和其他先天畸形[3],还与成人神经障碍的格林-巴利综合症(Guillain-Barré syndrome,GBS)相关[4]。自2015年传入巴西,寨卡病毒疫情暴发并迅速横跨美国,截至2018年1月,巴西已累计通报231 725例疑似病例,2 952例确诊先天畸形[5],婴儿小头症发生率增加20多倍。
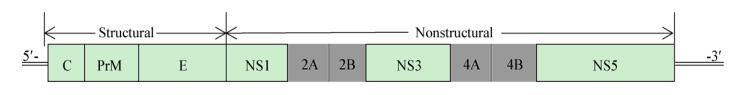
寨卡病毒基因组为长约11 kb的单股正链RNA,由两侧的非编码区(Untranslated region,UTR)和内部单一的开放读码框构成。其中开放读码框编码一个长的聚蛋白前体,经宿主及病毒蛋白酶切割后依次形成3个结构蛋白(C、prM/M和E)和7个非结构(Non-structural,NS)蛋白(NS1、NS2A、NS2B、NS3、NS4A、NS4B和NS5) (图 1)。结构蛋白主要参与病毒颗粒的组装、病毒的吸附和侵入,并且包含了主要的抗原表位。非结构蛋白主要调控病毒的基因组复制、翻译及调控宿主的免疫应答过程。由于寨卡病毒感染与胎儿小头症和睾丸损伤的关联[6]完全超越了此前对虫媒黄病毒的固有认识,迫切需要用新眼光来研究这个老病毒。
|
| 图 1 寨卡病毒的基因组结构示意图 Figure 1 The schematic diagram of the genome structure of Zika virus 注:灰色部分表示相应的蛋白质结构尚未解析 Note: The structure of the corresponding protein unresolved is in gray. |
|
|
寨卡病毒是直径约40 nm的包膜二十面体病毒,二十面体壳是由E蛋白和M蛋白的180拷贝组成,它们都通过其跨膜区域锚定在脂质膜中。寨卡病毒成熟颗粒的结构与DENV和WNV相似,脂质双分子层和包膜胞外域的径向距离与DENV-2相似[7]。冷冻电子显微镜结构研究表明,寨卡病毒颗粒具有2个独特的特征,即突出的N154糖基化位点和更紧凑的表面,具有较高的热稳定性[8]。N154位于E蛋白,E蛋白在病毒颗粒中呈现特征性的人字斜纹结构,30个两重轴顶点的每一个上面均有一个(E-M)2异源二聚体,还有60个(E-M)2异源二聚体在二十面体蛋白壳内的一般位置上。成熟ZIKV和DENV的E和M蛋白的等效Cα原子之间的均方根偏差为1.8 Å。病毒的等效Cα原子之间的最大差异(高达6 Å)是糖基化位点周围的区域(ZIKV中的Asn154和DENV中的Asn153)。ZIKV E蛋白具有单个糖基化位点(Asn154),而DENV E蛋白有2个位点(Asn67和Asn153)被糖基化[9]。鉴于树突状细胞特异性细胞间黏附分子-3-结合非整合素(DC-SIGN)和甘露醇受体可能是DENV的受体/辅助受体(与DENV聚糖结合)[10-12],因此N154糖基化位点可能是ZIKV宿主细胞的附着位点。
基于冷冻电镜解析了寨卡病毒完整的病毒颗粒结构;借助X射线晶体学解析了多个单个的病毒蛋白结构,且整体上具有很高的相似度,例如,多个团队都解析了NS5非结构蛋白。目前寨卡病毒结构蛋白C、M和E及非结构蛋白NS1、NS3和NS5的结构已被解析(表 1),对非结构蛋白NS2A、NS2B、NS4A、NS4B的结构尚缺乏认识,未来这些重要非结构蛋白的解析将为人们了解和战胜寨卡病毒奠定重要基础。
| 蛋白 Protein |
作者 Author |
日期 Date |
期刊 Journal |
蛋白质数据库 PDB |
方法 Methods |
备注 Comments |
| Capsid | Shang et al[13] | 2018.02.28 | Journal of Molecular Biology | 5YGH | X-ray Diffraction | |
| Envelope & prM | Prasad et al[14] | 2017.01.11 | Nature Structural & Molecular Biology | 5U4W | Cyro-EM | Firstly reported the immature structure |
| Kostyuchenko et al[8] | 2016.05.25 | Nature | 5IZ7 | Cyro-EM | Structure of the thermally stable | |
| Sirohi et al[9] | 2016.03.30 | Science | 5IRE | Cyro-EM | First ZIKV virion structure illuminated | |
| Envelope | Dai et al[15] | 2016.05.11 | Cell Host Microbe | 5JHM | X-ray Diffraction | In complex with mAb 2A10G6 |
| NS1 | Brown et al[16] | 2016.07.06 | Nature Structural & Molecular Biology | 5K6K | X-ray Diffraction | |
| Song et al[17] | 2016.04.13 | Nature Structural & Molecular Biology | 5IY3 | X-ray Diffraction | ||
| NS2B-NS3 | Lei et al[18] | 2016.07.06 | Science | 5LC0 | X-ray Diffraction | In complex with a peptidomimetic boronic acid inhibitor |
| Zhang et al[19] | 2016.12.14 | Science | 5H4I | X-ray Diffraction | ||
| NS5 | Zhang et al[20] | 2016.12.07 | Biochemical and Biophysical Research Communications | 5GP1 | X-ray Diffraction | Identified two conformations of the second base of the cap |
| Zhao et al[21] | 2017.03.29 | Nat Commun | 5U0B | X-ray Diffraction | Report the crystal structures of full-length NS5 | |
| Wang et al[22] | 2017.02.08 | Nat Commun | 5TMH | X-ray Diffraction | ||
| Godoy et al[23] | 2017.02.22 | Nat Commun | 5U04 | X-ray Diffraction | ||
| 注:PDB:蛋白质数据库. Note: PDB: Protein Data Bank. |
||||||
E蛋白是负责病毒与受体结合、膜融合以及宿主免疫识别的主要蛋白,本实验室和高福团队合作于2016年5月解析了寨卡病毒E蛋白膜外结构域在二聚体形式2.0 Å下的结构[15],目前完整E蛋白3.8 Å下的结构也已解析,包括4个结构域:负责锚定胞膜的茎-跨膜结构域和组成蛋白质剩余主要β链表面部分的结构域Ⅰ(Domain Ⅰ,DⅠ)、Ⅱ和Ⅲ,DⅡ连接DⅠ和DⅢ。DⅠ含有从胞膜表面突出的不同于其他黄病毒的单个糖基化位点(N154),可以作为寨卡病毒宿主细胞的附着位点;DⅠ具有一个较长的150环状区域(Loop region),可能影响寨卡病毒等黄病毒的传播和致病性。DⅡ的融合环在膜融合期间与宿主膜相互作用,DⅢ含有特定的受体结合位点,在膜融合中有重要作用[15]。与DENV相比,寨卡病毒E蛋白DⅢ C链末端的单残基插入可能增加病毒的整体稳定性[8]。在40 ℃下孵育时,寨卡病毒颗粒并没有聚集,且保持其感染性,而DENV-2和DENV-4毒株的感染性显著降低;随着孵育时间增加,寨卡病毒的感染性仅略有降低。其原因可能是高温下寨卡病毒膨胀成光滑的表面颗粒,使脂质包膜更易变,结构得以恢复到其正常状态。此外,在寨卡病毒结构中,5个A-C′二聚体接触的五重对称轴部位可能存在相互作用网络,导致更紧密的组装,从而防止这种结构转变。且寨卡病毒显示与TAM (Tyro3,Axl and Mer)结合但不结合TIM (T cell,immunoglobulin and mucin)受体,可见寨卡病毒比DENV-2和DENV-4具有更紧凑的表面。ZIKV的结构稳定性可能有助于其在精液[24]、唾液[25]和尿液[1]的不同环境下生存,研发破坏这种稳定结构的抗体或药物将减少疾病的发生或限制病毒的传播。
目前,靶向E蛋白已经开发了一系列特异识别寨卡病毒E蛋白的针对寨卡病毒感染的中和保护性单克隆抗体(Monoclonal antibodies,mAb)。第一个是本实验室发现的黄病毒广谱保护性鼠源单克隆抗体2A10G6,与DⅡ融合环中的保守序列结合,影响病毒与细胞之间的膜融合,与其他黄病毒相比,对寨卡病毒的中和效力相对较弱,但单次500 μg剂量可在感染的A129小鼠体内提供完全保护[15],可能是由于寨卡病毒具有高度的热稳定结构。随后从恢复的DENV感染患者中分离出2种人类中和mAb C8和C11,与DENV和寨卡病毒保守区中的四级结构依赖性表位结合,已被证明对寨卡病毒感染有效[26]。然后从寨卡病毒患者中分离出针对E蛋白的特异性中和抗体Z20、Z23和Z3L1[27]。最近通过免疫小鼠分离出了靶向DⅢ的4种寨卡病毒特异性鼠中和mAb:ZV-48、ZV-54、ZV-64和ZV-67,且ZV-54和ZV-67具有更高的中和效率,对于不同的寨卡病毒毒株活性范围也更广[28]。
2.2 C蛋白和prM蛋白C蛋白与病毒RNA结合形成二十面体的核衣壳结构,通过与细胞蛋白相互作用调节细胞代谢、细胞凋亡和免疫应答,在病毒感染过程中发挥重要作用。未成熟的寨卡病毒含有部分有序的C蛋白壳,但在成熟的寨卡病毒颗粒中缺乏,推测C蛋白壳在成熟过程中发生重排[14, 29]。C蛋白在1.9 Å下的晶体结构主要包含4个α螺旋和一个长的pre-α1环并形成二聚体。与西尼罗病毒和登革病毒不同,独特的长pre-α1环有助于形成更紧凑的二聚体,并在脂质双层界面呈现发散的疏水特性。更有意思的是,在寨卡病毒感染的细胞中,C蛋白聚集在脂滴(Lipid droplets,LDs)表面,但是pre-α1环关键残基突变后,C蛋白就完全不能在脂滴聚集,表明pre-α1环是膜融合的关键。此外,寨卡病毒C蛋白具有广泛的结合能力,能够结合不同的核酸类型,包括单链和双链的RNA或DNA。主要由α4螺旋形成的高电荷界面可能负责核酸的结合[13]。寨卡病毒C蛋白晶体结构的解析为靶向C蛋白抗寨卡病毒药物的设计提供新思路。
M蛋白隐藏在E蛋白层下,由N末端的M环、包含一个螺旋的茎和包含2个螺旋的跨膜区组成,这些结构利于将M蛋白锚定在脂膜上。在成熟病毒颗粒释放的过程中,prM蛋白被位于反式高尔基体网络中的弗林蛋白酶切割成pr肽和M蛋白,prM蛋白参与E蛋白折叠。引起此次寨卡病毒病暴发的流行株,其未成熟颗粒pr三聚体间的氨基酸片段与以往的毒株不同,推测其可能影响未成熟病毒颗粒的构象变化[14],本实验室进一步研究发现,S139N变异使寨卡病毒的神经毒力和致小颅畸形能力显著增强[30]。并提出其可能通过影响病毒颗粒的成熟及子代病毒的异质性而实现上述表型的改变。
3 寨卡病毒非结构蛋白的结构 3.1 NS1蛋白的结构及其特异性NS1蛋白的整体结构与DENV-2和WNV NS1的结构相似,具有相同的蛋白质折叠和结构域排列[16]。它们都形成同源二聚体,每个单体有3个结构域:β-发夹结构域、翼结构域和β-梯形结构域。NS1二聚体有内外2个表面,其中内表面上有一个不连续的线性疏水性突起,是膜相互作用的主要候选区域,有利于膜融合,而外表面是极性,含有高度保守的N130糖基化位点。在NS1六聚体结构中,3个二聚体与极性外表面组合,疏水性内表面指向内,利于与sNS1脂蛋白颗粒中的脂质分子相互作用。
NS1蛋白是病毒感染和复制的必需蛋白质,通过与宿主先天和适应性免疫系统的组分及其它宿主因子相互作用参与免疫逃逸和发病机制,也是黄病毒感染的主要抗原标志物。最近从寨卡病毒患者中分离的一组mAb识别NS1蛋白,主要是寨卡病毒特异性,而交叉反应性低,在预先暴露于DENV的供体中也是如此,说明寨卡病毒NS1蛋白可诱导出明显的寨卡病毒特异性免疫应答[31]。因此,开发针对寨卡病毒NS1的血清学诊断工具具有实际意义,NS1将成为疾病诊断的主要生物标志物。
与其他黄病毒相比,寨卡病毒NS1结构具有独特的静电势,主要表现在β-梯形结构域的环形表面[17],DENV-2和WNV的NS1结构在环形表面的中心区域分别显示带正电和负电,而对于寨卡病毒,环形表面呈现包含带正电荷和带负电荷的中心区域及朝向2个远端的带负电区域的复合表面。在NS1内表面,仅寨卡病毒显示负电荷,DENV-2和WNV显示中性电荷。此外,寨卡病毒NS1在翼结构域外表面的尖端区域带正电荷,WNV NS1的相应结构则带负电荷,而DENV-2 NS1结构既包含带正电荷区域,又包含带负电荷区域。鉴于寨卡病毒NS1具有发散静电势的界面,它可能含有改变与宿主因子的结合特性和抗黄病毒NS1的抗体;可能与寨卡病毒神经向性有关,有助于病毒跨越血脑屏障、血胎盘屏障、血眼屏障和血睾屏障;也可用于开发寨卡病毒感染的新型治疗和诊断工具[31]。当然这些都需要进一步的探索和试验。
3.2 NS2B-NS3复合体及其抑制剂NS2B蛋白激活NS3蛋白活性区(含催化三联体Ser135-His51-Asp75)并与之组成复合体发挥蛋白水解酶活性。病毒多聚蛋白前体经宿主细胞蛋白酶和病毒NS2B-NS3蛋白酶加工为成熟蛋白(3个结构蛋白和7个非结构蛋白)[32]。构建体(NS2B-NS3pro)的研究显示强的肽解活性[18, 33],具有良好排序的NS3pro核心和柔性NS2B部分的“开放构象”,与NS3pro有限的相互作用,与抑制剂结合诱导NS2B显著的构象变化,产生紧密的“封闭”形式。未连接的NS2B-NS3蛋白酶晶体结构显示,其采用封闭构象,NS2B与NS3结合形成空的底物结合位点,底物肽结合不诱导进一步的实质构象变化。人造接头引入空间位阻并改变底物(抑制剂)结合行为,说明寨卡病毒未连接NS2B-NS3蛋白酶可用于研究酶行为和抑制剂的发现,是具有吸引力的抗病毒药物靶点[19]。有研究报道了以C对N取向结合肽的黄病毒蛋白酶的共晶体结构,肽键的相反方向不能在蛋白酶活性位点形成四面体的中间体,因此不可裂解。尽管这可能是一个结晶过程中形成的非天然状态,但肽在蛋白酶口袋中的良好配合为开发新型肽模拟抑制剂(如环肽)提供了新的见解和方向。
3.3 NS5蛋白及其药物靶标NS5蛋白是分子量最大的非结构蛋白,包含N端甲基转移酶(Methyltransferase,MTase)结构域和C端RNA依赖性RNA聚合酶结构域(RNA-dependent RNA polymerase,RdRp),二者在RNA合成的起始和延伸过程中相互协作[20]。另外,有研究表明NS5蛋白抑制Ⅰ型干扰素信号通路以逃避宿主的抗病毒防御,NS5在病毒复制和免疫抑制中的作用使其成为抗病毒的理想靶点[34]。目前已经获得了寨卡病毒NS5全长的晶体结构,发现N端典型的S-腺苷甲硫氨酸依赖性的MTase区域位于C-端RdRps区域的顶端,两区域在域间并无广泛的接触,且MTase-RdRp结合也未引起MTase区域有意义的构象改变[21-23],寨卡等黄病毒NS5蛋白两交替构象的功能作用需要进一步的生物和细胞学研究。
ZIKV NS5 RdRp是具有手指、手掌和拇指子域的右手构象,相比于其他黄病毒的RdRp,拇指子域更多的旋转到手指子域[35],表明RNA模板入口隧道较窄。基于73种黄病毒RdRp序列比对的保守分析显示,引物环、酶活性位点、RNA模板进入隧道及锌口袋位点是最保守的区域[36],可能是广泛反应性药物的候选靶标,其他黄病毒的抑制剂是否可用于治疗寨卡病毒感染尚需进一步研究。
4 展望寨卡病毒持续在美洲和亚洲传播,迫切需要寨卡病毒相关疫苗和药物。最近的研究表明,减毒活疫苗、DNA疫苗等疫苗可以在不同的动物模型(小鼠和非人灵长类)中有效预防寨卡病毒的先天性感染和疾病,寨卡病毒蛋白晶体结构的解析为这些疫苗的研发提供了重要的科学依据。但目前对于NS2A、NS4A等膜锚定的非结构蛋白的三维结构与功能尚缺乏足够认识。例如,这些重要非结构蛋白在内质网中是如何组装成复制复合物,并行使其生物学功能的?再如,它们抑制宿主干扰素信号通路的结构基础是怎样的?这一系列关键科学问题的阐明将能够极大加深我们对寨卡病毒免疫学和发病机理的理解和认识,有助于研发安全有效的寨卡病毒疫苗和药物。
| [1] |
Shan C, Xie XP, Muruato AE, et al. An infectious cDNA clone of Zika virus to study viral virulence, mosquito transmission, and antiviral inhibitors[J]. Cell Host & Microbe, 2016, 19(6): 891-900. |
| [2] |
Zhang FC, Li XF, Deng YQ, et al. Excretion of infectious Zika virus in urine[J]. The Lancet: Infectious Diseases, 2016, 16(6): 641-642. DOI:10.1016/S1473-3099(16)30070-6 |
| [3] |
Brasil P, Pereira JP Jr, Moreira ME, et al. Zika virus infection in pregnant women in Rio de Janeiro[J]. The New England Journal of Medicine, 2016, 375(24): 2321-2334. DOI:10.1056/NEJMoa1602412 |
| [4] |
Brasil P, Sequeira PC, Freitas AD, et al. Guillain-Barré syndrome associated with Zika virus infection[J]. Lancet, 2016, 387(10026): 10026. |
| [5] |
PAHO, WHO. 泛美卫生组织(PAHO)和世界卫生组织(WHO)关于寨卡疫情的通报[EB/OL]. [2018-01-04]. http://www.paho.org/hq/index.php?option=com_content&view=article&id=12390&Itemid=42090&lang=en
|
| [6] |
Lazear HM, Govero J, Smith AM, et al. A mouse model of Zika virus pathogenesis[J]. Cell Host & Microbe, 2016, 19(5): 720-730. |
| [7] |
Zhang XK, Ge P, Yu XK, et al. Cryo-EM structure of the mature dengue virus at 3.5- resolution[J]. Nature Structural & Molecular Biology, 2013, 20(1): 105-110. |
| [8] |
Kostyuchenko VA, Lim EXY, Zhang SJ, et al. Structure of the thermally stable Zika virus[J]. Nature, 2016, 533(7603): 425-428. DOI:10.1038/nature17994 |
| [9] |
Sirohi D, Chen ZG, Sun L, et al. The 3.8 resolution cryo-EM structure of Zika virus[J]. Science, 2016, 352(6284): 467-470. DOI:10.1126/science.aaf5316 |
| [10] |
Navarro-Sanchez E, Altmeyer R, Amara A, et al. Dendritic-cell-specific ICAM3-grabbing non-integrin is essential for the productive infection of human dendritic cells by mosquito-cell-derived dengue viruses[J]. EMBO Reports, 2003, 4(7): 723-728. DOI:10.1038/sj.embor.embor866 |
| [11] |
Pokidysheva E, Zhang Y, Battisti AJ, et al. Cryo-EM reconstruction of dengue virus in complex with the carbohydrate recognition domain of DC-SIGN[J]. Cell, 2006, 124(3): 485-493. DOI:10.1016/j.cell.2005.11.042 |
| [12] |
Miller JL, deWet BJM, Martinez-Pomares L, et al. The mannose receptor mediates dengue virus infection of macrophages[J]. PLoS Pathogens, 2008, 4(2): e17. DOI:10.1371/journal.ppat.0040017 |
| [13] |
Shang ZF, Song H, Shi Y, et al. Crystal structure of the capsid protein from Zika virus[J]. Journal of Molecular Biology, 2018, 430(7): 948-962. DOI:10.1016/j.jmb.2018.02.006 |
| [14] |
Prasad VM, Miller AS, Klose T, et al. Structure of the immature Zika virus at 9 resolution[J]. Nature Structural & Molecular Biology, 2017, 24(2): 184-186. |
| [15] |
Dai LP, Song J, Lu XS, et al. Structures of the Zika virus envelope protein and its complex with a flavivirus broadly protective antibody[J]. Cell Host & Microbe, 2016, 19(5): 696-704. |
| [16] |
Brown WC, Akey DL, Konwerski JR, et al. Extended surface for membrane association in Zika virus NS1 structure[J]. Nature Structural & Molecular Biology, 2016, 23(9): 865-867. |
| [17] |
Song H, Qi JX, Haywood J, et al. Zika virus NS1 structure reveals diversity of electrostatic surfaces among flaviviruses[J]. Nature Structural & Molecular Biology, 2016, 23(5): 456-458. |
| [18] |
Lei J, Hansen G, Nitsche C, et al. Crystal structure of Zika virus NS2B-NS3 protease in complex with a boronate inhibitor[J]. Science, 2016, 353(6298): 503-505. DOI:10.1126/science.aag2419 |
| [19] |
Zhang ZZ, Li Y, Loh YR, et al. Crystal structure of unlinked NS2B-NS3 protease from Zika virus[J]. Science, 2016, 354(6319): 1597-1600. DOI:10.1126/science.aai9309 |
| [20] |
Zhang CY, Feng TT, Cheng JB, et al. Structure of the NS5 methyltransferase from Zika virus and implications in inhibitor design[J]. Biochemical and Biophysical Research Communications, 2017, 492(4): 624-630. DOI:10.1016/j.bbrc.2016.11.098 |
| [21] |
Zhao BY, Yi GH, Du FL, et al. Structure and function of the Zika virus full-length NS5 protein[J]. Nature Communications, 2017, 8: 14762. DOI:10.1038/ncomms14762 |
| [22] |
Wang BX, Tan XF, Thurmond S, et al. The structure of Zika virus NS5 reveals a conserved domain conformation[J]. Nature Communications, 2017, 8: 14763. DOI:10.1038/ncomms14763 |
| [23] |
Godoy AS, Lima GMA, Oliveira KIZ, et al. Crystal structure of Zika virus NS5 RNA-dependent RNA polymerase[J]. Nature Communications, 2017, 8: 14764. DOI:10.1038/ncomms14764 |
| [24] |
Mansuy JM, Dutertre M, Mengelle C, et al. Zika virus: high infectious viral load in semen, a new sexually transmitted pathogen?[J]. The Lancet: Infectious Diseases, 2016, 16(4): 405. |
| [25] |
Musso D, Roche C, Nhan TX, et al. Detection of Zika virus in saliva[J]. Journal of Clinical Virology, 2015, 68: 53-55. DOI:10.1016/j.jcv.2015.04.021 |
| [26] |
Barba-Spaeth G, Dejnirattisai W, Rouvinski A, et al. Structural basis of potent Zika-dengue virus antibody cross-neutralization[J]. Nature, 2016, 536(7614): 48-53. DOI:10.1038/nature18938 |
| [27] |
Wang QH, Yang HB, Liu XQ, et al. Molecular determinants of human neutralizing antibodies isolated from a patient infected with Zika virus[J]. Science Translational Medicine, 2016, 8(369): 369ra179. DOI:10.1126/scitranslmed.aai8336 |
| [28] |
Zhao HY, Fernandez E, Dowd KA, et al. Structural basis of Zika virus-specific antibody protection[J]. Cell, 2016, 166(4): 1016-1027. DOI:10.1016/j.cell.2016.07.020 |
| [29] |
Oliveira ERA, Mohana-Borges R, de Alencastro RB, et al. The flavivirus capsid protein: structure, function and perspectives towards drug design[J]. Virus Research, 2017, 227: 115-123. DOI:10.1016/j.virusres.2016.10.005 |
| [30] |
Yuan L, Huang XY, Liu ZY, et al. A single mutation in the prM protein of Zika virus contributes to fetal microcephaly[J]. Science, 2017, 358(6365): 933-936. DOI:10.1126/science.aam7120 |
| [31] |
Stettler K, Beltramello M, Espinosa DA, et al. Specificity, cross-reactivity, and function of antibodies elicited by Zika virus infection[J]. Science, 2016, 353(6301): 823-826. DOI:10.1126/science.aaf8505 |
| [32] |
Badshah SL, Naeem A, Mabkhot Y. The new high resolution crystal structure of NS2B-NS3 protease of Zika virus[J]. Viruses, 2017, 9(1): 7. DOI:10.3390/v9010007 |
| [33] |
Chen X, Yang KL, Wu C, et al. Mechanisms of activation and inhibition of Zika virus NS2B-NS3 protease[J]. Cell Research, 2016, 26(11): 1260-1263. DOI:10.1038/cr.2016.116 |
| [34] |
Grant A, Ponia SS, Tripathi S, et al. Zika virus targets human STAT2 to inhibit type Ⅰ interferon signaling[J]. Cell Host & Microbe, 2016, 19(6): 882-890. |
| [35] |
Duan WQ, Song H, Wang HY, et al. The crystal structure of Zika virus NS5 reveals conserved drug targets[J]. The EMBO Journal, 2017, 36(7): 919-933. DOI:10.15252/embj.201696241 |
| [36] |
Shi Y, Gao GF. Structural biology of the Zika virus[J]. Trends in Biochemical Sciences, 2017, 42(6): 443-456. DOI:10.1016/j.tibs.2017.02.009 |
 2018, Vol. 45
2018, Vol. 45