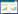扩展功能
文章信息
- 仇焕娜, 赵东东, 满淑丽, 毕昌昊, 朱欣娜, 张学礼
- QIU Huan-Na, ZHAO Dong-Dong, MAN Shu-Li, BI Chang-Hao, ZHU Xin-Na, ZHANG Xue-Li
- 大肠杆菌染色体上严谨型启动子的构建
- Construction of promoters with tight regulation on chromosome of Escherichia coli
- 微生物学通报, 2018, 45(8): 1693-1704
- Microbiology China, 2018, 45(8): 1693-1704
- DOI: 10.13344/j.microbiol.china.170847
-
文章历史
- 收稿日期: 2017-10-19
- 接受日期: 2017-12-28
- 网络首发日期(www.cnki.net): 2018-01-08
2. 中国科学院天津工业生物技术研究所 天津 300308;
3. 中国科学院系统微生物工程重点实验室 天津 300308
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3. Key Laboratory of Systems Microbial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
启动子是位于结构基因5′-上游的DNA序列,用于RNA聚合酶特异识别和结合。它控制着基因表达(转录)的起始时间和程度[1-2]。细菌的基因表达调控主要发生在转录水平上,而启动子在转录水平调控中居于关键的地位。随着合成生物学和代谢工程的发展,细菌启动子获得了越来越多的应用。利用启动子可以调控基因的表达强度,改造细菌的代谢通路,或者方便地表达目的蛋白,使其在细胞工厂和酶催化中应用广泛[3]。
诱导型启动子Plac、Ptac和Ptrc,其上含有lacO调控序列,当无诱导剂存在时,阻遏蛋白LacI和调控序列结合,抑制启动子的表达[4]。在常规的大肠杆菌中,LacI的表达量不高,导致非诱导条件下较高的表达,系统严谨性不强。虽然突变的阻遏蛋白LacIq能部分解除非诱导条件下的高表达,但仍然不够严谨[5]。常用的λ噬菌体PL是一个组成型启动子,其表达不需要诱导剂,但这种启动子的表达不受控制[6]。实际应用中生物合成途径中某些基因的表达,需要对表达时间和表达量进行控制,而不受控制的启动子很难实现这一点,因此,有必要构建出严谨型的启动子,使基因的表达在可调控下进行。
人们常常在质粒上研究启动子的强度,用于代谢途径中关键基因的表达,实现提高目标产品的生产能力[7]。然而质粒用于表达具有诸多弊端[8]:质粒的维持对宿主细胞会造成很大的代谢负荷,尤其是高拷贝的质粒;很多质粒的遗传稳定性不好,不利于工业化大规模的生产;只有低拷贝数质粒的复制和细胞的繁殖是同步进行的,因此也只有它们在所有的细胞中保持一致的拷贝数;很多化合物的合成需要在细胞中构建出一条复杂的代谢途径,因此需要引入包含多个基因的长DNA片段,而大部分质粒都比较难携带长DNA片段。
在质粒上研究启动子和基因的表达时,由于含质粒的工程菌株不稳定性,在确定了目标基因的表达水平后,最终的目标还是将启动子和目标基因整合于染色体上,但是整合于染色体上的基因表达强度会和质粒上有偏差,在某个启动子控制下基因在染色体上的表达不是最优状态。因此,通过在细胞染色体上直接研究启动子强度来调控基因表达的策略和质粒过表达相比有很多优势。
基因编辑技术CRISPR/Cas9已经成功地应用于大肠杆菌,实现了基因的插入、删除和替换[9-10]。我们课题组已经开发出一种高效的基因组编辑技术,只需一个质粒的构建和转化的CRISPR/Cas9[11]就能对基因进行编辑。CRISPR/Cas9系统不仅消除了基因组编辑选择标记,也提高了编辑工作效率[12]。在本研究中我们利用CRISPR/Cas9将启动子整合于染色体上进行表达分析。
基于严谨型启动子对于合成生物学和代谢工程中的重要作用,本研究基于4种调控元件四环素tetO、乳糖lacO、阿拉伯糖araC和鼠李糖rhaR的结合序列,设计和组合构建了6个启动子PtetO2、PtetO3、PlacO2、PlacO3、PlacO+ara和PlacO+rha。应用CRISPR/Cas9系统将这6个启动子序列整合到大肠杆菌ATCC 8739染色体上,利用绿色荧光蛋白GFP的表达分析了这6个启动子的相对表达强度和严谨型控制情况。
1 材料与方法 1.1 菌种来源本实验所用菌株和质粒见表 1。
| 菌株和质粒 Strains and plasmids |
遗传特征 Genetic characteristics |
来源 Source |
| Strains | ||
| E. coli ATCC 8739 | Wild type | Lab collection |
| M1-46 | ATCC 8739, FRT-km-FRT::M1-46::lacZ | [8] |
| S-PlacZ | ATCC 8739, ΔlacZ-PlacZ-gfp | This work |
| S-PlacO2 | ATCC 8739, ΔlacZ-PlacO2-gfp | This work |
| S-PlacO3 | ATCC 8739, ΔlacZ-PlacO3-gfp | This work |
| S-ΔaraBAD | ATCC 8739, ΔaraBAD | This work |
| S-PlacO+ara | ATCC 8739, ΔaraBAD, ΔlacZ-PlacO+ara-gfp, | This work |
| S-ΔrhaBAD | ATCC 8739, ΔrhaBAD | This work |
| S-PlacO+rha | ATCC 8739, ΔrhaBAD, ΔlacZ-PlacO+rha-gfp | This work |
| S-tetR | ATCC 8739, ΔrhaBAD::M1-46-tetR | This work |
| S-PtetO2 | ATCC 8739, ΔrhaBAD::M1-46-tetR, ΔlacZ-PtetO2-gfp | This work |
| S-PtetO3 | ATCC 8739, ΔrhaBAD::M1-46-tetR, ΔlacZ-PtetO3-gfp | This work |
| Plasmids | ||
| pUC57-Kan | plac, ColEl, kan | Lab collection |
| pACYC184-M | cat, replace tet with lacI and Ptrc of pTrc99A-M | [13] |
| pRed_Cas9_ΔpoxB300 | Derived from pKD46, exo, bet, gam, arabinose operon, Cas9, gRNA with N20 and with homologous arms | [11] |
| pCmGFP | A plasmid containing gfp gene | [14] |
| pSC101 | tet, SC101, repA | Lab collection |
| pGFP | A plasmid modified from pCmGFP by removing the Bsa Ⅰ restriction endonuclease site of gfp gene | This work |
| placZ | cat and P15A from pACYC184-M, N20-gRNA targeting lacZ gene expressed under constitute promoter, lacZ gene and its up and down homologous hand | This work |
| pΔlacZ-PlacZ-gfp | lacZ gene was replaced by gfp gene using placZ-N20 as template | This work |
| pΔlacZ-PlacO2-gfp | Pnative-lacZ promoter was replaced by PlacO2 promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔlacZ-PlacO3-gfp | Pnative-lacZ promoter was replaced by PlacO3 promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔlacZ-PlacO+ara-gfp | Pnative-lacZ promoter was replaced by PlacO+ara promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔlacZ-PlacO+rha-gfp | Pnative-lacZ promoter was replaced by PlacO+rha promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔlacZ-PlacO+tetO2-gfp | Pnative-lacZ promoter was replaced by PlacO+tetO2 promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔlacZ-PlacO+tetO3-gfp | Pnative-lacZ promoter was replaced by PlacO+tetO3 promoter using pΔlacZ-Pnative-lacZ-gfp as template | This work |
| pΔaraBAD-N20 | cat, P15A, N20-gRNA targeting araBAD gene expressed under constitute promoter, the up and down homologous sequence of araBAD gene, which used for deletion ara BAD gene | This work |
| pΔrhaBAD-N20 | cat, P15A, N20-gRNA targeting rhaBAD gene expressed under constitute promoter, the up and down homologous sequence of rhaBAD gene, which used for deletion rhaBAD gene | This work |
| pΔrhaBAD::M1-46-tetR | M1-46-tetR gene was inserted between the up and down homologous sequence of rhaBAD gene, which used for integrated tetR gene | This work |
| pRedCas9 | kan, PBAD-cas9, PBAD-Red-recA, repA101(Ts) | [11] |
LB培养基(g/L):酵母膏5.0,蛋白胨10.0,氯化钠10.0,琼脂15.0 (固体培养基)大肠杆菌于37 ℃、250 r/min培养。
1.3 主要试剂和仪器抗生素和诱导剂,Sigma-Aldrich公司。抗生素的使用浓度分别为:氨苄霉素100 μg/mL,卡那霉素50 μg/mL,氯霉素34 μg/mL。诱导剂:IPTG、脱水四环素、L(+)阿拉伯糖、L(+)鼠李糖。PCR高保真扩增Phusion酶、快速T4 DNA连接酶、限制性内切酶Bsa Ⅰ,NEB生物公司;Trans T1感受态细胞,全式金生物技术有限公司。离心机,Sigma实验室离心机公司;荧光显微镜,奥林巴斯公司;荧光强度分析酶标仪,TECAN酶标仪公司;全自动凝胶成像仪,Bio-rad公司。
1.4 启动子合成6条启动子PtetO2、PtetO3、PlacO2、PlacO3、PlacO+ara和PlacO+rha (含RBS结合位点,表 2)分别全基因合成于质粒pUC57-Kan上。为方便后续克隆,每条启动子的上下游加了20-30 bp的特殊序列:上游为5′-TGGTTAATGACAGTGGGACGggtctca-3′,下游为5′-tgagaccAACCTCGGCATTGCGTAA-3′,斜体小写为Bsa Ⅰ酶切位点。
| 引物及调控元件 Primers and regulatory parts |
序列 Sequences (5′→3′) |
| Construction of placZ | |
| N20-B-F1 | CCAGGTCTCAGTGCGCCATGAGAACGAACCATTG |
| N20-B-R1 | CCAGGTCTCAGCTAAGATCTGACTCCATAACAGAGTACTCGC |
| lacZ-N20-B-F2 | CCAGGTCTCATAGCTTTACAGGGCGGCTTCGTCT GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC |
| N20-B-R2 | CCAGGTCTCAACCGCTGGCTAAATACGGAAGGATCT |
| lacZ-F1 | CCAGGTCTCACGGT GGATACGACGATACCGAAGA |
| lacZ-R1 | CCAGGTCTCAGCAC TGACTTTCTCAATAAATGCCTCTAC |
| Construction of pGFP | |
| Gfp-GGA-F | GGACCACATGGTCCTTCTTGAGTT |
| gfp-GGA-R | CCATGTGGTCCCTCTTTTCGTTG |
| Construction of pΔlacZ-PlacZ-gfp | |
| lacZ-B-F2 | CCAGGTCTCA CCAG TAATAACCGGGCAGGCCAT |
| lacZ-B-R2 | CCAGGTCTCA AGCTGTTTCCTGTGTGAAATTGT |
| gfp-F | CCAGGTCTCAAGCT ATGGGTAAGGGAGAAGAACTTTTC |
| gfp-R | CCAGGTCTCACTGG TTATTTGTATAGTTCATCCATGCC |
| Constructing plasmid with designed promoter | |
| Promoter-F | TGGTTAATGACAGTGGGACGggtctca |
| Promoter-R | TTACGCAATGCCGAGGTTggtctca |
| GFP-B-P-F1 | CCAGGTCTCATACCATGGGTAAGGGAGAAGAACTTTTC |
| GFP-B-Plac-R1 | CCAGGTCTCATATATCACTGCCCGCTTTCCAG |
| GFP-B-Para-R1 | CCAGGTCTCATATGTCACTGCCCGCTTTCCAG |
| GFP-B-Prha-R1 | CCAGGTCTCAAGATTCACTGCCCGCTTTCCAG |
| GFP-B-Ptet-R1 | CCAGGTCTCAATTTTCACTGCCCGCTTTCCAG |
| Detecting promoter with gfp integration | |
| N20-B-YZ-F | TCGAGTAAGGATCTCCAGGC |
| lacZ-YZ-up | TGGTTGCCAACGATCAGA |
| gfp-YZ-down200 | AAGTCGTGCCGCTTCATA |
| araBAD-YZ-up | GATTTCTGCGTCGGTCATT |
| rhaBAD-YZ-up | AAAAGTGGTTACTGTCGCTGAA |
| Construction of pΔaraBAD-N20 | |
| araBAD-N20-B-F2 | CCAGGTCTCATAGCGCACATGGCGGTCGTGAGTTGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC |
| araBAD-F1 | CCAGGTCTCACGGTCGGACGAAAGTAAACCCACTG |
| araBAD-R1 | CCAGGTCTCACGGCAGTGTGACGCCGTGCAAATAAT |
| araBAD-F2 | CCAGGTCTCAGCACGGTTTCGATGCTAACCACGAC |
| araBAD-R2 | CCAGGTCTCAGCACGGTTTCGATGCTAACCACGAC |
| Construction of pΔrhaBAD-N20 | |
| rhaBAD-N20-B-F2 | CCAGGTCTCATAGCTGTTTCCGGTTTTGAAAACTGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGC |
| rhaBAD-F1 | CCAGGTCTCACGGTCTTTTACGCAGCAAGAGCAG |
| rhaBAD-R1 | CCAGGTCTCAAATG GAACGTGATGATGTTCACAATTT |
| rhaBAD-F2 | CCAGGTCTCA CATT ACTCAGTCCTGGTTTGTC |
| rhaBAD-R2 | CCAGGTCTCAGCACCGGGAATACCACCAGACACT |
| Instruction of pΔrhaBAD::M1-46-tetR | |
| rhaBAD-B-F1 | CCAGGTCTCACGCACATTACTCAGTCCTGGTTTGTC |
| rhaBAD-B-R1 | CCAGGTCTCAAGTGGAACGTGATGATGTTCACAATTT |
| M1-46-up-F | CCAGGTCTCACACTTTATCTCTGGCGGTGTTGAC |
| M1-46-down-R | CCAGGTCTCA AGCTGTTTCCTGGTTTAAACC |
| tetR-F | CCAGGTCTCAAGCTATGTCTAGATTAGATAAAAGTAAAGTGATTAAC |
| tetR-R | CCAGGTCTCATGCGTTAAGACCCACTTTCACATTTAAGT |
| p-up | TTATCTCTGGCGGTGTTGACA |
| Synthesis promoter | |
| PtetO2 | AAATCCCTATCAGTGATAGAGATTGACATCCCTATCAGTGATAGAGATACTGAGCACATCAGCAGGACGCACTGACCTTCACACAAGGAGATATACC (97 bp) |
| PtetO3 | AAATCCCTATCAGTGATAGAGATTGACATCCCTATCAGTGATAGAGATACTGAGCACATCCCTATCAGTGATAGAGATTCACACAAGGAGATATACC (97 bp) |
| PlacO2 | TATAAATTGTGAGCGGATAACATTGACATTGTGAGCGGATAACAAGATACTGACCACATCAGGAGGACGCACTGACCTTCACACAAGGAGATATACC (97 bp) |
| PlacO3 | TATAAATTGTGAGCGGATAACATTGACATTGTGAGCGGATAACAAGATACTGACCACATTGTGAGCGGATAACAACCTTCACACAAGGAGATATACC (97 bp) |
| PlacO+ara | CATAGCATTTTTATCCATAAGATTAGCGGATCCTAAGCTTGACATTGTGAGCGGATAACAATATAATGAGCACAATTGTGAGCGGATAACAACCTTCACACAAGGAGATATACC (114 bp) |
| PlacO+ara | ATCTTTCCCTGGTTGCCAATGGCCCATTTTCCTGTCAGTAACGAGAAGGTTTGACATTGTGAGCGGATAACAATATAATGAGCACAATTGTGAGCGGATAACAACCTTCACACAAGGAGATATACC (126 bp) |
| 注:下划线为靶向各基因的N20序列;小写ggtctc为Bsa Ⅰ酶切位点. Note: The underlines represent the N20 sequence of the targeting gene; The Bsa Ⅰ is represented by lowercase letters (ggtctc). |
|
质粒pΔlacZ-PlacZ-gfp含有在PlacZ启动子下的gfp基因,及针对lacZ基因的N20-gRNA。为构建质粒pΔlacZ-PlacZ-gfp,首先构建placZ质粒和pGFP质粒。
placZ质粒含有氯霉素抗性基因cat,复制子P15A,识别lacZ基因的lacZN20-gRNA序列,lacZ基因和该基因上下游同源臂序列。其中,氯霉素抗性基因cat和复制子P15A序列,以质粒pACYC184-M[13]为模板,用引物对N20-B-F1/ N20-B-R1扩增获得;lacZN20-gRNA的片段以质粒pRed_Cas9_ΔpoxB300[11]为模板,用引物lacZ-N20-B-F2/N20-B-R2扩增;lacZ基因及同源臂序列,以E. coli ATCC 8739 DNA为模板,用引物lacZ-F1/lacZ-R1扩增得到。这3个片段用Golden Gate技术策略[16]进行连接,转化TransT1感受态细胞,得到的克隆用引物N20-B-YZ-F/lacZ-R1进行验证,阳性克隆提取质粒DNA送样测序分析,得到正确的placZ质粒。
pGFP质粒含有gfp基因,为使gfp基因序列不含有Bsa Ⅰ酶切位点,从而方便运用Golden Gate策略,对出发质粒pCmGFP上gfp的Bsa Ⅰ酶切位点的序列用简并碱基进行了替换。以pCmGFP质粒为模板,用引物Gfp-GGA-F/Gfp-GGA-R (引物序列上含有改变的碱基,且两条引物上含有互补序列)进行PCR扩增,得到的片段用Dpn Ⅰ处理后,直接转化TransT1感受态细胞,提取质粒DNA测序分析,得到正确的pGFP质粒。
在上述2个质粒构建的基础上,构建pΔlacZ-PlacZ-gfp质粒。具体过程如下:以placZ质粒为模板,用引物lacZ-B-F2/lacZ-B-R2进行反向扩增,得到含有cat、P15A、lacZN20-gRNA、PlacZ和lacZ基因外上下游同源臂序列片段;以pGFP质粒为模板,用引物gfp-F/gfp-R扩增得到gfp基因序列。用同样的Golden Gate策略将这2个片段连接起来,转化TransT1感受态细胞,得到的克隆用引物lacZ-F1/gfp-YZ-down200进行验证,阳性克隆提取质粒DNA送样测序分析,得到正确的pΔlacZ-PlacZ-gfp质粒。
所用引物见表 2。
1.5.2 启动子表达质粒系列pΔlacZ-Pxx-gfp的构建分别以6个启动子PtetO2、PtetO3、PlacO2、PlacO3、PlacO+ara和PlacO+rha的全基因合成质粒为模板,用引物Promoter-F/Promoter-R进行PCR扩增得到6个启动子序列。
以pΔlacZ-PlacZ-gfp为模板,反向PCR扩增获得连接启动子的骨架片段。正向引物GFP-B-P-F1为扩增骨架通用引物。当反向引物为GFP-B-Plac-R1时,扩增用于连接PlacO2和PlacO3启动子的骨架片段;反向引物为GFP-B-Ptet-R1扩增用于连接PtetO2和PtetO3启动子的骨架片段;反向引物GFP-B-Para-R1和GFP-B-Prha-R1分别扩增用于连接PlacO+ara和PlacO+rha启动子的骨架片段。
用Golden Gate策略将骨架片段和相应的启动子片段进行连接,转化TransT1感受态细胞,得到的克隆用引物lacZ-F1/gfp-YZ-down200进行验证,阳性克隆提取质粒DNA送样测序分析,得到正确的pΔlacZ-PtetO2-gfp、pΔlacZ-PtetO3-gfp、pΔlacZ-PlacO2-gfp、pΔlacZ-PlacO3-gfp、pΔlacZ-PlacO+ara-gfp和pΔlacZ-PlacO+rha-gfp质粒。
1.6 CRSIPR/Cas9技术将启动子整合染色体上利用CRISPR/Cas9基因编辑技术将启动子整合到染色体上,替换lacZ基因的启动子;同时将lacZ基因替换为绿色荧光蛋白gfp基因(图 1)。
|
| 图 1 基于CRISPR/Cas9技术将启动子整合到染色体上 Figure 1 Promoter integration into chromosomes by CRISPR/Cas9 Note: A: Co-transformation of pRedCas9 and pΔlacZ-Pxx-gfp into target strain; B: With the help of pRedCas9, promoter with gfp was inserted and replaced lacZ gene. |
|
|
PlacO2和PlacO3启动子的整合:将pRedCas9和启动子表达质粒如pΔlacZ-PlacO2-gfp或pΔlacZ-PlacO3-gfp同时电转化于E. coli ATCC 8739感受态细胞中,于卡那霉素和氯霉素双抗平板上30 ℃生长。挑取单克隆转接到2 mL LB [含卡纳霉素和氯霉素;2% L(+)-阿拉伯糖],30 ℃、250 r/min过夜诱导重组和切割未发生重组的DNA。稀释涂LB卡纳霉素和氯霉素双抗平板[2% L(+)-阿拉伯糖] 30 ℃生长。挑取10个单克隆,用引物lacZ-YZ-up/gfp-YZ-down200进行菌落PCR验证。阳性克隆的PCR反应体系送样测序分析。获得PlacO2和PlacO3启动子整合菌株S-PlacO2和S-PlacO3。同时,利用相同的策略,将pΔlacZ-Plac-gfp中的PlacZ-gfp整合,得到对照菌株S-Plac。
对于PlacO+ara和PlacO+rha启动子整合,分别从S-ΔaraBAD和S-ΔrhaBAD菌株(材料与方法1.7)出发,利用相同的启动子整合策略,将PlacO+ara-gfp和PlacO+rha-gfp整合,得到启动子整合菌株S-PlacO+ara和S-PlacO+rha。
对于PtetO2和PtetO3启动子的整合,从S-tetR菌株(1.8节)出发,利用相同的启动子整合策略,将PtetO2-gfp和PtetO3-gfp整合,得到启动子整合菌株S-tetO2和S-tetO3。
1.7 araBAD和rhaBAD基因的敲除araBAD基因敲除质粒的构建:以质粒placZ为模板,用引物N20-B-F1/N20-B-R1进行反向扩增获得通用骨架片段,含有cat和P15A序列;以相同质粒为模板,用引物araBAD-N20-B-F2/N20-B-R2扩增,获得araBADN20-gRNA的片段;以E. coli ATCC 8739 DNA为模板,分别用引物对araBAD-F1/araBAD-R1和araBAD-F2/araBAD-R2获得araBAD基因上下游同源臂序列。4个片段用Golden Gate技术策略[16]进行组装,转化TransT1感受态细胞,得到的克隆用引物N20-B-YZ-F/araBAD-BsaI-R2进行验证,阳性克隆提取质粒DNA送样测序分析,正确质粒命名为pΔaraBAD-N20。从E. coli ATCC 8739出发,制备电转化感受态细胞,利用CRISPR/Cas9基因编辑系统,共转化pRedCas9和pΔaraBAD-N20,实现araBAD基因的敲除,得到菌株S-ΔaraBAD,具体过程同1.6所述。
rhaBAD基因敲除质粒pΔrhaBAD-N20的构建同上,相应引物名称用rhaBAD替换araBAD,其敲除技术方法同araBAD基因的敲除,得到菌株S-ΔrhaBAD。
1.8 tetR基因的整合以质粒pΔrhaBAD-N20为模板,用引物rhaBAD-B-BsaI-F1/rhaBAD-B-BsaI-R1进行反向扩增,得到骨架片段;以M1-46基因组为模板[8],用引物M1-46-up-F/M1-46-R扩增得到M1-46启动子;以质粒pSC101为模板,用引物tetR-F/tetR-R扩增tetR基因序列。用Golden Gate策略将这3个片段组装,转化TransT1感受态细胞,得到的克隆用引物N20-B-YZ-F/tetR-R进行验证,阳性克隆提取质粒DNA送样测序分析,得到正确的pΔrhaBAD::M1-46-tetR质粒。从E. coli ATCC 8739出发,制备电转化感受态细胞,利用CRISPR/Cas9基因编辑系统,共转化pRedCas9和pΔrhaBAD:: M1-46-tetR,实现tetR基因的整合,得到菌株S-tetR,具体过程同1.6所述。
1.9 启动子相对表达强度分析及荧光显微镜观察挑取单克隆到1 mL LB试管37 ℃、250 r/min培养过夜;按1%转接到1 mL LB试管,37 ℃、250 r/min培养1 h后加诱导剂IPTG (终浓度为0.1 mmol/L),继续培养3 h后测荧光和荧光显微镜观察。取200 μL到96孔黑色透明板,用酶标仪测荧光及OD550;荧光强度值=(样品荧光强度-培养基背景荧光强度)/OD。同时取5 μL培养液于载玻片,在100倍油镜下用奥林巴斯荧光显微镜进行观察;GFP荧光强度测试条件:激发光波长488 nm;发射光520 nm;Gain 60。Exposure值为400 s,Gain 10,Saturation 10。
2 结果与分析 2.1 启动子的设计启动子的表达,由RNA聚合酶和启动子的结合速率决定[17]。RNA聚合酶和启动子结合速率低,抑制蛋白就会竞争性结合,启动子的表达受到抑制。众所周知,这种类型的启动子即使不诱导,仍然会有泄露的表达,如Plac启动子。另外一种类型的启动子,在不存在诱导剂的情况下,RNA聚合酶和启动子的结合速率高,启动子强烈表达,缺点是这种启动子的表达不受控制。因此,有必要设计出一类严谨型启动子,在抑制蛋白存在时,RNA聚合酶不能有效地与启动子结合,启动子暂不表达;在有激活剂(诱导剂)存在时,RNA聚合酶能有效地与启动子结合,启动子表达。
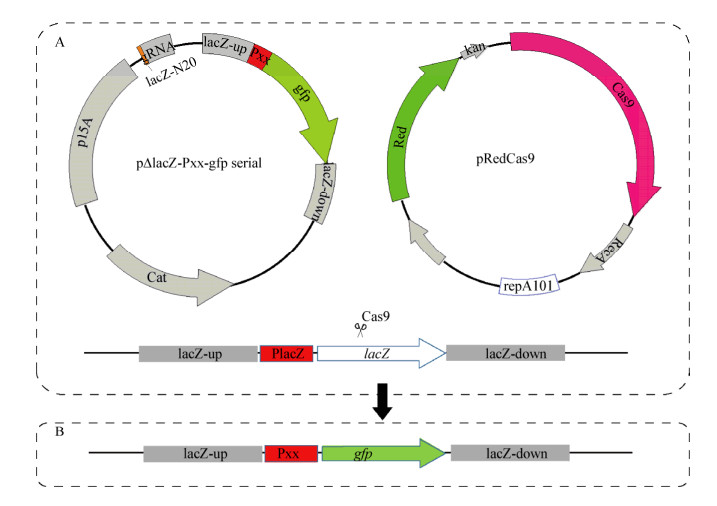
为更好地描述组合设计的启动子,我们对启动子上的序列相对于-10区和-35区的位置进行命名。将-35区上游的序列称为位置Ⅳ,-10区和-35区之间的序列称为位置Ⅲ,-10区下游的序列称为位置Ⅱ,核糖体RBS结合序列称为位置Ⅰ。我们对2种来源的启动子(PL和Plac)及4种调控元件tetO、lacO、ara和rha进行组合设计(图 2),组合的策略为将调控元件按一定的规律插入且替换位置Ⅳ、Ⅲ或者Ⅱ上的序列,以期从中得到具有严谨控制的启动子。λ噬菌体来源的PL启动子是一个强启动子,该启动子受cI蛋白的严格调控抑制[18]。用四环素操纵子的调控区序列tetO在位置Ⅳ和Ⅲ替换cI结合序列,得到97 bp的PtetO2;调控区序列tetO除替换启动子PL位置Ⅳ和Ⅲ外,还替换启动子下游序列位置Ⅱ,得到97 bp的PtetO3。类似的,用lacO调控序列替换PL启动子的位置Ⅳ,Ⅲ或者Ⅳ,Ⅲ和Ⅱ,分别得到97 bp的启动子PlacO2和PlacO3。Plac来源的启动子,将lacO调控序列替换到启动子Ⅱ和Ⅲ的位置,将ara调控序列替换到Ⅳ的位置得到114 bp的Plac+ara启动子;将rha调控序列替换到Ⅳ的位置得到126 bp的Plac+rha启动子。
|
| 图 2 启动子序列设计 Figure 2 Design promoters Note: The -10 and -35 hexamers are boxed and the transcriptional start site is dotted. Bars indicated the binding site of LacO (lacO), TetO (tetO), AraC (ara) and RhaR (rha) in promoters. |
|
|
利用CRISPR/Cas9基因编辑系统,本研究将6个合成的组合启动子序列PtetO2、PtetO3、PlacO2、PlacO3、PlacO+ara、PlacO+rha及对照启动子Plac,连同gfp基因同时整合于染色体上lacZ基因位点,替换野生型的PlacZ-lacZ基因元件(图 1)。具体的整合过程见1.6。
PlacO2、PlacO3和Plac启动子序列及gfp整合于E. coli ATCC 8739中;PtetO2和PtetO3启动子序列及gfp整合于S-tetR菌株中,该菌株在E. coli ATCC 8739菌株中整合了tetR基因,其编码蛋白TetR能调控含tetO序列类型启动子的表达;PlacO+ara启动子序列及gfp整合于E. coli S-araBAD菌株中,该菌株从E. coli ATCC 8739敲除了阿拉伯糖代谢途径基因araBAD,使阿拉伯糖作为诱导剂时不被细胞利用而消耗;PlacO+rha启动子序列及gfp整合到S-rhaBAD菌株中,该菌株从E. coli ATCC 8739敲除了鼠李糖代谢基因rhaBAD,使鼠李糖作为诱导剂时不被细胞利用而消耗。得到6个组合启动子整合菌株S-tetO2、S-tetO3、S-lacO2、S-lacO3、S-araBAD、S-rhaBAD和启动子对照菌株S-Plac。
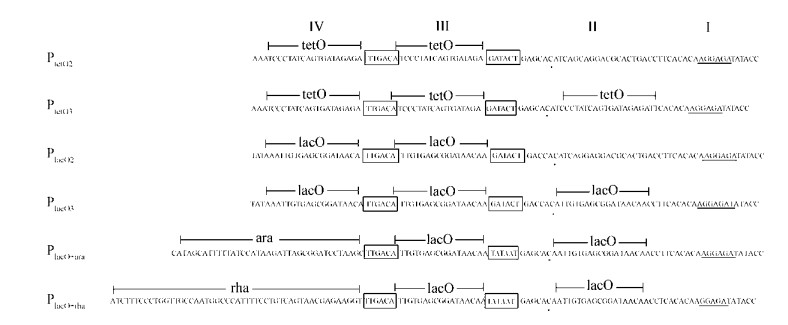
用引物lacZ-YZ-up/gfp-YZ-down200对整合后的组合启动子菌株进行PCR分析,得到大小约700 bp的目标条带(图 3)。并对PCR产物进行测序分析,测序分析结果显示得到正确的启动子的染色体整合菌株。
|
| 图 3 整合型启动子菌株的PCR鉴定 Figure 3 Determination of strains with promoter integration by PCR Note: M: DNA marker trans 2 000 plus; 1-7: PCR products from strains with promoter Plac, PtetO2, PtetO3, PlacO2, PlacO3, PlacO+ara, PlacO+rha. |
|
|
对染色体上的6个组合型启动子的表达强度进行了评价:用启动子控制下的gfp荧光强度作为指示,用酶标仪对荧光强度进行检测,将对照启动子PlacZ,在诱导剂下的表达强度值设为1,其他启动子的表达强度和PlacZ启动子的表达强度相比较得出。分析结果列于表 3。
| 启动子整合型菌株 Strains |
相对表达强度Relative strength | OD550 | 诱导剂 Inducer |
||
| 加诱导剂 With inducer |
不加诱导剂 Without inducer |
表达控制范围 Control range |
|||
| S-PlacZ | 1.00 | 0.80 | 1.4 | 0.1 mmol/L IPTG | |
| S-PtetO2 | 4.50 | 0.02 | 225.00 | 1.2 | 1 μg/mL Anhydrotetracycline |
| S-PtetO3 | 3.70 | 0.02 | 185.00 | 1.4 | 1 μg/mL Anhydrotetracycline |
| S-PlacO2 | 0.97 | 0.84 | 1.15 | 1.4 | 0.1 mmol/L IPTG |
| S-PlacO3 | 0.82 | 0.77 | 1.06 | 1.4 | 0.1 mmol/L IPTG |
| S-PlacO+ara | 6.00 | 0.60 | 10.00 | 1.4 | 0.1 mmol/L IPTG+0.2% Arabinose |
| S-PlacO+rha | 12.00 | 0.02 | 600.00 | 1.4 | 0.1 mmol/L IPTG+0.2% Rhamnose |
PlacO2和PlacO3组合型启动子,其表达在不加诱导剂的表达强度和加诱导剂IPTG的相对表达强度相差无几,为0.8-1.0左右,说明这对组合型启动子渗漏表达严重,即使在有诱导剂的情况下,也没有提高启动子的表达强度。因此,PlacO2和PlacO3这组由PL启动子和lacO调控元件组合而来的启动子,组合后并没有达到预期的目标,不可用于基因的严谨表达。
PtetO2和PtetO3启动子,其表达在不加诱导剂时的相对表达强度为0.02,加诱导剂脱水四环素后,PtetO2和PtetO3的相对表达强度分别为4.5和3.7,其表达控制范围分别为225倍和185倍。这组启动子在无诱导剂存在下表达强度低,在有诱导剂存在下表达较高。因此,PtetO2和PtetO3这组由PL和tetO调控元件组合而来的启动子,可用于基因的严谨型表达控制,为严谨型启动子。
PlacO+ara组合启动子在不加诱导剂时,相对表达强度为0.6,在加诱导剂时,相对表达强度为6。这个组合启动子虽然在加诱导剂时相对表达强度较高,但是不加诱导剂时也有渗漏表达较多。因此,由Plac和araC调控元件组合的PlacO+ara启动子,不能成为严谨型启动子。而PlacO+rha组合启动子,其在不加诱导剂时的相对表达强度为0.02,在有诱导剂存在时其相对表达强度为12,表达控制范围达到600倍。由Plac和rhaR调控元件组合的PlacO+rha组合启动子,在无诱导剂时基本上无渗漏表达,在有诱导剂时表达强度很高,达到了预期目标。因此,PlacO+rha是一个很好的严谨型启动子,可用于严谨型基因表达。
同时,用荧光显微镜对启动子的表达进行了观察,荧光显微镜观察的结果和用酶标仪测得的相对表达强度的结果具有相关性,启动子表达强度高的,绿色荧光亮度高。在此,只显示了3个严谨型启动子PtetO2、PtetO3和PlacO+rha的荧光观察情况(图 4)。从图 4中可以看出,相对于对照菌株S-PlacZ,菌株S-PtetO2和S-PtetO3的绿色荧光强度居中,S-PlacO+rha菌株的绿色荧光强度最高。

|
| 图 4 严谨型启动子荧光显微镜观察 Figure 4 Observation of promoter strength by fluorescence microscope |
|
|
本研究扩大了对基因功能的研究。首先,能严格控制基因的表达,如在生理条件下,某些如热激调控子σ32、伴侣蛋白DnaJ、在细胞分裂中的重要成分FtsZ等的基因低表达时对细胞影响的研究。第二,获得的严谨型启动子PtetO2和PlacO+rha元件,使单个或多个基因进行独立或组合调控,研究某个蛋白浓度的变化或协同表达后对表型的贡献。
启动子-调控序列的组合有以下原则:17-19 bp的调控序列能充分与阻遏蛋白结合,将这段序列置于间隔区-10和-35之间,能有效地阻止RNA聚合酶和启动子的结合,从而严格控制启动子的表达[18];另一个比较好的调控序列位置是-10区下游lacO调控区,这个位置虽不能很好抑制RNA聚合酶和启动子的结合,但组合起来利用也有一定的效果[19]。
PL启动子和tetO调控序列组合,将tetO序列置于PL启动子的位置Ⅲ Ⅳ或Ⅱ Ⅲ Ⅳ (图 2),得到组合型启动子PtetO2和PtetO3,这些启动子的活性受到TetR和脱水四环素的控制,启动子强度分别是野生型PlacZ的4.5倍和3.7倍。另外,脱水四环素是比四环素更为有效的诱导剂,它结合TetR的能力35倍于四环素,因此能在相当低的浓度下诱导启动子的表达[20],本研究中所用浓度在1 μg/mL就能充分诱导PtetO2和PtetO3的表达,且对细胞生长没有影响。PtetO3的强度略低于PtetO2,说明当tetO同时在位置Ⅱ Ⅲ Ⅳ时,对启动子的活性略有影响。
PlacO2和PlacO3启动子在有诱导剂和无诱导剂的情况下,表达强度和对照菌株相当。这可能是因为染色体上lacI基因为单拷贝,LacI的表达量不足,产生的阻遏蛋白不能充分地与PlacO2和PlacO3操纵子上的lacO序列结合,因此启动子泄露表达,从而这2个启动子都不能起到严谨控制的作用。若增加染色体上lacI基因的表达,提供足够的LacI阻遏蛋白,可能会实现PlacO2和PlacO3的严谨控制,相关研究正在探索。
为更好地应用利用阿拉伯糖和鼠李糖作为诱导剂,我们分别敲除了阿拉伯糖和鼠李糖代谢途径基因araBAD和rhaBAD,用于评价PlacO+ara和PlacO+rha启动子。这2个启动子来源于Plac启动子,除了在Ⅱ和Ⅲ位子上含lacO操纵子序列外,在-35区上游CRP/cAMP结合区用ara序列替换或rha结合序列替换。从诱导情况来看,这2个启动子都能较强地表达;从严谨型控制来看,Plac+rha的严谨型更好,在不添加诱导剂时几乎不表达,相对表达量为0.02,添加诱导剂后表达强度为12,表达差异范围为600。Lutz等[21]在不同拷贝数的质粒上对组合型启动子进行了研究,得到了具有严谨性控制的启动子PLtetO-1。遗憾的是,该启动子在不同拷贝数的质粒上表现出不一样的严谨型控制能力。另外,启动子用于质粒上进行基因的表达具有诸多弊端,深化应用于工业生产也有很大的局限性。不同于Lutz等的研究,本研究在染色体上对组合型启动子的严谨性进行了探索,得到了最佳严谨型启动子Plac+rha,本研究结果将为代谢工程和合成生物学中的精确调控基因表达奠定良好的应用基础。
| [1] |
Jin XM, Ma YB. Advances in Pichia pastoris promoters used for heterogeneous gene expression[J]. Journal of Microbiology, 2015, 35(3): 71-74. 金晓媚, 马雁冰. 用于异源基因表达的毕赤酵母启动子研究进展[J]. 微生物学杂志, 2015, 35(3): 71-74. |
| [2] |
Xu YQ, Ma CQ, Tao F, et al. Bacterial promoter recognition and application[J]. Chinese Journal of Biotechnology, 2010, 26(10): 1393-1403. 徐友强, 马翠卿, 陶飞, 等. 细菌启动子识别及应用研究进展[J]. 生物工程学报, 2010, 26(10): 1393-1403. |
| [3] |
Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(36): 12678-12683. DOI:10.1073/pnas.0504604102 |
| [4] |
Dykxhoorn DM, St Pierre R, Linn T. A set of compatible tac promoter expression vectors[J]. Gene, 1996, 177(1/2): 133-136. |
| [5] |
Arfman N, Worrell V, Ingram LO. Use of the tac Promoter and laciq for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis[J]. Journal of Bacteriology, 1992, 174(22): 7370-7378. DOI:10.1128/jb.174.22.7370-7378.1992 |
| [6] |
Kincade JM, deHaseth PL. Bacteriophage lambda promoters PL and PR sequence determinants of in vivo activity and of sensitivity to the DNA gyrase inhibitor, coumermycin[J]. Gene, 1991, 97(1): 7-12. DOI:10.1016/0378-1119(91)90003-T |
| [7] |
Jensen PR, Hammer K. Artificial promoters for metabolic optimization[J]. Biotechnology Bioengineering, 1998, 58(2/3): 191-195. |
| [8] |
Lu J, Tang JL, Liu Y, et al. Combinatorial modulation of galP and glk gene expression for improved alternative glucose utilization[J]. Applied Microbiology and Biotechnology, 2012, 93(6): 2455-2462. DOI:10.1007/s00253-011-3752-y |
| [9] |
Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas9 systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [10] |
Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J]. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [11] |
Zhao DD, Yuan SL, Xiong B, et al. Development of a fast and easy method for Escherichia coli genome editing with CRISPR/Cas9[J]. Microbial Cell Factories, 2016, 15(1): 205. DOI:10.1186/s12934-016-0605-5 |
| [12] |
Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J]. Metabolic Engineering, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006 |
| [13] |
Zhao J, Li QY, Sun T, et al. Engineering central metabolic modules of Escherichia coli for improving β-carotene production[J]. Metabolic Engineering, 2013, 17: 42-50. DOI:10.1016/j.ymben.2013.02.002 |
| [14] |
Srikhanta YN, Dowideit SJ, Edwards JL, et al. Phasevarions mediate random switching of gene expression in pathogenic Neisseria[J]. PLoS Pathogens, 2009, 5(4): e1000400. DOI:10.1371/journal.ppat.1000400 |
| [15] |
Sambrook J, Fritsch EF, and Maniatis T, Molecular Cloning: A Laboratory Manual[M]. C.S.H. Laboratory, Cold Spring Harbor, New York, USA, 3rd edition, 2001: 1595-1595
|
| [16] |
Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability[J]. PLoS One, 2008, 3(11): e3647. DOI:10.1371/journal.pone.0003647 |
| [17] |
Lanzer M, Bujard H. Promoters largely determine the efficiency of repressor action[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(23): 8973-8977. DOI:10.1073/pnas.85.23.8973 |
| [18] |
Knaus R, Bujard H. PL of coliphage lambda: an alternative solution for an efficient promoter[J]. The EMBO Journal, 1988, 7(9): 2919-2923. |
| [19] |
Kammerer W, Deuschle U, Gentz R, et al. Functional dissection of Escherichia coli promoters: information in the transcribed region is involved in late steps of the overall process[J]. The EMBO Journal, 1986, 5(11): 2995-3000. |
| [20] |
Takahashi M, Degenkolb J, Hillen W. Determination of the equilibrium association constant between tet repressor and tetracycline at limiting Mg2+ concentrations: A generally applicable method for effector-dependent high-affinity complexes[J]. Analytical Biochemistry, 1991, 199(2): 197-202. DOI:10.1016/0003-2697(91)90089-C |
| [21] |
Lutz R, Bujard H. Independent and tight regulation of transcriptional units in Escherichia coli via the LacR/O, the TetR/O and AraC/I1-I2 regulatory elements[J]. Nucleic Acids Research, 1997, 25(6): 1203-1210. DOI:10.1093/nar/25.6.1203 |
 2018, Vol. 45
2018, Vol. 45