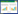扩展功能
文章信息
- 梁丽琴, 杜海燕, 段江燕, 谢丙炎
- LIANG Li-Qin, DU Hai-Yan, DUAN Jiang-Yan, XIE Bing-Yan
- 真菌小RNA的发生及其作用机制
- Research advance on the generation and mechanism of
small RNAs in fungi- 微生物学通报, 2018, 45(7): 1563-1573
- Microbiology China, 2018, 45(7): 1563-1573
- DOI: 10.13344/j.microbiol.china.170769
-
文章历史
- 收稿日期: 2017-09-27
- 接受日期: 2018-04-04
- 网络首发日期(www.cnki.net): 2018-04-16
2. 中国农业科学院蔬菜花卉研究所 农业部园艺作物生物学与种质创制重点实验室 北京 100081
small RNAs in fungi
2. Key Laboratory of Biology and Genetic Improvement of Horticultural Crops, Ministry of Agriculture, Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081, China
小RNA是一种非编码的单链RNA分子,大小一般在20−40 nt之间,主要通过染色质修饰、转录后水平降解mRNA或抑制mRNA翻译来调控基因表达,从而达到对真核生物的生长发育进行调控以抵御外来病毒入侵的目的。基于其重要性,小RNA的研究已成为当今生物学界的研究热点之一。哺乳动物中已有研究证明小RNA的发生与癌症密切相关,植物小RNA主要与适应逆境胁迫及抵御外来侵染物有关,病毒RNA主要与抵御宿主的防御相关。真菌在自然界中分布广泛,与人类的生产与生活密切相关,近年来,随着真菌基因组测序的不断增加及计算科学的发展,有关真菌小RNA的研究也取得了相应的进展。本文就目前真菌中已发现的小RNA种类、小RNA发生相关蛋白因子以及小RNA的具体作用机制进行全面综述,为进一步研究小RNA对真菌的生长发育提供参考。
1 真菌中已发现的小RNA种类目前已研究的真菌中主要发现了8种类型的小RNA,研究者根据其前体合成分子及合成机制进行命名,分别为siRNA (包括vsiRNA)、milRNA、qiRNA、disiRNA、esRNA、tRF、CPA-sRNA和rasiRNA。
在真核生物中,最常见的两种小RNA为siRNA[1]和miRNA[2],它们最早是在线虫中被发现,之后在大量的植物及动物种类中相继被发现。近年来,在脉胞菌(Neurospora crassa)、致病疫霉(Phytophthora infestans)、稻瘟菌(Magnaporthe oryzae)等少数真菌中也发现有siRNA的存在[3],却未发现miRNA的存在,但在脉胞菌中却发现有miRNA类似物的存在,由于其与动植物中的miRNA相似,因而得名milRNA (miRNA-like)[4]。此外,在脉胞菌中还发现有qiRNA (QDE-2-interacting small RNA)[5]及disiRNA (Dicer independent siRNA)[4]。研究发现DNA的损伤会诱导QDE-1 (DNA依赖的RNA聚合酶)以高度重复的rDNA位点为模板产生异常RNA (Aberrant RNAs,aRNAs),aRNAs又被Rdrps特异性地识别形成dsRNA,经Dicers剪切加工形成小的双链RNA。此外,DNA损伤还会诱导QDE-2 (Argonaute)的表达,qiRNA通过与QDE-2互作进行转录后基因沉默[5]。而disiRNA的产生则不需要RNAi组分的参与,与动植物中的endo-siRNA (Endogenous small interfering RNAs)和natsiRNA (Natural antisense short interfering RNA)相似[4]。
目前真菌小RNA的研究主要还是以脉胞菌为模式菌,除此之外,稻瘟菌及毛霉菌(Mucor circinelloides)中小RNA的研究也相对比较多。在稻瘟菌中,目前已发现的小RNA主要有tRFs (tRNA-derived RNA fragments)、esRNAs (Endogenous {Nicolas, 2010 #113}short RNAs)及CPA-sRNAs (5′-Methylguanosine-capped及-3′polyA small RNA)。tRFs是通过对稻瘟菌的高通量测序被发现,它起源于tRNA的5′或3′端,经反义密码环内部或与反义密码环接近的核糖核酸酶剪切形成了两部分,这两部分即为tRFs,另外的一些小片段可能是通过tRNA三叶草结构两臂的顶端暴露的核苷酸位点错误的核酸酶活性位点产生[6-8]。在人类HeLa细胞中Dicer负责把tRNAGln加工成20 nt的tRFs,生长活体研究证实tRFs在生长和发育中发挥重要的作用[9]。在稻瘟菌附着胞和孢子中的tRFs量相对于菌丝成比例地增加,说明tRFs在调节蛋白质合成过程中发挥重要作用,而菌丝组织的生长需要大量完整的tRNAs,这与附着胞和孢子中的情况正好相反[8]。esRNAs由Dicer剪切产生,普遍存在于动植物及真菌中,但是其类型并不完全相同,Nicolas等[10]研究证实esRNAs在稻瘟菌的生长表型发育中发挥重要的作用,在毛霉菌中由Dicer-2产生的esRNAs能定位于外显子上调节许多编码蛋白基因的表达。CPA-sRNAs与白色脉胞菌中的qiRNAs、milRNAs及disiRNAs没有相似性,其5′及3′端没有被修饰,推测其可能是由基因组或长的转录本经过一些非典型的RNA聚合酶如Pol Ⅳ (DNA-dependent RNA polymerase Ⅱ的同源物)或Pol Ⅲ加工所形成,必要时通过细胞质机制在5′末端加上甲基鸟苷,3′末端加上聚腺苷酸尾巴。大量的CPA-sRNAs能与rRNAs、tRNAs、snRNAs、转座元件及基因间隔区相匹配,主要与编码蛋白基因的起始处与终止处匹配,尤其是一些高表达的基因,诸如编码核糖体蛋白的基因,具体可能是通过与转录激活区结合形成RNA-DNA杂合区,从而对基因表达进行正向调节;CPA-sRNAs也可能会促进转录因子和RNA聚合酶结合,或作为RNA合成的引物;另外,CPA-sRNAs也有可能会组织进一步的转录并促进RNA聚合酶的释放;此外,也有可能CPA-sRNAs仅仅是反映了无效的转录起始位点[11]。
rasiRNA即重复相关小RNA (Repeat associated small interfering RNA),数量较miRNA少得多,其长度在不同物种中差异很大,与Dicer-dependent siRNA或miRNA相似或长度稍长些。rasiRNA最早被发现于果蝇(Drosophila melanogaster)和斑马鱼(Brachydanio rerio)的胚胎发育时期,之后发现在果蝇的生殖系细胞中也有,同piRNA (Piwi interacting RNA)一样,rasiRNA也与Piwi蛋白相结合,可能在生殖细胞中沉默逆转录转座子和重复元件来调节果蝇生殖系的发育,其沉默机制可能与piRNA的调节方式存在一定的关系[12-14]。在植物中Dicer-1加工生成21 nt的miRNAs和siRNAs,Dicer-3加工生成24 nt的rasiRNAs[15];在黑腹果蝇(D. melanogaster)中Dicer-1加工产生21 nt的miRNAs,Dicer-2加工产生21 nt的siRNAs,而24−27 nt之间不同大小的rasiRNAs以及它们的产生机制还有待研究[16-17];在单细胞的布氏锥虫(Trypanosoma brucei)[18]及裂殖酵母(Schizosaccharomyces pombe)[12]中也证实有rasiRNAs,rasiRNA是否广泛存在于其它真菌中以及由哪种Dicer同源物产生还有待进一步的研究。
近年来,随着小RNA测序技术的迅速发展,不同真菌的小RNA数据将逐步被解析。测序表明,寄生疫霉菌有大量的25−26 nt和少量21 nt小RNA,同时也累积一系列侵染阶段高度诱导表达的29 nt和33 nt tRNA来源的小RNA。25−26 nt小RNA和21 nt小RNA具有强烈的5′碱基偏好性,无链特异性地成簇分布在基因组上[19]。
以上真菌中8种类型小RNA的大小、前体结构、结构特点、发生相关蛋白及作用机制具体见表 1。真菌中除了这8种小RNA类型之外,诸如小鼠中存在的piRNA[20]及线虫中存在的微小非编码RNA (tncRNA)[21]是否也存在于真菌中?除此之外,真菌中是否还存在有其它小RNA类型,均有待于进一步研究。
| 种类 Species |
大小 Size (nt) |
前体结构 Precursor structure |
结构特点 Structural features |
发生蛋白 Generation proteins |
作用机制 Mechanism |
真菌种类 Fungal species |
| siRNA | 20−30 | Long dsRNA, mRNA, heterochromatin region, transposons, virus DNA | 5′-Monophosphates, 3′-Hydroxyl and overhangs two single-stranded nucleotides at their 3′ ends | Dicer, Argonaute, Rdrp | Posttranscriptional gene silencing (PTGS), inhibiting protein translation, infecting chromatin structure, inhibiting transposon jumping, resisting the virus infection[8] | Neurospora crassa[3], Magnaporthe oryzae[3], Phytophthora infestans[3] |
| esRNA | Unknown | Transposons and intergenic regions | Varied | Dicer, Rdrp | Eliminating mRNA transcripts of the protein coding genes from which they are produced | Magnaporthe oryzae[10], Mucor circinelloides[10] |
| rasiRNA | 22−24 | Antisense strand of long dsRNA | No hydroxyl at 2′ and 3′ end, and species diversity | Unknown | Participate in formatting and keeping the heterochromatin structure, controlling the transcription from repeat sequences | Schizosaccharomyces pombe[12] |
| disiRNA | Unknown | Overlapping sense and antisense transcripts | Have bias for 5′ U | Independent of RNAi components | Unknown | Neurospora crassa[4] |
| milRNA | 19−25 | Specific hairpin structure | Have U at the 5′ end | Dicer, QDE-2, QIP, RNaseIII domain-containing protein, MRPL3 | Inhibiting protein translation or degrading transcripts | Neurospora crassa[4] |
| qiRNA | 20−21 | Highly repetitive rDNA loc | Have bias for 5′ uridine | QDE-1, Dicer, Rdrp | Posttranscriptional gene silencing (PTGS) | Neurospora crassa[5] |
| tRFs | 22, 27−40 | 5′ end or 3′ end of tRNA | Have phosphate group at 5′ end and hydroxyl at 3′ end | RNaseZ, Dicer | Regulating the synthesis of protein | Mucor circinelloides[6-8], Aspergillus fumigatus[7] |
| CPA-sRNA | 16−218, average size of 41 | Genom or long transcript | Have methylation GMP at 5′ end and polyadenyl acid tail at 3′ end | Unknown | May be positive regulator of gene expression, or may be just an invalid transcription start site | Mucor circinelloides[11] |
小RNA的发生及其作用机制是个复杂的过程,不同物种及同一物种的不同小RNA的发生及作用机制均有差异。综合来看,真菌小RNA发生相关蛋白主要有Dicer、Argonaute及Rdrp。近年来,研究者们通过基因敲除的方式对真菌中的这3类蛋白进行了功能研究,取得了初步的研究进展。
2.1 DicerDicer是一种核酸内切酶,属于RNase Ⅲ家族,主要负责切割双链RNA成为各类21−25 nt的小RNA。目前发现真菌中仅含有1−2个Dicer (植物中含有4−7个Dicer),且一些真菌的2个Dicer之间没有功能重复,这说明真菌经过长期的进化,Dicer的功能已逐渐整合并分工明确。研究发现,粗糙脉胞菌的2个Dicer蛋白在RNA沉默途径中有功能重复[22];而在稻瘟病菌中Dicer-1及Dicer-2之间没有功能重复,其中Dicer-2负责siRNA产生[23],二者之间的功能分化可能是由于转录抑制及蛋白质特异化引起[24];毛霉菌的2个Dicer也没有功能重复,Dicer-1控制菌丝生长[25],Dicer-2负责小RNA的产生;在出芽酵母类中,芽殖酵母(Saccharomyces cerevisiae)及病原菌白色念珠菌(Candida albicans)中的Dicer与其它真菌及高等真核生物中的Dicer结构相差极大,典型的真菌Dicer包括有2个RNase Ⅲ结构域,而芽殖酵母及白色念珠菌的Dicer仅含有1个RNase Ⅲ结构域,但是RNase Ⅲ的活性位点却很保守[26],下游与dsRBD结构域(Double stranded RNA binding domain)相邻,此外下游C端还有1个dsRBD结构域[27]。白色念珠菌的Dicer除了负责产生siRNA之外,还与生长发育密切相关[28];栗疫病菌(Cryphonectria parasitica)中仅Dicer-2对抗病毒RNA沉默及vsRNA的产生是必需的,而Dicer-1则并不重要[29];在木霉菌(Trichoderma atroviride)中,Dicer-1调控生殖发育,Dicer-1和Dicer-2都调控营养生长,但是每个Dicer控制不同的生物学过程,如发育或代谢[30],具体的调控机制还有待进一步研究;研究证明,病原真菌灰葡萄孢菌(Botrytis cinerea)的2个Dicer被双敲除之后,突变体由于不再能产生小RNA致使其对拟南芥和番茄的致病力降低,另外拟南芥被敲除Argonaute-1之后对葡萄孢菌的敏感性降低,从而推测葡萄孢菌在侵染拟南芥时,分泌的小RNA通过与拟南芥体内小RNA竞争并劫持其RNAi机制,借助宿主的Argonaute-1做为载体选择性沉默宿主的免疫基因,此研究成果预示了植物与病原菌互作过程中,RNAi机制可能在更高水平上进行互作调控[31]。此项研究将小RNA与病原真菌的致病力相联系,推进了小RNA研究从分子调控理论到实践生物防治的迈进;分别将生防菌盾壳霉(Coniothyrium minitans)的Dicer-1和Dicer-2敲除后,突变株的生长速度、寄生致腐核盘菌(Sclerotinia sclerotiorum)菌核能力均显著低于野生型菌株,产孢量和产抗真菌物质抑制核盘菌菌丝生长的能力均显著高于野生型菌株,其中Dicer-2对影响外来病毒在宿主体内的积累起到积极作用[32];在禾谷镰刀菌(Fusarium graminearum)中,Dicer-2对分生孢子的萌发和芽管的产生具有一定的功能,并能影响毒素的合成[33];作者对尖孢镰刀菌(Fusarium oxysporum)的2个Dicer基因进行了单敲除及双敲除,结果表明尖孢镰刀菌的2个Dicer对菌丝的营养生长及发育繁殖均起到重要的调控作用[34];金龟子绿僵菌(Metarhizium anisopliae)的Dicer-1和Dicer-2单、双敲除突变体的菌落形态、菌丝形态、生长速率与野生型菌株相比均没有显著差异,突变菌株对NaCl、H2O2及SDS的敏感程度也无显著差异,说明Dicer缺失不会影响金龟子绿僵菌的抗逆性。但是,相比野生型,突变体的分生孢子产量和对大蜡螟的致病能力都有所降低,Dicer-2突变体产孢相关基因上调幅度变窄,microRNA表达水平下调较显著,这说明在金龟子绿僵菌中microRNA的生物起源可能依赖Dicer-2[35]。从已研究的真菌来看,真菌中的Dicer对小RNA的发生、生长发育及繁殖具有重要的调控作用,但是在不同的真菌中,Dicer行使的具体生物学功能及2个Dicer之间的功能是否重复则有差异,因而还需要对不同的真菌展开具体深入的研究,并根据研究结果结合已有的真菌分类进行深入分析,探索Dicer与真菌的协同进化。
小RNA的重要性决定了其发生蛋白Dicer在生物体的生长发育、免疫及逆境胁迫中的重要作用。目前的研究主要还是通过观察与分析基因敲除前后的表型、转录组与小RNA的变化来确定Dicer基因功能。随着研究的不断深入,Dicer基因的不同表达量是否会引起一些渐进表型?Dicer基因的表达量是如何被上游物质所调控的?这些都是将来要研究的内容。
2.2 ArgonauteArgonaute蛋白(Ago)最早在拟南芥中被鉴定,之后发现其与RNA沉默有关[36-37],Ago主要是通过参与形成RNA诱导的沉默复合体来介导RNA沉默[38-39],Hӧck等[40]和Hutvagner等[41]将AGO确定为小RNA在生物体细胞中发挥沉默机制的必要组分。
真菌中Ago蛋白的研究相较于Dicer蛋白要滞后。研究发现,裂殖酵母中Ago参与RNAi途径,介导转录和转录后沉默,在着丝粒区的转录沉默中发挥作用,而且可引起交配型位点的异染色质发生沉默。RNA诱导的沉默复合体(RITS,RNA-induced initiation of transcriptional gene silencing)包括Ago1、Chp1、Tas3和着丝粒siRNAs,为异染色质在着丝粒位点基因沉默所必需。裂殖酵母中存在一种Ago复合体(Argonaute siRNA分子伴侣,ARC),除了含有Ago1之外,还包含Arb1及Arb2两个蛋白,它们均参与组蛋白H3 Lys9甲基化,异染色质装配及siRNA的产生(图 1)。然而,RITS复合体中的siRNA大多都是单链,与ARC相关的siRNA大多都是双链,这说明Arb1及Arb2抑制了siRNA滞后链从Ago1上的释放。相反,纯化的Arb1在体外会抑制Ago1的剪切活性,而纯化的催化失活的Ago1仅包含双链siRNA。这说明双链siRNA到单链siRNA的转换为异染色质的形成所必需,而Arb在此转换过程中发挥了重要作用[42]。
在栗疫病菌中有4个Ago,但仅有Agl2 (Ago-like2)参与了病毒防卫反应,并激发RNA重组[43],这说明Argonaute基因(ago)在RNA沉默抗病毒防卫反应及促使病毒RNA重组中具有关键性的作用,这也为病毒编码的RNA沉默抑制子抑制真菌中RNA沉默组分的转录诱导机制提供了证据;在白色念珠菌中仅有1个ago基因,与其它真菌及高等真核生物中Argonaute的结构相差极大,研究发现敲除ago后白色念珠菌的生长发育基本不受影响[28];毛霉菌中有3个ago基因,其中只有ago-1参与了营养生长过程中的RNAi,并参与了转基因诱导的RNA沉默及积累来自于外显子的各类内源esRNAs过程。Ⅰ型和Ⅱ型ex-siRNAs结合到Ago-1上控制mRNA目标蛋白质编码基因的mRNA积累。Ⅲ型ex-siRNAs不会特异结合到Ago-1上,但是需要这种蛋白质作为它们的产物,这揭示了ex-siRNAs生物合成途径的复杂性。研究发现ago-1突变体的营养发育和营养压力诱导的自溶受到了影响,这表明ago-1参与了对环境信号的反应。此项研究结果说明仅有1个Ago蛋白通过不同的途径参与生产不同类型的esRNAs,也凸显了ex-siRNAs对真菌内源基因的调节作用,扩大了RNAi所调控生物功能的范围[44];将生防菌盾壳霉的ago敲除后,突变株的生长速度、寄生致腐核盘菌菌核能力均显著低于野生型菌株。产孢量、产抗真菌物质抑制核盘菌菌丝生长的能力均显著高于野生型菌株,孢子萌发速率显著高于野生型菌株[32];在禾谷镰刀菌中Ago-2参与芽管的延伸[33];在尖孢镰刀菌中,Ago-1与分生孢子形态发育相关,Ago-2与菌丝发育有关,Ago-1与Ago-2均抑制尖孢镰刀菌在生长早期产生分生孢子,促进菌丝的延伸生长[34];金龟子绿僵菌的Ago-1和Ago-2同Dicer一样,与菌落形态、菌丝形态、生长速率及抗逆性没有明显相关性[35];苹果树腐烂病菌有1个Ago蛋白,与粗糙脉孢菌和构巢曲霉的Ago蛋白蛋白具有高度的同源性,Ago-1和Ago-3对菌落、菌丝形态、菌落生长速率及对盐离子胁迫的响应没有明显影响,而Ago-3参与病菌对pH、H2O2胁迫的响应及致病力[45]。
综上可知,Ago蛋白作为RNA沉默的必要组分,在生物体的生长发育过程中发挥重要的调控作用,但就目前的研究结果来看,生物体缺少Ago蛋白后,除了尖孢镰刀菌外,还未观察到具体的生长表型变化,这可能是由于Ago数量较多,彼此之间存在着功能互补现象,从而导致敲除一个ago后另外一个ago互补所致,即使是某一个ago在起重要作用,也有可能敲除之后,其它ago会适应细胞生长需求而弥补其功能缺陷。真菌中Ago蛋白的研究还处于起步阶段,Ago蛋白的具体生物学功能及其在不同真菌中的功能分化还有待于进一步研究。
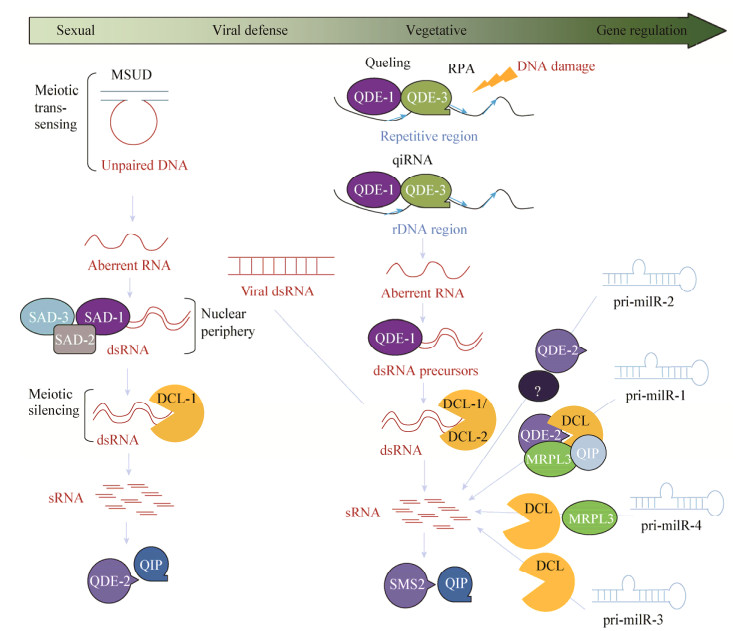
2.3 Rdrp蛋白(RNA dependent RNA polymerase)Rdrp是一种RNA聚合酶,它能依赖于RNA为模板合成互补链RNA分子,是RNAi的主要信号扩增分子。Rdrp主要分为两种,一种是在病毒中用于复制病毒基因组,一种是存在于真核生物中参与形成RNA沉默过程中涉及到的双链RNA。最早在番茄中分离到具有活性的Rdrp[46],之后研究者在脉胞菌中发现RNA沉默需要有QDE-1 (Rdrp-6)组分的参与,此QDE-1与番茄中的Rdrp基因序列相似,由1 402个氨基酸组成,分子量为158 004 Da,没有信号肽及跨膜区域,属于胞内蛋白,亲水性图说明QDE-1是可溶性蛋白[47],进一步研究证实QDE-1蛋白具有Rdrp活性[48],其晶体结构如图 2所示,这也是目前唯一的一个已观察到晶体结构的Rdrp基因[49]。在粟酒裂殖酵母中有一个Rdrp基因,它表达的Rdrp参与了裂殖酵母的着丝粒功能[13]及异染色质的形成和维护[50];毛霉菌中Rdrp-1负责正向转基因诱导的沉默产生dsRNA分子[10],但是对二级siRNAs的产生并无明显作用,Rdrp-2可能通过控制二级siRNA的产生负责有效dsRNA及正向转基因诱导的沉默[51];在木霉菌中,Rdrp-3调控生殖发育[30];在尖孢镰刀菌中,Rdrp-2及Rdrp-3与分生孢子的形态发生及繁殖方式相关[34]。
|
| 图 2 QDE-1的晶体结构[49] Figure 2 Crystal structure of QDE-1[49] Note: The picture was quoted from https://www.ncbi.nlm.nih.gov/Structure/pdb/2J7O |
|
|
对动植物及真菌中的161个rdrp基因进行进化分析,推测原始真核细胞仅有3种Rdrp,之后在进化过程中逐渐分化。Rdrpβ (动物和真菌Rdrp-3)及Rdrpα (植物Rdrp-3和真菌Rdrp-6)之间几乎没有同源性,说明它们在进化过程中可能发生了功能分化[52]。
Rdrp在真核生物的RNA沉默中发挥级联扩大效应的作用,此外与DNA复制之间也存在着一定的联系,今后还需要对多种真菌中的Rdrp功能进行广泛且深入的探讨,为系统解析Rdrp在真菌以及真核生物中发挥的作用奠定理论基础。
此外,真核生物中还有一些与RNA沉默相关的作用组分,如Exportin-5,它主要负责将pre-miRNAs从细胞核转运到细胞质中[53];QIP (QDE-2-interacting protein)是脉胞菌中发现的一种核酸外切酶,它主要负责特异性剪切与降解siRNA中的随从链[54];C3PO是果蝇中的一种核酸外切酶[55];HEN1与拟南芥中miRNA的甲基化及果蝇中的piRNA的甲基化相关[56-57];RITS复合物能够与RDRC (RNA-directed RNA polymerase complex)及ARC (Argonaute siRNA)互作去沉默异染色质RNA的转录[42, 57-58]。这些组分是否也存在于真菌中或有其它相应组分的存在还有待进一步发现。
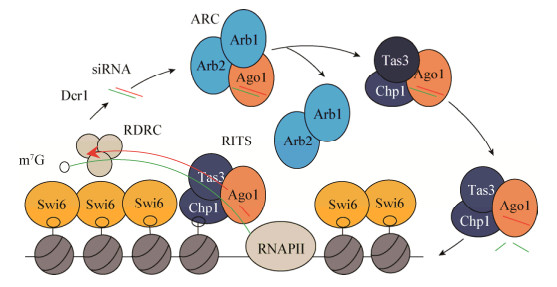
3 真菌小RNA的生成及其作用机制目前,真菌中脉胞菌的小RNA生成及其作用机制的研究相对成熟,已初步得到阐明的路径主要有4种(图 3):siRNA、qiRNA的生成及其介导的抑制现象;milRNA的生成及其介导的基因表达调控机制;减数分裂沉默抑制;外源双链RNA诱发的真菌RNAi防卫机制[59]。
在营养生长阶段,重复的转座子及核糖体DNA位点能够通过QDE-1和QDE-3产生异常的aRNAs,单链的aRNAs被QDE-1转换成dsRNA前体,Dicer在ATP作用下将dsRNA解旋,并切割成5′端为磷酸基、3′端为羟基,还有2 nt突出的双链siRNA。siRNA在胞内RNA解旋酶的作用下解链成正义链及反义链,正义链与mRNA发生交换,反义链与RISC (含有QDE-2/QIP)结合并激活RISC,通过碱基配对定位于同源mRNA转录本上,然后由核酸内切酶在距离siRNA 3′端12 bp的位置切割mRNA,随后mRNA迅速降解,并诱发宿主细胞对这些mRNA进行降解。siRNA不仅能引导RISC切割同源单链mRNA,而且可作为引物与靶RNA结合并在Rdrp作用下通过类似PCR扩增合成更多新的dsRNA,新合成的dsRNA再由Dicer切割产生大量的次级siRNA,从而使RNAi的作用进一步放大,最终将靶mRNA完全降解[59]。
针对Rdrp如何识别aRNAs的问题,Nolan等[60]以脉胞菌为研究材料,发现Rdrp (QDE-1)能与DNA复制机制中的元件复制蛋白A (RPA)互作,QDE-1与RPA都是核内蛋白,QDE-1能被特异结合到重复转基因座位点,并推测QDE的此定位点能以转基因座初始转录本为模板原位产生dsRNA。这2个蛋白之间有相关性的支持证据是siRNAs (沉默的标志)的积累依赖于连续的DNA合成。QDE-1与RPA之间的互作对于进一步研究与理解Rdrp的特异性很重要,并第一次将PTGs组分与DNA复制机制相联系。
真菌中的milRNA同动植物的miRNA一样,主要是对基因表达进行调控。脉胞菌中milRNA由4种不同的途径合成,每种途径所需要的蛋白元件不同,主要有Dicers、QDE-2、QIP、含有RNase Ⅲ结构域的蛋白及MRPL3。这4种milRNA中,只有milR-3的形成机制与植物相似,仅需要Dicer就可以把pre-milRNA形成milRNA。milR-4的形成仅部分依赖于Dicers,另外还需要有其它的核酸酶。milR-1在脉胞菌基因组中的产生位点最多,需要有Dicers、QDE-2以及有催化活性的QIP,首先,Dicer将初级pri-milR-1加工成pre-milR-1,然后,QDE-2结合到pre-milR-1上,在QIP的作用下将pre-milR-1加工成成熟的milR-1,QDE-2在这里起到了一个加工平台的作用。milR-2的形成完全不依赖于Dicers,而是需要有催化活性的QDE-2[59]。milR-2的生物合成途径是一个不依赖Dicer而依赖Ago合成sRNA的典型机制,在斑马鱼和小鼠中miR-451也由同样的机制产生[61-62],说明此机制是一个保守的sRNA生物合成途径,更说明了真核生物miRNAs形成的进化多样性。
减数分裂沉默(Meiotic silencing by unpaired DNA,MSUD)现象最初是在研究脉胞菌中Ascospore maturation 1 gene (asm-1)时发现[63-65],MSUD发生在减数分裂的前期1阶段,由于基因缺失、复制、转座等引起同源基因未配对从而发生MSUD,MSUD相关蛋白主要有6种,分别是:SAD-1 (Suppressor of ascus dominance 1),一种Rdrp;DCL-1,一种Dicer-like RNase Ⅲ酶;SMS-2 (Suppressor of meiotic silencing 2),一种Argonaute家族蛋白同源物;SAD-2,能够使SAD-1正确定位于细胞核周围;QIP,一种核酸外切酶,它们存在于细胞核周围;SAD-3,推断是一种解螺旋酶,MSUD及产生有性孢子所必需,SAD-3的直系同源物包括粟酒裂殖酵母中的Hrr1,RNAi诱导的异染色质形成所必需的一种解螺旋酶。SAD-3与Hrr1都能与RNA介导的RNA聚合酶及Argonaute互作,这说明在这两种真菌中沉默复合体形成的特定方面是保守的[66]。MSUD负责将未配对片段形成单链异常RNA,这个RNA被转运到细胞核周围后,作为SAD-1介导的双链RNA合成模板形成双链RNA,然后DCL-1剪切dsRNA为siRNA,siRNA指导SMS-2去识别并剪切与自身完全互补的mRNA,在此过程中,SAD-2及SAD-3可与SAD-1互作,并帮助转运到细胞核周围,而QIP作为一种外切酶移去siRNA双链中的滞后链[67-72]。
稻瘟菌中RNA沉默机制主要有Dicer依赖的RNAi途径以及Dicer非依赖的RNAi途径,其作用不仅局限于防御外来核酸片段的入侵,并通过产生内源小RNA调控基因表达,从而参与调控特殊的生理及发育过程来应对外界环境信号[73]。
除了稻瘟病菌外,研究发现在脉胞菌、粟疫病菌及构巢曲霉(Aspergillus nidulans)中存在着与植物类似的抗病毒机制,当外源病毒RNA侵入真菌体内时,会诱导真菌通过RNA沉默激发免疫机制[29, 44]。近年来,研究发现在植物与病原真菌互作过程中,小RNA作为密使发挥着重要作用。一方面,宿主诱导的基因沉默机制(Host induced gene silencing,HIGS)会影响丝状真菌的基因表达;另一方面,真菌诱导的基因沉默机制Filamentous organism-induced gene silencing,FIGS)会影响宿主的防卫基因表达[74]。
4 展望小RNA在真核生物中发挥着重要作用,除了目前发现的8种小RNA之外,是否还存在其它小RNA?如piRNA、tncRNA等还有待进一步研究。另外,目前仅得到了小RNA对真菌的生长发育具有调控作用的初步结果,而有关具体的调控机制还有待深入研究,其中涉及到小RNA的具体作用机制,与小RNA作用相关的各种蛋白因子的表达、结构与功能及其进化机制[75],小RNA与信号转导之间的联系,小RNA与病原菌致病性的相关性,这些都还有待于进一步深入的研究。随着计算科学、生物信息学以及生物技术的全面快速发展,生物学研究已进入了全基因组时代,相信在不久的将来,小RNA对真菌的调控机制将逐步被阐明,而随着不同真菌中小RNA沉默机制的阐明,以RNA沉默机制类型为分类依据对植物病原真菌进行遗传学分类也不无可能,这将为人类从更高水平了解不同真菌之间的进化关系,甚至真菌与动物和植物之间的进化关系提供理论依据。
| [1] |
Hannon GJ. RNA interference[J]. Nature, 2002, 418(6894): 244-251. DOI:10.1038/418244a |
| [2] |
Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843-854. DOI:10.1016/0092-8674(93)90529-Y |
| [3] |
Nakayashiki H. RNA silencing in fungi: Mechanisms and applications[J]. FEBS Letters, 2005, 579(26): 5950-5957. DOI:10.1016/j.febslet.2005.08.016 |
| [4] |
Lee HC, Li LD, Gu WF, et al. Diverse pathways generate microRNA-like RNAs and Dicer-independent small interfering RNAs in fungi[J]. Molecular Cell, 2010, 38(6): 803-814. DOI:10.1016/j.molcel.2010.04.005 |
| [5] |
Lee HC, Chang SS, Choudhary S, et al. qiRNA is a new type of small interfering RNA induced by DNA damage[J]. Nature, 2009, 459(7244): 274-277. DOI:10.1038/nature08041 |
| [6] |
Kawaji H, Nakamura M, Takahashi Y, et al. Hidden layers of human small RNAs[J]. BMC Genomics, 2008, 9(1): 157. DOI:10.1186/1471-2164-9-157 |
| [7] |
Jӧchl C, Rederstorff M, Hertel J, et al. Small ncRNA transcriptome analysis from Aspergillus fumigatus suggests a novel mechanism for regulation of protein synthesis[J]. Nucleic Acids Research, 2008, 36(8): 2677-2689. DOI:10.1093/nar/gkn123 |
| [8] |
Cole C, Sobala A, Lu C, et al. Filtering of deep sequencing data reveals the existence of abundant dicer-dependent small RNAs derived from tRNAs[J]. RNA, 2009, 15(12): 2147-2160. DOI:10.1261/rna.1738409 |
| [9] |
Nunes CC, Gowda M, Sailsbery J, et al. Diverse and tissue-enriched small RNAs in the plant pathogenic fungus, Magnaporthe oryzae[J]. BMC Genomics, 2011, 12: 288. DOI:10.1186/1471-2164-12-288 |
| [10] |
Nicolas FE, Moxon S, de Haro JP, et al. Endogenous short RNAs generated by Dicer 2 and RNA-dependent RNA polymerase 1 regulate mRNAs in the basal fungus Mucor circinelloides[J]. Nucleic Acids Research, 2010, 38(16): 5535-5541. DOI:10.1093/nar/gkq301 |
| [11] |
Gowda M, Nunes CC, Sailsbery J, et al. Genome-wide characterization of methylguanosine-capped and polyadenylated small RNAs in the rice blast fungus Magnaporthe oryzae[J]. Nucleic Acids Research, 2010, 38(21): 7558-7569. DOI:10.1093/nar/gkq583 |
| [12] |
Reinhart BJ, Bartel DP. Small RNAs correspond to centromere heterochromatic repeats[J]. Science, 2002, 297(5588): 1831. DOI:10.1126/science.1077183 |
| [13] |
Volpe TA, Kidner C, Hall IM, et al. Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi[J]. Science, 2002, 297(5588): 1833-1837. DOI:10.1126/science.1074973 |
| [14] |
Aravin AA, Lagos-Quintana M, Yalcinet A, et al. The small RNA profile during Drosophila melanogaster development[J]. Developmental Cell, 2003, 5(2): 337-350. DOI:10.1016/S1534-5807(03)00228-4 |
| [15] |
Xie ZX, Johansen LK, Gustafson AM, et al. Genetic and functional diversification of small RNA pathways in plants[J]. PLoS Biology, 2004, 2(5): E104. DOI:10.1371/journal.pbio.0020104 |
| [16] |
Lee YS, Nakahara K, Pham JW, et al. Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways[J]. Cell, 2004, 117(1): 69-81. DOI:10.1016/S0092-8674(04)00261-2 |
| [17] |
Pham JW, Pellino JL, Lee YS, et al. A Dicer-2-dependent 80s complex cleaves targeted mRNAs during RNAi in Drosophila[J]. Cell, 2004, 117(1): 83-94. DOI:10.1016/S0092-8674(04)00258-2 |
| [18] |
Djikeng A, Shi H, Tschudi C, et al. RNA interference in Trypanosoma brucei: cloning of small interfering RNAs provides evidence for retroposon-derived 24-26-nucleotide RNAs[J]. RNA, 2001, 7(11): 1522-1530. |
| [19] |
Jia JB. Diversity of small RNAs and their potential roles in the regulation of gene expression in Phytophthora parasitica[D]. Yangling: Doctoral dissertation of Northwest Agriculture & Forestry University, 2017 (in Chinese) 贾津布. 寄生疫霉菌小RNA多样性及其参与基因表达调控的探索研究[D]. 杨凌: 西北农林科技大学博士学位论文, 2017 |
| [20] |
Lau NC, Seto AG, Kim J, et al. Characterization of the piRNA complex from rat testes[J]. Science, 2006, 313(5785): 363-367. DOI:10.1126/science.1130164 |
| [21] |
Ambros V, Lee RC, Lavanway A, et al. MicroRNAs and other tiny endogenous RNAs in C. elegans[J]. Current Biology, 2003, 13(10): 807-818. DOI:10.1016/S0960-9822(03)00287-2 |
| [22] |
Catalanotto C, Pallotta M, ReFalo P, et al. Redundancy of the two Dicer genes in transgene-induced posttranscriptional gene silencing in Neurospora crassa[J]. Molecular and Cellular Biology, 2004, 24(6): 2536-2545. DOI:10.1128/MCB.24.6.2536-2545.2004 |
| [23] |
Kadotani N, Nakayashiki H, Tosa Y, et al. One of the two Dicer-like proteins in the filamentous fungi Magnaporthe oryzae genome is responsible for hairpin RNA-triggered RNA silencing and related small interfering RNA accumulation[J]. The Journal of Biological Chemistry, 2004, 279(43): 44467-44474. DOI:10.1074/jbc.M408259200 |
| [24] |
Kadotani N, Murata T, Quoc NB, et al. Transcriptional control and protein specialization have roles in the functional diversification of two dicer-like proteins in Magnaporthe oryzae[J]. Genetics, 2008, 180(2): 1245-1249. DOI:10.1534/genetics.108.093922 |
| [25] |
Nicolás FE, de Haro JP, Torres-Martínez S, et al. Mutants defective in a Mucor circinelloides dicer-like gene are not compromised in siRNA silencing but display developmental defects[J]. Fungal Genetics and Biology, 2007, 44(6): 504-516. DOI:10.1016/j.fgb.2006.09.003 |
| [26] |
Weinberg DE, Nakanishi K, Patel DJ, et al. The inside-out mechanism of Dicers from budding yeasts[J]. Cell, 2011, 146(2): 262-276. DOI:10.1016/j.cell.2011.06.021 |
| [27] |
Drinnenberg IA, Weinberg DE, Xie KT, et al. RNAi in budding yeast[J]. Science, 2009, 326(5952): 544-550. DOI:10.1126/science.1176945 |
| [28] |
Bernstein DA, Vyas VK, Weinberg DE, et al. Candida albicans Dicer (CaDcr1) is required for efficient ribosomal and spliceosomal RNA maturation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(2): 523-528. DOI:10.1073/pnas.1118859109 |
| [29] |
Segers GC, Zhang XM, Deng FY, et al. Evidence that RNA silencing functions as an antiviral defense mechanism in fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(31): 12902-12906. DOI:10.1073/pnas.0702500104 |
| [30] |
Carreras-Villase or N, Esquivel-Naranjo EU, Villalobos-Escobedo JM, et al. The RNAi machinery regulates growth and development in the filamentous fungus Trichoderma atroviride[J]. Molecular Microbiology, 2013, 89(1): 96-112. DOI:10.1111/mmi.2013.89.issue-1 |
| [31] |
Weiberg A, Wang M, Lin FM, et al. Fungal small RNAs suppress plant immunity by hijacking host RNA interference pathways[J]. Science, 2013, 342(6154): 118-123. |
| [32] |
Zeng LS. Functional analysis of genes of small RNA pathways in Coniothyrium minitans[D]. Wuhan: Master's Thesis of Huazhong Agriculture University, 2012 (in Chinese) 曾丽斯. 盾壳霉小RNA相关通路基因的功能研究[D]. 武汉: 华中农业大学硕士学位论文, 2012 |
| [33] |
Wang Y. Functional analysis of genes associated with small RNA pathways in Fusarium graminearum[D]. Wuhan: Master's Thesis of Huazhong Agriculture University, 2013 (in Chinese) 王颖. 禾谷镰刀菌小RNA通路相关基因的功能研究[D]. 武汉: 华中农业大学硕士学位论文, 2013 |
| [34] |
Liang LQ. Characterization of small RNAs biogenesis related genes Dicer, Argonaute and Rdrp in Fusarium oxysporum[D]. Beijing: Doctoral dissertation of China Agriculture University, 2014 (in Chinese) 梁丽琴. 尖孢镰刀菌小RNA发生相关Dicer, Argonaute和Rdrp基因功能研究[D]. 北京: 中国农业大学博士学位论文, 2014 |
| [35] |
Meng HM, Wang ZX, Wang YL, et al. Characterization of small RNAs biogenesis related genes Dicer, Argonaute and Rdrp in Metarhizium anisopliae[A]//2015 Academic Conference Paper Abstract Book of Chinese Institute Of Fungi (CBC)[C]. Beijing, 2015 (in Chinese) 孟慧敏, 汪章勋, 王玉龙, 等. 金龟子绿僵菌小RNA通路相关Dicer, Argonaute基因的功能研究[A]//中国菌物学会2015年学术年会论文摘要集[C]. 北京, 2015 |
| [36] |
Grishok A, Pasquinelli AE, Conte D, et al. Genes and mechanisms related to RNA interference regulate expression of the small temporal RNAs that control C. elegans developmental timing[J]. Cell, 2001, 106(1): 23-24. DOI:10.1016/S0092-8674(01)00431-7 |
| [37] |
Tabara H, Sarkissian M, Kelly WG, et al. The rde-1 gene, RNA interference, and transposon silencing in C. elegans[J]. Cell, 1999, 99(2): 123-132. DOI:10.1016/S0092-8674(00)81644-X |
| [38] |
Hammond SM, Bernstein E, Beach D, et al. An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells[J]. Nature, 2000, 404(6775): 293-296. DOI:10.1038/35005107 |
| [39] |
Hammond SM, Boettcher S, Caudy AA, et al. Argonaute2, a link between genetic and Biochemical analyses of RNAi[J]. Science, 2001, 293(5532): 1146-1150. DOI:10.1126/science.1064023 |
| [40] |
Hӧck J, Meister G. The Argonaute protein family[J]. Genome Biology, 2008, 9(2): 210. DOI:10.1186/gb-2008-9-2-210 |
| [41] |
Hutvagner G, Simard MJ. Argonaute proteins: key players in RNA silencing[J]. Nature Reviews Molecular Cell Biology, 2008, 9(1): 22-32. DOI:10.1038/nrm2321 |
| [42] |
Buker SM, Iida T, Bühler M, et al. Two different Argonaute complexes are required for siRNA generation and heterochromatin assembly in fission yeast[J]. Nature Structural & Molecular Biology, 2007, 14(3): 200-207. |
| [43] |
Sun Q, Choi GH, Nuss DL. A single Argonaute gene is required for induction of RNA silencing antiviral defense and promotes viral RNA recombination[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(42): 17927-17932. DOI:10.1073/pnas.0907552106 |
| [44] |
Cervantes M, Vila A, Nicolás FE, et al. A single Argonaute gene participates in exogenous and endogenous RNAi and controls cellular functions in the basal fungus Mucor circinelloides[J]. PLoS One, 2013, 8(7): e69283. DOI:10.1371/journal.pone.0069283 |
| [45] |
Feng H, Liu YY, Dong RQ, et al. Preliminary function analysis of Argonaute genes from Valsa mali[J]. Journal of Agricultural University of Hebei, 2016, 39(4): 57-62. 冯浩, 刘洋洋, 董茹青, 等. 苹果树腐烂病菌Argonaute基因鉴定及功能初步分析[J]. 河北农业大学学报, 2016, 39(4): 57-62. |
| [46] |
Schiebel W, Pélissier T, Riedel L, et al. Isolation of an RNA-directed RNA polymerase-specific cDNA clone from tomato[J]. Plant Cell, 1998, 10(12): 2087-2101. |
| [47] |
Cogoni C, Macino G. Gene silencing in Neurospora crassa requires a protein homologous to RNA-dependent RNA polymerase[J]. Nature, 1999, 399(6732): 166-169. DOI:10.1038/20215 |
| [48] |
Tijsterman M, Ketting RF, Plasterk RHA. The genetics of RNA silencing[J]. Annual Review of Genetics, 2002, 36(1): 489-519. DOI:10.1146/annurev.genet.36.043002.091619 |
| [49] |
Salgado PS, Koivunen MRL, Makeyev EV, et al. The structure of an RNAi polymerase links RNA silencing and transcription[J]. PLoS Biology, 2006, 4(12): e434. DOI:10.1371/journal.pbio.0040434 |
| [50] |
Sugiyama T, Cam H, Verdel A, et al. RNA-dependent RNA polymerase is an essential component of a self-enforcing loop coupling heterochromatin assembly to siRNA production[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(1): 152-157. DOI:10.1073/pnas.0407641102 |
| [51] |
Calo S, Nicolás FE, Vila A, et al. Two distinct RNA-dependent RNA polymerases are required for initiation and amplification of RNA silencing in the basal fungus Mucor circinelloides[J]. Molecular Microbiology, 2012, 83(2): 379-394. DOI:10.1111/mmi.2012.83.issue-2 |
| [52] |
Zong J, Yao X, Yin JY, et al. Evolution of the RNA-dependent RNA polymerase (RdRP) genes: duplications and possible losses before and after the divergence of major eukaryotic groups[J]. Gene, 2009, 447(1): 29-39. DOI:10.1016/j.gene.2009.07.004 |
| [53] |
Yi R, Qin Y, Macara IG, et al. Exportin-5 mediates the nuclear export of pre-microRNAs and short hairpin RNAs[J]. Genes & Development, 2003, 17(24): 3011-3016. |
| [54] |
Maiti M, Lee HC, Liu Y. QIP, a putative exonuclease, interacts with the Neurospora Argonaute protein and facilitates conversion of duplex siRNA into single strands[J]. Genes & Development, 2007, 21(5): 590-600. |
| [55] |
Liu Y, Ye XC, Jiang F, et al. C3PO, an endoribonuclease that promotes RNAi by facilitating RISC activation[J]. Science, 2009, 325(5941): 750-753. DOI:10.1126/science.1176325 |
| [56] |
Yu B, Yang ZY, Li JJ, et al. Methylation as a crucial step in plant microRNA biogenesis[J]. Science, 2005, 307(5711): 932-935. DOI:10.1126/science.1107130 |
| [57] |
Saito K, Sakaguchi Y, Suzuki T, et al. Pimet, the Drosophila homolog of HEN1, mediates 2′-O-methylation of Piwi-interacting RNAs at their 3′ ends[J]. Genes & Development, 2007, 21(13): 1603-1608. |
| [58] |
Motamedi MR, Verdel A, Colmenares SU, et al. Two RNAi complexes, RITS and RDRC, physically interact and localize to noncoding centromeric RNAs[J]. Cell, 2004, 119(6): 789-802. DOI:10.1016/j.cell.2004.11.034 |
| [59] |
Chang SS, Zhang ZY, Liu Y. RNA interference pathways in fungi: Mechanisms and functions[J]. Annual Review of Microbiology, 2012, 66(1): 305-323. DOI:10.1146/annurev-micro-092611-150138 |
| [60] |
Nolan T, Cecere G, Mancone C, et al. The RNA-dependent RNA polymerase essential for post-transcriptional gene silencing in Neurospora crassa interacts with replication protein A[J]. Nucleic Acids Research, 2008, 36(2): 532-538. DOI:10.1093/nar/gkm1071 |
| [61] |
Cheloufi S, Dos Santos CO, Chong MMW, et al. A Dicer-independent miRNA biogenesis pathway that requires Ago catalysis[J]. Nature, 2010, 465(7298): 584-589. DOI:10.1038/nature09092 |
| [62] |
Cifuentes D, Xue HL, Taylor DW, et al. A novel miRNA processing pathway independent of Dicer requires Argonaute2 catalytic activity[J]. Science, 2010, 328(5986): 1694-1698. DOI:10.1126/science.1190809 |
| [63] |
Aramayo R, Metzenberg RL. Meiotic transvection in fungi[J]. Cell, 1996, 86(1): 103-113. |
| [64] |
Shiu PKT, Metzenberg RL. Meiotic silencing by unpaired DNA: properties, regulation and suppression[J]. Genetics, 2002, 161(14): 1483-1495. |
| [65] |
Shiu PKT, Raju NB, Zickler D, et al. Meiotic silencing by unpaired DNA[J]. Cell, 2001, 107(7): 905-916. DOI:10.1016/S0092-8674(01)00609-2 |
| [66] |
Hammond TM, Xiao H, Boone EC, et al. SAD-3, a putative helicase required for meiotic silencing by unpaired DNA, interacts with other components of the silencing machinery[J]. G3: Genes, Genomes, Genetics, 2011, 1(5): 369-376. |
| [67] |
Alexander WG, Raju NB, Xiao H, et al. DCL-1 colocalizes with other components of the MSUD machinery and is required for silencing[J]. Fungal Genetics and Biology, 2008, 45(5): 719-727. DOI:10.1016/j.fgb.2007.10.006 |
| [68] |
Shiu PKT, Zickler TD, Raju NB, et al. SAD-2 is required for meiotic silencing by unpaired DNA and perinuclear localization of SAD-1 RNA-directed RNA polymerase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(7): 2243-2248. DOI:10.1073/pnas.0508896103 |
| [69] |
Lee DW, Pratt RJ, McLaughlin M, et al. An argonaute-like protein is required for meiotic silencing[J]. Genetics, 2003, 164(2): 821-828. |
| [70] |
Xiao H, Alexander WG, Hammond TM, et al. QIP, a protein that converts duplex siRNA into single strands, is required for meiotic silencing by unpaired DNA[J]. Genetics, 2010, 186(1): 119-126. DOI:10.1534/genetics.110.118273 |
| [71] |
Lee DW, Millimaki R, Aramayo R. QIP, a component of the vegetative RNA silencing pathway, is essential for meiosis and suppresses meiotic silencing in Neurospora crassa[J]. Genetics, 2010, 186(1): 127-133. DOI:10.1534/genetics.110.118422 |
| [72] |
Kelly WG, Aramayo R. Meiotic silencing and the epigenetics of sex[J]. Chromosome Research, 2007, 15(5): 633-651. DOI:10.1007/s10577-007-1143-0 |
| [73] |
Torres-Martínez S, Ruiz-Vázquez RM. RNAi pathways in Mucor: A tale of proteins, small RNAs and functional diversity[J]. Fungal Genetics and Biology, 2016, 90: 44-52. DOI:10.1016/j.fgb.2015.11.006 |
| [74] |
Weiberg A, Jin HL. Small RNAs-the secret agents in the plant-pathogen interactions[J]. Current Opinion in Plant Biology, 2015, 26: 87-94. DOI:10.1016/j.pbi.2015.05.033 |
| [75] |
Chand SK, Nanda S, Mishra R, et al. Multiple garlic (Allium sativum L.) microRNAs regulate the immunity against the basal rot fungus Fusarium oxysporum f. sp. Cepae[J]. Plant Science, 2017, 257: 9-21. DOI:10.1016/j.plantsci.2017.01.007 |
 2018, Vol. 45
2018, Vol. 45