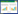扩展功能
文章信息
- 赖爱兰, 黄运红, 龙中儿
- LAI Ai-Lan, HUANG Yun-Hong, LONG Zhong-Er
- 翻译延伸因子EF-P的结构和功能及其研究进展
- Progress in studies on structure and function of elongation factor P
- 微生物学通报, 2018, 45(7): 1555-1562
- Microbiology China, 2018, 45(7): 1555-1562
- DOI: 10.13344/j.microbiol.china.170803
-
文章历史
- 收稿日期: 2017-10-09
- 接受日期: 2018-01-24
- 网络首发日期(www.cnki.net): 2018-03-22
EF-P (Elongation factor P)是一种蛋白质翻译延伸因子,由Glick等于1975年从大肠杆菌中首次分离得到,与真核生物的eIF5A同源[1]。EF-P可作用于核糖体的肽基转移中心,刺激翻译延伸阶段肽键的生成,促使fMet-tRNAfMet和aa-tRNA结构类似物嘌呤霉素(Puromycin,PMN)之间形成肽键。随着研究的深入,已证实EF-P广泛存在于各种细菌体内,具有蛋白质翻译延伸功能,即当蛋白质多肽链序列中含有聚脯氨酸(Polyproline)残基及各种Diproly基序时,会导致翻译的延宕。EF-P结合于翻译延宕的核糖体上肽基-tRNA位点(P位点)和tRNA退出位点(E位点)之间,刺激肽基转移酶的活性,从而使翻译继续进行。本文综述了细菌EF-P的结构、功能及二者之间相互关系的研究进展。
1 蛋白质翻译的延宕核糖体是合成蛋白质的场所。在细菌蛋白质合成的延伸阶段,核糖体沿着mRNA移动,一次移动一个密码子,平均速度约每秒10−20个密码子。蛋白质延伸的每个周期包括3个连续的步骤:(1)氨酰tRNA与暴露在核糖体A位上的配对密码子结合;(2) A位上的氨酰tRNA和P位上肽基tRNA形成肽键;(3) 2个转运RNA的易位,从A位到P位以及由P位到E位。上述过程需要有多种翻译延伸因子(Elongation factor,EF)辅助,其中EF-Tu加速氨酰tRNA与核糖体的连接,EF-Ts是EF-Tu的鸟苷酸交换因子,EF-G促进转运RNA的易位。EF-Tu确保不同的aa-tRNAs以同样的速率到达核糖体,这一性能可以保证翻译过程不受mRNA密码子的影响而以较快速度进行。尽管有EF-Tu的均衡效应,翻译依然是一个不连续的过程,存在翻译延宕的现象。原因有多种,如核糖体、新生肽链以及参与的辅因子或者化合物都影响核糖体上的蛋白质翻译延伸功能,其中新生肽链上存在的Pro残基是一个重要原因。Pro和Gly可导致肽基转移翻译延宕,这2种氨基酸比其他任何氨基酸形成蛋白质的速度都要慢,甚至导致翻译中断。
2 EF-P的结构EF-P是由ef-p基因编码的一个21 kD的蛋白质,大肠杆菌ef-p基因位于染色体上94.3 min的位置[2-3]。嗜热链球菌的EF-P由3个β筒形结构域组成,类似一个tRNA分子的形状和大小[4-6](图 1A),C端区域Ⅲ与区域Ⅱ有相同的折叠可能是因为部分基因重复造成的。其真核生物同源因子eIF5A也同样有一个类似tRNA的形状(图 1D)。eIF5A有64%的序列与EF-P的域Ⅰ和域Ⅱ组成部分相似,它没有域Ⅲ的原因并不清楚。曾有研究者提出eIF5A二聚形成了一个类似EF-P单体分子。
|
| 图 1 EF-P、tRNA及eIF5A的结构比较[4, 7] Figure 1 Structural comparison of EF-P, tRNA and eIF5A[4, 7] 注:A:嗜热链球菌EF-P结构(PDB 1UEB);B:酿酒酵母tRNAphe结构(PDB 1EVV);C:大肠杆菌(PDB 3A5Z) EF-P的结构域Ⅰ、Ⅱ和Ⅲ以及Lys34残基;D:酿酒酵母(PDB 3ER0) eIF5A的结构域Ⅰ和Ⅱ及其保守的Lys51残基. Note: A: EF-P from T. thermophilus (PDB 1UEB); B: tRNAPhe from Saccharomyces cerevisiae (PDB 1EVV); C: EF-P from E. coli (PDB 3A5Z), showing the domains Ⅰ, Ⅱ, Ⅲ and the Lys34 residue; D: eIF5A from S. cerevisiae (PDB 3ER0) with domains Ⅰ and Ⅱ and the conserved Lys51 residue. |
|
|
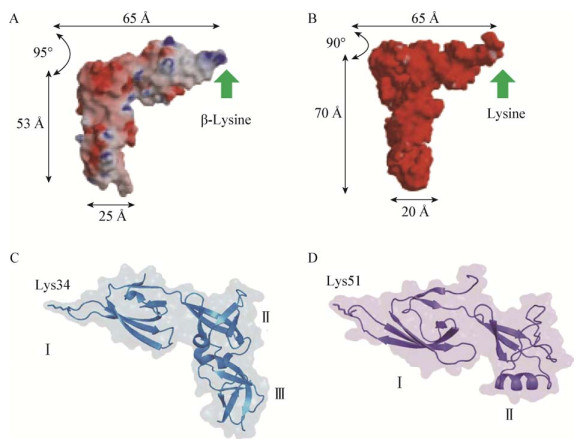
各种细菌的EF-P一级结构高度相似,它们的N端域Ⅰ的保守环上有一个带正电荷的氨基酸,和tRNAs的氨基酸受体位点有一个保守的Lys/Arg残基相似[8](图 2A)。EF-P的活性取决于这个带正电荷氨基酸的翻译后修饰。实验发现,EF-P修饰系统突变体的表型与EF-P缺失突变体相似,同时可使poly(Pro)的翻译速度降低[9]。
|
| 图 2 EF-P的修饰通路[10] Figure 2 Post-translational modification system of EF-P[10] 注:A:大肠杆菌、希瓦氏菌、嗜根考克氏菌EF-P的结构域Ⅰ、Ⅱ和Ⅲ以及N末端功能性氨基酸残基(a:大肠杆菌K-12 Lys34;b:希瓦氏菌MR-1 Arg32;c:嗜根考克氏菌DC2201 Lys32);B:大肠杆菌、沙门氏菌和福氏志贺菌EF-P (Lys34)的β-赖氨酸化及羟基化修饰;C:希瓦氏菌、铜绿假单胞菌和脑膜炎双球菌中EF-P (Arg32)鼠李糖修饰系统. Note: A: EF-P from E. coli, Shewanella oneidensis and Kocuria rhizophila, showing the domains Ⅰ, Ⅱ, Ⅲ and functional amino acid residues at the end of N (a: E. coli K-12 Lys34; b: Shewanella oneidensis MR-1 Arg32; c: Kocuria rhizophila DC2201 Lys32); B: EF-P (Lys34) β-lysylation and subsequent hydroxylation in E. coli, S. enterica and S. flexneri; C: Rhamnosyl modification of EF-P (Arg32) in S. oneidensis, P. aeruginosa and N. meningitides. |
|
|
大肠杆菌和沙门氏菌EF-P的翻译后修饰需要3种酶的参与:EpmA (也称为YjeA、PoxA或GenX)、EpmB (也称为YjeK)和EpmC (原名YfcM)[11],它们使EF-P Lys34带上一个羟基化的β-Lys官能团(图 2B)[12-13]。
EpmB是Lys-2, 3氨基变位酶(Lysine-2, 3- aminomutase,LAM)的同源体,但缺乏多聚化域。LAM蛋白通常促使(S)-α-Lys转化成(S)-β-Lys,而大肠杆菌的EpmB将(S)-α-Lys转化为(R)-β-Lys,这正是EF-P修饰路径的第一步。EpmA是赖氨酰tRNA合成酶(Lys-RS2)的间接同源体,指导Lys-tRNA到Lys位置。EpmA和Lys-RS2催化中心的序列与结构非常相似,但没有反密码子结合域,也没有与Lys相关的任何其他氨酰化tRNA。EpmA能利用EpmB催化生成的(R)-β-Lys形成一个活化的赖氨酸腺苷酸,同时有部分Lys转移到EF-P中Lys34的ε氨基,取代成为EF-P的特异性底物(图 2B)[8, 12, 14]。EpmC催化对EF-P Lys34的C5 (δ)位置进行羟化[11],从而完成整个EF-P Lys34的翻译修饰路径。
3.2 EF-P中Arg32的鼠李糖基化修饰EmpAB(C)修饰酶及其同源基因只存在于约25%的细菌中。其他大部分细菌,如脑膜炎双球菌、铜绿假单胞菌和希瓦氏菌等都不存在Lys34 β-赖氨酸化修饰途径。
2015年,Lassak等通过生物信息学分析发现,希瓦氏菌的EF-P在与大肠杆菌EF-P Lys34的同等位置变成了严格保守的Arg32。EF-P修饰途径也由Lys34 β-赖氨酸化变成了Arg32-鼠李糖基化[10, 15-16],并将参与修饰的糖基转移酶命名为EarP[16]。通过分析缺失dTDP-鼠李糖生物合成的突变体以及糖基化体外实验,确认EF-P Arg32的鼠李糖基化可加速含聚Pro蛋白质的合成。值得注意的是,只有当宿主生存需要时,rmlABCD基因才存在[15]。例如,专性细菌捕食者Micavibrio aeruginosavorus可在捕食期夺取铜绿假单胞菌的dTDP鼠李糖[15]。
临床上的致病菌,如淋球菌属、百日咳杆菌、绿脓杆菌也是通过EarP修饰酶激活EF-P活性[15-16]。Lassak等证实ef-p基因或是相应修饰酶EarP的缺失,可导致细菌的重要毒力因子如鼠李糖脂或含有聚脯氨酸的绿脓菌素毒性降低,甚至完全丧失[16]。事实上,缺少任一EF-P或EarP基因的绿脓杆菌突变株不仅毒性降低,而且对靶向细胞壁的抗生素敏感性也增加,推测可能是由于含三联子Pro蛋白MexA的翻译受到损伤的缘故[15, 17]。
EF-P的修饰不仅提高了与核糖体的亲和力,同时也加大了肽键的合成速率。但目前发现EF-P修饰酶只存在于约35%的细菌中,还有很大一部分细菌,例如Kocuria rhizophila还未发现有任何修饰系统,这些细菌可能存在未知的修饰途径,也可能随着生物的进化而产生了其他应对策略。
4 EF-P的作用机理Glick等首次揭示了EF-P的作用机理,它不是以一般方式提高肽基转移酶活性,而是作用于特定氨酰基tRNA,例如转运Gly的tRNA[18]。另一方面,EF-P不会刺激poly(Phe)或poly(Lys)的合成,在重构的体外翻译系统中模型蛋白质合成时,EF-P又变成了非必要因素。在大肠杆菌中,虽然EF-P与核糖体形成1:1的复合物,但EF-P仅是核糖体数量的10%,使得EF-P不太可能参与每一轮的肽键形成。这些结果暗示,EF-P只促进第一个肽键的合成,这一观点随后因为真核生物同源蛋白eIF5A功能的揭示而受到挑战。
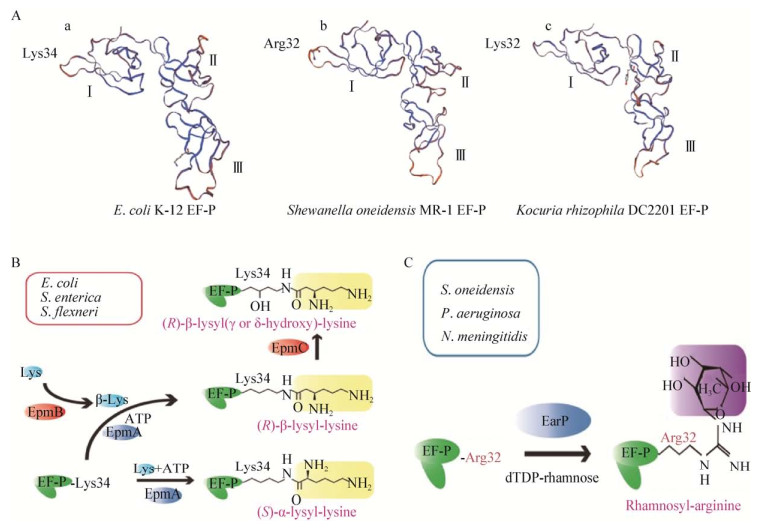
在P位上带有fMet-tRNA的嗜热细菌EF-P晶体结构表明,EF-P结合于P位与A位之间的30S与50S亚基的界面上,EF-P的域Ⅲ、Ⅱ、Ⅰ分别与fMet-tRNA的反密码子茎环、D环以及接近肽基转移酶中心的位置相互作用(图 3)[19-20]。EF-P和fMet-tRNA的作用网似乎是为了稳定tRNA,也可能是这个因子在核糖体上的功能之一。在核糖体-EF-P复合物中,50S亚基的动力元件L1移到E位与EF-P域Ⅱ连接,参与tRNA的易位和释放。EF-P-L1在EF-P的补充或者功能方面的作用并不清楚。需要注意的是,不同细菌EF-P的N端顶部的保守氨基酸各不相同,嗜热细菌的EF-P是一个Arg残基,而大肠杆菌EF-P则是一个保守的Lys残基(34位置),这个残基是翻译后通过赖氨酸化和羟基化修饰而形成的,可能进一步深入到肽基转移酶中心内部(图 3B)。
|
| 图 3 tRNA二级结构及核糖体-EF-P复合物的晶体结构[13] Figure 3 Secondary structure of tRNAfMet and crystal structure of the ribosom-EF-P complex[13] 注:A:嗜热细菌(PDB 3HUW和3HUX)的EF-P结合于核糖体E位和P位之间并连接tRNAfMet,其中核糖体的50S和30S亚基分别显示亮灰色与暗灰色,EF-P显示深绿色,tRNAfMet显示为紫色,核糖体蛋白L1显示为暗红色;B:赖氨酸化的EF-P与核糖体结合的模型图,颜色标识同A图,浅绿色表示23S rRNA残基在核糖体的催化核心(A2451、U2506、U2585和A2602);C:嗜热细菌tRNAfMet的二级结构. Note: A: The EF-P from T. thermophilus (PDB 3HUW and 3HUX) is combined with the E and the P site of the ribosome and contacts to the tRNAfMet, the 50S and 30S subunits of ribosome are colored in light and dark gray respectively, EF-P is shown in dark green and tRNAfMet in purple; Ribosomal protein L1 is shown in dark red; B: A model of lysinylated EF-P bound to the ribosome, color coded as in A, and 23S rRNA residues at the catalytic core of the ribosome (A2451, U2506, U2585 and A2602) is colored in light green; C: Secondary structure of T. thermophilus tRNAfMet. |
|
|
蛋白质合成过程中,几个连续Pro残基间肽键的形成会导致核糖体延宕,EF-P可减轻核糖体的延宕,同时促进Pro残基快速掺入多肽使翻译延续(图 4)。连续的3个Pro残基足以引起核糖体停滞在第2个Pro密码子位置,而第3个Pro残基的掺入会使其受到损害。人工导入PPP或PPG基序到原本不含poly(Pro)序列的蛋白质中并迅速合成,结果发现EF-P的存在可以使失速减缓,甚至恢复正常的合成速率。
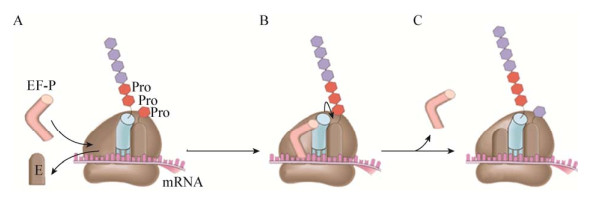
|
| 图 4 EF-P与核糖体作用的机理[7] Figure 4 Mechanism of EF-P action on the ribosome[7] 注:A:Pro或Gly作为下一个进来的氨基酸与2个连续Pro残基结合后,核糖体停滞,去酰基化tRNA解离后,E位点可与EF-P结合;B:EF-P可容纳于E位点和P位点之间,促进肽键形成;C:EF-P解离后,翻译恢复. Note: A: The ribosome stalls after incorporation of two consecutive Pro residues with Pro or Gly as the next incoming amino acid; The E site becomes free after dissociation of deacylated-tRNA, which allows for binding of EF-P; B: EF-P accommodates between E and P sites and promotes rapid peptide bond formation; C: After the dissociation of EF-P, the translation is restored. |
|
|
转译融合及体外转译实验结果均表明,当含有3个或更多连续Pro残基的蛋白质在翻译到Pro基序时易导致核糖体的延宕,而EF-P能够减缓蛋白质翻译时核糖体的延宕现象[21]。另有研究发现,EF-P可选择性刺激肽基转移酶活性[1, 22]。Doerfel等[21]和Woolstenhulme等[22]系统确定了构成二肽的fMet和任一与Pmn作用的20种蛋白氨基酸之间肽键形成的动力学过程[21-22],发现EF-P存在时fMet-Pro-Pmn的生成速度加快了90倍,而对于不含Pro的肽只有轻微的刺激作用。事实上,当多肽含有3个连续的Pro时,只有EF-P存在时才能生成完整的产物。
5.2 EF-P作用于各种含Diproly基序蛋白质的有效翻译Peil等通过蛋白质组学方法发现,EF-P不仅对含Pro三联子(PPP)的蛋白质翻译至关重要,还可作用于各种Diproly基序(X-/PP-/-X)[23]。体内外实验数据分析发现,各种含Diproly基序蛋白的翻译对EF-P的依赖程度排序如下:由最强的失速三联子如PPP、D/PP/D、PPW、APP、G/PP/G和PPN (一个字母代码一个氨基酸)到弱阻滞肽如L/PPL、CPP和HPP[23]。在缺失ef-p基因的沙门氏菌株中,含有PPP、PPG、APP和DPP基序的蛋白质合成量相应减少[24]。
5.3 EF-P在翻译Pro密码子过程中起维持正确开放阅读框的作用Gamper等研究结果表明,当mRNA上有不稳定的Pro密码子如CC[C/U]-[C/U]时,EF-P参与维持开放阅读框的准确性[25]。+1移码突变曾证实是由于相应GGG甲基化和UGG tRNAPro分离受体N1的G37(m1G37)[26]抑制。现在Gamper等说明了易位后的复合物与CCC-C序列+1移码突变主要由UGG tRNA的m1G37阻止,而EF-P主要作用于GGG tRNA。值得注意的是,当核糖体暂停在P位tRNAPro,以及CCC-C mRNA的基序与起始密码子相邻时,移码抑制尤为明显[25]。因此,EF-P补充了位于tRNAPro的m1G37的功能,同时维持蛋白质合成开放阅读框的正确。
6 EF-P的生物学意义 6.1 EF-P对细菌适应性的影响虽然EF-P不是细菌生存的必要蛋白,但EF-P及其修饰酶EmpAB(C)和EarP的缺失可导致多样化的表型变化,如生长速度和活力降低,对高渗、酸、洗涤剂、抗生素等胁迫条件的敏感性降低,以及代谢增强、毒力减弱等[27-33]。不同的细菌,如大肠杆菌、鼠伤寒沙门氏菌、农杆菌、枯草芽孢杆菌、流产布鲁氏菌、铜绿假单胞菌等均有类似的现象[12, 27, 29-35]。进一步的研究结果表明,上述表型的变化不是细菌基因转录的原因,而是翻译受到影响[30, 34],造成许多如代谢蛋白、膜相关蛋白、转运蛋白和双组分调控系统,特别是那些参与耐药性和运动性的蛋白表达水平的改变[12, 27, 30],而属于基础转译系统的蛋白几乎不受影响。
6.2 EF-P对细菌毒力的影响EF-P或其修饰酶的缺失对细菌的毒力效应影响较大。一方面,EF-P可直接作用于毒性相关的蛋白影响其翻译合成。例如,肠道出血性大肠杆菌和致病性大肠杆菌(EHEC和EPEC)[36-37]感染中的关键因子EspF,在ef-p缺失菌株中翻译阻断于PPPP基序,使得EHEC和EPEC毒性大大减弱。另一方面,通过改变蛋白质组的整体平衡,EF-P或其修饰酶间接地影响细菌毒力。在EmpA缺失的鼠伤寒沙门氏菌菌株中,双组分体系PhoP/PhoQ的毒力因子PhoP[30]过表达,打破了整个蛋白质组的平衡,使得鼠伤寒沙门氏菌毒力和活力都受损。这些因缺乏EF-P或其修饰酶导致表达下调的蛋白不胜枚举,如鼠伤寒沙门氏菌ef-p缺失菌株的外膜蛋白KdgM和SlyB[28]、铜绿假单胞菌ef-p缺失菌株的RND家族流出泵组件MexX蛋白[27]、鼠伤寒沙门氏菌EmpA缺失菌株的ManX(YZ)[38]等等。
7 结论与展望EF-P是广泛存在于细菌中的一类蛋白质翻译延伸因子,与真核生物的eIF5A同源,其类似tRNA的构象使它能够深入核糖体肽基合成的中心位置,参与蛋白质的翻译延伸。而EF-P的活性主要取决于其修饰系统,目前已知的有β-赖氨酸化修饰或鼠李糖基化修饰,作用位点都是类似tRNA氨基酸受体位点的保守氨基酸残基。通过相应的修饰,EF-P不仅可减缓蛋白质合成过程中poly(Pro)的延宕效应,同时对细菌自身的适应性以及一些致病菌的毒性有重要的作用。细菌EF-P的这一特点使它有望成为抗菌药物筛选的新型药靶。
纵观近几十年来研究EF-P的文献,有关细菌EF-P的研究集中在它的来源、结构、功能、作用机理及其生物学意义,至今未发现有关细胞内EF-P自身表达调控的文献报道。
本实验室在前期研究工作中获得一种能下调细菌EF-P表达的抗生素JX[39-40],这为研究细菌EF-P自身的表达调控提供了重要材料。通过iTRAQ实验发现在抗生素JX胁迫作用下(1/2 MIC),嗜根考克氏菌(Kocuria rhizophila) EF-P蛋白下调倍数为0.782 5,这一结果通过实时荧光定量PCR得到印证,嗜根考克氏菌EF-P的mRNA相对含量为0.92,小于1[41]。下一步我们将从ef-p自身的表达调控出发,鉴定并验证调控ef-p基因表达的各种元件,并以此为契机筛选与调控元件相关的互作蛋白,通过各种转录调控研究技术探求细菌细胞内ef-p自身的表达调控规律及其分子机制,为进一步揭示细菌体内蛋白质的生物合成规律,进而为靶向细菌蛋白质合成的新药研究提供理论和实践依据。
| [1] |
Glick BR, Ganoza MC. Identification of a soluble protein that stimulates peptide bond synthesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1975, 72(11): 4257-4260. DOI:10.1073/pnas.72.11.4257 |
| [2] |
An G, Glick BR, Friesen JD, et al. Identification and quantitation of elongation factor EF-P in Escherichia coli cell-free extracts[J]. Canadian Journal of Biochemistry, 1980, 58(11): 1312-1314. DOI:10.1139/o80-177 |
| [3] |
Aoki H, Adams SL, Chung DG, et al. Cloning, sequencing and overexpression of the gene for prokaryotic factor EF-P involved in peptide bond synthesis[J]. Nucleic Acids Research, 1991, 19(22): 6215-6220. DOI:10.1093/nar/19.22.6215 |
| [4] |
Hanawa-Suetsugu K, Sekine SI, Sakai H, et al. Crystal structure of elongation factor P from Thermus thermophilus HB8[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(26): 9595-9600. DOI:10.1073/pnas.0308667101 |
| [5] |
Choi S, Choe J. Crystal structure of elongation factor P from Pseudomonas aeruginosa at 1.75 resolution[J]. Proteins: Structure, Function, and Bioinformatics, 2011, 79(5): 1688-1693. DOI:10.1002/prot.v79.5 |
| [6] |
Kristensen O, Laurberg M, Gajhede M. Crystallization of a stringent response factor from Aquifex aeolicus[J]. Acta Crystallographica Section D: Biological Crystallography, 2002, 58(7): 1198-1200. DOI:10.1107/S0907444902008417 |
| [7] |
Doerfel LK, Rodnina MV. Elongation factor P: Function and effects on bacterial fitness[J]. Biopolymers, 2013, 99(11): 837-845. DOI:10.1002/bip.22341 |
| [8] |
Katz A, Solden L, Zou SB, et al. Molecular evolution of protein-RNA mimicry as a mechanism for translational control[J]. Nucleic Acids Research, 2014, 42(5): 3261-3271. DOI:10.1093/nar/gkt1296 |
| [9] |
Sasaki K, Abid R, Miyazaki M. Deoxyhypusine synthase gene is essential for cell viability in the yeast Saccharomyces cerevisiae[J]. FEBS Letters, 1996, 384(2): 151-154. DOI:10.1016/0014-5793(96)00310-9 |
| [10] |
Yanagisawa T, Takahashi H, Suzuki T, et al. Neisseria meningitidis translation elongation factor P and its active-site arginine residue are essential for cell viability[J]. PLoS One, 2016, 11(2): e0147907. DOI:10.1371/journal.pone.0147907 |
| [11] |
Peil L, Starosta AL, Virum e K, et al. Lys34 of translation elongation factor EF-P is hydroxylated by YfcM[J]. Nature Chemical Biology, 2012, 8(8): 695-697. DOI:10.1038/nchembio.1001 |
| [12] |
Navarre WW, Zou SB, Roy H, et al. PoxA, YjeK, and elongation factor P coordinately modulate virulence and drug resistance in Salmonella enterica[J]. Molecular Cell, 2010, 39(2): 209-221. DOI:10.1016/j.molcel.2010.06.021 |
| [13] |
Blaha G, Stanley RE, Steitz TA. Formation of the first peptide bond: The structure of EF-P bound to the 70S ribosome[J]. Science, 2009, 325(5943): 966-970. DOI:10.1126/science.1175800 |
| [14] |
Yanagisawa T, Sumida T, Ishii R, et al. A paralog of lysyl-tRNA synthetase aminoacylates a conserved lysine residue in translation elongation factor P[J]. Nature Structural & Molecular Biology, 2010, 17(9): 1136-1143. |
| [15] |
Rajkovic A, Erickson S, Witzky A, et al. Cyclic rhamnosylated elongation factor P establishes antibiotic resistance in Pseudomonas aeruginosa[J]. mBio, 2015, 6(3): e00823-5. |
| [16] |
Lassak J, Keilhauer EC, Fürst M, et al. Arginine-rhamnosylation as new strategy to activate translation elongation factor P[J]. Nature Chemical Biology, 2015, 11(4): 266-270. DOI:10.1038/nchembio.1751 |
| [17] |
Li XZ, Nikaido H, Poole K. Role of mexA-mexB-oprM in antibiotic efflux in Pseudomonas aeruginosa[J]. Antimicrobial Agents and Chemotherapy, 1995, 39(9): 1948-1953. DOI:10.1128/AAC.39.9.1948 |
| [18] |
Glick BR, Chládek S, Ganoza MC. Peptide bond formation stimulated by protein synthesis factor EF-P depends on the aminoacyl moiety of the acceptor[J]. European Journal of Biochemistry, 1979, 97(1): 23-28. DOI:10.1111/ejb.1979.97.issue-1 |
| [19] |
Ude S, Lassak J, Starosta AL, et al. Translation elongation factor EF-P alleviates ribosome stalling at polyproline stretches[J]. Science, 2013, 339(6115): 82-85. DOI:10.1126/science.1228985 |
| [20] |
Ganoza MC, Aoki H. Peptide bond synthesis: Function of the efp gene product[J]. Biological Chemistry, 2000, 381(7): 553-559. |
| [21] |
Doerfel LK, Wohlgemuth I, Kothe C, et al. EF-P is essential for rapid synthesis of proteins containing consecutive proline residues[J]. Science, 2013, 339(6115): 85-88. DOI:10.1126/science.1229017 |
| [22] |
Woolstenhulme CJ, Parajuli S, Healey DW, et al. Nascent peptides that block protein synthesis in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(10): E878-E887. DOI:10.1073/pnas.1219536110 |
| [23] |
Peil L, Starosta AL, Lassak J, et al. Distinct XPPX sequence motifs induce ribosome stalling, which is rescued by the translation elongation factor EF-P[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(38): 15265-15270. DOI:10.1073/pnas.1310642110 |
| [24] |
Hersch SJ, Wang MC, Zou SB, et al. Divergent protein motifs direct elongation factor P-mediated translational regulation in Salmonella enterica and Escherichia coli[J]. mBio, 2013, 4(2): e00180-13. |
| [25] |
Gamper HB, Masuda I, Frenkel-Morgenstern M, et al. Maintenance of protein synthesis reading frame by EF-P and m1G37-tRNA[J]. Nature Communications, 2015, 6: 7226. DOI:10.1038/ncomms8226 |
| [26] |
Bj rk GR, Wikstr m PM, Bystr m AS. Prevention of translational frameshifting by the modified nucleoside 1-methylguanosine[J]. Science, 1989, 244(4907): 986-989. DOI:10.1126/science.2471265 |
| [27] |
Balibar CJ, Iwanowicz D, Dean CR. Elongation factor P is dispensable in Escherichia coli and Pseudomonas aeruginosa[J]. Current Microbiology, 2013, 67(3): 293-299. DOI:10.1007/s00284-013-0363-0 |
| [28] |
Zou SB, Hersch SJ, Roy H, et al. Loss of elongation factor P disrupts bacterial outer membrane integrity[J]. Journal of Bacteriology, 2012, 194(2): 413-425. DOI:10.1128/JB.05864-11 |
| [29] |
Iannino F, Ugalde JE, de Lannino NI. Brucella abortus efp gene is required for an efficient internalization in HeLa cells[J]. Microbial Pathogenesis, 2012, 52(1): 31-40. DOI:10.1016/j.micpath.2011.09.008 |
| [30] |
Bearson SMD, Bearson BL, Brunelle BW, et al. A mutation in the poxA gene of Salmonella enterica serovar Typhimurium alters protein production, elevates susceptibility to environmental challenges, and decreases swine colonization[J]. Foodborne Pathogens and Disease, 2011, 8(6): 725-732. DOI:10.1089/fpd.2010.0796 |
| [31] |
Behshad E, Ruzicka FJ, Mansoorabadi SO, et al. Enantiomeric free radicals and enzymatic control of stereochemistry in a radical mechanism: The case of lysine 2, 3-aminomutases[J]. Biochemistry, 2006, 45(42): 12639-12646. DOI:10.1021/bi061328t |
| [32] |
Peng WT, Banta LM, Charles TC, et al. The chvH locus of Agrobacterium encodes a homologue of an elongation factor involved in protein synthesis[J]. Journal of Bacteriology, 2001, 183(1): 36-45. DOI:10.1128/JB.183.1.36-45.2001 |
| [33] |
Kaniga K, Compton MS, Curtiss Ⅲ R, et al. Molecular and functional characterization of Salmonella enterica serovar Typhimurium poxA gene: Effect on attenuation of virulence and protection[J]. Infection and Immunity, 1998, 66(12): 5599-5606. |
| [34] |
Aoki H, Dekany K, Adams SL, et al. The gene encoding the elongation factor P protein is essential for viability and is required for protein synthesis[J]. Journal of Biological Chemistry, 1997, 272(51): 32254-32259. DOI:10.1074/jbc.272.51.32254 |
| [35] |
Abratt VR, Mbewe M, Woods DR. Cloning of an EF-P homologue from Bacteroides fragilis that increases B. fragilis glutamine synthetase activity in Escherichia coli[J]. Molecular and General Genetics MGG, 1998, 258(4): 363-372. DOI:10.1007/s004380050742 |
| [36] |
Holmes A, Mühlen S, Roe AJ, et al. The EspF effector, a bacterial pathogen's Swiss army knife[J]. Infection and Immunity, 2010, 78(11): 4445-4453. DOI:10.1128/IAI.00635-10 |
| [37] |
Zou SB, Roy H, Ibba M, et al. Elongation factor P mediates a novel post-transcriptional regulatory pathway critical for bacterial virulence[J]. Virulence, 2011, 2(2): 147-151. |
| [38] |
Bowden SD, Rowley G, Hinton JCD, et al. Glucose and glycolysis are required for the successful infection of macrophages and mice by Salmonella enterica serovar typhimurium[J]. Infection and Immunity, 2009, 77(7): 3117-3126. DOI:10.1128/IAI.00093-09 |
| [39] |
Long ZE, Zhu YJ, Huang YH, et al. Isolation and purification of antibiotic from the fermentation broth of Micromonospora carbonacea JXNU-1 and its physical-chemical properties[J]. Microbiology China, 2008, 35(9): 1450-1454. 龙中儿, 朱跃进, 黄运红, 等. 炭样小单孢菌JXNU-1广谱抗生素产物的分离及其理化性质[J]. 微生物学通报, 2008, 35(9): 1450-1454. |
| [40] |
Peng WM. Studies on the antibacterial activity of a new nucleoside antibiotic from a strain of Micromonospora cabonacea[D]. Nanchang: Master's Thesis of Jiangxi Normal University, 2013 (in Chinese) 彭伟梦. 一株炭样小单孢菌产新型核苷类抗生素的抗菌作用研究[D]. 南昌: 江西师范大学硕士学位论文, 2013 |
| [41] |
Chen P. The study on antagonism mechanism of the antibiotics JX from Micromonospora carbonacea JXNU-1 against Kocuria rhizophila[D]. Nanchang: Master's Thesis of Jiangxi Normal University, 2016 (in Chinese) 陈鹏. 炭样小单孢菌JXNU-1产抗生素JX对嗜根考克氏菌的拮抗作用机理研究[D]. 南昌: 江西师范大学硕士学位论文, 2016 |
 2018, Vol. 45
2018, Vol. 45