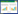扩展功能
文章信息
- 李琳, 王磊, 王文静, 张瑞良, 徐军, 王瑞, 赵霞
- LI Lin, WANG Lei, WANG Wen-Jing, ZHANG Rui-Liang, XU Jun, WANG Rui, ZHAO Xia
- 基于串联质谱标签法和平行反应监测技术的氟喹诺酮耐药沙门菌蛋白质组学分析
- Fluoroquinolone resistant Salmonella proteomics analysis based on tandem mass tag and parallel reaction monitoring techniques
- 微生物学通报, 2018, 45(7): 1535-1545
- Microbiology China, 2018, 45(7): 1535-1545
- DOI: 10.13344/j.microbiol.china.170835
-
文章历史
- 收稿日期: 2017-12-27
- 接受日期: 2018-04-20
- 网络首发日期(www.cnki.net): 2018-04-24
沙门菌(Salmonella)是重要的人畜共患病原菌,可引起伤寒、副伤寒和败血症等,严重危害养殖业和食品安全[1]。氟喹诺酮类药物(Fluoroquinolones,FQs)是一类用于治疗沙门菌感染的常用药物,但由于其不规范使用导致了近年来FQs的耐药性不断增加[2-4]。沙门菌的FQ耐药机制非常复杂,主要包括基因突变、产生水解酶或钝化酶、药物靶位改变、细胞膜通透性改变、主动外排功能增强和获得外源耐药基因等。目前,已有基因组学、转录组学研究沙门菌耐药性的相关报道[5-6],而从蛋白质组学的角度研究诱导耐药沙门菌的FQ耐药机理尚未见报道。
高通量蛋白质组学技术的应用为研究细菌耐药性提供了有效途径。串联质谱标签(Tandem mass tag,TMT)是利用化学标签通过串联质谱对不同样品中的蛋白进行同时鉴定和定量的研究方法,已广泛应用于蛋白质组学研究[7]。传统的蛋白质组验证手段如蛋白质免疫印迹法(Western blot)和酶联免疫吸附测定(ELISA)等由于抗体等要求,使得验证对象和数量受到了限制,而平行反应监测(Parallel reaction monitoring,PRM)是一种靶向的蛋白质组学技术,具有灵敏度高、分辨率高、可同时检测多个目的蛋白、兼具定性定量且无需抗体等优点,主要用于后期差异蛋白质的规模化验证[8],是理想的验证手段。本研究利用TMT定量蛋白组学结合PRM技术验证,对鼠伤寒沙门菌FQ耐药性获得前后进行蛋白质组学分析,为深入研究沙门菌的FQ耐药机理奠定基础。
1 材料与方法 1.1 菌株鼠伤寒沙门菌ATCC13311 (标准株)由中国农业大学动物医学院兽医药理学与毒理学实验室惠赠,沙门菌环丙沙星耐药株由本实验室诱导并保存。
1.2 主要试剂和仪器TMT标记试剂盒,赛默飞世尔科技公司;胰蛋白酶,Promega公司;BCA试剂盒,碧云天生物技术有限公司;乙腈,Fisher Chemical公司;碘代乙酰胺、二硫苏糖醇和尿素等试剂,Sigma- Aldrich公司;环丙沙星标准品,中国食品药品检定研究院;其余常规试验试剂均为国产分析纯。
EASY-nLC 1000超高效液相系统、Orbitrap Fusion Lumos质谱仪,赛默飞世尔科技公司。
1.3 培养基MH肉汤培养基(Mueller Hinton),杭州微生物试剂有限公司;LB培养基(g/L):蛋白胨10.0,酵母提取物5.0,NaCl 10.0。
1.4 沙门菌环丙沙星耐药性诱导使用浓度梯度递增法诱导耐药菌株[9],将鼠伤寒沙门菌标准株接种于MH培养基中,37 ℃、180 r/min振荡培养过夜,菌液接种于环丙沙星(MIC值为0.004 mg/L)的MH肉汤中培养。然后,不断以相同方式接种ATCC13311于药物浓度依次翻倍的MH肉汤中,直至菌株可以在环丙沙星浓度为32 mg/L的培养基中生长。耐药菌株在空白培养基中传5代,然后进行药敏实验以确定其并非是适应性生长。参照CLSI标准对鼠伤寒沙门菌耐药株进行环丙沙星的MIC值测定。
1.5 TMT定量蛋白组学方法和PRM靶向蛋白验证由杭州景杰生物科技有限公司提供并完成。
1.5.1 蛋白提取和定量分别挑取沙门菌标准株与耐药株的单菌落,接种于LB液体培养基中,37 ℃、180 r/min振荡培养12 h,再转接于相应的LB培养基培养至对数前期(OD600约0.4)。菌液于4 ℃、8 000 r/min离心5 min,去上清液得菌体沉淀。再用0.01 mol/L PBS洗涤菌体沉淀并同上离心,重复2次,各组样品分别加入300 μL裂解缓冲液(8 mol/L尿素,1%蛋白酶抑制剂,2 mmol/L EDTA),超声裂解(功率900 W,超声3 min),4 ℃、20 000×g离心10 min,上清液转移至新的离心管,取5 μL蛋白样品,使用BCA试剂盒测定蛋白浓度。
根据蛋白浓度测定结果,每个样品取30 μg蛋白到离心管中,加入5 μL 4×Loading buffer,再加入2% SDS使总体积为20 μL,金属浴100 ℃热变性5 min;依次上样1 μL预染蛋白Marker和20 μL蛋白样品,样品相邻的空白孔上样20 μL 1×Loading buffer封闭;浓缩胶80 V电泳20 min,分离胶120 V电泳到溴酚蓝指示带到胶底端;胶取出后于考马斯亮蓝R250染液中室温染色2 h,然后加入脱色液,脱色至背景无色、条带清晰。
1.5.2 样品的胰酶酶解、TMT标记蛋白及HPLC分级蛋白溶液中加入二硫苏糖醇使其终浓度为5 mmol/L,56 ℃还原30 min。之后加入碘代乙酰胺使其终浓度为11 mmol/L,室温避光孵育15 min。最后将样品的尿素浓度稀释至低于2 mol/L。以1׃50的质量比例(胰酶׃蛋白)加入胰酶,37 ℃酶解过夜。再以1׃100的质量比例(胰酶׃蛋白)加入胰酶,继续酶解4 h;胰酶酶解的肽段用Strata X C18 (Phenomenex)除盐后真空冷冻干燥。以0.5 mol/L TEAB肽段用高pH反向HPLC分级,色谱柱为Agilent 300 Extend C18 (5 μm粒径,4.6 mm内径,250 mm长)。根据TMT试剂盒操作说明标记肽段,标记试剂解冻后用乙腈溶解,与肽段混合后室温孵育2 h,标记后的肽段混合后除盐,真空冷冻干燥;分级梯度为8%−32%乙腈,pH 9.0,60 min分离60个组分,随后肽段合并为5个组分,合并后的组分经真空冷冻干燥后进行后续操作。
1.5.3 液相色谱-质谱联用分析肽段用液相色谱流动相A相(0.1%,甲酸水溶液体积比)溶解后使用EASY-nLC 1000超高效液相系统进行分离。流动相A为含0.1%甲酸的水溶液;流动相B为含0.1%甲酸的乙腈溶液。液相梯度设置:0−40 min,6%−20% B;40−52 min,20%−30% B;52−56 min,30%−80% B;56−60 min,80% B,流速维持在350 nL/min。
肽段经由超高效液相系统分离后被注入NSI离子源中进行电离,然后使用Orbitrap Fusion Lumos质谱仪进行分析。离子源电压设置为2.4 kV,肽段母离子及其二级碎片都使用高分辨的Orbitrap进行检测和分析。数据采集模式使用数据依赖型扫描(DDA)程序。为了提高质谱的有效利用率,自动增益控制(AGC)设置为5E4,信号阈值设置为5 000 ions/s,最大注入时间设置为100 ms,串联质谱扫描的动态排除时间设置为30 s,避免母离子的重复扫描。
1.5.4 数据库搜索二级质谱数据使用MaxQuant (V1.5.2.8)进行检索。检索参数设置:数据库为Uniprot Salmonella typhimurium (4 743条序列),添加了反库以计算随机匹配造成的假阳性率(FDR),并且在数据库中加入了常见的污染库,用于消除鉴定结果中污染蛋白的影响;酶切方式设置为Trypsin/P;漏切位点数设为2;肽段最小长度设置为7个氨基酸残基;肽段最大修饰数设为5;First search和Main search的一级母离子质量误差容忍度分别设为0.02 g/L和0.005 g/L,二级碎片离子的质量误差容忍度为0.02 Da;将半胱氨酸烷基化设置为固定修饰,可变修饰为甲硫氨酸的氧化和蛋白N端的乙酰化;定量方法设置为TMT-6plex,蛋白鉴定、PSM (Peptide spectrum matching)鉴定的FDR都设置为1%。
1.5.5 PRM靶向蛋白验证提取样品蛋白,用胰酶酶解后,肽段用液相色谱流动相A相(0.1%,甲酸水溶液体积比)溶解后使用EASY-nLC 1000超高效液相系统进行分离。流动相A为含0.1%甲酸和2%乙腈的水溶液;流动相B为含1%甲酸和90%乙腈的水溶液。液相梯度设置:0−38 min,7%−25% B;38−52 min,23%−40% B;52−56 min,40%−80% B;56−60 min,80% B,流速维持在700 nL/min。
肽段经由超高效液相系统分离后被注入NSI离子源中进行电离,然后使用Q ExactiveTM Plus质谱进行分析。离子源电压设置为2.0 kV,肽段母离子及其二级碎片都使用高分辨率的Orbitrap进行检测和分析。数据采集模式使用数据非依赖型扫描(DIA)程序,HCD碰撞池的碎裂能量设置为27。一级质谱自动增益控制(AGC)设置为3E6,最大离子注入时间(Maxumum IT)设置为50 ms;二级质谱自动增益控(AGC)设置为1E5,最大离子注入时间(Maxumum IT)设置为100 ms,隔离窗口(Isolation window)设置为2.0 m/z。
肽段参数:蛋白酶设置为Trypsin[KR/P],最大漏切位点数设置为0,肽段长度设置为7–25个氨基酸残基,设置半胱氨酸烷基化为固定修饰。Transition参数:母离子电荷设置为2、3,子离子电荷设置为1,离子类型设置为b、y,离子匹配的质量误差容忍度设置为0.02 Da。
1.6 生物信息学分析将蛋白质组学的数据进行分析处理后,以2倍为变化阈值(Fold change≥2或≤0.5),P < 0.05为标准[10]。将筛选出来的差异蛋白进行蛋白注释、蛋白质功能富集等。
2 结果与分析 2.1 药敏实验结果药敏实验结果显示,鼠伤寒沙门菌标准株经环丙沙星诱导后,对环丙沙星的耐药值为32 mg/L。
2.2 蛋白鉴定总览本实验共鉴定了2 517个蛋白质,其中2 265个蛋白质具有定量信息。其中分别以变化阈值为1.2、1.3、1.5和2.0,P < 0.05为标准,差异蛋白数量见表 1。
| 比较组 Compare group |
耐药株/标准株 Drug resistant strains/Standard strains |
|
| 上调 Up regulated |
下调 Down regulated |
|
| Fold change ≥ 1.2 | 489 | 471 |
| Fold change ≥ 1.3 | 413 | 374 |
| Fold change ≥ 1.5 | 305 | 263 |
| Fold change ≥ 2.0 | 159 | 159 |
由图 1可知,在上调蛋白中,参与的生物过程有刺激反应、信号处理、定位过程、运动反应等。分子功能主要表现在催化活性、信号传感器活动、转运活性等。细胞组分主要集中在细胞器、胞外区、大分子复合物、细胞膜。
|
| 图 1 上调蛋白在GO二级注释中的分布情况(耐药株/标准株) Figure 1 Distribution of up-regulated proteins in GO secondary annotation (drug resistant strains/standard strains) |
|
|
由图 2可知,在159个下调蛋白中,参与的生物过程有运动反应、信号处理、刺激反应、定位过程等。分子功能主要表现在核酸结合转录因子的活动、分子传感器活动、信号传感器活动、转运活性等。细胞组分主要集中在细胞器、大分子复合物、细胞膜。

|
| 图 2 下调蛋白在GO二级注释中的分布情况(耐药株/标准株) Figure 2 Distribution of down-regulated proteins in GO secondary annotation (drug resistant strains/standard strains) |
|
|
对鉴定到的全体蛋白的注释以及差异表达蛋白的筛选,进行GO、KEGG、蛋白结构域(Domain)等功能注释类型的富集分析,运用Fisher’s exact test (费希尔精确检验)得到的p值进行负对数(−log10)转换,即得到转换后的值越大则此功能类型的富集越显著。
2.4.1 GO富集由图 3可知,在上调蛋白参与的生物过程中,富集显著的有趋药性、运动反应、对外刺激反应、化学反应、信号转导等方面;分子功能中富集显著的有信号传感器活动;细胞组分中富集显著的有细胞突触、膜的有机组分、膜固有组分以及膜组分。

|
| 图 3 上调蛋白GO富集结果(耐药株/标准株) Figure 3 Up-regulation of protein GO enrichment results (drug resistant strains/standard strains) |
|
|
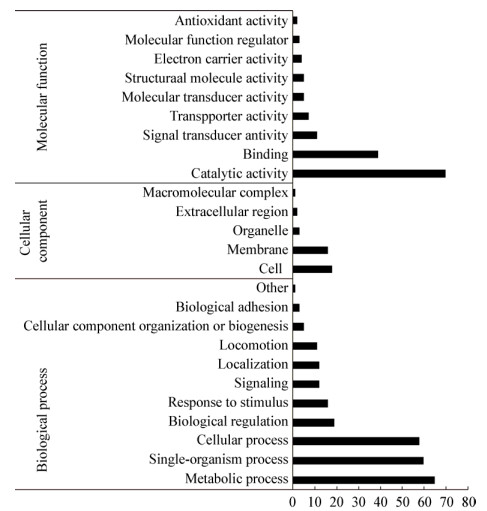
由图 4可知,在下调蛋白参与的生物过程中,富集显著的有定位过程、转运、糖的运输及合成、丙酸的分解及代谢等方面;分子功能中富集显著的有糖的跨膜转运活性、膜孔蛋白活性等方面。
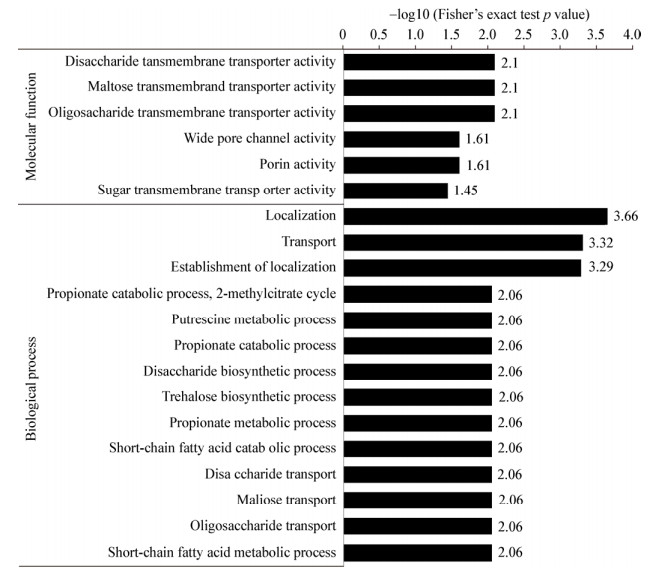
|
| 图 4 下调蛋白GO富集结果(耐药株/标准株) Figure 4 Down-regulation of protein GO enrichment results (drug resistant strains/standard strains) |
|
|
KEGG (Kyoto encyclopedia of genes and genomes)是连接已知分子间相互作用的信息网络,如代谢通路、复合物、生化反应。KEGG通路主要包括:代谢、遗传信息处理、环境信息处理、细胞过程等。
由图 5和图 6可知,上调蛋白富集显著的KEGG通路有细菌趋化性、双组分系统、链霉素的生物合成、组氨酸的代谢和酮化合物糖单位的生物合成;下调蛋白富集显著的KEGG通路有ABC转运系统、淀粉与蔗糖的代谢、细菌群体感应、β-内酰胺类抗性、氨基糖与核苷酸糖代谢、阳离子抗菌肽抗性。细菌趋化性KEGG通路和双组分系统KEGG通路见图 7和图 8 (红色表示上调蛋白,绿色表示下调蛋白)。

|
| 图 5 上调蛋白的KEGG通路富集结果(耐药株/标准株) Figure 5 Up-regulation of protein KEGG pathway enrichment results (drug resistant strain/standard strain) |
|
|
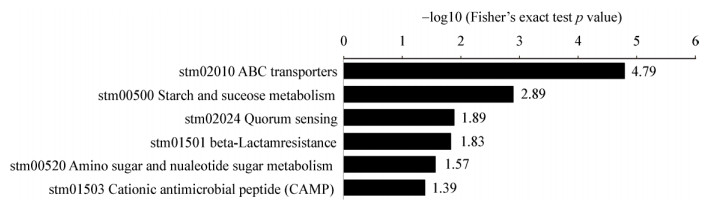
|
| 图 6 下调蛋白的KEGG通路富集结果(耐药株/标准株) Figure 6 Down-regulation of protein KEGG pathway enrichment results (drug resistant strains/standard strains) |
|
|

|
| 图 7 细菌趋化性KEGG通路 Figure 7 KEGG pathway of bacterial chemotaxis |
|
|

|
| 图 8 双组分系统KEGG通路(部分) Figure 8 KEGG pathway of two-component system (part) |
|
|
由表 2可知,在15个目的蛋白的PRM验证中,受限于部分蛋白的特性及其表达丰度,定量到了13个目的蛋白,PRM验证结果与TMT定量结果趋势一致。
| 蛋白名称 Protein name |
蛋白编号 Protein accession No. |
蛋白描述 Protein description |
TMT | PRM |
| AcrA | A0A0D6H824 | Multidrug transporter | 4.319 | 5.057 |
| HisJ | A0A0D6FAY9 | Histidine ABC transporter substrate-binding protein HisJ | 2.174 | 2.506 |
| Cse4 | A0A0D6IH30 | CRISPR-associated protein Cse4 | 2.238 | 2.960 |
| TolC | A0A0M2J547 | Membrane protein | 3.140 | 5.237 |
| bepG_1 | A0A0D6H9C4 | Efflux pump membrane transporter | 3.550 | 7.088 |
| CheW | A0A0D6FN54 | Chemotaxis protein CheW | 11.371 | 40.983 |
| OmpC | C7EXM1 | Membrane protein | 0.128 | 0.010 |
| OmpR | A0A0J6DVR3 | Osmolarity response regulator | 0.398 | 0.319 |
| PhoQ | A0A0W5X4D4 | Sensor protein PhoQ | 0.382 | 0.122 |
| PhoP | A0A0J5IUL5 | Transcriptional regulator | 0.371 | 0.095 |
| RstA | A0A0D6G7Y0 | Transcriptional regulator | 0.383 | 0.054 |
| FliC | A0A0W5C546 | Flagellin (Fragment) | 10.309 | INF |
| Hfq | A0A0B5KAT0 | RNA-binding protein Hfq | 7.808 | INF |
| 注:INF表示在PRM实验中该蛋白只在耐药菌中被鉴定到. Note: INF indicates that the protein was identified only in drug-resistant bacteria in PRM experiments. |
||||
本研究利用TMT蛋白定量结合PRM靶向蛋白验证技术,对鼠伤寒沙门菌标准株和耐药株进行了蛋白质组学分析,共鉴定到2 517个蛋白质,其中2 265个具有定量信息,标记效率达到了90%,标记效果良好,共筛选出差异蛋白318个(上调159个、下调159个)。结果显示,鼠伤寒沙门菌在获得FQ耐药性之后,差异表达蛋白显著富集在趋药性、双组分系统、ABC转运等途径;筛选了15个差异显著的蛋白做PRM靶向蛋白验证,成功定量到13个差异蛋白且与TMT结果相符。
3.1 外排泵与沙门菌FQ耐药性外排泵是细菌耐药的主要调控途径,沙门菌中AcrAB-TolC属于RND家族,是参与FQs耐药调控的主要外排泵,同时也是沙门菌对FQs产生高水平耐药的重要机制,其过量表达可以降低细菌细胞内的药物浓度[11]。Chen等[12]研究表明,在环丙沙星的选择压力下,诱导菌株对环丙沙星的值可达到256 μg/mL,但当AcrB失活后突变株对环丙沙星的MIC值降到0.25 μg/mL。Blair等[13]研究表明,当细菌存在AcrAB-TolC时,其他外排泵不会对细菌的多重耐药性产生显著影响。Ricci等[14]在体外诱导实验中表明,当鼠伤寒沙门菌缺失了tolC时,筛选不到突变株。本实验中AcrA、TolC等外排泵相关蛋白显著上调,说明AcrAB-TolC外排系统是沙门菌FQs耐药性的重要调控机制。
3.2 趋化性、双组分系统与沙门菌FQ耐药性细菌趋化性(Chemotaxis)是指具有运动能力的细菌通过感应环境信号和调整鞭毛运动方向来达到对有利环境的趋附和对有害环境的避离,当趋化物为药物时即为趋药性。Szurmant等[15]研究显示,双组分系统(Two-component system,TCS)是细菌完成趋药性的主要途径,TCS的跨膜组氨酸激酶负责感受化学效应物刺激,而胞浆应答调节蛋白则将接受到的化学刺激转换为有效的鞭毛信号,进而产生趋化运动。TCS是普遍存在于原核生物中的信号转导系统,它是细菌感受和应答外界信号的基础,与细菌的致病性、耐药性等有着密切关系[16-17]。Li等[18]通过对嗜水气单胞菌的耐药株与标准株进行定量蛋白组实验,发现耐药株的CheA、CheR、CheW等趋药性相关蛋白和FliS、FliG等鞭毛蛋白的表达均有显著差异。Shprung等[19]研究表明,PhoP/PhoQ双组分系统的表达可调控沙门菌的耐药性。本研究中,沙门菌在获得FQ耐药性之后,趋药性相关蛋白MCPs、CheA、CheW、CheY、CheB、CheV以及鞭毛蛋白FliC均显著上调,其KEGG通路显著富集,说明沙门菌FQ耐药性的调控过程中,趋药性起到重要作用。PhoP、PhoQ、RstA、BasR等双组分系统相关蛋白表达差异显著,其KEGG通路显著富集,提示双组分系统通过自我调节及与其他系统的相互作用来调控FQ耐药相关蛋白的表达。
3.3 其他差异蛋白与沙门菌FQ耐药性外膜蛋白(Outer membrane protein,Omp)是细菌细胞膜的重要组分,参与药物向细胞内的转运,当外膜蛋白异常时,细胞膜的通透性会下降,影响药物转运可导致耐药。本研究中,OmpC和OmpR等外膜蛋白的表达显著下调,说明外膜蛋白也参与了沙门菌FQ耐药性的调控。
细菌中的非编码小RNA (Small non-cading RNA,sRNA)是一个重要的基因表达调控层次,能够参与细菌mRNA的翻译和稳定性调节、RNA的加工与修饰及蛋白质的运输等[20]。目前已有研究表明sRNA能够参与细菌耐药性的调控[21-22]。Hfq是细菌中高度保守的六聚体蛋白质,可以通过sRNA与mRNA的结合来调控靶基因的表达[23-24]。Sittka等[25]研究表明沙门菌sRNA表达量变化与hfq的缺失有关。本研究显示耐药菌与敏感菌相比,Hfq蛋白表达差异显著,提示Hfq-sRNA参与沙门菌FQ耐药性的调控。
本研究通过TMT蛋白定量结合PRM靶向蛋白验证技术,筛选出鼠伤寒沙门菌FQ耐药相关蛋白及KEGG通路,主要包括外排泵相关蛋白、双组分相关蛋白、外膜蛋白及细菌趋化性、双组分系统、ABC转运系统、细菌群体感应、β-内酰胺类抗性等通路。其中,AcrA/TolC等外排泵相关蛋白、PhoP/Q等双组分相关蛋白、OmpC等外膜蛋白和双组分系统、ABC转运系统、细菌群体感应等通路的差异情况与以往报道的沙门菌耐药性研究相符[13, 19, 26]。而本研究筛选到的MCPs、CheA、CheW、Hfq、Cse4等蛋白和细菌趋化性、β-内酰胺类抗性等通路在沙门菌中首次报道,提示上述差异蛋白是沙门菌潜在的耐药性调控靶点。
Li等[18]发现嗜水气单胞菌在获得耐药性后MCPs、CheA、CheW的表达发生显著变化;Hfq蛋白对耐药性的影响在肺炎克雷伯菌中有过报道[27];Sampson等[28]发现CRISPR相关蛋白Cas9对弗朗西斯菌的耐药性具有增强作用;而本研究筛选的差异蛋白MCPs、CheA、CheW、Hfq和CRISPR相关蛋白Cse4等蛋白与沙门菌耐药性的关系为首次报道。在KEGG通路中,嗜水气单胞菌标准株与耐药株的蛋白质组定量结果显示,细菌趋化性通路差异显著[18];细菌群体感应系统对细菌的生物被膜形成和革兰氏阴性菌的RND家族外排泵的表达具有调控作用[29-30];Dreier等[31]研究显示,绿脓杆菌中β-内酰胺类抗性与其外排泵表达和耐药性有关。而本研究发现细菌趋化性、β-内酰胺类抗性通路等均有显著差异,提示这些通路参与沙门菌对FQ耐药性的调控。
在后续研究中,我们将通过编码目标蛋白基因缺失株的构建等进一步探究这些蛋白和通路对耐药性的调控作用和机制,为深入研究沙门菌FQ耐药机理提供依据和研究思路。
| [1] |
Patel S, McCormick BA. Mucosal inflammatory response to Salmonella typhimurium infection[J]. Frontiers in Immunology, 2014, 5: 311. |
| [2] |
Shi HP, Zhou XY, Wang YX, et al. Correlation of biofilm-forming ability and antimicrobial resistance of salmonella strains isolated from layers[J]. China Poultry, 2017, 39(2): 22-27. 师红萍, 周雪雁, 王勇祥, 等. 蛋鸡沙门菌分离株生物被膜与耐药相关性研究[J]. 中国家禽, 2017, 39(2): 22-27. |
| [3] |
Andoh LA, Dalsgaard A, Obiri-Danso K, et al. Prevalence and antimicrobial resistance of Salmonella serovars isolated from poultry in Ghana[J]. Epidemiology and Infection, 2016, 144(15): 3288-3299. DOI:10.1017/S0950268816001126 |
| [4] |
Wang Y, Yang BW, Wu Y, et al. Molecular characterization of Salmonella enterica serovar Enteritidis on retail raw poultry in six provinces and two national cities in China[J]. Food Microbiology, 2015, 46: 74-80. DOI:10.1016/j.fm.2014.07.012 |
| [5] |
Deekshit VK, Ballamoole KK, Rai P, et al. Draft genome sequence of multidrug resistant Salmonella enterica serovar Weltevreden isolated from seafood[J]. Journal of Genomics, 2015, 3: 57-58. DOI:10.7150/jgen.11253 |
| [6] |
Li L, Dai XY, Wang Y, et al. RNA-seq-based analysis of drug-resistant Salmonella enterica serovar Typhimurium selectedin vivo and in vitro[J]. PLoS One, 2017, 12(4): e0175234. DOI:10.1371/journal.pone.0175234 |
| [7] |
Duarte TT, Spencer CT. Personalized proteomics: the future of precision medicine[J]. Proteomes, 2016, 4(4): 29. DOI:10.3390/proteomes4040029 |
| [8] |
Peterson AC, Russell JD, Bailey DJ, et al. Parallel reaction monitoring for high resolution and high mass accuracy quantitative, targeted proteomics[J]. Molecular and Cellular Proteomics, 2012, 11(11): 1475-1488. DOI:10.1074/mcp.O112.020131 |
| [9] |
Feng S, Wang Y, Wang BT, et al. The establishment of the drug-resistance inducing model in vivo of Salmonella enterica typhimurium in the Caenorhabditis elegans and dissimilarity analysis of drug-resistance inducing S. typhimurium in vivo and in vitro[J]. Chinese Journal of Veterinary Science, 2014, 34(12): 1926-1930. 冯帅, 汪莹, 王彬婷, 等. 沙门菌体内耐药性诱导模型的建立和体内外诱导耐药菌基因差异分析[J]. 中国兽医学报, 2014, 34(12): 1926-1930. |
| [10] |
Huang YL, Fu Q, Huang DL, et al. Comparative analysis of proteomics at different developmental stages of buffalo testicular seminiferous tubules by TMT-Coupled 2D LC-MS/MS[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(8): 1265-1273. 黄愉淋, 付强, 黄德伦, 等. 利用TMT标记结合2D LC-MS/MS技术分析不同时期水牛睾丸曲精细管差异蛋白质组[J]. 畜牧兽医学报, 2014, 45(8): 1265-1273. |
| [11] |
Piddock LJV, White DG, Gensberg K, et al. Evidence for an efflux pump mediating multiple antibiotic resistance in Salmonella enterica serovar Typhimurium[J]. Antimicrobial Agents and Chemotherapy, 2000, 44(11): 3118-3121. DOI:10.1128/AAC.44.11.3118-3121.2000 |
| [12] |
Chen S, Cui SH, McDermott PF, et al. Contribution of target gene mutations and efflux to decreased susceptibility ofSalmonella enterica serovar Typhimurium to fluoroquinolones and other antimicrobials[J]. Antimicrobial Agents and Chemotherapy, 2007, 51(2): 535-542. DOI:10.1128/AAC.00600-06 |
| [13] |
Blair JMA, Smith HE, Ricci V, et al. Expression of homologous RND efflux pump genes is dependent upon AcrB expression: implications for efflux and virulence inhibitor design[J]. Journal of Antimicrobial Chemotherapy, 2015, 70(2): 424-431. DOI:10.1093/jac/dku380 |
| [14] |
Ricci V, Tzakas P, Buckley A, et al. Ciprofloxacin-resistant Salmonella enterica serovar Typhimurium strains are difficult to select in the absence of AcrB and TolC[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(1): 38-42. DOI:10.1128/AAC.50.1.38-42.2006 |
| [15] |
Szurmant H, Ordal GW. Diversity in chemotaxis mechanisms among the bacteria and archaea[J]. Microbiology and Molecular Biology Reviews, 2004, 68(2): 301-319. DOI:10.1128/MMBR.68.2.301-319.2004 |
| [16] |
Koretke KK, Lupas AN, Warren PV, et al. Evolution of two-component signal transduction[J]. Molecular Biology and Evolution, 2000, 17(12): 1956-1970. DOI:10.1093/oxfordjournals.molbev.a026297 |
| [17] |
Stock AM, Robinson VL, Goudreau PN. Two-component signal transduction[J]. Annual Review of Biochemistry, 2000, 69(1): 183-215. DOI:10.1146/annurev.biochem.69.1.183 |
| [18] |
Li WX, Ali F, Cai QL, et al. Quantitative proteomic analysis reveals that chemotaxis is involved in chlortetracycline resistance of Aeromonas hydrophila[J]. Journal of Proteomics, 2018, 172: 143-151. DOI:10.1016/j.jprot.2017.09.011 |
| [19] |
Shprung T, Peleg A, Rosenfeld Y, et al. Effect of PhoP-PhoQ activation by broad repertoire of antimicrobial peptides on bacterial resistance[J]. Journal of Biological Chemistry, 2012, 287(7): 4544-4551. DOI:10.1074/jbc.M111.278523 |
| [20] |
Bojer MS, Jakobsen H, Struve C, et al. Lack of the RNA chaperone Hfq attenuates pathogenicity of several Escherichia coli pathotypes towards Caenorhabditis elegans[J]. Microbes and Infection, 2012, 14(12): 1034-1039. DOI:10.1016/j.micinf.2012.06.002 |
| [21] |
Ramos CG, Grilo AM, da Costa PJP, et al. MtvR is a global small noncoding regulatory RNA in Burkholderia cenocepacia[J]. Journal of Bacteriology, 2013, 195(16): 3514-3523. DOI:10.1128/JB.00242-13 |
| [22] |
Yu J, Schneiders T. Tigecycline challenge triggers sRNA production in Salmonella enterica serovar Typhimurium[J]. BMC Microbiology, 2012, 12(1): 195. DOI:10.1186/1471-2180-12-195 |
| [23] |
Vogel J, Luisi BF. Hfq and its constellation of RNA[J]. Nature Reviews Microbiology, 2011, 9(8): 578-589. DOI:10.1038/nrmicro2615 |
| [24] |
Nishino K, Yamasaki S, Hayashi-Nishino M, et al. Effect of overexpression of small non-coding DsrA RNA on multidrug efflux in Escherichia coli[J]. Journal of Antimicrobial Chemotherapy, 2011, 66(2): 291-296. DOI:10.1093/jac/dkq420 |
| [25] |
Sittka A, Lucchini S, Papenfort K, et al. Deep sequencing analysis of small noncoding RNA and mRNA targets of the global post-transcriptional regulator, Hfq[J]. PLoS Genetics, 2008, 4(8): e1000163. DOI:10.1371/journal.pgen.1000163 |
| [26] |
Molecular basis of multidrug transport by ABC transporters[J]. Biochimica et Biophysica Acta-proteins and Proteomics, 2009, 1794(5): 725-737
|
| [27] |
Li XR. Effect of Hfq on Klebsiella pneumoniae resistance and virtual screening of Hfq inhibitors[D]. Changchun: Doctoral Dissertation of Jilin University, 2016 (in Chinese) 李欣燃. Hfq对肺炎克雷伯菌耐药性影响和机制初探及Hfq抑制剂虚拟筛选[D]. 长春: 吉林大学博士学位论文, 2016 |
| [28] |
Sampson TR, Napier BA, Schroeder MR, et a1. A CRISPR cas system enhances envelope integrity mediating antibiotic resistance and inflammasome evasion[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 11. |
| [29] |
Dandekar AA, Greenberg EP. Microbiology: Plan B for quorum sensing[J]. Natural Chemical Biology, 2013, 9(5): 292-293. DOI:10.1038/nchembio.1233 |
| [30] |
Pumbwe L, Skilbeck CA, Wexler HM. Presence of quorum-sensing systems associated with multidrug resistance and biofilm formation in Bacteroides fragilis[J]. Microbial Ecology, 2008, 56(3): 412-419. DOI:10.1007/s00248-007-9358-3 |
| [31] |
Dreier J, Ruggerone P. Interaction of antibacterial compounds with RND efflux pumps inPseudomonas aeruginosa[J]. Front Microbiol, 2015, 6: 660. |
 2018, Vol. 45
2018, Vol. 45