扩展功能
文章信息
- 盖婧璇, 韩铁生, 刘文秀, 武策, 汪志军, 苟丽霞
- GE Jing-Xuan, HAN Tie-Sheng, LIU Wen-Xiu, WU Ce, WANG Zhi-Jun, GOU Li-Xia
- 卡西霉素生物合成调控基因calR2的功能
- Function of the regulatory gene calR2 involved in calcimycin biosynthesis
- 微生物学通报, 2018, 45(7): 1518-1526
- Microbiology China, 2018, 45(7): 1518-1526
- DOI: 10.13344/j.microbiol.china.180127
-
文章历史
- 收稿日期: 2018-02-09
- 接受日期: 2018-04-20
- 网络首发日期(www.cnki.net): 2018-05-09
2. 华北理工大学公共卫生学院 河北省煤矿卫生与安全重点实验室 河北 唐山 063210;
3. 上海交通大学生命科学技术学院 微生物代谢国家重点实验室 上海 200240
2. Hebei Province Key Laboratory of Occupational Health and Safety for Coal Industry, School of Public Health, North China University of Science and Technology, Tangshan, Hebei 063210, China;
3. State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China
卡西霉素(Calcimycin)是由教酒链霉菌Streptomyces chartreusis NRRL3882产生的聚醚类抗生素,化学结构包括吡咯环、螺旋环及含有取代基团的苯并噁唑环[1](图 1A)。卡西霉素独特的分子结构赋予其多样的生物活性,最显著的特点是能特异螯合二价阳离子,被广泛应用于增加细胞内游离二价金属离子浓度等细胞生物属性的研究中[2]。此外,卡西霉素还具有抑菌活性,而且是哺乳动物细胞氧化磷酸化的解偶联剂及ATP酶的抑制剂,近几年研究表明其具有诱导哺乳动物精子顶体反应、辅助卵子激活等功能[3-4]。鉴于卡西霉素具有广泛的生物活性,而目前市场上销售的卡西霉素及其衍生物主要以化学及半化学合成为主,成本较高,因此在了解卡西霉素生物合成途径基础上,提高卡西霉素产生菌的产素水平具有重要的实际应用价值。

|
| 图 1 卡西霉素化学结构(A)及生物合成基因簇(B) Figure 1 Structure (A) and biosynthetic gene cluster (B) of calcimycin |
|
|
前期工作中,我们已从教酒链霉菌NRRL3882总DNA中获得了卡西霉素的生物合成基因簇(图 1B),鉴定了卡西霉素生物合成基因簇中部分结构基因的功能。通过对基因簇上的calB1及calB3基因进行敲除,以及突变株体外喂养实验证实这2个基因参与苯并噁唑环结构单元的前体物3-羟基邻氨基苯甲酸的生物合成[5]。对后修饰基因calM进行体内外功能研究,证实calM负责苯并噁唑环上的N-甲基转移功能[6]。对calD基因进行敲除、回补及突变株代谢产物分析表明其参与苯并噁唑环结构单元3位羟基上的氧化反应[7]。本研究在此基础上,进一步对卡西霉素生物合成基因簇上的调控基因进行功能研究,揭示调控基因对Calcimycin产量的影响。
根据NCBI提供的检索引擎,对卡西霉素基因簇上的基因序列进行比对分析,发现卡西霉素生物合成基因簇上共有3个可能的转录调控基因:calR1 (TylR家族)、calR2 (LuxR家族)和calR3 (TetR家族)。近期通过体内遗传学实验及体外反应已证实calR3在卡西霉素的生物合成中起负调控作用[8]。本研究选取calR2为研究对象,生物信息学比对分析发现CalR2蛋白属于LuxR家族同源蛋白,N端具有ATP结合位点,C端具有保守的螺旋-转角-螺旋DNA结合结构域。随着基因组测序技术的迅速发展,在许多细菌中都发现了LuxR家族同源蛋白,但大部分该类蛋白的功能尚未见报道。我们通过对calR2基因进行中断、回补,对突变菌株及回补菌株的代谢产物及突变菌株和野生菌株的基因转录水平进行比较分析,揭示了calR2基因对Calcimycin生物合成的影响,为Calcimycin生物合成调控网络的研究提供了基础,为利用分子生物技术手段从基因水平上对Calcimycin产生菌进行定向遗传育种以提高抗生素产量提供理论依据。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒实验所用的菌株和质粒见表 1,Escherichia coli BW25113/pIJ790用于基因中断,ET12567/pUZ8002用于大肠杆菌与链霉菌两亲本接合转移,pJTU2170由pIB139衍生而来(用氨苄霉素和卡那霉素抗性基因取代阿泊拉霉素抗性基因),用于突变菌株的回补。
| 菌株/质粒 Strain or plasmid |
特征 Characteristics |
来源 Reference or source |
| Strains | ||
| Streptomyces chartreusis strains | ||
| NRRL3882 | Calcimycin producer, wild type | NRRL |
| GLX25 (ΔcalR2) | calR2-deletion mutant | This work |
| GLX28 (ΔcalR2:calR2) | ΔcalR2 complementation strain | This work |
| Escherichia coli strains | ||
| DH10B | F- recA lacZ ΔM15 | Invitrogen |
| ET12567(pUZ8002) | CmL, Kan, dam dcm hsdS Tra+ CmL | [9] |
| Plasmids | ||
| pJTU2170 | aac(3)IV, lacz, reppuc, attФC31, oriT | [10] |
| p16F9 | CmL | [1] |
| pJTU3790 | p16F9 derived plasmid carrying an apramycin resistance gene and a defective calR2 | This work |
| pJTU2170-R2 | pJTU2170 derived plasmid carrying calR2 for expression in Streptomyces | This work |
LB (Luria-Bertani)培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,氯化钠5.0,pH 7.0;LA培养基:在LB培养基中加入琼脂粉,使其终浓度为1.5%,LA及LB培养基用于大肠杆菌的培养;SFM固体培养基[6]用于教酒链霉菌孢子收集及接合转移;SFM液体培养基[6]用于教酒链霉菌的发酵;TSBY液体培养基[6]用于链霉菌总DNA及RNA的提取。
rTaq,TaKaRa公司;KOD plus,TOYOBO公司;限制性内切酶,New England Biolabs公司;DNA胶回收试剂盒、RNA提取试剂盒、FastQuant RT Kit及SuperReal qPCR PreMix (SYBR Green)试剂盒,天根生化科技有限公司;卡那霉素(50 mg/mL)、氨苄霉素(100 mg/mL)、氯霉素(25 mg/mL)、阿泊拉霉素(30 mg/mL)及乙酸乙酯,天津润泰生物公司;卡西霉素标准品及甲醇,Sigma-Aldrich公司。HPLC、LC/MS及ZORBAX SB-C18 (2.1 mm×150 mm,3.5 μm),安捷伦科技有限公司;ScanDrop200,德国耶拿分析仪器股份公司;ABI7900HT,美国应用生物系统公司。
1.2 方法 1.2.1 calR2基因的生物信息学分析CalR2的结构域及其功能预测采用NCBI的CDD工具[11];同源序列比对采用Clustal Omega工具[12],比对后将各同源蛋白相关同源区域的保守氨基酸标记在下方。
1.2.2 calR2基因中断突变菌株的构建通过PCR-targeting的方法,选用从pIJ773载体中双酶切(Hind Ⅲ和EcoR I)并回收的含有Apramycin抗性基因及oriT的片段为模板设计引物R2-F1和R2-F2 (表 2),扩增用于替换calR2基因的PCR cassette序列。将PCR扩增回收的片段,利用BW25113/pIJ790的重组系统与携带有calR2基因的柯斯质粒p16F9,通过同源臂交换获得含有Apramycin抗性基因及oriT且同时calR2基因中断的重组质粒pJTU3790,最后将含有重组质粒pJTU3790的ET12567/pUZ8002与野生型教酒链霉菌NRRL3882进行两亲本接合转移,30 ℃、220 r/min培养16 h后用阿泊拉霉素及萘啶酮酸进行覆盖,继续培养4 d后筛选具有阿泊拉霉素抗性的接合转移子,将筛选的接合子扩大培养后提取基因组DNA,利用验证引物R2-F3和R2-F4 (表 2)对其进行PCR验证。
| 引物 Primers |
序列 Primers sequence (5′→3′) |
用途 Usage |
| R2-F1 | GTGACGCATCGGCCTTCGGCCCAGGAGCGCGCGGTGTCGATTCCGGGGATCCGTCGACC | Disruption of calR2 |
| R2-F2 | CGTCCGTTCCGCGGTCCTGCGGGCATCGGCCGCGCCCGGTGTAGGCTGGAGCTGCTTC | |
| R2-F3 | CGCCGACTGCCTTTCCG | Confirmation of calR2 disruption in ΔcalR2 mutant |
| R2-F4 | CGATGAGCCGTCCCCGTC | |
| R2-F5 | GGAGTTCCATATGTTGGAACGTCGGGGGACGCACA | Complementation of GLX25 (ΔcalR2) |
| R2-F6 | CCGGAATTCAGGAGATACTCGCCGACATCGTCAGA | |
| RT16S-F1 | CTCCTCAGCGTCAGTATCGGC | internal reference |
| RT16S-F2 | CGGGCAGGCTAGAGTTCGGTAG | |
| RTA1-F1 | ACCCGACCGCCAGGAGATT | calA1 RT-PCR |
| RTA1-F2 | CAGCAGACGCCCGAAGGAG | |
| RTR1-F1 | TGGACGAGGTGCTGCACGAT | calR1 RT-PCR |
| RTR1-F2 | TGGATCAGCCCGTGGAGGTA | |
| RTM-F1 | ACCCTATGAACAGCCTGCCG | calM RT-PCR |
| RTM-F2 | CGTCTACACCAAGGATCTGGAGC | |
| RTR2-F1 | GTTCTCCTCCCTCAGCGAACTG | calR2 RT-PCR |
| RTR2-F2 | AGTACCGCAGCCGCCGTA | |
| RTB4-F1 | CGTCGTGTACTTCCTGCCTACCT | calB4 RT-PCR |
| RTB4-F2 | TCCAGTGCGCCGTTCGTG | |
| RTN1-F1 | CGTTCACCGACTGCCACGT | calN1 RT-PCR |
| RTN1-F2 | GCTCCATCAACCCGAGGTAGAC | |
| RTU2-F1 | GACGCGGGCAAGATCAACAC | calU2 RT-PCR |
| RTU2-F2 | CCCGAGAAGGAAGTCCAGGT | |
| RTA3-F1 | ACACCCAGTGCACCTCCTCGAT | calA3 RT-PCR |
| RTA3-F2 | TCGGCAACGACATGACGCAG | |
| RTR3-F1 | CGAGCATCCGGGGAAGATCA | calR3 RT-PCR |
| RTR3-F2 | TCGATCGTCGTCGCCTCGTA | |
| RTA5-F1 | TGTCGGGGAAGTCGTATGTGC | calA5 RT-PCR |
| RTA5-F2 | ATCGCCAACGCCTCACGC | |
| RTU3-F1 | CAGTCCCGCCATCTGCTGAT | calU3 RT-PCR |
| RTU3-F2 | GCTTCGCCCAGTCGTACAGA | |
| RTG-F1 | GTGCGTGGTCGACGAGGAAGAAGT | calG RT-PCR |
| RTG-F2 | AAGGCGAGCCATGACGAGGTAC |
使用高保真KOD-plus聚合酶,以教酒链霉菌NRRL3882的总DNA为模板,使用引物R2-F5和R2-F6 (表 2)扩增完整的calR2基因,通过EcoR I及Nde I双酶切后克隆到由pIB139衍生来的携带有红霉素强启动子的整合型载体pJTU2170 (氨苄霉素和卡那霉素抗性)上,获得回补质粒pJTU2170-R2。然后通过接合转移的方法将回补质粒引入到calR2基因缺失的突变株中,进一步利用卡那霉素抗性筛选回补菌株,并通过引物PCR验证回补菌株的正确性。
1.2.4 菌株发酵及代谢产物分析将20%甘油保存的野生菌株、突变菌株及回补菌株的孢子按1:1 000的比例接种于TSBY培养基中,30 ℃、220 r/min培养3 d,之后将种子液按1:50的比例转接于100 mL液体SFM培养基中扩大培养,30 ℃、220 r/min发酵培养7 d。用2倍体积的乙酸乙酯对所得发酵液进行萃取,之后4 000 r/min常温离心5 min,将所得上清液进行旋转蒸发,所得沉淀物用1 mL甲醇溶液进行溶解,过滤后用于HPLC或LC/MS检测分析。使用Agilent 1100 System及ZORBAX SB-C18分析柱对样品进行检测。流动相A相选用含有0.1%的三氟乙酸(TFA)的水溶液,B相为甲醇(70%-100%),流速为0.3 mL/min,检测波长为280 nm。
1.2.5 链霉菌总RNA的提取和反转录将野生菌株及突变菌株在TSBY培养基中30 ℃、220 r/min培养4 d,分别吸取1 mL菌液,12 000 r/min离心1min弃上清,然后按照RNA提取试剂盒操作说明提取RNA,并根据说明书用DNase I对所获得RNA进行去除基因组DNA处理, 最后用ScanDrop200及琼脂糖凝胶对所得RNA的浓度和质量进行检测,并设计引物RT16S-F1及RT16S-F2 (表 2)扩增16S rRNA基因作为内参基因来检测DNA是否消化完全。确认所得RNA没有DNA残留后,根据FastQuant RT Kit操作说明书,将RNA (2 μg)反转录为cDNA。
1.2.6 野生菌株与突变菌株中卡西霉素生物合成相关基因转录水平检测采用荧光定量PCR (Real-Time PCR)比较野生菌株及突变菌株的Calcimycin生物合成相关基因的转录水平差异,以反转录的cDNA为模板,16S rRNA基因为内参,根据公式ΔΔCt=(Ct目标基因–Ct内参基因)样品组–(Ct目标基因–Ct内参基因)参照组,对Ct值进行比较,计算出目标基因的相对变化量。根据基因簇上基因的功能及位置,随机筛选了12个基因检测其在突变株中的转录水平较野生菌株对应基因的转录差异,目标基因及内参基因引物见表 2。Real-time PCR反应根据SuperReal qPCR PreMix (SYBR Green)试剂盒配制20 μL反应体系:80 ng模板cDNA,300 nmol/L引物;10 μL SuperReal SYBR Green PreMix,参照试剂盒说明书的两步法进行:95 ℃ 15 min;95 ℃ 10 s,60 ℃ 30 s,40个循环。
2 结果与分析 2.1 CalR2的生物信息学分析通过NCBI的BLASTp序列比对分析表明CalR2属于LAL (Large ATP-binding regulators of the LuxR family)转录调控家族,该家族为LuxR转录调控家族的一个分支。CalR2由1 040个氨基酸组成,与麦芽糖操纵子调控蛋白MalT家族有较高的一致性(一致性约为29%,图 2A)。通过与其它链霉菌来源的LAL家族蛋白序列进行比对分析表明,CalR2的N端具有该家族的ATP/GTP结合结构域Walker A和Walker B,C端具有LuxR家族典型的HTH (helix-turn-helix) DNA结合结构域(图 2B,保守氨基酸如图底所示),说明CalR2对卡西霉素的生物合成起调节作用。

|
| 图 2 LAL家族CalR2蛋白序列同源比对 Figure 2 In silico analysis of LAL regulator CalR2 注:A:CalR2结构域预测;B:CalR2与其它LAL家族蛋白的保守结构域同源比对. Walker A、B:NTP结合基序;HTH:LuxR家族HTH-DNA结合结构域. MonH来源于S. cinnamonensis;AveR来源于S. avermitilis K139;PikD来源于S. venezuelae;SlnR来源于S. albus XM211;GdmRI和GdmRII来源于S. hygroscopicus 17997;OlmRI和OlmRII来源于S. avermitilis. Note: A: Conserved functional domain prediction of CalR2; B: Alignment of the conserved motifs of CalR2 with other LAL regulators from various Streptomyces. Walker A, B: NTP binding motifs; HTH: Lux R-family HTH DNA-binding domains. MonH from S. cinnamonensis; AveR from S. avermitilis K139; PikD from S. venezuelae; SlnR from S. albus XM211; GdmRI and GdmRII from S. hygroscopicus 17997; OlmRI and OlmRII from S. avermitilis. |
|
|
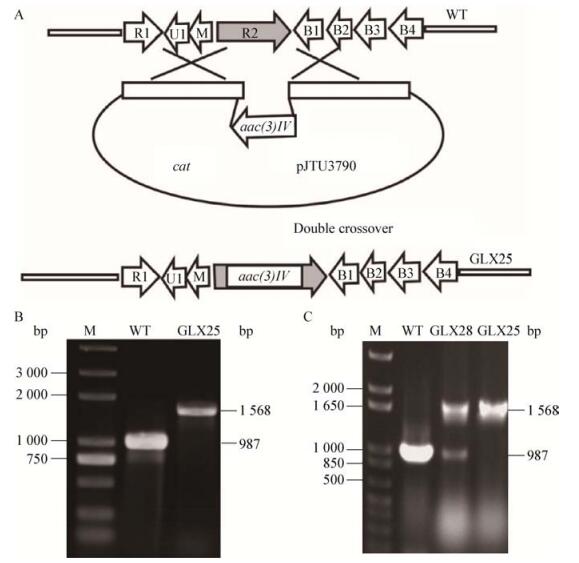
为了确定calR2基因在卡西霉素生物合成中的功能,通过PCR介导的REDIRECTR Technology技术对卡西霉素产生菌染色体上的calR2基因进行中断,通过同源臂交换构建calR2基因被阿泊拉霉素抗性基因置换的重组质粒pJTU3790 (图 3A)。通过两亲本接合转移将pJTU3790导入教酒链霉菌NRRL3882中,通过同源重组构建calR2基因中断突变菌株。提取野生菌株及突变菌株的基因组DNA进行PCR扩增验证,野生菌株的PCR片段大小为987 bp,对应的突变株GLX25(ΔcalR2)的扩增片段大小为1 568 bp (图 3B),说明突变菌株GLX25为calR2基因中断的双交换突变株。
|
| 图 3 calR2基因中断突变菌株的构建及验证 Figure 3 Construction and verification of the calR2 disruption mutant 注:A:通过基因置换构建calR2基因被阿泊拉霉素抗性基因置换的calR2基因中断突变菌株GLX25;B:calR2基因中断突变株GLX25的PCR验证;C:calR2基因回补菌株GLX28的PCR验证. Note: A: A scheme showing the disruption of calR2 by the apramycin resistance gene cassette aac(3)IV to generate the mutant GLX25; B: The ΔcalR2 mutant GLX25 was confirmed by PCR amplification; C: The calR2 gene complementation strain GLX28 was confirmed by PCR amplification. |
|
|
将构建的含有完整calR2基因的整合型回补质粒pJTU2170-R2通过接合转移的方法导入到突变菌株GLX25 (ΔcalR2)中获得回补菌株GLX28 (ΔcalR2:calR2),所得回补菌株则应同时含有被阿泊拉霉素抗性基因片段中断的calR2基因及整合型回补质粒pJTU2170-R2携带的完整calR2基因,因此以回补菌株GLX28所提取的DNA为模板,PCR扩增验证得到两条带,包括1 568 bp大小的重组条带及987 bp的野生条带(图 3C)。
2.3 野生菌株、突变菌株GLX25与回补菌株GLX28的代谢产物分析将野生菌株、突变菌株GLX25与回补菌株GLX28摇瓶发酵培养7 d,用乙酸乙酯对发酵液进行初步萃取,用HPLC对代谢产物进行分析。用卡西霉素标准品为阳性对照,HPLC结果显示(图 4),calR2基因中断的突变菌株GLX25丧失产生卡西霉素的能力,暗示calR2基因在卡西霉素的生物合成过程中起正调控的功能。对突变株GLX25进行calR2基因回补后,回补菌株GLX28恢复产生卡西霉素的能力(约为野生型产量的78%),进一步说明calR2基因在卡西霉素生物合成中的正调控作用。

|
| 图 4 HPLC分析野生菌株、calR2基因中断突变株GLX25及回补菌株GLX28的代谢产物 Figure 4 HPLC analysis of the fermentation extracts of wild-type, GLX25 (ΔcalR2) and GLX28 (ΔcalR2:calR2) 注:卡西霉素(Calcimycin)标准品做参照. Note: Calcimycin standard as control. |
|
|
为了分析calR2基因对卡西霉素生物合成基因簇上基因的转录水平影响,提取处于平台期早期(96 h) [8]的突变菌株及野生菌株的总RNA,根据基因功能及位置不同,选择了12个基因(calA1、calR1、calM、calR2、calB4、calN1、calU2、calA3、calT、calR3、calA5及calG),测定其在突变菌株中相对于野生菌株的转录差异。结果如图 5所示,突变菌株内与卡西霉素结构单元螺旋环合成相关的calA1、calA3及calA5基因,苯并噁唑环结构单元合成相关的calB4基因,后修饰基因calM及抗性基因calT的转录水平显著降低,未知功能基因calU2以及calR2基因自身的转录水平也略有下降。相比之下,调控基因calR1、calR3和吡咯环结构单元合成相关基因calN1及功能未知的Ⅱ型硫酯酶基因calG的转录水平较野生菌株相比变化不明显。这些重要骨架基因转录水平的显著降低与突变菌株GLX25中卡西霉素产生能力的丧失相一致,进一步说明calR2在卡西霉素生物合成过程中起正调控作用。

|
| 图 5 野生型菌株与GLX25 (ΔcalR2)的卡西霉素生物合成相关基因转录水平差异检测 Figure 5 The transcriptional difference detection between the wild type NRRL 3882 and the calR2 disrupted strain GLX25 |
|
|
LuxR家族蛋白是一类起着重要生理功能的转录调控蛋白,广泛存在于多种细菌中,主要参与细菌的群体感应机制、细菌的毒力因子调控及微生物次级代谢产物的调控等多种生物学功能[13-15]。生物信息学分析表明CalR2的C端具有LuxR家族典型的HTH DNA结合结构域,暗示其可能具有LuxR家族成员相似的转录调控功能。LuxR家族蛋白作为正调控因子调控抗生素的生物合成已在其它链霉菌中被证实,例如将S. cinnamonensis菌株的LuxR家族同源基因monH敲除后,突变菌株失去产生莫能霉素(Monensin)的能力[16];同样,将S. avermitilis菌株的LuxR家族同源基因olmRI及olmRII敲除后,突变株不再产生寡霉素(Oligomycin)[17]。
本研究通过PCR-targeting的方法,构建了calR2中断的突变菌株GLX25,HPLC分析结果显示calR2基因中断的突变株丧失产生卡西霉素的能力,通过体内基因回补构建的回补菌株GLX28恢复产生卡西霉素的能力;荧光定量RT-PCR实验也证实calR2基因中断的突变菌株中,卡西霉素合成相关的结构基因转录水平显著降低。这说明CalR2在卡西霉素生物合成中起正调控作用。
LuxR家族蛋白普遍接受某种信号分子作为配体,通过与配体结合改变蛋白构象,调节其结合DNA的强度,进而调控下游基因的表达。典型例子如Vibrio fischeri的LuxR蛋白能够与高丝氨酸内酯(AHL)结合,激活下游基因表达[18],以及Streptomyces coelicolor中的ScbR蛋白与链霉菌群体感应分子γ-丁酸内酯(GBL)结合调控抗生素基因表达等[19]。CalR2蛋白的中间区域功能尚属未知,可能含有与信号分子结合的功能域,因此进一步筛选CalR2的配体分子,并测定其DNA识别位点等,对进一步阐明CalR2的调控功能及卡西霉素的调控网络,并最终确定提高抗生素产量的生物合成调控策略将有重要意义。
| [1] |
Wu QL, Liang JD, Lin SJ, et al. Characterization of the biosynthesis gene cluster for the pyrrole polyether antibiotic calcimycin (A23187) in Streptomyces chartreusis NRRL 3882[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(3): 974-982. DOI:10.1128/AAC.01130-10 |
| [2] |
Reed PW. Effects of divalent cation ionophore A23187 on potassium permeability of rat erythrocytes[J]. Journal of Biological Chemistry, 1976, 251(11): 3489-3494. |
| [3] |
Tateno H, Krapf D, Hino T, et al. Ca2+ ionophore A23187 can make mouse spermatozoa capable of fertilizing in vitro without activation of cAMP-dependent phosphorylation pathways[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(46): 18543-18548. DOI:10.1073/pnas.1317113110 |
| [4] |
Bloemberg D, Quadrilatero J. Caspase activity and apoptotic signaling in proliferating C2C12 cells following cisplatin or A23187 exposure[J]. Data Brief, 2016, 7: 1024-1030. DOI:10.1016/j.dib.2016.03.032 |
| [5] |
Gou LX, Wu QL, Lin SJ, et al. Mutasynthesis of pyrrole spiroketal compound using calcimycin 3-hydroxy anthranilic acid biosynthetic mutant[J]. Applied Microbiology and Biotechnology, 2013, 97(18): 8183-8191. DOI:10.1007/s00253-013-4882-1 |
| [6] |
Wu QL, Gou LX, Lin SJ, et al. Characterization of the N-methyltransferase CalM involved in calcimycin biosynthesis by Streptomyces chartreusis NRRL 3882[J]. Biochimie, 2013, 95(7): 1487-1493. DOI:10.1016/j.biochi.2013.03.014 |
| [7] |
Li YL, Gou LX, Wu QL, et al. Functional analysis of calD involved in calcimycin postsynthetic modification by Streptomyces chartreusis NRRL 3882[J]. Microbiology China, 2015, 42(1): 148-156. 李园莉, 苟丽霞, 吴秋林, 等. 教酒链霉菌NRRL 3882中钙霉素生物合成后修饰基因calD的功能分析[J]. 微生物学通报, 2015, 42(1): 148-156. |
| [8] |
Gou LX, Han TS, Wang XX, et al. A novel TetR family transcriptional regulator, CalR3, negatively controls calcimycin biosynthesis in Streptomyces chartreusis NRRL 3882[J]. Frontiers in Microbiology, 2017, 8: 2371. DOI:10.3389/fmicb.2017.02371 |
| [9] |
Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces genetics[J]. Norwich, United Kingdom: The John Innes Foundation, 2000, 25. |
| [10] |
Huang TT, Wang YM, Yin J, et al. Identification and characterization of the pyridomycin biosynthetic gene cluster of Streptomyces pyridomyceticus NRRL B-2517[J]. Journal of Biological Chemistry, 2011, 286(23): 20648-20657. DOI:10.1074/jbc.M110.180000 |
| [11] |
Marchler-Bauer A, Derbyshire MK, Gonzales NR, et al. CDD: NCBI's conserved domain database[J]. Nucleic Acids Research, 2015, 43(Database issue): D222-D226. |
| [12] |
Sievers F, Higgins DG. Clustal Omega for making accurate alignments of many protein sequences[J]. Protein Science, 2018, 27(1): 135-145. DOI:10.1002/pro.3290 |
| [13] |
Nasser W, Reverchon S. New insights into the regulatory mechanisms of the LuxR family of quorum sensing regulators[J]. Analytical and Bioanalytical Chemistry, 2007, 387(2): 381-390. DOI:10.1007/s00216-006-0702-0 |
| [14] |
Smith RS, Iglewski BH. P. aeruginosa quorum-sensing systems and virulence[J]. Current Opinion in Microbiology, 2003, 6(1): 56-60. DOI:10.1016/S1369-5274(03)00008-0 |
| [15] |
Li QL, Wang LF, Xie YY, et al. SsaA, a member of a novel class of transcriptional regulators, controls sansanmycin production in Streptomyces sp. strain SS through a feedback mechanism[J]. Journal of Bacteriology, 2013, 195(10): 2232-2243. DOI:10.1128/JB.00054-13 |
| [16] |
Tang ZK, Li XM, Pang AP, et al. Characterization of three pathway-specific regulators for high production of monensin in Streptomyces cinnamonensis[J]. Applied Microbiology and Biotechnology, 2017, 101(15): 6083-6097. DOI:10.1007/s00253-017-8353-y |
| [17] |
Yu Q, Bai LQ, Zhou XF, et al. Inactivation of the positive LuxR-type oligomycin biosynthesis regulators OlmRI and OlmRII increases avermectin production in Streptomyces avermitilis[J]. Chinese Science Bulletin, 2012, 57(8): 869-876. DOI:10.1007/s11434-011-4865-5 |
| [18] |
Fuqua C, Parsek MR, Greenberg EP. Regulation of gene expression by cell-to-cell communication: acyl-homoserine lactone quorum sensing[J]. Annual Review of Genetics, 2001, 35(1): 439-468. DOI:10.1146/annurev.genet.35.102401.090913 |
| [19] |
Takano E, Chakraburtty R, Nihira T, et al. A complex role for the gamma-butyrolactone SCB1 in regulating antibiotic production in Streptomyces coelicolor A3(2)[J]. Molecular Microbiology, 2001, 41(5): 1015-1028. |
 2018, Vol. 45
2018, Vol. 45