扩展功能
文章信息
- 黄鹏伟, 龚大春, 戴传超, 梅艳珍
- HUANG Peng-Wei, GONG Da-Chun, DAI Chuan-Chao, MEI Yan-Zhen
- 基因组装技术在合成生物学中的应用
- Application of gene assemblies in synthetic biology
- 微生物学通报, 2018, 45(6): 1358-1368
- Microbiology China, 2018, 45(6): 1358-1368
- DOI: 10.13344/j.microbiol.china.170548
-
文章历史
- 收稿日期: 2017-07-17
- 接受日期: 2018-03-28
- 网络首发日期(www.cnki.net): 2018-04-11
2. 三峡大学生物与制药学院 湖北省天然产物研究与利用重点实验室 湖北 宜昌 443002
2. Hubei Key Laboratory of Natural Products Research & Development, College of Biological & Pharmaceutical, China Three Gorges University, Yichang, Hubei 443002, China
合成生物学, 即综合应用科学、技术和工程学来促进和加速生物体中遗传物质的设计、改造和修饰的一门学科。该技术突破自然进化的限制, 以“人工设计与基因组编辑”为核心, 从工程学角度设计表达元件或模块, 可用于生产特定的目标产物。通过设计合理的元件或模块对现有生物体系进行改造和优化, 从而得到人工可控的生物体系。2015年12月底以合成生物学发展战略为主题的第552次香山科学会议上, 30多位专家研讨如何将“可以像组装机器一样组配生物”的设想变为现实。合成生物学技术已成为绿色制造的核心技术, 广泛应用于生物医药、生物能源、大宗化学品及食品添加剂等[1]。与传统的代谢工程和化学合成相比, 有如下优势:(1)合成生物学可以激活或优化天然表达体系中沉默的基因或基因簇, 从而生产生物活性物质[2]; (2)可将动植物源的代谢途径构建到微生物体系中, 最终实现目标产物的异源表达[1]; (3)可人工设计非天然存在的代谢途径, 进而合成所需目标产物或者实现特定的功能等[1]。基因组装技术是合成生物学技术领域近年来发展起来的关键技术之一, 本文旨在阐述基因组装技术在合成生物学应用中的设计原理、关键策略及其应用举例。
1 基于同源重组的体内DNA assembler技术的设计原理与关键策略DNA assembler技术的核心是去除目的基因前原有的受到复杂代谢调控的启动子, 替换成研究透彻且可诱导的组成型启动子[3]。使用OE-PCR[4]将启动子和目的基因连接成表达盒, 再将这些表达盒以及含有筛选标记的片段共转化进入酿酒酵母(Saccharomyces cerevisiae)体内, S. cerevisiae识别这些DNA片段在衔接处的同源序列, 通过同源重组机制将其组装成一个完整的基因通路[5]。
DNA assembler技术主要包含如下几部分:目标物质相关基因簇的分析, 启动子的选择, 表达模块的组装, 辅助模块的设计及一步转化等。如图 1所示, DNA assembler技术设计的简要过程原理如下:(1)基于测序的基因组或宏基因组数据分析, 推测可能的生物合成基因簇及目的产物; (2)选择一系列合适的启动子; (3) PCR扩增或化学合成相应的DNA片段, 采用OE-PCR将不同的启动子和相应的结构基因组装成表达模块; (4)将表达盒与辅助模块共转化S. cerevisiae, 组装成环化的表达载体; (5)在S. cerevisiae或其他宿主体内异源表达目标化合物; (6)目标产物的检测及分析[2] (图 1)。
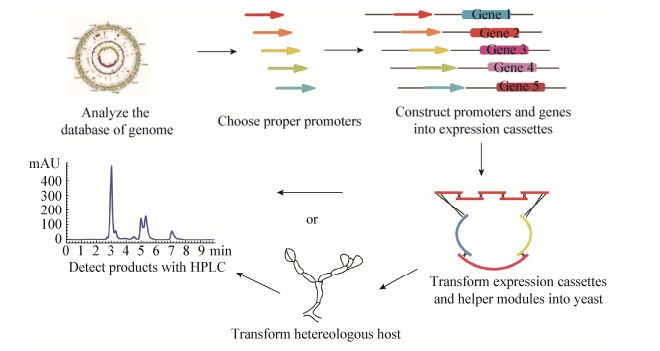
|
| 图 1 沉默目标产物生物合成基因簇的激活表达过程 Figure 1 The workflow of the activation of cryptic gene clusters for target products |
|
|
基于微生物基因组或宏基因组测序数据分析, 越来越多的次级代谢产物生物合成基因簇被发现或预测。然而, 大多数次级代谢产物生物合成受到负调控, 目标产物不表达或表达量很低。基因簇分析相关分析工具的发展, 为发现新型次级代谢产物及激活沉默的次级代谢产物生物合成基因簇提供了理论基础。通常用于挖掘基因组数据的方法有:基因组分析常用的注释工具NR (NCBI的一个综合的蛋白注释数据库)、COG (NCBI的注释同源蛋白的数据库)、GO (基因本体联合会建立的描述基因产物功能的数据库)、SwissPlot (蛋白高级结构预测及同源建模数据库)等, 还可利用数据库如KEGG (可预测代谢通路和蛋白分子互作)、antiSMASH (可预测细菌、真菌和植物体内生物合成基因簇)等对基因组数据进行分析, 预测出可能的生物合成基因簇, 选定合适的次级代谢产物。
Mei等[6-7]利用比较基因组学将一株莓实假单胞菌(Pseudomonas fragi)与其他3株荧光假单胞菌、恶臭假单胞菌、铜绿假单胞菌进行了比较研究, 发现了6个新的次级代谢生物合成基因簇, 同时发现该菌具有不同的信号转导系统, 揭示了其对环境响应的机制。这些数据为相关莓实假单胞菌次级代谢产物生物合成基因簇或基因功能的研究奠定了坚实的基础。
1.2 启动子的选择启动子通常决定基因的表达活性, 影响目标产物的产量。直接将一个完整的基因簇从野生宿主转入异源宿主体内时由于启动子无法被识别导致表达失败的例子十分常见[8]。常见的策略是将基因前的启动子置换成研究透彻的启动子或适合于该宿主的强启动子[9]。启动子的选择与应用原则是:(1)选择在目标表达宿主体内的强启动子, 可以从其内源性看家基因的潜在启动子区入手; (2)选择其他和目标表达宿主紧密联系的物种相应的强启动子; (3)将启动子插入各基因CDS区上游; (4)启动子的类型有诱导型和组成型, 一般选择组成型启动子。特殊情况下, 可选择诱导型启动子, 如终产物或者生物合成中间物对表达宿主有毒性时, 可添加外源诱导剂或改变培养条件, 从而控制基因的表达。该方法可有效降低异源表达宿主被过量生产的次级代谢产物致死的危险[3]。蒋钰瑶等[9]综述了大肠杆菌高效表达载体pHsh的构建与应用, pHsh及其衍生质粒是近年发展起来的新型大肠杆菌表达载体, 其通过热激启动子调控外源基因表达, 仅需改变培养温度即可控制相关基因的表达, 具有易操作和成本低廉等特点。
1.3 表达模块的组装表达模块包含启动子、结构基因和终止子。其组装过程包括:启动子的克隆、目标基因(簇)的克隆、启动子与目标基因(簇)的连接等。首先从基因组中克隆出DNA片段, 然后利用OE-PCR将启动子与目标基因(簇)连接成独立的表达模块, 这是关键步骤之一, 避免了重复的酶切酶连操作。OE-PCR采用具有互补末端的引物, 通过重叠区退火形成互补双链将不同来源的扩增片段连接起来, 最后利用两端引物扩增产生完整的融合双链DNA[10-11], 过程如图 2所示。在第一轮PCR反应中, 分别设计引物独立扩增相应片段。引物F1R的前20 bp和F2F的前20 bp反向互补配对, F2R和F3F也是如此, 因此, 片段A-3′端和片段B的5′-端完全相同, 同理, 片段B-3′端和片段C的5′-端也完全相同。在第二轮PCR反应中, 分别加入片段A、片段B和片段C使融合成一条全长的嵌合DNA片段即一个完整独立的表达模块, 最后再用两侧的引物F1F和F3R扩增出大量的全长融合DNA片段[12-13]。
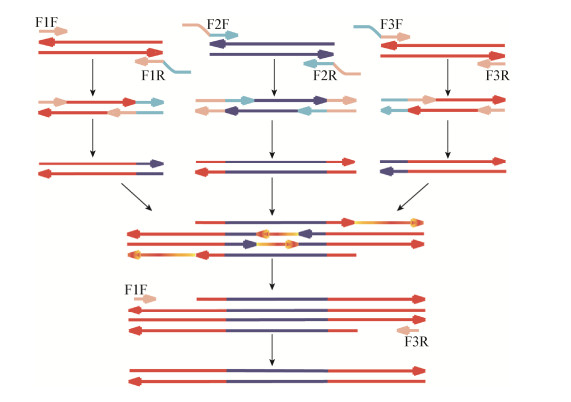
|
| 图 2 OE-PCR技术原理 Figure 2 The principle of OE-PCR technique |
|
|
重叠PCR应用原则或要求:(1)引物长度约为50 bp; (2)引物位于2个相邻片段的衔接处; (3)引物退火温度一般为58 ℃; (4)适用于0.5-5 kb的DNA片段的组装。
1.4 辅助模块的设计辅助模块是在S. cerevisiae (DNA组装宿主)、大肠杆菌(Escherichia coli) (DNA富集宿主)和目标表达宿主体内维持DNA稳定和DNA复制的遗传元件, 分别克隆自相应的载体[3]。具有互补末端的PCR引物使相邻片段间产生重叠, 这些片段共转化进入S. cerevisiae后通过同源重组组装成一个完整的DNA分子。随后提取出质粒转入E. coli富集并鉴定, 最后将正确组装的DNA转入宿主体内异源表达目标通路[3, 8]。
辅助模块主要包括复制起始元件和筛选标记。起始因子和复制起始元件结合并招募复制装置的组成蛋白起始DNA的复制, 筛选标记用于筛选转入了供体基因的细胞, 可以节约时间和资源[14-15]。酿酒酵母和大肠杆菌中常用的筛选标记如表 1所示[16-17]。
| 菌株 Strains |
筛选标记 Selection markers |
基因 Genes |
酶 Enzymes |
| S.cerevisiae | Fluoroacetate | dehH1 | Haloacetate dehydrogenase |
| Glyphosate | aroA-tADH1 | EPSP synthase | |
| Hygromycin B | hph | Hygromycin B phosphotransferase | |
| Methotrexated | R.dhfr(-tTRP5) | Dihydrofolate reductase | |
| Leu | leu2 | Beta-isopropylmalate dehydrogenase | |
| His | his3 | Imidazole glycerol phosphate dehydratase | |
| Trp | trp1 | Tryptophan synthetase | |
| Ura | ura3 | Orotidine-5ʹ-phosphate decarboxylase | |
| E.coli | Ampicillin | amp | β-lactamase |
| Kanamycin | kan | Aminoglycoside phosphotransferase | |
| Chloromycetin | cm | Chloramphenicol acetyl transferase | |
| Streptomycin | str | Streptomycin acetyl transferase |
在完成表达模块和辅助模块的操作后, 可在酵母中通过一步转化法实现完整生物合成基因簇的组装。Shao等[5]率先使用带有同源臂的引物扩增DNA片段, 每个片段取200-300 ng, 等比例混合, 电转化酵母感受态细胞, 挑选长出的单菌落5-10个, 提取质粒, 酶切和测序验证组装正确的质粒或生物合成基因簇片段。例如, 该技术应用于变铅青链霉菌(Streptomyces lividans)合成聚环类抗生素(Polycyclic tetramate macrolactam, PTM)取得良好效果。
PTMs是一类广泛分布、有重要生物活性的天然产物, 具有抗真菌、细菌和抗氧化的活性[18-19]。无论在天然宿主或异源宿主体内, PTMs生物合成基因簇都保持沉默。该基因簇由SGR810 (预测的细胞色素P450)、SGR811 (预测的氧化还原酶)、SGR812 (预测的FAD依赖的氧化还原酶)、SGR813 (预测的FAD依赖的氧化还原酶)、SGR814 (Ⅰ型非核糖体多肽聚酮合酶融合蛋白)、SGR815 (脂肪酸脱氢酶)共6个基因组成。Luo等[2]采用DNA assembler技术在基因前面分别添加组成型启动子gapdhp(KR)、gapdhp(SG)、rpsLp(XC)、rpsLp(TP)、rpsLp(SG)和rpsLp(CF)激活了沉默的PTMs生物合成基因簇, 在链霉菌中成功合成了PTMs。其过程如下:首先以灰色链霉菌(Streptomyces griseus)基因组DNA为模板分别扩增出启动子和目标基因片段, 利用OE-PCR将基因和相应的启动子连接, 再以质粒pRS416为模板扩增出S. cerevisiae辅助元件(含尿嘧啶合成营养缺陷筛选标记), 以质粒pAE4为模板扩增出E.coli (含阿泊拉霉素抗性筛选标记)和变铅青链霉菌(S. lividans) (含2, 6-二氨基庚二酸筛选标记)辅助元件, 然后利用DNA assembler技术在酿酒酵母中完成质粒的组装。从S. cerevisiae体内提取并验证正确组装的质粒, 将其转入S. lividans体内异源表达PTMs, 最后采用HPLC-MS检测PTMs目标产物。
Shao等[5]又在酿酒酵母中实现了玉米黄质的生物合成, 其过程是:以质粒pCAR-ΔCrtX为模板分别克隆玉米黄质生物合成通路中的一系列基因, 以S. cerevisiae基因组DNA为模板分别克隆出相应的启动子和终止片段, 然后利用OE-PCR将启动子、基因和终止片段连接成5个表达盒, 分别是TEF1p-CrtE-PGIt、HXT7P-CrtB-TPI1t、TEF2p-CrtI-FBA1t、FBA1p-CrtY-ENO2t、PDC1p- CrtZ-TDH2t, 最后利用DNA assembler技术将表达盒与BamHⅠ线性化处理后的质粒pRS416m[20]组装成一个环状质粒。质粒pRS416m上含有S. cerevisiae辅助元件(含尿嘧啶合成营养缺陷筛选标记)和E. coli (含氨苄青霉素抗性筛选标记)辅助元件。正确组装的质粒在S. cerevisiae中表达后, 可检测到玉米黄质。
链霉菌中的Spectinabilin是一个含有硝基苯的聚酮化合物, 有抗疟疾和抗病毒的活性[21]。在正常的培养条件下其生物合成途径保持沉默。Shao等[8]选择链霉菌体内看家基因(包括RNA聚合酶亚基、延伸因子、核糖体蛋白、糖酵解酶类和氨酰-tRNA合成酶等)上游的强启动子, 应用该技术在酵母体内将启动子、结构基因、终止子、辅助元件组装成完整质粒。将带有完整基因簇的质粒导入目标异源宿主, 实现了Spectinabilin在链霉菌中的生物合成(图 3)。
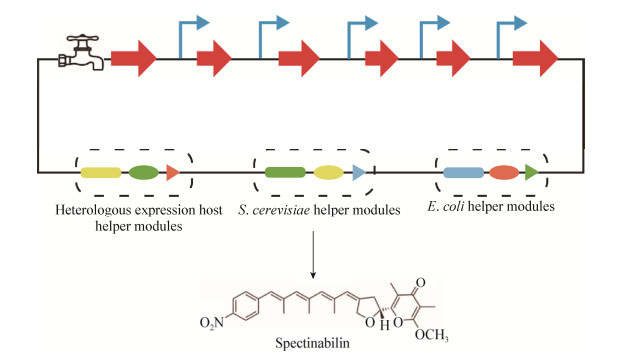
|
| 图 3 DNA assembler技术激活链霉菌的Spectinabilin生物合成基因簇 Figure 3 The activation of spectinabilin biosynthetic pathway in Streptomycetes by DNA assembler |
|
|
该策略与传统方法相比, 简化了实验流程, 还可利用该策略在异源宿主体内表达天然宿主体内受到抑制的生物合成途径。NorG将莽草酸途径中的分支酸转变成氨基苯甲酸, 是Spectinabilin生物合成通路的限速酶, Herai等[22]在NorG前添加可被ε-己内酰胺诱导的启动子nitAp, 增强了基因表达水平, 实现了Spectinabilin的有效合成。许多次级代谢产物大多在稳定期合成, 会对异源表达宿主产生毒性, 因此在稳定期添加外源诱导剂可以缓解初级代谢的压力。
以上实例是DNA assembler技术应用的典型代表, 该技术可重构次级代谢产物生物合成基因簇或激活沉默的生物合成基因簇。这使得利用廉价的底物生物合成一系列有价值的药物或者大宗化学品成为可能, 同时也避免了化学合成带来的污染问题。
2 体外基因组装技术 2.1 Gibson组装Gibson组装最早由Gibson等[23]在2009年提出, 其原理如图 4所示。该技术通过PCR的方法在DNA片段的两端加上同源序列, 其长度通常为15-40 bp, PCR扩增出不同DNA片段。然后, 将这些DNA片段和一种含有3种酶的混合溶液孵育1 h即可。外切酶, 从5′-端开始对DNA进行消化, 产生长的黏性末端, 这样便于与另外的同源末端进行配对结合; 聚合酶, 用于修补Gap; DNA连接酶, 实现无痕拼接, 形成完整的DNA分子。该方法的优势在于这3种酶都可以在同一个温度下很好地发挥功能, 可一步完成组装, 组装后的质粒可直接用于转化感受态细胞, 无需限制性内切酶, 通常该方法可组装不超过6个片段。

|
| 图 4 Gibson技术的组装原理 Figure 4 The principle of Gibson assembly |
|
|
2010年, Gibson等[24]使用Gibson组装和酿酒酵母体内同源重组人工合成了1.08 Mb的丝状支原体基因组并转入去核的山羊支原体, 新移殖的细胞表现出了丝状支原体的性状。2016年, Hutchison等[25]先用PCR合成出一系列丝状支原体基因组片段, 每个DNA片段长1.4 kb, 然后每5个分成一组, 再利用Gibson组装技术将5个一组的DNA片段连接成7 kb的单片段, 最后组装成完整的基因组。作者只保留了必需基因, 将1.08 Mb的基因组缩短到了含有473个基因的531 kb。2017年, Esvelt等[26]将Gibson组装技术与CRISPR/Cas9技术结合构建了一个PAM质粒文库, 用于评估鉴定来自不同物种的一系列不同的Cas9蛋白的活性。该方法在体外组装技术中应用广泛。
2.2 Golden Gate组装Golden Gate组装技术是基于非同源重组的代表性技术, 其利用Ⅱ型限制性酶BsaⅠ在识别位点外部切割的特性, 设计特异的突出序列同时组装多个片段, 且酶切和酶连可以同时进行[27], 其原理如图 5所示。首先, 扩增目的片段, 在两端加上BsaⅠ识别序列, 同时在识别序列内侧加上不同的4 nt突出部分, 相邻片段衔接处的4 nt反向互补配对, 然后将片段分别插入酶切前的中间载体, 因此一共可以设计256个突出的末端。利用E. coli富集含有不同目的片段的中间载体, 将这些载体与最终的载体(含有2个相邻的BsaⅠ酶切位点)混合, 加入BsaⅠ和DNA连接酶, 同时进行酶切和酶连组装多达9个DNA片段。
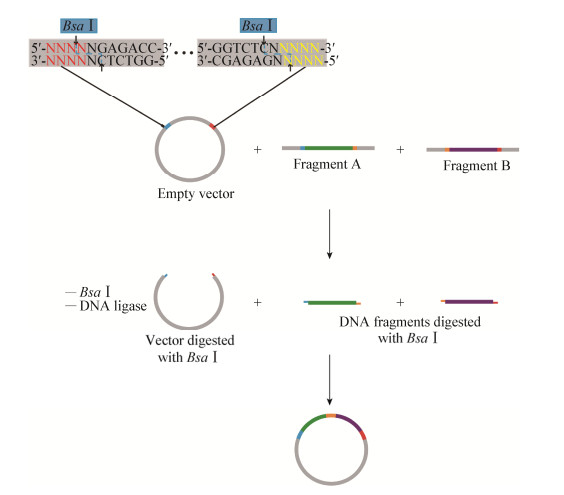
|
| 图 5 Golden Gate技术的组装原理 Figure 5 The principle of Golden Gate assembly |
|
|
2011年, Cermak等[28]利用Golden Gate高效组装了TALEN敲除系统中的重复序列, 整个过程只需要两步:第一步先将最多10个RVDs按顺序组装, 每个RVDs可识别1个核苷酸; 第二步再将这些RVDs和TALEN系统的其它功能元件如NLS、AD等组装成最终的敲除质粒。2015年, Lauressergues等[29]使用Golden Gate构建microRNAs表达质粒, 探究了苜蓿、拟南芥中2条microRNAs的转录调控作用及其它5条microRNAs编码的具有调控作用的短肽对植物主根生长的影响。同年, Xie等[30]将该技术与CRISPR/Cas9结合, 将tRNA基因和sgRNA序列间隔排列, 利用tRNA的转录和加工系统在产生tRNA的同时生成大量的sgRNAs, 构建了一个高效的CRISPR/Cas9敲除系统, 可以同时对水稻基因组中的多个基因进行靶向敲除。
2.3 成对引物无酶DNA组装成对引物无酶DNA组装(Twin-primer non- enzymatic DNA assembly, TPA)[31]的原理如图 6所示, 其过程不需要限制性内切酶、DNA连接酶等, 没有序列依赖性, 使用2对引物扩增同一个DNA片段, 一个产物含有与前面DNA同源的序列, 另一个产物含有与后面DNA同源的序列, 两者1:1混合后变性退火即可形成一条首尾含有粘性末端的DNA片段用于后续的DNA组装, 显示出方便快捷的应用潜力。

|
| 图 6 TPA技术的组装原理 Figure 6 The principle of TPA assembly |
|
|
BioBrick是合成生物学领域一个经典的基因组装技术, 2003年, 麻省理工的Tom Knight对BioBrick进行了详细介绍并制定了一系列标准。BioBrick的原理如图 7所示, 其利用同尾酶Xba Ⅰ和Spe Ⅰ酶切产生相同的粘性末端, 酶连后片段连接处会产生86 p的疤痕, Xba Ⅰ和Spe Ⅰ都不再识别连接处的疤痕, 这样即可在不损失酶切位点的情况下不断组装DNA片段。2014年, 国内学者Liu等[32]利用归位内切酶Ⅰ-Sce Ⅰ和PI-Psp Ⅰ可产生相同粘性末端的特点, 模仿BioBrick原理设计出一种名为iBrick的DNA组装方法。归位内切酶识别的序列比普通的限制性内切酶长, 在DNA中出现的频率极低, 因此iBrick相比于BioBrick对DNA片段序列几乎没有限制。但较长的识别序列在酶切酶连组装的同时也产生了更长的疤痕, 为了解决这个问题, Li等[33]在2016年报道了一种应用了CRISPR-Cas系统的DNA组装方法C-Brick。C-Brick利用Ⅴ型CRISPR/Cas系统蛋白Cpf1核酸内切酶, 在PAM位点切割产生5 nt的粘性末端。该方法利用sgRNA识别长序列的特点避免了对待组装片段序列的要求, 同时可灵活设计sgRNA以识别不同的序列, 只产生5 bp的疤痕, 完美地规避了BioBrick的缺点。
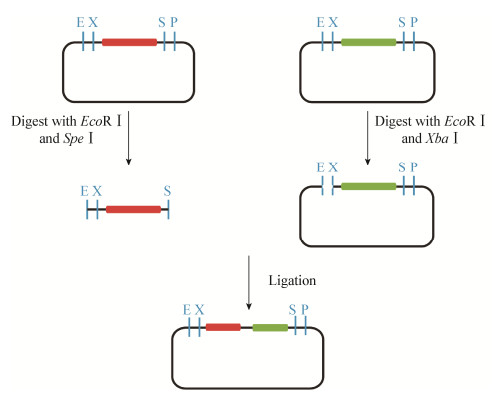
|
| 图 7 BioBrick技术的组装原理 Figure 7 The principle of BioBrick assembly |
|
|
除了以上基因组装技术, 近年来也发展起来很多其他DNA组装方法, 如:SLIC[34]、UNS[35-36]、PaperClip[37]和MASTER[38]等。SLIC和UNS都是通过核酸外切酶消化DNA片段的5′-端或-3′端, 产生-3′或5′-同源粘性末端, 然后在重组酶、DNA连接酶和DNA聚合酶的作用下组装成完整的DNA分子。PaperClip则利用加在DNA片段两端的桥式寡核苷酸进行组装, 不需要限制性酶切或增加同源序列的特定引物。MASTER连接法利用识别甲基化序列mCNNR (R=A或G)的核酸内切酶MspJⅠ对片段进行酶切, 切割位点在mCNNR之外, 可以通过在引物中添加mCNNR将识别序列加入PCR产物两端, 然后酶切酶连实现无缝拼接。2016年, Zhou等[39]报道了一种结合了CRISPR/Cas9和同源重组的基因组装方法CasHRA, 首先利用CRISPR/Cas9系统将质粒上待组装的DNA片段切割下来, 切下的DNA片段和线性化处理过的载体在S. cerevisiae体内通过同源重组组装成环化质粒, 然后使用半乳糖诱导CRISPR系统破坏含有sgRNA的质粒使完成组装的质粒在S. cerevisiae体内稳定存在, 以便用于下一轮的组装。
3 总结不同的基因组装技术各有优势, 如何根据实际情况选择合适的方法是其关键, 各基因组装技术的优缺点比较如表 2所示。主要体现在:(1) DNA assembler体内同源重组技术可同时完成多基因片段的整合和组装, 还可以对目的基因特定位点进行点突变或优化密码子, 与SLIC和Domino方法相比, DNA assembler技术是一种快速有效地构建较大的重组DNA分子的方法, 而且能够应用于染色体基因敲除或敲入; (2) Gibson体外同源重组技术利用3种酶在同一个温度下一步组装DNA分子, 方便快捷; (3) Golden-Gate适合于少数片段或较短片段的组装; (4)成对引物无酶DNA组装技术(TPA)是一种不使用任何酶直接组装PCR片段为质粒的方法, 适合较大片段的组装。笔者认为, 这4种方法的有效组合可完成绝大多数DNA分子的有效组装。
| 技术 Technologies |
原理 Principles |
组装片段大小 The size of DNA (kb) |
限制性内切酶 Restriction endonuclease |
参考文献 References |
| DNA assembler | Homologous recombination in vivo | 52 | None | [8] |
| Gibson | Homologous recombination in vitro | 583 | None | [23] |
| Golden Gate | Digest with BsaⅠ | 33 | BsaⅠ | [27] |
| TPA | Twin primers pairs | 31 | None | [31] |
| SLIC | Digest from -3′ to 5′- | 10.2 | None | [34] |
| UNS | Digest from 5′- to -3′ | 64 | None | [35] |
| PaperClip | Pairs of oligonucleotides | 6 | None | [37] |
基因组装技术在微生物合成生物学领域的应用展现了无与伦比的魅力, 在过去的几十年中取得了重要突破, 但还是存在着诸多问题。例如, 基因组装技术依赖于酶基因结构和功能的清楚解析、不同宿主高效启动子的使用及筛选标记的选择等, 深入开发和挖掘其功能研究, 有利于次级代谢产物的生物合成。除此之外, 我们还需要发展相关的基因调控技术如CRISPR/Cas9和CRISPR/dCas9技术, 以调控关键节点酶基因的表达水平、解除反馈抑制和平衡代谢流等。微生物生长快速, 易于培养, 对其进行基因操作简单方便, 特别是能够生产化学合成不能实现的天然产物、多肽、酶制剂等, 合成生物学将凭借其优势在人类的发展进程中扮演不可或缺的角色。
| [1] |
Xiao WH, Wang Y, Yuan YJ. Core technology in chemicals green manufacturing:synthetic biology[J]. CIESC Journal, 2016, 67(1): 119-128. 肖文海, 王颖, 元英进. 化学品绿色制造核心技术—合成生物学[J]. 化工学报, 2016, 67(1): 119-128. |
| [2] |
Luo YZ, Huang H, Liang J, et al. Activation and characterization of a cryptic polycyclic tetramate macrolactam biosynthetic gene cluster[J]. Nature Communications, 2013, 4: 2894. DOI:10.1038/ncomms3894 |
| [3] |
Shao ZY, Rao GD, Li C, et al. Refactoring the silent spectinabilin gene cluster using a plug-and-play scaffold[J]. ACS Synthetic Biology, 2013, 2(11): 662-669. DOI:10.1021/sb400058n |
| [4] |
Zhang PP, Ding YY, Liao WT, et al. A simple, universal, efficient PCR-based gene synthesis method:Sequential OE-PCR gene synthesis[J]. Gene, 2013, 524(2): 347-354. DOI:10.1016/j.gene.2013.03.126 |
| [5] |
Shao ZY, Luo YZ, Zhao HM. DNA assembler method for construction of zeaxanthin-producing strains of Saccharomyces cerevisiae[A]//Barredo JL. Methods in Molecular Biology, Vol. 898[M]. Totowa, NJ: Humana Press, 2012: 251-262
|
| [6] |
Mei YZ, Huang PW, Liu Y, et al. Cold stress promoting a psychrotolerant bacterium Pseudomonas fragi P121 producing trehaloase[J]. World Journal of Microbiology and Biotechnology, 2016, 32(8): 134-142. DOI:10.1007/s11274-016-2097-1 |
| [7] |
Mei YZ, Liu Y, Xu XT, et al. Complete genome sequence of a bacterium Pseudomonas fragi P121, a strain with degradation of toxic compounds[J]. Journal of Biotechnology, 2016, 224: 68-69. DOI:10.1016/j.jbiotec.2016.03.019 |
| [8] |
Shao ZY, Zhao HM. Manipulating natural product biosynthetic pathways via DNA assembler[J]. Current Protocols in Chemical Biology, 2014, 6(2): 65-100. |
| [9] |
Jiang YY, He JR, Wang WW, et al. The approach to high production of recombinant protein via pHsh vectors for Escherichia coli[J]. Microbiology China, 2012, 39(3): 394-400. 蒋钰瑶, 何嘉荣, 王未未, 等. 新型大肠杆菌高效表达载体pHsh的构建与应用[J]. 微生物学通报, 2012, 39(3): 394-400. |
| [10] |
Shi YR, Sun YH. Progress in DNA cloning and assembly techniques[J]. Microbiology China, 2015, 42(11): 2229-2237. 史晏榕, 孙宇辉. DNA克隆和组装技术研究进展[J]. 微生物学通报, 2015, 42(11): 2229-2237. |
| [11] |
Zhao J, Wang X, Li BZ, et al. DNA assembly in synthetic biology[J]. Chinese Bulletin of Life Sciences, 2013, 25(10): 983-992. 赵鹃, 王霞, 李炳志, 等. 合成生物学中的DNA组装技术[J]. 生命科学, 2013, 25(10): 983-992. |
| [12] |
Nagy ZB, Varga-Orvos Z, Szakál B, et al. Assembling and cloning genes for fusion proteins using reverse transcription one-step overlap extension PCR method[J]. Analytical Biochemistry, 2006, 351(2): 311-313. DOI:10.1016/j.ab.2006.01.049 |
| [13] |
Wei HL, Hu J, Wang L, et al. Rapid gene splicing and multi-sited mutagenesis by one-step overlap extension polymerase chain reaction[J]. Analytical Biochemistry, 2012, 429(1): 76-78. DOI:10.1016/j.ab.2012.06.027 |
| [14] |
Yang HB, Wu ZF, Liu JF, et al. Activation of a dormant replication origin is essential for Haloferax mediterranei lacking the primary origins[J]. Nature Communications, 2015, 6: 8321. DOI:10.1038/ncomms9321 |
| [15] |
Herzog E, Frisch M. Selection strategies for marker-assisted backcrossing with high-throughput marker systems[J]. Theoretical and Applied Genetics, 2011, 123(2): 251-260. DOI:10.1007/s00122-011-1581-0 |
| [16] |
van den Berg MA, Steensma HY. Expression cassettes for formaldehyde and fluoroacetate resistance, two dominant markers in Saccharomyces cerevisiae[J]. Yeast, 1997, 13(6): 551-559. DOI:10.1002/(ISSN)1097-0061 |
| [17] |
Le YL, Sun Y, Wang HC, et al. Advances in selection markers and their bio-safety in applications of transformed microorganisms[J]. Microbiology China, 2016, 43(8): 1814-1821. 乐易林, 孙宇, 王洪成, 等. 微生物遗传转化筛选标记及其生物安全性的研究进展[J]. 微生物学通报, 2016, 43(8): 1814-1821. |
| [18] |
Cao SG, Blodgett JAV, Clardy J. Targeted discovery of polycyclic tetramate macrolactams from an environmental Streptomyces strain[J]. Organic Letters, 2010, 12(20): 4652-4654. DOI:10.1021/ol1020064 |
| [19] |
Blodgett JAV, Oh DC, Cao SG, et al. Common biosynthetic origins for polycyclic tetramate macrolactams from phylogenetically diverse bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(26): 11692-11697. DOI:10.1073/pnas.1001513107 |
| [20] |
Lee FWF, Da Silva NA. Sequential δ-integration for the regulated insertion of cloned genes in Saccharomyces cerevisiae[J]. Biotechnology Progress, 1997, 13(4): 368-373. DOI:10.1021/bp970055d |
| [21] |
Choi YS, Johannes TW, Simurdiak M, et al. Cloning and heterologous expression of the spectinabilin biosynthetic gene cluster from Streptomyces spectabilis[J]. Molecular BioSystems, 2010, 6(2): 336-338. DOI:10.1039/B923177C |
| [22] |
Herai S, Hashimoto Y, Higashibata H, et al. Hyper-inducible expression system for Streptomycetes[J]. Proceedings of the National Academy of Science of the United States of America, 2004, 101(39): 14031-14035. DOI:10.1073/pnas.0406058101 |
| [23] |
Gibson DG, Lei Y, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases[J]. Nature Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318 |
| [24] |
Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome[J]. Science, 2010, 329(5987): 52-56. DOI:10.1126/science.1190719 |
| [25] |
Hutchison CA Ⅲ, Chuang RY, Noskov VN, et al. Design and synthesis of a minimal bacterial genome[J]. Science, 2016, 351(6280): aad6253. DOI:10.1126/science.aad6253 |
| [26] |
Esvelt KM, Mali P, Braff JL, et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing[J]. Nature Methods, 2013, 10(11): 1116-1121. DOI:10.1038/nmeth.2681 |
| [27] |
Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability[J]. PLoS One, 2008, 3(11): e3647. DOI:10.1371/journal.pone.0003647 |
| [28] |
Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Research, 2011, 39(12): e82. DOI:10.1093/nar/gkr218 |
| [29] |
Lauressergues D, Couzigou JM, Clemente HS, et al. Primary transcripts of microRNAs encode regulatory peptides[J]. Nature, 2015, 520(7545): 90-93. DOI:10.1038/nature14346 |
| [30] |
Xie KB, Minkenberg B, Yang YN. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): 3570-3575. DOI:10.1073/pnas.1420294112 |
| [31] |
Liang J, Liu ZH, Low XZ, et al. Twin-primer non-enzymatic DNA assembly:an efficient and accurate multi-part DNA assembly method[J]. Nucleic Acids Research, 2017, 45(11): e94. DOI:10.1093/nar/gkx132 |
| [32] |
Liu JK, Chen WH, Ren SX, et al. iBrick:A new standard for iterative assembly of biological parts with homing endonucleases[J]. PLoS One, 2014, 9(10): e110852. DOI:10.1371/journal.pone.0110852 |
| [33] |
Li SY, Zhao GP, Wang J. C-Brick:a new standard for assembly of biological parts using Cpf1[J]. ACS Synthetic Biology, 2016, 5(12): 1383-1388. DOI:10.1021/acssynbio.6b00114 |
| [34] |
Li MZ, Elledge SJ. Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC[J]. Nature Methods, 2007, 4(3): 251-256. DOI:10.1038/nmeth1010 |
| [35] |
Torella JP, Boehm CR, Lienert F, et al. Rapid construction of insulated genetic circuits via synthetic sequence-guided isothermal assembly[J]. Nucleic Acids Research, 2014, 42(1): 681-689. DOI:10.1093/nar/gkt860 |
| [36] |
Torella JP, Lienert F, Boehm CR, et al. Unique nucleotide sequence-guided assembly of repetitive DNA parts for synthetic biology applications[J]. Nature Protocols, 2014, 9(9): 2075-2089. DOI:10.1038/nprot.2014.145 |
| [37] |
Trubitsyna M, Michlewski G, Cai YZ, et al. PaperClip:rapid multi-part DNA assembly from existing libraries[J]. Nucleic Acids Research, 2014, 42(10): e154. |
| [38] |
Chen WH, Qin ZJ, Wang J, et al. The MASTER (methylation-assisted tailorable ends rational) ligation method for seamless DNA assembly[J]. Nucleic Acids Research, 2013, 41(8): e93. DOI:10.1093/nar/gkt122 |
| [39] |
Zhou JT, Wu RH, Xue XL, et al. CasHRA (Cas9-facilitated Homologous Recombination Assembly) method of constructing megabase-sized DNA[J]. Nucleic Acids Research, 2016, 44(14): e124. DOI:10.1093/nar/gkw475 |
 2018, Vol. 45
2018, Vol. 45




