扩展功能
文章信息
- 向慧平, 关统伟, 赵顺先, 张习超, 欧梦莹, 林宜锦, 王鹏昊
- XIANG Hui-Ping, GUAN Tong-Wei, ZHAO Shun-Xian, ZHANG Xi-Chao, OU Meng-Ying, LIN Yi-Jin, WANG Peng-Hao
- 新疆巴里坤、七角井和台特玛盐湖沉积物中免培养放线菌群落组成与离子成分分析
- Actinobacterial community and ionic composition in sediment of Xinjiang saline lakes: Barkol, Qijiaojing and Taitema
- 微生物学通报, 2018, 45(6): 1228-1236
- Microbiology China, 2018, 45(6): 1228-1236
- DOI: 10.13344/j.microbiol.china.170559
-
文章历史
- 收稿日期: 2017-07-20
- 接受日期: 2017-12-11
- 网络首发日期(www.cnki.net): 2018-04-17
我国是世界盐湖最多的国家之一,据统计面积大于1 km2的盐湖有813个[1],但对我国盐湖进行过生物学调查的却不足50个[2]。盐湖环境蕴藏着丰富的放线菌类群,目前大多数研究集中在放线菌的纯培养方面[3-5],同时在盐湖环境中发现了一些新的放线菌物种,如Streptoalloteichus[6]、Haloactinospora alba[7]、Myceligenerans salitolerans[8]等;另外,大量活的但不可培养的放线菌资源也被探索到[6, 9-10]。这些研究为盐湖放线菌资源的深度挖掘提供了很有价值的参考数据,但关于盐湖放线菌群落结构与化学离子的关联性分析的研究却很少。盐湖中存在着丰富的化学离子,如Na+、K+、Mg2+、Cl−、SO42−等,那么这些盐湖化学离子成分对放线菌的群落结构产生怎样的影响,这些离子分别对哪些放线菌有显著影响等人们了解得还不是很清楚。为此,本研究应用免培养技术和基于16S rRNA基因序列的生物信息学分析对台特玛盐湖(氯化物型)、巴里坤盐湖(硫酸镁亚型)和七角井盐湖(硫酸钠亚型)[11]进行放线菌的免培养研究,进一步了解盐湖放线菌的群落结构;同时将盐湖沉积物8种主要的化学离子成分(Na+、K+、Ca2+、Mg2+、Cl−、SO42−、HCO3−、CO32−)与放线菌群落组成进行了化学离子因子关联性分析,从而有利于系统保护盐湖放线菌物种资源和探索群体多样性演化机制,进而为深度挖掘活的新型放线菌资源提供有价值的参考。
1 材料与方法 1.1 材料样品来自于新疆天山盆地盐湖中的巴里坤盐湖(92°43′10"−92°52′58"E,43°36′30"−43°42′50"N)和七角井盐湖(91°26′43"−91°38′45"E,43°26′15"−43°26′45"N),以及塔里木盆地盐湖中的台特玛盐湖(88°22′40"E,39°27′08"N),采样时间为夏季,每个盐湖采集沉积物5份,样品深度0−40 cm,其pH为6.5−8.0,样品采集后放于4 ℃保存,运抵实验室后立即做免培养分析[12]。以上操作过程均进行严格的无菌操作,尽量排除操作污染。
1.2 主要试剂和仪器蛋白酶K,德国Merck公司;PCR纯化试剂盒,凯杰生物技术有限公司;DNA marker,生工生物工程(上海)股份有限公司;pMD18-T Vector、Taq酶、dNTPs、UNIQ-10柱式DNA胶回收试剂盒,宝生物工程有限公司;其他化学试剂(苯酚、氯仿、异戊醇等)均购于成都迪维乐普科技有限公司。离子色谱仪,赛默飞世尔科技有限公司;凝胶成像仪、PCR扩增仪、电泳仪,美国Bio-Rad公司。
1.3 样品离子组成检测将土样60 ℃烘干,等量混合5份土样过150目筛用于理化因子测定,参照DZ/T0064.51-1993,用离子色谱仪测定Cl−、SO42−;参照GB/T15454-2009,用离子色谱仪测定Na+、K+、Ca2+、Mg2+;用滴定法测定CO32−、HCO3−。
1.4 样品总DNA的提取和PCR扩增主要采用赵勇等[13]描述使用的SDS-CTAB抽提法提取盐湖沉积物中的微生物总DNA。采用放线菌的特异性引物[14] S-C-Act-0235-a-S-20(S20) (5′-CGC GGCCTATCAGCTGTTG-3′)和S-C-Act0878-a-A- 19(A19) (5′-CCGTACTCCCCAGGCGGGG-3′)对样品总DNA进行PCR扩增,PCR反应条件参照Stach等[14]的方法进行,扩增体系为50 μL,扩增片段大小约640 bp。然后将目的条带按照UNIQ-10柱式DNA胶回收试剂盒的说明对PCR产物进行纯化处理。
1.5 克隆文库构建及测序纯化产物通过T4 DNA连接酶与载体进行连接,并转化大肠杆菌DH5α感受态细胞[15]。转化产物涂布在含有氨苄青霉素(100 mg/L)的LB平板,随机挑取白色克隆子划线培养,并构建克隆文库。PCR扩增插入的放线菌16S rRNA基因片段后用HaeⅢ酶切3 h,每个样品随机挑选220个克隆子,经过Hae Ⅲ酶切筛选后,选出酶切带谱不同的阳性克隆子送到生工生物工程(上海)股份有限公司进行测序。
1.6 生物信息学分析测得的原始序列使用QIIME平台去除低质量序列,根据标签序列分开来自不同样品中的序列,利用USEARCH软件在97%相似度下进行聚类,对聚类后的序列进行嵌合体过滤后得到可用于物种分类的OTU[16]。使用QIIME平台软件从OTU中提取一条Tag序列作为代表序列,使用RDP (Ribosomal database project)方法与NCBI数据库比对(Https://blast.ncbi.nlm.nih.gov/Blast),相似性在95%以上即可认为在同一个属。通过SPADE软件和稀释曲线分析构建的16S rRNA基因文库,并制作稀释曲线。将测得的样品离子组成与样品放线菌多样性进行基于RDA (Redundancy analysis)的关联性分析。
2 结果与分析 2.1 盐湖沉积物化学离子组成盐湖沉积物离子成分检测结果如表 1所示。4种阳离子组成中,巴里坤盐湖Mg2+含量显著高于其他两湖,台特玛盐湖Ca2+显著高于其他两湖,而七角井盐湖Na+含量最高。4种阴离子组成中,七角井盐湖与巴里坤盐湖较为接近,台特玛盐湖中的Cl−显著低于七角井盐湖和巴里坤盐湖,而台特玛盐湖中的HCO3−要显著高于另外两湖。台特玛盐湖在20世纪60年代初,湖水干涸演变为干盐湖,后来重新汇入塔里木河水,因此沉积物离子浓度最高。此外,巴里坤和七角井盐湖都是浅水湖,受到气候等因素的影响,正逐渐演化成干盐湖。
| 样品 Sample |
Na+ | K+ | Ca2+ | Mg2+ | Cl− | SO42− | HCO3− | CO32− |
| 巴里坤 Barkol |
103.644 | 151.262 | 59.964 | 122.818 | 259.880 | 19.620 | 1.446 | 0.015 |
| 七角井 Qijiaojing |
143.702 | 63.689 | 64.228 | 49.997 | 254.484 | 16.378 | 0.255 | 0.086 |
| 台特玛 Taitema |
47.206 | 206.990 | 118.679 | 20.107 | 125.127 | 14.502 | 5.104 | 0.752 |
在3个盐湖沉积物样品中测序得到381个克隆序列,聚类结果显示,从巴里坤盐湖样品中得到了34个OTU;从七角井盐湖中得到了45个OTU;从台特玛盐湖样品中得到了64个OTU,使用维恩图(Venn diagram)来表征3个盐湖沉积物样品间放线菌OTU的分布情况(图 1)。台特玛盐湖独有的OTU数占总OTU数的37.50%,显著高于巴里坤盐湖的14.71%和七角井盐湖的17.78%,同时从台特玛盐湖沉积物中提取的OTU最多,表明台特玛盐湖沉积物放线菌独有类群显著高于另外两湖。巴里坤盐湖和七角井盐湖相互共有18个OTU,占到巴里坤盐湖的52.94%和七角井盐湖的40%,两湖的共有率很高,放线菌类群相似性较高。而3个盐湖共有的OTU数为10个,占到巴里坤盐湖的29.41%、七角井的22.22%和台特玛盐湖的15.63%,表明3个盐湖样品中的放线菌多样性是十分丰富的。

|
| 图 1 巴里坤(B)、七角井(Q)和台特玛盐湖(T)放线菌类群关于OTU的Venn图 Figure 1 The Venn was based on the OTUs which extracted from actinobacteria in the sediment of the three lakes: Barkol lake (B), Qijiaojing lake (Q) and Taitema lake (T) |
|
|
通过SPADE软件和稀有度统计方法分析构建的16S rRNA基因文库,结果显示在置信度为95%时,3个盐湖沉积物放线菌的物种覆盖度都达到99.3%−99.9%,说明样本容量足够,并且稀释曲线(Rarefaction curve)趋于平缓(图 2),认为库容(Library size)已经足够。

|
| 图 2 巴里坤、七角井和台特玛盐湖沉积物放线菌类群稀释曲线 Figure 2 Rarefaction curve for the three lakes: Barkol lake, Qijiaojing lake and Taitema lake which based on the OTUs of 16S rRNA gene clones from actinobacteria |
|
|
在NCBI的BLAST比对结果显示共获得37个放线菌属(图 3),其中酸土单胞菌属(Aciditerrimonas)、Aquihabitans、拟无枝酸菌属(Amycolatopsis)、Demequina、迪茨氏菌属(Dietzia)和沉积岩杆菌属(Ilumatobacter)是3个盐湖的共有放线菌属;棒状杆菌属(Corynebacterium)、微杆菌属(Microbacterium)、链霉菌属(Streptomyces)和嗜热油菌属(Thermoleophilum)是巴里坤与台特玛盐湖的共有属;两面神菌属(Janibacter)为巴里坤与七角井盐湖的共有属。值得注意的是,由于区域性环境的差异性也形成了区域性的特有放线菌物种。如巴里坤盐湖有5个独有的放线菌属:放线多孢菌属(Actinopolyspora)、伦兹氏菌属(Lentzea)、贫养杆菌属(Modestobacter)、丙酸杆菌属(Propionibacterium)和罗思氏菌属(Rothia);七角井盐湖有7个特有的放线菌属:隐孢囊菌属(Cryptosporangium)、乔治菌属(Georgenia)、东大分子与细胞所菌属(Iamia)、姜氏菌属(Jiangella)、考克氏菌属(Kocuria)、微球菌属(Micrococcus)和鸟氨酸微菌属(Ornithinimicrobium);台特玛盐湖独有的放线菌属最多,达到了14个,分别为节杆菌属(Arthrobacter)、Amnibacterium、芽球菌属(Blastococcus)、纤维单胞菌属(Cellulomonas)、弗莱德门氏菌属(Friedmanniella)、糖霉菌属(Glycomyces)、栖白蚁菌属(Isoptericola)、大理石雕菌属(Marmoricola)、拟诺卡氏菌属(Nocardiopsis)、类诺卡氏菌属(Nocardioides)、植物栖居菌属(Phytohabitans)、Pontimonas、链单孢菌属(Streptomonospora)和链孢囊菌属(Streptosporangium)。

|
| 图 3 巴里坤盐湖(B)、七角井盐湖(Q)和台特玛盐湖(T)放线菌类群在属水平群落组成结构差异 Figure 3 The actinobacterial community composition in the genus level which extracted from the sediment of the three lakes |
|
|
巴里坤盐湖、七角井盐湖和台特玛盐湖沉积物中放线菌类群在属水平上的组成结构差异如图 3所示,在巴里坤盐湖中的主要优势放线菌为Aquihabitans(10.59%)、酸土单胞菌属(Aciditerrimonas) (5.29%)、拟无枝酸菌属(Amycolatopsis) (3.93%)、嗜热油菌属(Thermoleophilum) (3.81%)、丙酸杆菌属(Propionibacterium) (3.26%)和链霉菌属(Streptomyces) (3.22%)。在七角井盐湖中的主要优势放线菌为拟无枝酸菌属(Amycolatopsis) (13.44%)、Aquihabitans (7.27%)、酸土单胞菌属(Aciditerrimonas) (3.02%)、沉积岩杆菌属(Ilumatobacter) (2.60%)、姜氏菌属(Jiangella) (2.46%)和隐孢囊菌属(Cryptosporangium) (2.37%)。而在台特玛盐湖中放线菌多样性最丰富,主要的优势放线菌为糖霉菌属(Glycomyces) (5.13%)、类诺卡氏菌属(Nocardioides) (4.69%)、栖白蚁菌属(Isoptericola) (2.87%)、迪茨氏菌属(Dietzia) (2.68%)、酸土单胞菌属(Aciditerrimonas) (1.71%)、Aquihabitans (1.88%)、沉积岩杆菌属(Ilumatobacter) (2.06%)、拟无枝酸菌属(Amycolatopsis) (2.31%)、嗜热油菌属(Thermoleophilum) (2.45%)、链单孢菌属(Streptomonospora) (1.71%)、拟诺卡氏菌属(Nocardiopsis) (1.76%)和链孢囊菌属(Streptosporangium) (2.17%)。从放线菌群落结构上看,台特玛盐湖与其他两湖差异性都很大,而巴里坤盐湖与七角井盐湖相对更近,但仍存在放线菌类群差异性。3个盐湖中都存在大量与现今有效发表的所有类群无较近亲缘性的OTU,巴里坤盐湖多达47.59%,七角井盐湖达到53.07%,台特玛盐湖达到51.53%。表明盐湖中含有大量的未知放线菌类群,等待研究者的深入挖掘。
2.4 盐湖沉积物放线菌群落多样性与化学离子关联性分析结合沉积物离子成分检测和盐湖放线菌多样性分析,可以在一定程度上解释不同离子对盐湖沉积物放线菌群落具体的影响方式,采用RDA分析的结果如图 4所示。Na+与链霉菌属(Streptomyces)和微杆菌属(Microbacterium)呈显著负相关(P < 0.05);K+与链霉菌属(Streptomyces)和微杆菌属(Microbacterium)呈显著正相关,与Demequina和姜氏菌属(Jiangella)呈显著负相关(P < 0.05);Ca2+和CO32−与拟诺卡氏菌属(Nocardiopsis)、链孢囊菌属(Streptosporangium)、迪茨氏菌属(Dietzia)、链霉菌属(Streptomyces)、节杆菌属(Arthrobacter)和糖霉菌属(Glycomyces)呈显著正相关(P < 0.05);HCO3−与链孢囊菌属(Streptosporangium)和迪茨氏菌属(Dietzia)呈显著正相关,与两面神菌属(Janibacter)和Aquihabitans呈显著负相关(P < 0.05);Mg2+和SO42−与罗思氏菌(Rothia)、放线多孢菌属(Actinopolyspora)、酸土单胞菌属(Aciditerrimonas)、丙酸杆菌属(Propionibacterium)、伦兹氏菌属(Lentzea)和贫养杆菌属(Modestobacter)呈显著正相关(P < 0.05);Cl−与两面神菌属(Janibacter)和Aquihabitans呈显著正相关(P < 0.05)。这些菌属的组成和含量在不同盐湖中的发生了较大的变化(图 3),盐湖离子对盐湖放线菌群落组成具有多方面的影响,相互交叉共同作用。
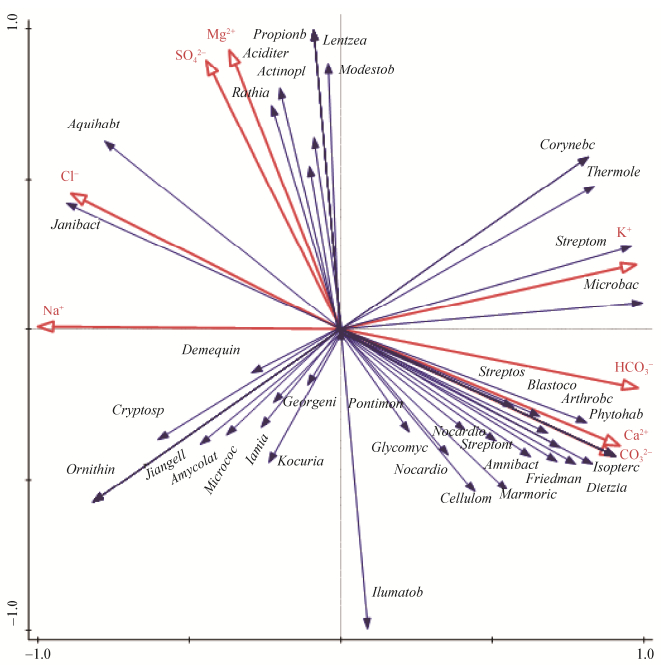
|
| 图 4 巴里坤湖(B)、七角井湖(Q)和台特玛湖(T)放线菌类群与沉积物离子关联性分析 Figure 4 The correlative analysis of ions with the actinobacterial community composition in sediment of three salt lakes |
|
|
极端环境微生物被认为是地球上尚未开发的极具应用潜能的一大类生物类群。嗜盐微生物(Halophile)在环境生物治理、酶制剂、生物电子和医药工业等领域具有极大的利用价值,促使各国学者不断探索更多的高盐环境微生物[17-18]。本研究采用免培养技术对巴里坤、七角井、台特玛3个盐湖沉积物中的放线菌类群进行了研究,挖掘出37个属的放线菌类群,展示了新疆盐湖丰富的放线菌多样性,而生命世界最令人难忘的特征之一就是独特的地理分布造就极端生命的充分多样性。本研究还首次在新疆盐湖中挖掘到Aquihabitans、Amnibacterium、节杆菌属(Arthrobacter)、芽球菌属(Blastococcus)、隐孢囊菌属(Cryptosporangium)、棒状杆菌属(Corynebacterium)、弗莱德门氏菌属(Friedmanniella)、伦兹氏菌属(Lentzea)、大理石雕菌属(Marmoricola)、鸟氨酸微菌属(Ornithinimicrobium)、Pontimonas、植物栖居菌属(Phytohabitans)和嗜热油菌属(Thermoleophilum)的类群,这与夏占峰等[6]、关统伟等[9]在艾丁湖和硝尔库勒湖发现的放线菌存在明显的群落差异。这些差异可能是基于地理隔离和遗传漂变的介导,使种群产生了对异质性环境的局域适应,并逐渐趋异快速分化为不同的新物种而形成的[19]。那么这些放线菌是如何适应高盐环境的,具有怎样的生理特性和特殊功能还有待深入挖掘。而获得的其他24个属已有研究报道过,如吕杰等[20]采用免培养技术在新疆艾比湖同样发现了丙酸杆菌属(Propionibacterium)、纤维素单胞菌属(Cellulomonas)、微杆菌属(Microbacterium)、节杆菌属(Arthrobacter)、考克氏菌属(Kocuria)和罗氏菌属(Rothia)。另外,研究发现47.59%−53.07%的放线菌克隆相似性小于97%,为新的放线菌资源,表明盐湖环境是挖掘放线菌新物种的天然场所。通常,隔离程度高的区域其特有物种区域分布也更丰富,而且这个区域被隔离的越久则它的特有性物种的分类地位也就越高,异域物种分化形成新物种的概率也越大。本研究的3个盐湖气候极端干旱、高盐、营养贫瘠,四周被高山环绕,是地理隔离程度极高的封闭性内陆盐湖,也是异域分化产生放线菌新物种资源的天然实验室。高水平的空间地理隔离也保持了较少的竞争对手,从而展现出更为丰富的极端微生物多样性。然而,这些丰富的微生物多样性也可能被高估,根据Sun等[21]研究发现基于16S rRNA基因的细菌多样性普遍存在高估(3.0%−12.9%)的现象,且V4−V5区域具有最低的高估率,其中放线菌门在V3区也具有极低的高估率。本研究也采用细菌16S rRNA基因的V3−V5区来扩增和表征盐湖环境土壤放线菌多样性,因此,研究结果中放线菌多样性也具有较低的高估率。此外,国内外学者使用纯培养和免培养技术同样在类似环境中发现了大量的放线菌多样性及其新类群[22-28],说明盐湖中丰富的放线菌是普遍存在的。由于极端环境具有和原始地球类似的环境,盐湖中那些令人兴奋的大量未知的不可培养放线菌资源,则有可能为研究生命进化提供新的证据。
沉积物离子检测发现3个盐湖的Na+、K+、Mg2+浓度极高,但依然多达37个放线菌属被探测到,表明盐湖放线菌对Na+、K+、Mg2+具有广泛的适应性,这与邹静等[29]的研究结果基本一致。RDA分析发现与Na+、Cl−和K+相比,Mg2+、Ca2+、SO42−、HCO3−和CO32−与盐湖放线菌类群的相关关系要更加紧密。唐蜀昆等[30]关于嗜盐放线菌生物学特性初步研究中指出K+和Mg2+对Na+具有一定的替代作用,并提出了存在专性嗜Mg2+的嗜盐放线菌的假设。另外,Foti等[31]研究发现盐湖中SO42−对硫酸盐还原菌具有富集效应,SO42−对能够还原硫酸盐的放线菌可能具有类似的作用,这一结论对于利用镁盐和硫酸盐发展新的培养基来筛选嗜盐放线菌有一定的指导意义。但需要思考的是盐湖中除了本研究阐述的8种化学离子成分外,还具有Si+、AL3+、Mn2+、Fe2+、Cu2+、Mo2+、Zn2+、F−、PO43−、NO3−、Br−、NH4+等成分。以微生物耐受NaCl的量来界定嗜盐菌[32]很可能是一个严重的思想禁锢,使得研究者习惯性地在培养基中添加不同浓度的NaCl来分离嗜盐放线菌,往往获得的放线菌种群较少。因此,接下来我们还将扩大盐湖样本,深入研究其他化学离子对盐湖放线菌多样性的影响,为更好地挖掘盐湖放线菌资源提供参考依据。
| [1] |
Zheng XY, Zhang MG, Xu C, et al. Saline Lake of China[M]. Beijing: Science Press, 2002, 4-5. 郑喜玉, 张明刚, 徐昶, 等. 中国盐湖志[M]. 北京: 科学出版社, 2002, 4-5. |
| [2] |
Pan HL, Zhou C, Wang HL, et al. Diversity of halophilic archaea in Hypersaline lakes of Inner Mongolia, China[J]. Acta Microbiologica Sinica, 2006, 46(1): 1-6. 潘海莲, 周成, 王红蕾, 等. 内蒙古锡林浩特地区嗜盐古菌多样性的研究[J]. 微生物学报, 2006, 46(1): 1-6. |
| [3] |
Cui HL, Yang Y, Dilbr, et al. Biodiversity of halophilic archaea isolated from two salt lakes in Xinjiang region of China[J]. Acta Microbiologica Sinica, 2006, 46(2): 171-176. 崔恒林, 杨勇, 迪丽拜尔·托乎提, 等. 新疆两盐湖可培养嗜盐古菌多样性研究[J]. 微生物学报, 2006, 46(2): 171-176. |
| [4] |
Xu XW, Wu M, Wu YH, et al. Culturable halophilic archaea of Ayakekumu lake located in Xinjiang[J]. Acta Ecologica Sinica, 2007, 27(8): 3119-3123. 许学伟, 吴敏, 吴月红, 等. 新疆阿牙克库木湖可培养嗜盐古菌的种群结构[J]. 生态学报, 2007, 27(8): 3119-3123. |
| [5] |
Dai X, Wang BJ, Huang Y, et al. Bacterial diversity in the sediments of Taihu lake by using traditional nutrient medium and dilution nutrient medium[J]. Acta Microbiologica Sinica, 2015, 45(2): 161-165. 戴欣, 王保军, 黄燕, 等. 普通和稀释培养基研究太湖沉积物可培养细菌的多样性[J]. 微生物学报, 2015, 45(2): 161-165. |
| [6] |
Xia ZF, Guan TW, Ruan JS, et al. Studies on the actinobacterial diversity in aiding lake sediments[J]. Acta Microbiologica Sinica, 2011, 51(8): 1023-1031. 夏占峰, 关统伟, 阮继生, 等. 艾丁湖沉积物放线菌多样性[J]. 微生物学报, 2011, 51(8): 1023-1031. |
| [7] |
Tang SK, Tian XP, Zhi XY, et al. Haloactinospora alba gen. nov., sp. nov., a halophilic filamentous actinomycete of the family Nocardiopsaceae[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58: 2075-2080. DOI:10.1099/ijs.0.65531-0 |
| [8] |
Guan TW, Wu N, Tang SK, et al. Myceligenerans salitoler-ans sp.nov, a halotolerant actinomycete isolated from a salt lake in Xinjiang, China[J]. Extremophiles, 2013, 17(1): 147-152. DOI:10.1007/s00792-012-0502-8 |
| [9] |
Guan TW, Wu JY, Zhi XY, et al. Actinobacterial diversity of a sediment sample from Xiaoerkule lake[J]. Acta Microbiologica Sinica, 2008, 48(7): 851-856. 关统伟, 吴晋元, 职晓阳, 等. 硝尔库勒湖沉积物中非培养放线菌多样性[J]. 微生物学报, 2008, 48(7): 851-856. |
| [10] |
Jiang HC, Dong HL, Zhang GX, et al. Microbial diversity in water and sediment of Lake Chaka, an athalassohaline lake in northwestern China[J]. Applied and Environmental Microbiology, 2006, 72(6): 3832-3845. DOI:10.1128/AEM.02869-05 |
| [11] |
Zheng XY, Li BX, Gao ZH, et al. Saline Lake of Xinjiang[M]. Beijing: Science China Press, 1995. 郑喜玉, 李秉孝, 高章洪, 等. 新疆盐湖[M]. 北京: 科学出版社, 1995. |
| [12] |
Brambilla E, Hippe H, Hagelstein A, et al. 16S rDNA diversity of cultured and uncultured prokaryotes of a mat sample from lake Fryxell, McMurdo Dry Valleys, Antarctica[J]. Extremophiles, 2001, 5(1): 23-33. DOI:10.1007/s007920000169 |
| [13] |
Zhao Y, Zhou ZH, Li W, et al. DNA extraction from soil for molecular microbial community analysis[J]. Journal of Agro-Environment Science, 2005, 24(5): 854-860. 赵勇, 周志华, 李武, 等. 土壤微生物分子生态学研究中总DNA的提取[J]. 农业环境科学学报, 2005, 24(5): 854-860. |
| [14] |
Stach JE, Maldonado LA, Ward AC, et al. New primers for the class actinobacteria: application to marine and terrestrial environments[J]. Environmental Microbiology, 2003, 5(10): 828-841. DOI:10.1046/j.1462-2920.2003.00483.x |
| [15] |
Ma MC, Jiang X, Li J, et al. Analysis of bacterial community composition by 16S rDNA clone library sampling from constructed rapid infiltration system (CRI)[J]. Microbiology China, 2008, 35(5): 731-736. 马鸣超, 姜昕, 李俊, 等. 应用16S rDNA克隆文库解析人工快速渗滤系统细菌种群多样性[J]. 微生物学通报, 2008, 35(5): 731-736. |
| [16] |
Wang Q, Garrity GM, Tiedie JM, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [17] |
Zhao BS, Wang H, Mao XW. The recent advances in halophilic microorganisms for environmental bioremediation[J]. Microbiology China, 2007, 34(6): 1209-1212. 赵百锁, 王慧, 毛心慰. 嗜盐微生物在环境修复中的研究进展[J]. 微生物学通报, 2007, 34(6): 1209-1212. |
| [18] |
Zhuang Y, Peng C, Ye BP. Research advances on the functions and applications of extremophile[J]. Pharmaceutical Biotechnology, 2016, 23(5): 442-446. 庄原, 彭程, 叶波平. 极端微生物及其功能和应用研究进展[J]. 药物生物技术, 2016, 23(5): 442-446. |
| [19] |
Flaxman SM, Feder JL, Nosil P. Genetic hitchhiking and the dynamic buildup of genomic divergence during speciation with gene flow[J]. Evolution, 2013, 67(9): 2577-2591. DOI:10.1111/evo.2013.67.issue-9 |
| [20] |
Lv J, Lv GH, Ma Y. Actinobacterial diversity in sediments of Ebinur lake, Xinjiang, China[J]. Acta Microbiologica Sinica, 2016, 56(9): 1426-1433. 吕杰, 吕光辉, 马媛. 新疆艾比湖沉积物中免培养放线菌的多样性[J]. 微生物学报, 2016, 56(9): 1426-1433. |
| [21] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [22] |
Vijayakumar R, Muthukumar C, Thajuddin N, et al. Studies on the diversity of actinomycetes in the palk strait region of Bay of Bengal, India[J]. Actinomycetologica, 2007, 21(2): 59-65. DOI:10.3209/saj.SAJ210203 |
| [23] |
Yadav AK, Vardhan S, Kashyap S, et al. Actinomycetes diversity among rRNA gene clones and cellular isolates from Sambhar Salt Lake, India[J]. The Scientific World Journal, 2013, 781301. |
| [24] |
Jiang HC, Dong HL, Zhan GX, et al. Microbial diversity in water and sediment of lake Chaka, an athalassohaline lake in north-western China[J]. Applied and Environmental Microbiology, 2006, 72(6): 3832-3845. DOI:10.1128/AEM.02869-05 |
| [25] |
Stevens H, Brinkhoff T, Rink B, et al. Diversity and abundance of gram positive bacteria in a tidal flat ecosystem[J]. Environmental Microbiology, 2007, 9(7): 1810-1822. DOI:10.1111/j.1462-2920.2007.01302.x |
| [26] |
Cao LL, Wang Y, Tang SK, et al. Biodiversity and enzyme screening of actinomycetes from Hami Lake[J]. Acta Microbiologica Sinica, 2009, 49(3): 287-293. 曹兰兰, 王芸, 唐蜀昆, 等. 新疆哈密地区盐湖放线菌的多样性及其功能酶的筛选[J]. 微生物学报, 2009, 49(3): 287-293. |
| [27] |
Wu JY, Guan TW, Jiang HC, et al. Diversity of actinobacterial community in saline sediments from Yunnan and Xinjiang, China[J]. Extremophiles, 2009, 13(4): 623-632. DOI:10.1007/s00792-009-0245-3 |
| [28] |
Guan TW, Che ZM, Xiang WL, et al. Actinobacterial community structure in sediment from Lop Nur Salt Lake[J]. Chinese Journal of Applied and Environmental Biology, 2013, 19(3): 466-470. 关统伟, 车振明, 向文良, 等. 罗布泊盐湖沉积物放线菌的群落结构[J]. 应用与环境生物学报, 2013, 19(3): 466-470. |
| [29] |
Zou J, Mi Q, Ma YG, et al. Study effect on salts concentration of Na+, K+, Mg2+ and Ca2+ to growth of actinomyces[J]. Science and Technology of Qinghai Agriculture and Forestry, 2005(3): 1-3. 邹静, 米琴, 马永贵, 等. Na+, K+, Mg2+, Ca2+浓度对嗜盐放线菌生长的影响[J]. 青海农林科技, 2005(3): 1-3. |
| [30] |
Tang SK, Li WJ, Zhang YG, et al. Studies on biological characteristics of halophilic actinomycetes[J]. Microbiology China, 2003, 30(4): 15-19. 唐蜀昆, 李文均, 张永光, 等. 嗜盐放线菌生物学特性初步研究[J]. 微生物学通报, 2003, 30(4): 15-19. |
| [31] |
Foti M, Sorokin DY, Lomans B, et al. Diversity, activity, and abundance of sulfate-reducing bacteria in saline and hypersaline soda lakes[J]. Applied and Environmental Microbiology, 2007, 73(7): 2093-2100. DOI:10.1128/AEM.02622-06 |
| [32] |
Kushner, DJ. Life in high salt and solute concentrations: halophilic bacteria[A]//Microbial Life in Extreme Environments[M]. Lodon Academic Press, 1978: 317-368
|
 2018, Vol. 45
2018, Vol. 45




