扩展功能
文章信息
- 郭军玲, 张帆, 黄楠, 周丽, 周哲敏
- GUO Jun-Ling, ZHANG Fan, HUANG Nan, ZHOU Li, ZHOU Zhe-Min
- Tyr78-loop对鱼腥藻来源苯丙氨酸脱氨酶活性的影响
- Influence of Tyr78-loop on phenylalanine ammonia-lyase from Anabaena variabilis
- 微生物学通报, 2018, 45(6): 1171-1179
- Microbiology China, 2018, 45(6): 1171-1179
- DOI: 10.13344/j.microbiol.china.170701
-
文章历史
- 收稿日期: 2017-09-06
- 接受日期: 2017-12-06
- 网络首发日期(www.cnki.net): 2017-12-26
2. 江南大学工业生物技术教育部重点实验室 江苏 无锡 214122
2. Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi, Jiangsu 214122, China
苯丙氨酸脱氨酶(Phenylalanine ammonia-lyase,PAL,EC:4.3.1.5)在pH 8.0−9.0条件下能催化L-苯丙氨酸非氧化脱氨形成反式肉桂酸,在pH 10.0−11.0以及浓氨条件下可催化逆向反应[1]。PAL可用于工业生产阿斯巴甜的前体L-苯丙氨酸[2]和通过拆分L, D-苯丙氨酸制备高光学纯度的D-苯丙氨酸[3],在治疗癌症和苯丙氨酸代谢障碍疾病苯丙酮尿症(Phenylketonuria,PKU)方面也展示了很好的潜力[4-6]。其中,鱼腥藻(Anabaena variabilis)来源的PAL (AvPAL)稳定性优异,包括热稳定性以及体内抵抗蛋白酶降解能力和极端肠胃pH能力,而且在体内引起的抗原性低,最具有医药应用的前景[6-7]。同时,AvPAL的底物谱宽,在合成绿色非天然手性氨基酸方面有较大的应用前景[8]。然而,AvPAL酶活显著低于真核生物来源的PAL,在一定程度上限制了该酶的应用[6]。
在4-Methylideneimidazol-5-one (MIO)依赖型酶中[9],催化中心的Tyr具有接收底物L-苯丙氨酸上的质子推进催化进行的作用,它所在的柔性区Tyr-loop对酶学性质起着相当重要的作用,该区的柔性在一定程度上直接影响这类酶的脱氨活性[6, 10-12]。比较AvPAL (PDB ID: 3CZO)和冬红酵母(Rhodotorula toruloides)来源的PAL (RtPAL,PDB ID:1T6J)以及欧芹(Petroselinum crispum)来源的PAL (PcPAL,PDB ID:1w27)的晶体结构,发现AvPAL的Tyr-loop比后两者的柔性都要低。与此同时,AvPAL的酶活比酵母和植物来源的PAL低[13-15]。另外,研究结果表明[10-11],通过提高反应温度以及定点突变等方式提高相应Tyr-loop的柔性,可以使苯丙氨酸氨基变位酶(Phenylalanine aminomutase,PAM,MIO依赖型酶)催化脱氨活性明显提高。但目前还没有通过改造苯丙氨酸脱氨酶相应的Tyr-loop来改变其催化性质的研究报道。
本研究组在前期研究中获得了鱼腥藻的AvPAL基因[16],在大肠杆菌中实现了高效表达并对其酶学性质进行了系统的研究。因为研究发现粘红酵母(Rhodotorula glutinis)来源RgPAL和玉米(Zea mays)来源ZmPAL的PAL酶活分别比AvPAL高出1倍和3倍[17],本研究通过比对AvPAL、RgPAL和ZmPAL的Tyr-loop氨基酸序列,将AvPAL的Tyr78-loop进行一系列改造,包括将整个Tyr78-loop替换成RgPAL的Tyr-loop,以及分别突变RgPAL和ZmPAL的Tyr-loop相应氨基酸位点的定点突变,以提高AvPAL的Tyr-loop柔性,最终提高其酶活性并研究突变体的酶学性质。
1 材料与方法 1.1 材料 1.1.1 菌种、质粒和引物菌种Escherichia coli JM109、E. coli BL21(DE3)购自Novangen公司;重组质粒pET28a(+)-pal由本实验室构建并保存。
本文所用引物由生工生物工程(上海)股份有限公司合成。
1.1.2 培养基LB培养基(g/L):胰蛋白胨10.0,酵母膏5.0,NaCl 10.0,琼脂粉20.0;2YT培养基(g/L):胰蛋白胨16.0,酵母膏10.0,NaCl 10.0,琼脂粉20.0。
1.1.3 主要试剂和仪器工具酶、DNA marker和Protein marker购自宝生物工程(大连)有限公司;质粒提取试剂盒购自天根生化科技有限公司;胶回收试剂盒、PCR产物纯化试剂盒及异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、卡那霉素购自生工生物工程(上海)股份有限公司;酵母提取物、蛋白胨购自Oxford公司;其余试剂均为国产分析纯。
结合缓冲液(mmol/L):Na2HPO4 50、NaH2PO4 50、NaCl 500、Imidazole 20。
AKTA蛋白纯化系统、HisTrap HP 1 mL镍柱和Superdex 75 10/300GL凝胶柱均为通用电气医疗器械集团产品。
1.2 方法 1.2.1 突变体的构建用软件Primer 5.0设计引物(表 1),以重组质粒pET28a(+)-pal为模板进行全质粒PCR。
| 引物名称 Primers name |
序列 Sequences(5′→3′) |
| Loop up | TGTGATTACATTAATAATGCTGTTGAATCTGACAACAGTG TCTACGGTGTCACGACCGGCTTCGGCGGCTCGGCAGACAC CCGGACGGAGGACGCGATCTCTGAACTCCAAACCAACTTA |
| Loop down | GAACCAAACTAAGTTGGTTTGGAGTTCAGAGATCGCGTCC CCGTCCGGGTGTCTGCCGAGCCGCCGAAGCCGGTCGTGA CACCGTAGACACTGTTGTCAGATTCAACAGCATTATTAAT |
| S73N up S73N down |
TACATTAATAATGCTGTTGAAAATGGGGAACCAATT CACTCCATAAATTGGTTCCCCATTTTCAACAGCATT |
| E75T up | AATAATGCTGTTGAATCTGGGACTCCAATTTATGGA |
| E75T down | AGATGTCACTCCATAAATTGGAGTCCCAGATTCAAC |
| P76D up | AATGCTGTTGAATCTGGGGAAGATATTTATGGAGTG |
| P76D down | ACCAGATGTCACTCCATAAATATCTTCCCCAGATTC |
| I77V up | GCTGTTGAATCTGGGGAACCAGTTTATGGAGTGACA |
| I77V down | AAAACCAGATGTCACTCCATAAACTGGTTCCCCAGA |
| I77S up | GCTGTTGAATCTGGGGAACCATCATATGGAGTGACA |
| I77S down | AAAACCAGATGTCACTCCATATGATGGTTCCCCAGA |
| I77A up | GCTGTTGAATCTGGGGAACCAGCATATGGAGTGACA |
| I77A down | AAAACCAGATGTCACTCCATATGCTGGTTCCCCAG |
| I77G up | GCTGTTGAATCTGGGGAACCAGGTTATGGAGTGACA |
| I77G down | AAAACCAGATGTCACTCCATAACCTGGTTCCCCAGA |
| S82T up | GAACCAATTTATGGAGTGACAACTGGTTTTGGCGGT |
| S82T down | ATTGGCCATACCGCCAAAACCAGTTGTCACTCCATA |
| M87S up | GTGACATCTGGTTTTGGCGGTTCAGCCAATGTTGCC |
| M87S down | ACGGGATATGGCAACATTGGCTGAACCGCCAAAACC |
| M87T up | GTGACATCTGGTTTTGGCGGTACTGCCAATGTTGCC |
| M87T down | ACGGGATATGGCAACATTGGCAGTACCGCCAAAACC |
| N89D up | TCTGGTTTTGGCGGTATGGCCGATGTTGCCATATCC |
| N89D down | TTGTTCACGGGATATGGCAACATCGGCCATACCGCCA |
| N89H up | TCTGGTTTTGGCGGTATGGCCCATGTTGCCATATCC |
| N89H down | TTGTTCACGGGATATGGCAACATGGGCCATACCGCCA |
| V90T up | GGTTTTGGCGGTATGGCCAATACTGCCATATCCCGT |
| V90T down | TGCTTGTTCACGGGATATGGCAGTATTGGCCATACC |
| V90R up | GGTTTTGGCGGTATGGCCAATCGTGCCATATCCCGT |
| V90R down | TGCTTGTTCACGGGATATGGCACGATTGGCCATACC |
| A91R up | TTTGGCGGTATGGCCAATGTTCGTATATCCCGTGAA |
| A91R down | AGATGCTTGTTCACGGGATATACGAACATTGGCCAT |
| I92T up | GGCGGTATGGCCAATGTTGCCACTTCCCGTGAACAA |
| I92T down | TTCAGATGCTTGTTCACGGGAAGTGGCAACATTGGC |
| S93E up | GGTATGGCCAATGTTGCCATAGAACGTGAACAAGCA |
| S93E down | GAGTTCAGATGCTTGTTCACGTTCTATGGCAACATT |
| S93R up | GGTATGGCCAATGTTGCCATACGTCGTGAACAAGCA |
| S93R down | GAGTTCAGATGCTTGTTCACGACGTATGGCAACATT |
| R94D up | ATGGCCAATGTTGCCATATCCGATGAACAAGCATCT |
| R94D down | TTGGAGTTCAGATGCTTGTTCATCGGATATGGCAAC |
| R94T up | ATGGCCAATGTTGCCATATCCACTGAACAAGCATCT |
| R94T down | TTGGAGTTCAGATGCTTGTTCAGTGGATATGGCAAC |
| E95A up | GCCAATGTTGCCATATCCCGTGCACAAGCATCTGAA |
| E95A down | GGTTTGGAGTTCAGATGCTTGTGCACGGGATATGGC |
| E95K up | GCCAATGTTGCCATATCCCGTAAACAAGCATCTGAA |
| E95K down | GGTTTGGAGTTCAGATGCTTGTTTACGGGATATGGC |
| E95V up | GCCAATGTTGCCATATCCCGTGTTCAAGCATCTGAA |
| E95V down | GGTTTGGAGTTCAGATGCTTGAACACGGGATATGGC |
| E95G up | GCCAATGTTGCCATATCCCGTGGTCAAGCATCTGAA |
| E95G down | GGTTTGGAGTTCAGATGCTTGACCACGGGATATGGC |
| Q96I up | AATGTTGCCATATCCCGTGAAATTGCATCTGAACTC |
| Q96I down | GTTGGTTTGGAGTTCAGATGCAATTTCACGGGATATG |
| Q96E up | AATGTTGCCATATCCCGTGAAGAAGCATCTGAACTC |
| Q96E down | GTTGGTTTGGAGTTCAGATGCTTCTTCACGGGATATG |
| A97S up | GTTGCCATATCCCGTGAACAATCATCTGAACTCCAA |
| A97S down | TAAGTTGGTTTGGAGTTCAGATGATTGTTCACGGGA |
| A97G up | GTTGCCATATCCCGTGAACAAGGTTCTGAACTCCAA |
| A97G down | TAAGTTGGTTTGGAGTTCAGAACCTTGTTCACGGGA |
PCR反应体系:5×PrimeSTAR buffer (5 mmol/L Mg2+ plus) 10 μL,dNTP mixture (2.5 mmol/L) 4 μL,上、下游引物(10 mmol/L)各1 μL,pET28a(+)-pal质粒模板(1 ng/μL) 1 μL,PrimeSTAR HS DNA polymerase (2.5 U/μL) 0.5 μL,超纯水补至50 μL。
PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,58 ℃ 30 s,72 ℃ 8 min,25个循环;72 ℃ 10 min;4 ℃保温。
将PCR产物用胶回收试剂盒纯化后用DpnⅠ消化除去模板。将消化后的质粒转化入E. coli JM109感受态细胞,在含有50 mg/L卡那霉素的LB固体培养基中37 ℃培养筛选阳性克隆菌株,并提取质粒转入E. coli BL21(DE3)感受态细胞。
1.2.2 AvPAL及其突变体的表达从平板上挑取单菌落,分别接种至5 mL含有卡那霉素(50 mg/L)的液体LB培养基,37℃、200 r/min培养8−10 h。取1 mL种子液接种至100 mL含有卡那霉素的2YT培养基中,37℃、200 r/min培养至菌体OD600为0.8−1.0,加入IPTG至终浓度为0.4 mmol/L,20 ℃诱导表达16−18 h后,5 000×g 离心10 min收集清洗菌体,并保存于−80℃备用。
1.2.3 AvPAL的纯化将重组菌体悬浮于20 mL结合缓冲液,冰水浴超声破碎(工作3 s,间隔7 s,工作时间40 min),13 000×g离心25 min除去不溶性杂质获得粗酶液,上清用0.22 μm滤膜过滤,用HisTrap HP的1 mL镍柱对粗酶液进行纯化。用10倍柱体积的结合缓冲溶液平衡镍柱,用20倍柱体积的结合缓冲液洗去非特异性吸附的蛋白,分别用8倍柱体积的100 mmol/L和300 mmol/L咪唑分别洗脱杂蛋白和目的蛋白,将目的蛋白进行透析除盐,最后收集样品用SDS-PAGE分析鉴定,并用Bradford方法[18]测定蛋白浓度。
1.2.4 AvPAL酶活测定将3 μg纯酶加入总反应体系为400 μL的含底物L-Phe终浓度为10 mmol/L的100 mmol/L pH 7.5 Tris-HCl缓冲液中,37 ℃反应20 min,加入400μL甲醇终止反应。用分光光度计在290 nm下测定吸光值,并通过标准曲线确定生成肉桂酸的量,计算PAL酶活。
酶活定义:每分钟催化生成1 μmol反式肉桂酸所需要的PAL量定义为1 U。
1.2.5 最适反应pH以及pH稳定性测定最适反应pH的测定:在总反应体系为400 μL的不同pH缓冲液中加入3 μg纯化后的酶,加底物L-苯丙氨酸至10 mmol/L,在37 ℃反应20 min,确定相应酶活,作出pH-酶活曲线,得到最适pH。不同pH缓冲溶液分别是:pH 6.0−7.0的100 mmol/L KH2CO3-K2HCO3缓冲液和pH 7.0−9.5的100 mmol/L Tris-HCl缓冲液。
pH稳定性的测定:将野生酶和突变体在不同pH缓冲溶液中37 ℃保存12 h后,在37 ℃、pH 8.5下测定残留酶活。
1.2.6 最适反应温度以及热稳定性测定最适反应温度的测定:测定纯酶在30、35、40、45、50、55、60及65 ℃下的酶活,确定最适反应温度。
热稳定性的测定:将野生酶和E75L于70 ℃保温,适时取样,测定残留酶活。
1.2.7 动力学参数测定在总反应体系为200 μL的含有不同底物浓度(0.01−0.03 mmol/L L-苯丙氨酸)的pH 8.5 Tris-HCl缓冲液中加入0.5 μg纯酶,在37℃利用酶标仪连续12 min监测生成的肉桂酸量。利用软件GraphPad Prism 5.0进行非线性拟合,计算动力学参数(Km、kcat)。
1.2.8 突变酶的结构分析以AvPAL (PDB ID:3CZO)为模板,利用SWISS- MODEL对突变酶进行同源建模,利用Discovery studio 2.0软件对蛋白质的三级结构进行分析。
2 结果与分析 2.1 AvPAL基因突变位点的选择及突变子筛选由于RgPAL的酶活显著高于AvPAL[17],将AvPAL的Tyr78-loop (73−97氨基酸)替换成RgPAL的Tyr-loop (105−127氨基酸)编码基因(图 1)。

|
| 图 1 PAL的Tyr-loop氨基酸序列比对 Figure 1 Alignment of amino acid sequences of the Tyr-loop of PALs 注:红框氨基酸为Tyr催化因子;黑框氨基酸不被替换. Note: The amino acids of red box is Tyr catalytic factor; The amino acids of black box is not be replaced. |
|
|
进一步将AvPAL、RgPAL和ZmPAL相应的Loop序列进行比对(图 1)。结果表明,AvPAL的78−85位(除了Ser82)均为保守氨基酸,而Loop两端的氨基酸序列不保守。将AvPAL上这些不保守位点突变为RgPAL或ZmPAL上相应的氨基酸。此外,AvPAL的Ile77和Glu95对应于RgPAL中相应位点处为空间位阻较小的Val和Ala,空间位阻小则多肽链可能灵活性更高,因此进一步将AvPAL的Ile77和Glu95分别突变为空间位阻较小的Gly、Ala或Val,构建了I77G、I77A、E95G以及E95V突变体。
初步测定突变酶粗酶液相对酶活的结果表明,AvPAL整个Loop替换后,酶活几乎完全丧失。根据反应机理,Tyr78为催化因子,整个Tyr78-loop替换后可能造成了空间结构的紊乱,使得Tyr78在催化过程中不能发挥其应有的接受底物上的一个质子促进催化反应的路易斯碱作用,从而使该突变体失去PAL酶活性。
将Tyr78-loop上的氨基酸--突变后,发现大部分突变体保持酶活(图 2),特别是两端氨基酸突变后保持的酶活较高,选择粗酶液酶活保持在原酶酶活80%以上的突变体进行纯化,进一步测定突变体S73N、I77G、R94D、R94T、E95A、E95K、E95V、E95G、Q96I、Q96E、A97S和A97G的纯酶酶活(图 3)。如图 3所示,S73N、E95V和E95K的纯酶酶活明显提高,对S73N、E95V和E95K进行组合突变构建的突变体S73N/E95V和S73N/E95K其相应纯酶的比活都有所提高(图 3)。在37 ℃、pH 8.5条件下,比活提高最明显的4个突变体S73N、E95V、E95K和S73N/E95K的酶活分别由原酶的1.57 U/mg提高到了2.11、2.04、1.85和2.12 U/mg,后续对这4个突变体展开研究。

|
| 图 2 突变酶粗酶液相对酶活 Figure 2 Relative activity of crude mutant enzymes |
|
|

|
| 图 3 突变酶纯酶液相对酶活 Figure 3 Relative activity of purified mutant enzymes |
|
|
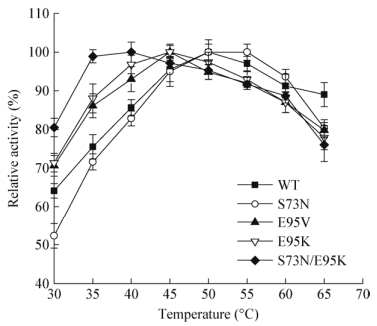
为了测定突变酶的最适反应温度,在pH 8.5的条件下,分别测定其在不同温度下(35−65 ℃)的酶活。如图 4所示,野生酶和S73N均在50℃时达到最高酶活,E95V和E95K在45℃时达到最高酶活,组合突变体S73N/E95K的最适反应温度较原酶降低10−40 ℃。与野生酶相比,突变体普遍在较低温度下就能达到高酶活,说明Tyr78-loop在较低温度下已经具有一定的灵活度。该结果与Chesters等[11]报道的在一定温度范围内,MIO依赖型酶随温度的升高Tyr-loop柔性增强、酶活提高的现象相一致。
|
| 图 4 突变酶的最适反应温度 Figure 4 The optimum temperature of mutant enzymes |
|
|
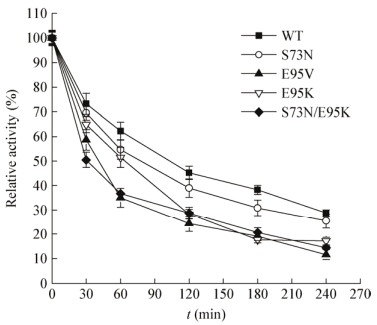
为了确定各酶的热稳定性,将稀释至相同浓度的酶液保存于70 ℃中,适时取样,测定残留酶活。从图 5可以看出,相对于野生酶,S73N、E95V、E95K以及组合突变S73N/E95K的热稳定性均有下降,半衰期由120 min分别降至了98、56、72和56 min。虽然AvPAL突变体的热稳定性有所降低,但是仍显著高于RgPAL的稳定性(RgPAL在60 ℃处理20 min仅保持10%的酶活)[15]。
|
| 图 5 突变酶的热稳定性 Figure 5 Thermal stability of mutant enzymes |
|
|
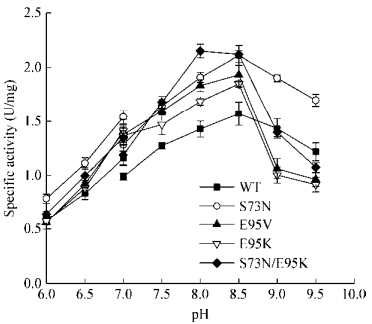
S73N、E95V、E95K以及组合突变体S73N/E95K的pH-活力曲线如图 6所示。S73N/E95K的最适反应pH向酸性范围偏移了0.5个单位至8.0,其他突变体的最适pH均未发生改变,但E95V和E95K在pH大于8.5时酶活急剧下降,说明Glu95突变体的活性对碱性pH比较敏感。
|
| 图 6 突变酶的最适反应pH Figure 6 The optimum pH of mutant enzymes |
|
|
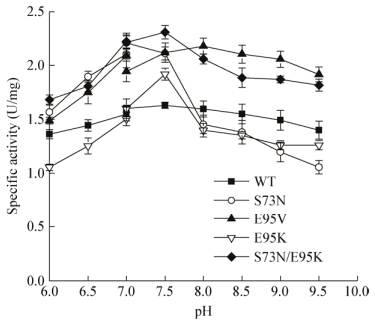
在37 ℃下,用不同pH值的缓冲液(6.5−9.5)分别处理S73N、E95V、E95K以及组合突变体S73N/E95K 12 h后测定酶活。结果如图 7所示,野生酶在pH 6.5−9.5条件下处理12 h后,野生酶均维持在80%以上,突变体E95V保持了野生酶较优异的pH稳定性,而其他突变体的pH稳定性均有一定程度的降低,其中E95K在酸性环境中的稳定性以及S73N在碱性环境中的稳定性下降明显。Loop交接处的Ser73和Glu95都属于表面氨基酸且位于活性中心周围,当把Ser73突变成Asn73时,局部疏水性减弱,推测活性中心附近表面区域疏水性的减弱使得蛋白更易受到周边不良环境的影响;同理,E95K的pH稳定性有所降低,而E95V保持了较好的稳定性[19]。
|
| 图 7 突变酶的pH稳定性 Figure 7 pH-stability of mutant enzymes |
|
|
在37℃下,以不同浓度的L-苯丙氨酸为底物测定突变酶的动力学参数。如表 2所示,与原始酶相比,S73N和S73N/E95K的Km几乎没有改变,但它们的kcat分别提高了26.7%和45.3%,最终催化效率kcat/Km分别提高了16.4%和27.2%。突变体E95V和E95K的Km分别提高了57.1%和35.0%,说明底物与酶的亲和性降低,而kcat却分别比突变前提高了93.9%和61.4%,在一定程度上抵消了底物与酶对催化效率kcat/Km的影响,最终催化效率分别比原酶增大了23.4%和7.0%。
| 酶 Enzyme |
Km (mmol/L) | kcat (s–1) | kcat/Km ((mmol·s)–1) |
| WT | 0.070±0.006 | 1.650±0.12 | 23.57 |
| E95V | 0.110±0.009 | 3.201±0.14 | 29.10 |
| E95K | 0.105±0.010 | 2.663±0.10 | 25.24 |
| S73N | 0.076±0.008 | 2.091±0.09 | 27.44 |
| S73N/E95K | 0.080±0.006 | 2.398±0.13 | 29.98 |
同源建模发现,在一定程度上,这4个突变体的Tyr78-loop柔性比原酶要大,主要由于Loop两端的氨基酸与附近氨基酸的成键情况发生了改变。如图 8A、B所示,在AvPAL原酶中,Ser73与Asn69形成2对极性作用力,突变后Asn73只与Asn69形成1对作用力,73位氨基酸刚好位于Loop与α螺旋的交接处,其与附近氨基酸作用力的改变使得Loop灵活性得到增加,甚至进一步带动了附近氨基酸的运动。同理,如图 8C、D所示,在AvPAL原酶中,Glu95与Ser93、Gln96共形成了3对作用力,而当其突变成Val或是Lys后,整个氨基酸并不与附近任何氨基酸成键,使得Loop末端的结构更加不稳定,柔性增加。Tyr78-loop灵活性的提高与其酶活性的提升(表 2)相对应,该结果也与文献[10-11]的报道结果相一致。同时,Loop柔性的增加在一定程度上也使得Tyr78 (路易斯碱)更容易随温度的升高而挣脱有效的空间位置,即蛋白的最适反应温度及热稳定性都有所降低(图 4、5)。
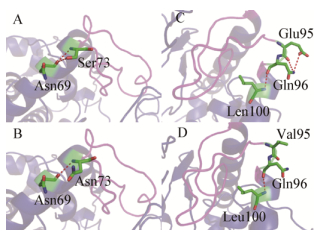
|
| 图 8 WT (A)、S73N (B)以及WT (C)、E95V (D)三维结构模拟 Figure 8 Simulated three-dimension structure of WT (A), S73N (B), WT (C) and E95V (D) |
|
|
现代工业PAL生产中应用较广泛的是真核生物粘红酵母来源的RgPAL和冬红酵母来源的RtPAL,但是其稳定性较原核生物来源的PAL差[16]。原核生物鱼腥藻来源的AvPAL稳定性等性能突出[6, 20-23],然而酶活性较真核生物来源的PAL低,一定程度上限制了其应用。
本研究将AvPAL的Tyr78-loop进行定点突变,分别突变为RgPAL和ZmPAL的Tyr-loop相应的氨基酸序列[17]。结果表明,相较于Tyr78-loop上的氨基酸残基,替换位于Tyr78-loop两端的Ser73位、Glu95位氨基酸可更有效地提高酶活(表 2)。这可能是因为位于Loop两端的氨基酸可影响整个Loop的摆动,能更有效地提高Loop的灵活性;而Loop上的氨基酸由于与Tyr78催化中心距离过近,其替换多导致酶活的降低甚至丧失。该结果为蛋白质Loop灵活性的改造以及MIO依赖型酶活性的改造提供了参考。
通过蛋白三级结构模拟发现,AvPAL的S733N和E95K突变均使Tyr78-loop的束缚力减少而灵活性增强(图 8),导致S733N/E95K突变体的最适反应温度降至40 ℃左右(图 4),更接近人体温度(37 ℃),更符合医药领域应用的需求。同时,Tyr78-loop灵活性的提高导致突变酶对热的耐受性降低(图 5),这也与PAL野生型酶中Tyr-loop灵活性与其热稳定性呈反相关的现象相一致。有研究表明,与AvPAL相比,RgPAL和ZmPAL的Tyr-loop具有较高灵活性,而其热稳定性低于AvPAL[13-15]。
此外,本实验室还系统改造了AvPAL活性中心Tyr78附近蛋白表面的氨基酸,成功将AvPAL的最适反应pH由8.5降至7.5,且酶活性比原酶提高了30%,热稳定性也显著提高[16, 24]。将这些突变位点与本研究结果进行组合,有望进一步改善AvPAL的性质,使其更适用于医药应用。
| [1] |
MacDonald MJ, D'Cunha GB. A modern view of phenylalanine ammonia lyase[J]. Biochemistry and Cell Biology, 2007, 85(3): 273-282. DOI:10.1139/O07-018 |
| [2] |
Miao YY, Liu CJ, Yang SK, et al. Core cDNA cloning and structural analysis of phenylalanine ammonia-lyase of Rhodotorula glutinis CIBAS A 1401[J]. Chinese Journal of Applied Environmental Biology, 2005, 11(6): 694-698. 缪元颖, 刘成君, 杨顺楷, 等. 粘红酵母(Rhodotorula glutinis CIBAS A 1401)苯丙氨酸解氨酶cDNA核心序列的克隆与分析[J]. 应用与环境生物学报, 2005, 11(6): 694-698. |
| [3] |
Huang JP, Yin RC, Zhang PP, et al. Method for manufacturing D-phenylalanine by Bio-enzyme asymmetric transformation: China, CN101671716[P]. 2010-03-17 (in Chinese) 黄建坡, 尹若春, 张萍萍, 等. 生物酶不对称转化生产D-苯丙氨酸的方法: 中国, CN101671716[P]. 2010-03-17 |
| [4] |
Babich OO, Pokrovsky VS, Anisimova NY, et al. Recombinant L-phenylalanine ammonia lyase from Rhodosporidium toruloides as a potential anticancer agent[J]. Biotechnology and Applied Biochemistry, 2013, 60(3): 316-322. DOI:10.1002/bab.2013.60.issue-3 |
| [5] |
Sarkissian CN, Gámez A, Wang L, et al. Preclinical evaluation of multiple species of PEGylated recombinant phenylalanine ammonia lyase for the treatment of phenylketonuria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(52): 20894-20899. DOI:10.1073/pnas.0808421105 |
| [6] |
Wang L, Gamez A, Archer H, et al. Structural and biochemical characterization of the therapeutic Anabaena variabilis phenylalanine ammonia lyase[J]. Journal of Molecular Biology, 2008, 380(4): 623-635. DOI:10.1016/j.jmb.2008.05.025 |
| [7] |
Kong JQ. ChemInform abstract: phenylalanine ammonia-lyase, a key component used for phenylpropanoids production by metabolic engineering[J]. ChemInform, 2015, 46(37): 62587-62603. |
| [8] |
Parmeggiani F, Lovelock SL, Weise NJ, et al. Synthesis of D-and L-phenylalanine derivatives by phenylalanine ammonia lyases: a multienzymatic cascade process[J]. Angewandte Chemie International Edition, 2015, 54(15): 4608-4611. DOI:10.1002/anie.201410670 |
| [9] |
Cooke HA, Christianson CV, Bruner SD. Structure and chemistry of 4-methylideneimidazole-5-one containing enzymes[J]. Current Opinion in Chemical Biology, 2009, 13(4): 460-468. DOI:10.1016/j.cbpa.2009.06.013 |
| [10] |
Bartsch S, Wybenga GG, Jansen M, et al. Redesign of a phenylalanine aminomutase into a phenylalanine ammonia lyase[J]. ChemCatChem, 2013, 5(7): 1797-1802. DOI:10.1002/cctc.v5.7 |
| [11] |
Chesters C, Wilding M, Goodall M, et al. Thermal bifunctionality of bacterial phenylalanine aminomutase and ammonia lyase enzymes[J]. Angewandte Chemie International Edition, 2012, 51(18): 4344-4348. DOI:10.1002/anie.201200669 |
| [12] |
Pilbák S, Tomin A, Rétey J, et al. The essential tyrosine-containing loop conformation and the role of the C-terminal multi-helix region in eukaryotic phenylalanine ammonia-lyases[J]. The FEBS Journal, 2006, 273(5): 1004-1019. DOI:10.1111/ejb.2006.273.issue-5 |
| [13] |
Moffitt MC, Louie GV, Bowman ME, et al. Discovery of two cyanobacterial phenylalanine ammonia lyases: kinetic and structural characterization[J]. Biochemistry, 2007, 46(4): 1004-1012. DOI:10.1021/bi061774g |
| [14] |
Ritter H, Schulz GE. Structural basis for the entrance into the phenylpropanoid metabolism catalyzed by phenylalanine ammonia-lyase[J]. The Plant Cell, 2004, 16(12): 3426-3436. DOI:10.1105/tpc.104.025288 |
| [15] |
Zhu LB, Cui WJ, Fang YQ, et al. Cloning, expression and characterization of phenylalanine ammonia-lyase from Rhodotorula glutinis[J]. Biotechnology Letters, 2013, 35(5): 751-756. DOI:10.1007/s10529-013-1140-7 |
| [16] |
Huang N, Zhu LB, Zhou L, et al. Gene cloning and expression of phenylalanine ammonia lyase from Anabaena variabilis and modification of the optimal reaction pH[J]. Microbiology China, 2015, 42(7): 1208-1215. 黄楠, 朱龙宝, 周丽, 等. 鱼腥藻苯丙氨酸脱氨酶的基因克隆、表达及最适反应pH改造[J]. 微生物学通报, 2015, 42(7): 1208-1215. |
| [17] |
Zang Y, Jiang T, Cong Y, et al. Molecular characterization of a recombinant Zea mays phenylalanine ammonia-Lyase (ZmPAL2) and its application in trans-cinnamic acid production from L-phenylalanine[J]. Applied Biochemistry and Biotechnology, 2015, 176(3): 924-937. DOI:10.1007/s12010-015-1620-4 |
| [18] |
Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [19] |
Martin A, Sieber V, Schmid FX. In-vitro selection of highly stabilized protein variants with optimized surface[J]. Journal of Molecular Biology, 2001, 309(3): 717-726. DOI:10.1006/jmbi.2001.4698 |
| [20] |
Kang TS, Wang L, Sarkissian CN, et al. Converting an injectable protein therapeutic into an oral form: phenylalanine ammonia lyase for phenylketonuria[J]. Molecular Genetics and Metabolism, 2010, 99(1): 4-9. DOI:10.1016/j.ymgme.2009.09.002 |
| [21] |
Heberling MM, Wu B, Bartsch S, et al. Priming ammonia lyases and aminomutases for industrial and therapeutic applications[J]. Current Opinion in Chemical Biology, 2013, 17(2): 250-260. DOI:10.1016/j.cbpa.2013.02.013 |
| [22] |
Bartsch S, Bornscheuer UT. Mutational analysis of phenylalanine ammonia lyase to improve reactions rates for various substrates[J]. Protein Engineering, Design and Selection, 2010, 23(12): 929-933. DOI:10.1093/protein/gzq089 |
| [23] |
Renard G, Guilleux JC, Bore C, et al. Synthesis of L-phenylalanine analogs by Rhodotorula glutinis. Bioconversion of cinnamic acids derivatives[J]. Biotechnology Letters, 1992, 14(8): 673-678. DOI:10.1007/BF01021641 |
| [24] |
Zhang F, Huang N, Zhou L, et al. Modulating the pH activity profiles of phenylalanine ammonia lyase from Anabaena variabilis by modification of center-near surface residues[J]. Applied Biochemistry and Biotechnology, 2017, 183(3): 699-711. DOI:10.1007/s12010-017-2458-8 |
 2018, Vol. 45
2018, Vol. 45




