扩展功能
文章信息
- 刘瑞华
- LIU Rui-Hua
- 林可霉素生物合成的研究进展
- Proceedings of lincomycin biosynthesis
- 微生物学通报, 2018, 45(5): 1138-1145
- Microbiology China, 2018, 45(5): 1138-1145
- DOI: 10.13344/j.microbiol.china.171039
-
文章历史
- 收稿日期: 2017-12-08
- 接受日期: 2018-02-12
- 网络首发日期(www.cnki.net): 2018-02-13
林可霉素(图 1)是林可链霉菌(Streptomyces lincolnensis)产生的林可酰胺类抗生素,作用于敏感菌核糖体,通过与50S亚基23S rRNA基因的中心环相结合,阻止肽链的延长,从而抑制细菌细胞的蛋白质合成。临床上,林可霉素主要用于治疗革兰氏阳性菌引起的感染性疾病。林可霉素还用于半合成制备克林霉素,是林可霉素的半合成的氯化衍生物,适用于革兰氏阳性菌和厌氧菌引起的各种感染性疾病,也可以用于治疗耐甲氧西林金黄色葡萄球菌引起的感染。克林霉素抗感染能力强、治疗范围广,被认为是20种最重要的抗生素之一[1]。从结构上看,林可霉素是由林可酰胺(Lincosamide,LSM)和丙基脯氨酸(Propylproline,PPL)两部分缩合并后修饰而成。近年来,林可霉素生物合成机理研究取得了实质性成果,生物合成途径日渐清晰。
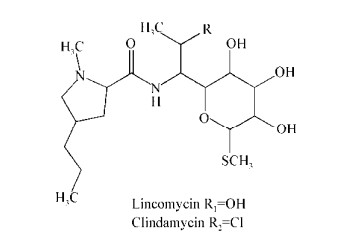
|
| 图 1 林可霉素和克林霉素分子结构 Figure 1 Structure of lincomycin and clindamycin |
|
|
1995年德国Peschke等克隆并测序了高产菌株林可链霉菌78-11的林可霉素生物合成基因簇(Lincomycin biosythetic gene cluster,lmb)[2]。2008年捷克Koběrská等对林可链霉菌ATCC25466的lmb进行测序[3],2017年上海交通大学白林泉等公开了林可链霉菌NRRL2936的基因组测序和lmb序列[4]。这3个菌株的lmb组织结构是基本一致的,有25或26个功能基因和调节基因、3个抗性基因,如图 2所示。序列分析显示,ATCC25466与NRRL2936中lmb的序列相似性为99%,但与林可链霉菌78-11相比,大多数lmb基因编码产物差异较小,个别基因差异很大。在林可链霉菌ATCC25466中lmbI和lmbH基因融合为一个基因lmbIH,lmbN、lmbT和lmbW基因序列变长了,lmrB在5ʹ-端截断而变短,而lmbD和lmbZ基因编码的氨基酸序列差异达11%。
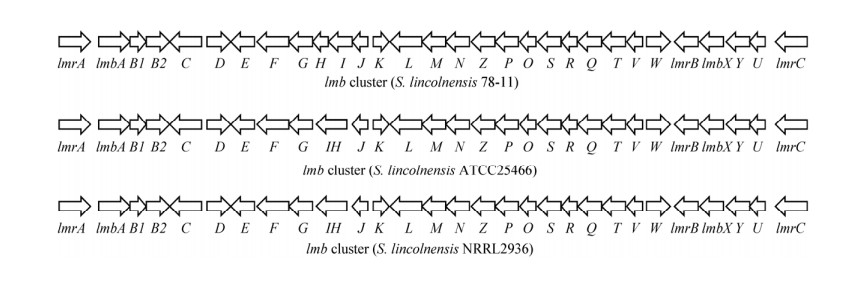
|
| 图 2 林可霉素的生物合成基因簇 Figure 2 The lincomycin biosynthetic gene clusters |
|
|
通过生物信息学分析、催化功能、遗传学研究,已经明确了lmb的部分基因功能,汇总结果见表 1。
| 生物合成 Biosynthesis |
基因名称 Gene name |
功能 Function |
| LSM | lmbR | Transaldolase, generating octulose |
| lmbN | Bio-functional enzyme, 1, 2-isomerase, generating 8-phosphate D-octose | |
| lmbP | Phosphate kinase, generating 1, 8-biphosphate octose | |
| lmbK | Phosphatase, generating 1-phosphate octose | |
| lmbO | Guanylyl transferase, generating GDP-octose | |
| lmbL | Putative UDP-glucose dehydrogenase | |
| lmbM | Putative glucose 4, 6-dehydrase | |
| lmbS | Putative aminotransferase | |
| lmbZ | Putative oxidoreductase | |
| PPL | lmbB2 | L-tyrosine-3-hydroxylase, generating L-dopa |
| lmbB1 | L-dopa-2, 3-dioxygenase, catalysing ring extradiol cleavage and intramolecular cyclization | |
| lmbW | C-methyltransferase, key to formation of lincomycin A | |
| lmbA | Glutamyltransferase, possibly catalyzing C–C bond cleavage | |
| lmbX | Putative isomerase | |
| lmbY | Putative F-420-dependent reductase | |
| lmbJ | N-methyltransferase for N-methylation of PPL or N-demethyl-LSM | |
| Condensation and post modification | lmbC | Adenylyltransferase for activation of PPL with ATP |
| lmbT | Glycosyltransferase for activation of LSM by EGT | |
| lmbN | Peptidyl carrier protein, condensation scaffold | |
| lmbD | Condensation enzyme for assembly of activated PPL and LSM | |
| lmbF | Pyridoxal-5ʹ-phosphate -dependent lyase, S–C bond cleavage | |
| lmbV | Glycosyltransferase, replacing EGT with MSH | |
| lmbE | Amidase, converting MSH S-conjugate into GlcN-Ins and mercapturic acid derivative | |
| lmbG | S-methyltransferase, S-methylation of lincosamide | |
| Resistance | lmrA | Transmembrance transporter, invovled in secretion of lincomycin |
| lmrB | 23S rRNA methyltransferase, increasing resistance of ribosome | |
| lmrC | ABC transporters, involved in exflux of lincomycin | |
| Regulation | lmbU | Positive regulation confirmed by genetic experiment |
| lmbIH | Putative regulation | |
| lmbQ | Putative regulation |
研究者对LSM生物合成做了很多研究。1984年Brahme等[5]通过同位素标记饲喂实验结合自旋偶联方法,提出由1个5-磷酸戊糖和1个7-磷酸景天庚酮糖经过碳链断裂和转移,生成1个4-磷酸赤藓糖和8-磷酸辛酮糖,而后8-磷酸辛酮糖经过多步反应而生成甲硫基林可酰胺(Methylthiolincosamide,MTL)。1995年Peschke等[2]通过对lmb的生物信息学分析,提出了可能参与MTL合成的基因。2012年Sasaki等[6]采用体外酶催反应确定了LSM合成中重要反应步骤及产物的构型。第一步由转醇醛酶LmbR催化,C5受体5-磷酸D-核糖和C3供体6-磷酸果糖或7-磷酸景天庚糖的缩合,生成8-磷酸辛酮糖。第二步由C1–C2异构化酶LmbN催化,生成中间产物8-磷酸辛糖。磷酸激酶LmbP催化8-磷酸辛糖的C1位羟基磷酸化,转化为1, 8-双磷酸辛糖。2014年Lin等[7]对LmbK和LmbO蛋白进行了催化反应功能研究,磷酸酶LmbK催化1, 8-双磷酸辛糖脱磷酸转化为1-磷酸辛糖;再经过lmbO编码的1-磷酸鸟苷酸转移酶将其转化为GDP-辛糖。随后发生转氨(LmbS)、脱水(LmbM)、还原(LmbZ)等反应,但还没有得到生化反应的证实。LSM生物合成途径见图 3。
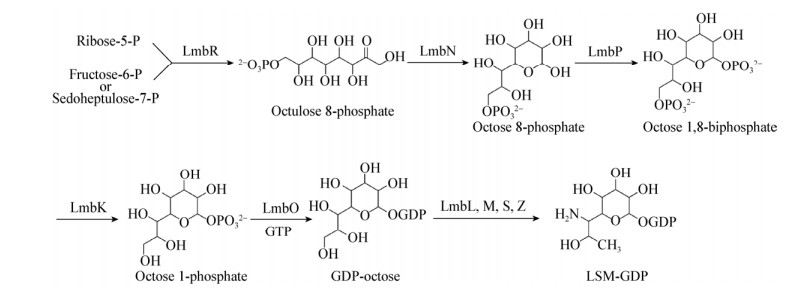
|
| 图 3 LSM生物合成途径 Figure 3 LSM biosynthetic pathway |
|
|
碳同位素标记显示,L-酪氨酸是PPL的合成前体。安曲霉素、托马霉素、西伯里亚霉素、胞诺斯霉素、侯马霉素结构中含有丙基脯氨酸(Propylproline,PPL)或其类似结构,由于具有较高的氨基酸同源性,推测lmbA、lmbB1、lmbB2、lmbW、lmbX、lmbY等参与PPL合成。
LmbB2是一种亚铁血红素型的L-酪氨酸羟化酶[8],催化PPL合成途径第一步反应,使L-酪氨酸3位羟基化,转变成L-多巴[9]。LmbB1是L-多巴邻位双加氧酶,催化PPL反应的第二步反应,使L-多巴2, 3位加氧裂解开环[10]。Colabroy等[11]通过停流技术、前稳定态、进度曲线等进一步研究了L-多巴双加氧酶的动力学四步反应机制。L-多巴首先被LmbB1酶催化成线性的半醛,然后环化为终产物,但遗憾是没有鉴定出该产物的结构[11-12]。
天津大学赵广荣教授课题组[13]通过基因敲除和回补实验表明,LmbW是第一个甲基转移酶,负责PPL中侧链的甲基转移反应。缺失lmbW的菌株,只能合成林可霉素B,没有林可霉素A。可见lmbW是林可霉素A和林可霉素B组分的决定基因。LmbY是氧化还原酶,参与PPL途径中双键的还原反应。敲除lmbX基因后,突变菌株不产林可霉素,其PPL的侧链中出现了不饱和双键;而加入PPL后,菌株又恢复了合成林可霉素的能力;加入丁基-脯氨酸和戊烷基-脯氨酸后,能合成丙基林可霉素和丙基戊烷基林可霉素[14]。Jiraskova等[15]对敲除lmbA、lmbX和lmbY基因菌株的中间代谢物进行了分析和结构鉴定,推定了可能的生化反应。如LmbW催化底物可能是脯氨酸双氢丙酮酸,LmbA可能催化醛酸链C–C键的裂解反应,LmbX可能是异构酶。综合截至目前的研究进展,提出了PPL的生物合成的基本途径(图 4)。

|
| 图 4 PPL生物合成途径 Figure 4 PPL biosynthetic pathway |
|
|
早期的研究推测是MTL与PPL缩合生成林可霉素,但近几年的实验显示,更有可能是LSM与PPL的缩合,然后是修饰。中国科学院上海有机化学研究所刘文教授课题组[16]研究了两种小分子硫醇分子的功能,解决了长期以来八碳糖单元是如何被活化、转移、硫来源及其修饰等问题。麦角硫因(Ergothioneine,EGT)作为载体,被转移酶LmbT催化,发生亲和置换反应,将林可酰胺从GDP上转移到EGT上[16],生成EGT S-林可酰胺,使LSM成为缩合的活性形式。PPL被活化成PPL-AMP,进入缩合反应。
LSM与PPL的缩合反应很独特,催化该反应的酶是非核糖体多肽酶[17]。lmbC基因编码的LmbC是腺苷化蛋白[18],使PPL活化形成PPL-AMP。lmbD基因编码的蛋白是缩合酶。LmbN蛋白在N末端有一个肽酰载蛋白结构域,起缩合反应载体支架的作用。分别敲除lmbC、lmbN和lmbD基因,发酵产物均为PPL和EGT-S林可酰胺。LmbC激活了PPL和ATP,并转移到LmbN的肽酰载蛋白结构域上。再由LmbD催化PPL和EGT S-林可酰胺的缩合[16],生成EGT S-结合物。
lmbV编码的蛋白属于DinB-2超级家族,拥有保守的类似DinB-2的结构域,参与不同低分子量硫醇的反应。敲除lmbV基因,突变菌株不再产林可霉素,而回补菌株一定程度上恢复了生产林可霉素的能力。由于LmbV蛋白难以纯化出来,用LmbV的同源蛋白CcbV (存在于天青菌素生物合成基因簇中,相似性57%)以缩合产物EGT S-结合物为底物,与放线硫醇(Mycothiol,MSH)进行体外反应实验,生成的产物为MSH S-结合物。因此可见LmbV是转移酶,将EGT置换为MSH[16]。MSH为林可霉素的合成提供了硫元素。
LmbE是一种酰胺酶,lmbE基因的敲除使突变菌株合成林可霉素的能力大大降低,但积累了MSH S-林可霉素衍生物。通过基因组分析和回补实验表明,林可霉素生物合成基因簇外的lmbE3457是lmbE的同功基因,其编码酶水解MSH S-结合物,生成硫醇尿酸基S-结合生物,释放氨基葡萄糖-肌醇(GlcN-Ins)。这证实了LmbE酶的水解作用,参与了MSH的再生过程[16]。硫醇尿酸基S-结合物如何脱去乙酰基生成半胱氨酰S-结合物仍然不清楚。
LmbJ是第二个甲基转移酶,失活lmbJ基因,阻断了突变株N-去甲基林可霉素的合成[19]。最近研究表明[20]以SAM为甲基供体,LmbJ催化生成半胱氨酰S-结合物中PPL的N-甲基化反应,生成N-甲基丙基脯氨酸(N-methylpropylproline)。
LmbF是磷酸吡哆醛依赖性酶[21-22],催化半胱氨酸S-结合物的S–C键切割反应,生成了高反应活性的硫醇中间体(去甲基林可霉素)以及副产物丙酮酸和氨。最后一步,在甲基转移酶LmbG催化下[21-22],以SAM为甲基供体,对巯基甲基化生成林可霉素A。LSM和PPL缩合及其后修饰生成林可霉素A的过程见图 5。
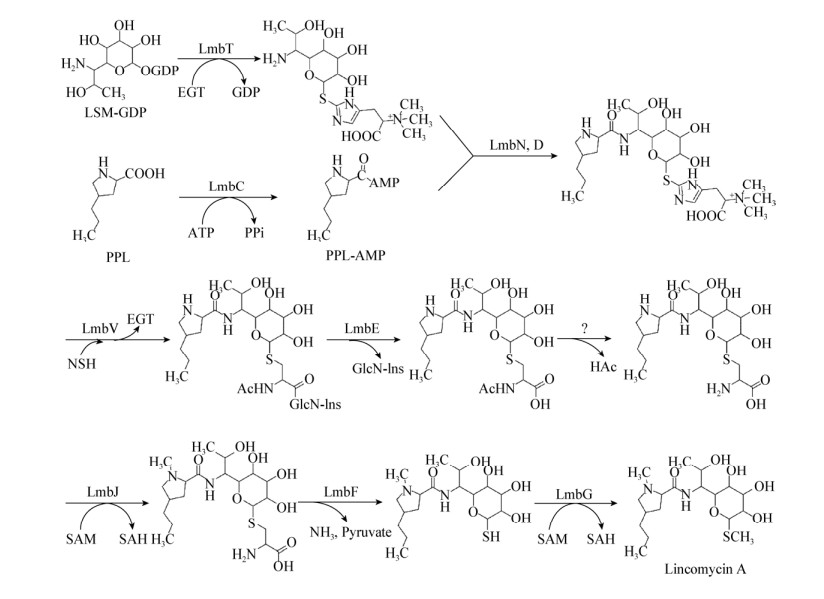
|
| 图 5 林可霉素A生物合成途径 Figure 5 Lincomycin A biosynthetic pathway from LSM and PPL |
|
|
林可霉素生物合成基因簇中除了结构基因,还包括3个可能的调控基因lmbU、lmbQ和lmbIH,以及3个抗性基因lmrA、lmrB和lmrC。
lmbU基因是一个正调控基因,与氨基香豆素类抗生素生物合成基因簇中的novE、couE有一定的同源性。华东理工大学张惠展教授研究组[23]敲除林可链霉菌中的lmbU基因,菌株不再产生林可霉素,表明lmbU基因是一个正调控基因。LmbU能激活lmbA、lmbC、lmbJ和lmbW基因的转录,阻遏lmbK和lmbU转录,是一个复杂的调控因子。由于lmbIH、lmbQ基因编码产物难以通过生物信息学预测其催化功能,从而推测可能具有调节功能[24]。
LmrA蛋白属于MFS超家族(Major facilitator superfamily),包括12个可能的跨膜疏水区域[25],负责将胞内合成的抗生素分泌到发酵液中。在变铅青霉菌中过表达lmrA基因,菌株能够产生林可霉素抗性。LmrB蛋白与核糖体50S亚基的23S rRNA基因的腺嘌呤N6上的单甲基化或双甲基化酶具有高度的相似性[25],可加强核糖体rRNA修饰,提高产生菌的自身抗性。LmrC蛋白属于ABC转运蛋白家族,包括2个ATP结合位点。我们实验证明了lmrC是抗性相关基因[26],当敲除lmrC后林可霉素产量显著下降,同时降低了抗性;而过表达lmrC能显著增强林可霉素的抗性。
6 结语与展望借助于基因敲除和过表达的体内研究策略,结合生化反应的离体研究,建立了林可霉素生物合成途径基本框架。林可霉素的两个直接前体林可酰胺和丙基脯氨酸合成过程的关键反应、缩合、硫来源、甲基化等已经明确,为林可霉素高产和高品质育种提供了理论指导。
在抗生素菌种改良中,增加主路通量和阻断支路是非常有效的策略。在磷酸戊糖和糖酵解途径的交汇处生成了林可霉素的两种前体。以5-磷酸核糖和6-磷酸果糖或7-磷酸景天庚酮糖为底物,发生转醛醇反应,生成磷酸辛酮糖,是整个林可酰胺合成的限制性步骤。相应地,4-磷酸赤藓糖和磷酸烯醇式丙酮酸缩合的芳香氨基酸途径合成了丙基脯氨酸。由于1分子林可酰胺和1分子丙基脯氨酸发生缩合反应,从糖代谢角度看,如何通过发酵平衡调控这两个前体合成是很具挑战性的工艺研究工作。已有研究表明[27]发酵培养基中添加丙酮酸和三甲胺,分别抑制了丙酮酸激酶基因和色氨酸生物合成基因的转录,提高了林可霉素产量。因此,丙酮酸激酶基因、色氨酸和苯丙氨酸生物合成支路基因,可作为高产林可霉素菌种改造的有效靶点。
林可霉素B的活性只有林可霉素A的25%,且毒性较大,是发酵的副产物,也是产品的杂质。国家药典规定,林可霉素B的含量超过林可霉素总含量的5%则不能用于药品生产。LmbW催化的甲基化反应是林可霉素A合成的决定性步骤,过表达lmbW和腺苷甲硫氨酸合成酶基因metK,不仅提高了林可霉素A的产量,同时有效降低了林可霉素B的含量[13]。
林可霉素生物合成基因簇中有3种抗性基因,其作用机理不同。我们研究表明[28]过表达3个抗性基因,都能提高林可霉素产量。因此,针对工业生产菌种增加抗性基因拷贝数,将是提高产量的一条有效技术途径。
林可霉素发酵过程,加大供氧不仅增加能耗,而且使酪氨酸生成黑色素,减少了林可霉素前体的供应。我们采用基因替换技术[29],将透明颤菌血红蛋白基因取代黑色素合成基因,不仅提高了产量,而且简化下游除色素的工艺。
BldD是链霉菌中普遍存在的形态分化及抗生素合成的正调控因子[30],糖多孢红霉菌bldD基因不仅可提高红霉素产量[31],还能提高跳跃链霉菌合成诺西肽的产量[32]。已有林可链霉菌基因组被测序,但全局调控因子未见报道。
在未来的研究中,需要鉴定出包括BldD在内的全局转录调控因子,确认LmbIH和LmbQ的途径调控功能,从全局和途径特异性两个维度研究林可霉素生物合成的调控机理。深度解析林可霉素生物合成过程的精细生化反应机理,如在PPL合成中的LmbW和LmbA、在LSM合成中的LmbLMSZ、PPL和LSM缩合后的修饰反应。针对林可霉素工业发酵过程特点和技术需求,采用新兴的合成生物学技术,通过基因组的精准编辑,把前体供应、抗性、供氧、层级调控等关键基因进行多效叠加,将理论研究成果应用于企业实际,可望减少林可霉素B组分,进一步提高我国林可霉素产量和质量。
| [1] |
Spizek J, Rezanka T. Lincosamides: chemical structure, biosynthesis, mechanism of action, resistance, and applications[J]. Biochemical Pharmacology, 2017, 133: 20-28. DOI:10.1016/j.bcp.2016.12.001 |
| [2] |
Peschke U, Schmidt H, Zhang HZ, et al. Molecular characterization of the lincomycin-production gene cluster of Streptomyces lincolnensis 78-11[J]. Molecular Microbiology, 1995, 16(6): 1137-1156. DOI:10.1111/mmi.1995.16.issue-6 |
| [3] |
Koběrská M, Kopeck J, Olšovská J, et al. Sequence analysis and heterologous expression of the lincomycin biosynthetic cluster of the type strain Streptomyces lincolnensis ATCC 25466[J]. Folia Microbiologica, 2008, 53(5): 395-401. DOI:10.1007/s12223-008-0060-8 |
| [4] |
Meng ST, Wu H, Wang L, et al. Enhancement of antibiotic productions by engineered nitrate utilization in actinomycetes[J]. Applied Microbiology and Biotechnology, 2017, 101(13): 5341-5352. DOI:10.1007/s00253-017-8292-7 |
| [5] |
Brahme NM, Gonzalez JE, Mizsak S, et al. Biosynthesis of the lincomycins. 2. Studies using stable isotopes on the biosynthesis of methylthiolincosaminide moiety of lincomycin A[J]. Journal of the American Chemical Society, 1984, 106(25): 7878-7883. DOI:10.1021/ja00337a039 |
| [6] |
Sasaki E, Lin CI, Lin KY, et al. Construction of the octose 8-phosphate intermediate in lincomycin A biosynthesis: characterization of the reactions catalyzed by LmbR and LmbN[J]. Journal of the American Chemical Society, 2012, 134(42): 17432-17435. DOI:10.1021/ja308221z |
| [7] |
Lin CI, Sasaki E, Zhong AS, et al. In vitro characterization of lmbK and lmbO: identification of GDP-D-erythro-α-D-gluco-octose as a key intermediate in lincomycin A biosynthesis[J]. Journal of the American Chemical Society, 2014, 136(3): 906-909. DOI:10.1021/ja412194w |
| [8] |
Novotna J, Olsovska J, Novak P, et al. Lincomycin biosynthesis involves a tyrosine hydroxylating heme protein of an unusual enzyme family[J]. PLoS One, 2013, 8(12): e79974. DOI:10.1371/journal.pone.0079974 |
| [9] |
Neusser D, Schmidt H, Spizèk J, et al. The genes lmbB1 and lmbB2 of Streptomyces lincolnensis encode enzymes involved in the conversion of L-tyrosine to propylproline during the biosynthesis of the antibiotic lincomycin A[J]. Archives of Microbiology, 1998, 169(4): 322-332. DOI:10.1007/s002030050578 |
| [10] |
Novotná J, Honzátko A, Bednář P, et al. L-3, 4-dihydroxyphenyl alanine-extradiol cleavage is followed by intramolecular cyclization in lincomycin biosynthesis[J]. European Journal of Biochemistry, 2004, 271(18): 3678-3683. DOI:10.1111/ejb.2004.271.issue-18 |
| [11] |
Colabroy KL, Smith IR, Vlahos AHS, et al. Defining a kinetic mechanism for L-DOPA 2, 3 dioxygenase, a single-domain type Ⅰ extradiol dioxygenase from Streptomyces lincolnensis[J]. Biochimica et Biophysica Acta, 2014, 1844(3): 607-614. DOI:10.1016/j.bbapap.2013.12.005 |
| [12] |
Colabroy KL, Hackett WT, Markham AJ, et al. Biochemical characterization of L-DOPA 2, 3-dioxygenase, a single-domain type Ⅰ extradiol dioxygenase from lincomycin biosynthesis[J]. Archives of Biochemistry and Biophysics, 2008, 479(2): 131-138. DOI:10.1016/j.abb.2008.08.022 |
| [13] |
Pang AP, Du L, Lin CY, et al. Co-overexpression of lmbW and metK led to increased lincomycin A production and decreased byproduct lincomycin B content in an industrial strain of Streptomyces lincolnensis[J]. Journal of Applied Microbiology, 2015, 119(4): 1064-1074. DOI:10.1111/jam.12919 |
| [14] |
Ulanova D, Novotna J, Smutna Y, et al. Mutasynthesis of lincomycin derivatives with activity against drug-resistant Staphylococci[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(2): 927-930. DOI:10.1128/AAC.00918-09 |
| [15] |
Jiraskova P, Gazak R, Kamenik Z, et al. New concept of the biosynthesis of 4-alkyl-L-proline precursors of lincomycin, hormaomycin, and pyrrolobenzodiazepines: Could a γ-glutamyltransferase cleave the C–C Bond?[J]. Frontiers in Microbiology, 2016, 7: 276. |
| [16] |
Zhao QF, Wang M, Xu DX, et al. Metabolic coupling of two small-molecule thiols programs the biosynthesis of lincomycin A[J]. Nature, 2015, 518(7537): 115-119. DOI:10.1038/nature14137 |
| [17] |
Janata J, Kadlcik S, Koberska M, et al. Lincosamide synthetase-a unique condensation system combining elements of nonribosomal peptide synthetase and mycothiol metabolism[J]. PLoS One, 2015, 10(3): e0118850. DOI:10.1371/journal.pone.0118850 |
| [18] |
Kadlčík S, Kučera T, Chalupská D, et al. Adaptation of an L-proline adenylation domain to use 4-propyl-L-proline in the evolution of lincosamide biosynthesis[J]. PLoS One, 2013, 8: e0084902. |
| [19] |
Najmanová L, Kutejová E, Kadlec J, et al. Characterization of N-demethyllincosamide methyltransferases LmbJ and CcbJ[J]. ChemBioChem, 2013, 14(17): 2259-2262. DOI:10.1002/cbic.v14.17 |
| [20] |
Kamenik Z, Kadlcik S, Radojevic B, et al. Deacetylation of mycothiol-derived 'waste product' triggers the last biosynthetic steps of lincosamide antibiotics[J]. Chemical Science, 2016, 7(1): 430-435. DOI:10.1039/C5SC03327F |
| [21] |
Wang M, Zhao QF, Zhang QL, et al. Differences in PLP-dependent cysteinyl processing lead to diverse S-functionalization of lincosamide antibiotics[J]. Journal of the American Chemical Society, 2016, 138(20): 6348-6351. DOI:10.1021/jacs.6b01751 |
| [22] |
Ushimaru R, Lin CI, Sasaki E, et al. Characterization of enzymes catalyzing transformations of cysteine S-conjugated intermediates in the lincosamide biosynthetic pathway[J]. ChemBioChem, 2016, 17(17): 1606-1611. DOI:10.1002/cbic.v17.17 |
| [23] |
Hou BB, Lin YW, Wu HZ, et al. The novel transcriptional regulator lmbU promotes lincomycin biosynthesis through regulating expression of its target genes in Streptomyces lincolnensis[J]. Journal of Bacteriology, 2018, 200(2): e00447-17. |
| [24] |
Janata J, Najmanová L, Novotná J, et al. Putative lmbI and lmbH genes form a single lmbIH ORF in Streptomyces lincolnensis type strain ATCC25466[J]. Antonie Van Leeuwenhoek, 2001, 79(3/4): 277-284. DOI:10.1023/A:1012034421088 |
| [25] |
Zhang HZ, Schmidt H, Piepersberg W. Molecular cloning and characterization of two lincomycin-resistance genes, lmrA and lmrB, from Streptomyces lincolnensis 78-11[J]. Molecular Microbiology, 1992, 6(15): 2147-2157. DOI:10.1111/mmi.1992.6.issue-15 |
| [26] |
Xu JJ, Wu H, Meng ST, et al. Functional analysis of lincomycin transporter gene lmrC in Streptomyces lincolnensis LC-G[J]. Journal of Shanghai Jiaotong University, 2014, 48(2): 159-163. 徐晶晶, 吴杭, 孟思童, 等. 林可霉素转运蛋白基因lmrC的功能分析[J]. 上海交通大学学报, 2014, 48(2): 159-163. |
| [27] |
Zhang HD, Ye RF, Mao QG, et al. Lincomycin fermentation in response to alanine and trimethylamine application and transcription level of related gene[J]. Jiangsu Journal of Agricultural Sciences, 2014, 30(5): 1003-1009. 张海丹, 叶蕊芳, 毛全贵, 等. 丙氨酸和三甲胺对林可霉素发酵的影响及部分相关基因的转录水平[J]. 江苏农业学报, 2014, 30(5): 1003-1009. |
| [28] |
Du L, Liu RH, Ying L, et al. An efficient intergeneric conjugation of DNA from Escherichia coli to mycelia of the lincomycin-producer Streptomyces lincolnensis[J]. International Journal of Molecular Sciences, 2012, 13(4): 4797-4806. DOI:10.3390/ijms13044797 |
| [29] |
Yang HT, Liu RH, He JY, et al. Cloning of Vitreoscilla hemoglobin gene and its expression in Streptomyces lincolnensis[J]. Journal of Shenyang Pharmaceutical University, 2009, 26(8): 657-662. 杨洪涛, 刘瑞华, 何建勇, 等. 透明颤菌血红蛋白基因的克隆及其在林可链霉菌中的表达[J]. 沈阳药科大学学报, 2009, 26(8): 657-662. |
| [30] |
Chng CP, Lum AM, Vroom JA, et al. A key developmental regulator controls the synthesis of the antibiotic erythromycin in Saccharopolyspora erythraea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(32): 11346-11351. DOI:10.1073/pnas.0803622105 |
| [31] |
He JJ, Huang XD, Song P, et al. Effect of bldD gene overexpression on erythromycin output and spore formation of Saccharopolyspora erythraea[J]. Bulletin of the Academy of Military Sciences, 2010, 34(3): 251-254. 何晶晶, 黄训端, 宋平, 等. bldD基因过量表达对红色糖多孢菌红霉素产量及孢子形成的影响[J]. 军事医学科学院院刊, 2010, 34(3): 251-254. |
| [32] |
Qin HJ, Huang XD, Yuan L, et al. Effects of bldD gene from Saccharopolyspora erythraea on nosiheptide production in Streptomyces actuosus[J]. Militory Medicine Sciences, 2012, 36(11): 847-850. 秦汉俊, 黄训端, 袁莉, 等. 糖多孢红霉菌bldD基因提高活跃链霉菌诺西肽产量的研究[J]. 军事医学, 2012, 36(11): 847-850. |
 2018, Vol. 45
2018, Vol. 45




