扩展功能
文章信息
- 林秋健, 郑平, 丁阿强
- LIN Qiu-Jian, ZHENG Ping, DING A-Qiang
- 羧酶体的组成、结构、功能和检测及其在脱氮细菌中的意义
- Composition, structure, function and detection of carboxysome and their significance to nitrogen-removal bacteria
- 微生物学通报, 2018, 45(5): 1127-1137
- Microbiology China, 2018, 45(5): 1127-1137
- DOI: 10.13344/j.microbiol.china.170593
-
文章历史
- 收稿日期: 2017-07-31
- 接受日期: 2017-12-01
- 网络首发日期(www.cnki.net): 2017-12-27
氮素污染是水体富营养化的主要原因,生物脱氮已成为废水处理和水体修复的重要手段。由于处理效果好、运行成本低、二次污染风险小,自养型生物脱氮广受青睐[1-3]。自养型脱氮细菌是自养型生物脱氮技术的功能之源,它可将有污染的氮素逐步转化为无污染的氮气,如硝化细菌可将氨氮转化为亚硝酸盐和硝酸盐,继而反硝化细菌将亚硝酸盐和硝酸盐转化为氮气;厌氧氨氧化细菌则可利用氨氮和亚硝酸盐直接转化为氮气。研究自养型脱氮细菌将有助于强化自养型生物脱氮过程。
碳素是构成细胞的主要元素,也是支持自养型脱氮细菌生长的基石。自养型脱氮细菌的生长有赖于其对CO2的同化,在低C/N废水或无机废水中,CO2供应不足容易成为自养型脱氮细菌生长的瓶颈。羧酶体(Carboxysome)是一种具有CO2浓缩功能的“类细胞器”,它存在于部分自养型微生物中,通过浓缩CO2,可促进自养型脱氮细菌的生长和代谢。探明羧酶体的组成、结构和功能,并建立适合的检测方法,将推进自养型细菌的研究与应用。
本文基于文献检索和自身研究,对细菌羧酶体的组成、结构、功能、检测等内容进行综述,探讨羧酶体对自养型脱氮细菌,尤其是对厌氧氨氧化细菌的意义,以供同行参考。
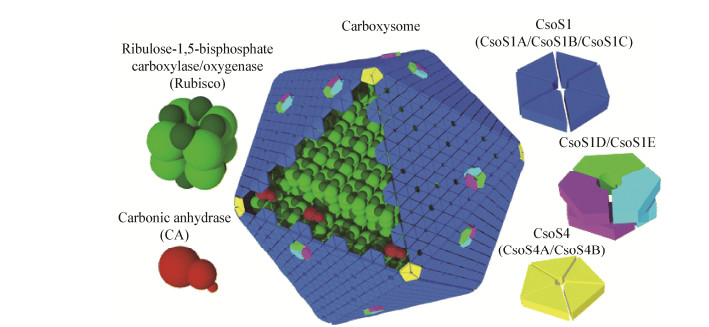
1 羧酶体的组成羧酶体是一种广泛存在于原核细胞中类似真核细胞细胞器的结构,又称为“类细胞器”结构,它具有蛋白质外壳组成的正面体结构,内部又含有Rubisco酶、碳酸酐酶等多种功能酶。1956年羧酶体被首次发现存在于钩状席藻(Phormidium uncinatum)中,目前已发现存在羧酶体的细菌涵盖了23个门[4]。根据羧酶体内部所含酶的种类不同,羧酶体可分为两类,即α-羧酶体(α-Carboxysome)和β-羧酶体(β-Carboxysome)。迄今为止,对羧酶体的研究大多集中在模式细菌(那不勒斯硫杆菌Halothiobacillus neapolitanus和集胞球藻Synechocystis sp. PCC 6803)上[5]。H. neapolitanus含有典型的α-羧酶体,Synechocystis sp. PCC 6803则含有典型的β-羧酶体。α-羧酶体存在于多种化能自养型细菌中[6](如自养型硝化和反硝化细菌常含有α-羧酶体结构),而β-羧酶体仅存在于蓝细菌内。据报道,α-羧酶体和β-羧酶体的功能并无显著差别[5]。
羧酶体的主要成分是蛋白质,不含脂质[7-10]。普遍认为羧酶体中不含核酸[11]。
1.1 羧酶体中的蛋白质羧酶体的内含物和外壳均由蛋白质组成。羧酶体至少含有12种蛋白质,其中核酮糖-1, 5-二磷酸羧化酶——Rubisco酶(羧酶体内含物)和一种糖蛋白(羧酶体外壳)占羧酶体总质量的60%以上[10]。据Heinhorst等报道,Rubisco酶的亚基CbbL (51 kD)和CbbS (9 kD)可占羧酶体总质量的70%以上;在每个羧酶体内,Rubisco酶的数量约270个[12],另一种内含酶CsoSCA (又名CsoS3,60 kD)也约有80个[13],占羧酶体总质量的2%[14]。组成羧酶体外壳的3种主要蛋白是CsoS1A (5 kD)、CsoS1B (15 kD)和CsoS1C (5 kD),约占羧酶体总质量的17%[15]。在每个羧酶体中,CsoS1A、CsoS1B、CsoS1C共约3 510个,Rubisco酶的大小亚基共4 320个[5]。近年来发现,CsoS1D (33 kD)、CsoS1E [16]、CsoS2A (85 kD)和CsoS2B (130 kD)也是组成羧酶体外壳的成分[17],但占比相对较少[18]。CsoS4A和CsoS4B是羧酶体结构的顶点蛋白,分布在羧酶体正二十面体的12个顶点上,分子质量约为11 kD[19]。
组成羧酶体的主要蛋白质种类见表 1。除了表 1所列的几种主要组成蛋白外,在羧酶体中还发现了一些含量很少但不可或缺的辅助蛋白。这些辅助蛋白有助于羧酶体的组装和功能的发挥。
| 编码基因 Genes |
编码蛋白 Proteins |
分子量 Molecular weight (kD) |
蛋白质数量 Protein numbers |
备注 Comments |
| cbbS | CbbS | 9 | 2 160 | Rubisco酶大亚基 Large subunit of Rubisco |
| cbbL | CbbL | 51 | 2 160 | Rubisco酶小亚基 Small subunit of Rubisco |
| csoS1A | CsoS1A | 5 | 共约3 510 Total 3 510 |
六聚体壳蛋白 Hexameric shell protein |
| csoS1B | CsoS1B | 15 | ||
| csoS1C | CsoS1C | 5 | ||
| csoS1D | CsoS1D | 33 | - - |
伪六聚体双层壳蛋白 Pseudo-hexameric shell protein |
| csoS1E | CsoS1E | 33 | ||
| csoS2 | CsoS2A | 85 | 143 | 外壳蛋白Shell protein |
| csoS2 | CsoS2B | 130 | 186 | |
| csoS3 | CsoSCA | 60 | 80 | 二聚体碳酸酐酶 Dimeric carbonic anhydrase |
| csoS4A csoS4B |
CsoS4A CsoS4B |
11 11 |
共约60 Total 60 |
五聚体壳蛋白 Pentameric shell protein |
| 注:蛋白质数量为组成羧酶体的各种蛋白质在一个羧酶体中的平均数量. -:未提及.
Note: The values of protein numbers in carboxysome is the average. –: Non-described. |
||||
真核细胞的细胞器(如叶绿体和线粒体)含有核酸。作为原核生物的类细胞器,羧酶体是否也含有核酸呢?1979年,Westphal等从硝化杆菌Nitrobacter winogradskyi的羧酶体中提取了总长约14 μm的环状DNA[20]。但7年后Holthuijzen等推翻了羧酶体含有核酸的结论,他们认为Westphal等检测到的DNA是粘附在羧酶体外壳上的细胞质DNA[21]。现已达成共识,羧酶体中不含有核酸。
2 羧酶体的结构和功能羧酶体具有正二十面体的几何结构(图 1),大小为100-160 nm,质量为100-350 MD [23]。羧酶体蛋白主要有外壳蛋白和内含酶蛋白。外壳蛋白CsoS1A、CsoS1B、CsoS1C、CsoS1D、CsoS1E、CsoS4A和CsoS4B有序排列成面,构成羧酶体外壳。外壳厚度约为4 nm[24],不足羧酶体直径的5%。羧酶体内部包裹着Rubisco羧化酶、CA酶(碳酸酐酶)和一些辅助蛋白,各蛋白结构与功能见表 2。
| 分类 Classification |
名称 Name |
一级结构 Primary structure |
二级结构 Secondary structure |
主要功能 Main functions |
|
| α螺旋 α-Helices |
β折叠 β-Strands |
||||
| 外壳蛋白 Shell protein |
CsoS1A | 98 | 3 | 4 | 组成羧酶体外壳,并控制物质进出 Form carboxysome shell and control the substrate in and out of the carboxysome |
| CsoS1B | 110 | ||||
| CsoS1C | 98 | ||||
| CsoS1D | 256 | 6 | 8 | ||
| CsoS1E | 200 | ||||
| CsoS2A | 870 | - | - | 羧酶体组装Assemble carboxysome | |
| CsoS2B | 870 | ||||
| CsoS4A | 82 | 1 | 5 | 组成羧酶体外壳,并控制物质进出 Form carboxysome shell and control the substrate in and out of the carboxysome |
|
| CsoS4B | 82 | ||||
| 内含酶蛋白 Encapsulated enzyme |
CsoSCA | 514 | 8 | 10 | 催化碳酸氢盐产生二氧化碳 Catalyze bicarbonate to carbon dioxide |
| Rubisco | 4 784 | 96 | 136 | 固定二氧化碳Fix carbon dioxide | |
| 辅助蛋白 Accessory protein |
CsoCbbQ | 270 | 9 | 5 | ATP酶ATPase |
| CPII | 194 | 6 | 12 | 感知环境中的碳酸氢盐 Sense bicarbonate availability |
|
| acRAFs | 164 | - | - | 羧酶体组装Assemble carboxysome | |
| 注:一级结构指氨基酸数量,二级结构指α螺旋、β折叠结构数量;-:未提及.
Note: The values of primary structure indicate the total number of amino acids in each protein, the values of secondary structure indicate the total number of α-helices/β-strands in each protein; -: Non-described. |
|||||
CsoS1是羧酶体外壳的主要成分。CsoS1A和CsoS1C的初级结构各含98个氨基酸残基,而CsoS1B的初级结构含有110个氨基酸残基。CsoS1A、CsoS1B、CsoS1C之间的氨基酸序列相似性高达80%[25]。CsoS1A、CsoS1B、CsoS1C蛋白均含有3个α螺旋和4个β折叠结构,其相互之间的结构和功能极为相似。6个CsoS1A、CsoS1B或CsoS1C单体组成一个六聚体,每个六聚体中间有直径约4 Å的小孔,可控制碳酸氢盐(HCO3-)、甘油酸-3-磷酸(3-PGA)和1, 5-二磷酸核酮糖(RUBP)等进出羧酶体[25]。这些小孔含有带正电的赖氨酸和精氨酸残基,使得带负电的碳酸氢盐(HCO3-)相对于不带电的CO2和O2而言更易被转运。小孔周围还有一层水化层,导致CO2和O2较难进入羧酶体[26]。这些六聚体具有凹凸正反面结构,凹凸一致的成分结合形成平面,再构成羧酶体的面状结构[27]。羧酶体的外壳不仅有构成羧酶体基本骨架的功能,还有控制物质进出的功能。CsoS1D和CsoS1E也是羧酶体外壳的蛋白成分,它们首先被发现于原绿球藻中[17]。CsoS1D约有256个氨基酸残基,CsoS1E约有200个氨基酸残基。CsoS1D、CsoS1E包含一对蛋白结构域:羧基端和氨基端[28]。两者在结构和功能上十分相似。CsoS1D、CsoS1E是三聚体,三聚体两两聚合,形成类似CsoS1A组成的六聚体的伪六聚体(图 1),伪六聚体两两堆叠,形成双层壳蛋白。这种双层蛋白的孔径约为14 Å,允许大分子物质进出羧酶体,但独特的双层构造可通过侧链的旋转开启或关闭小孔,对大分子物质有一定的选择性[28]。因此,CsoS1D、CsoS1E在控制物质进出羧酶体中起着重要且独特的作用[29]。
CsoS2对羧酶体的形成和维持起着重要的作用,csoS2基因被敲除后则无法形成羧酶体。csoS2基因通过移码突变[30]可以编码CsoS2A和CsoS2B两种结构和功能不同的蛋白。用电镜观察发现,CsoS2大多位于羧酶体的边缘,其主要成分是CsoS2B[30]。CsoS2有3个蛋白结构域:氨基端、中段和羧基端。中段的结构较氨基端和羧基端的结构复杂,这是因为CsoS2中段的结构形式具有很高的灵活性,一般都有1-2个α螺旋和4-6个β折叠[31]。α-羧酶体通过CsoS2B羧基端的作用,与羧酶体外壳紧密结合于羧酶体外壳内部边缘,并通过CsoS2B、CsoS2A与Rubisco酶之间的相互作用,将Rubisco酶封装在羧酶体中。在羧酶体组装过程中,β-羧酶体恰恰相反,其先通过Rubisco酶的自聚集成核,再将外壳蛋白连接成平面[32-33]。
CsoS4A、CsoS4B是组成羧酶体外壳的顶点蛋白。CsoS4A单体通过羧基末端相互结合,组成对称的五面体。五面体边长约42 Å,厚度约30-35 Å,圆盘中心有一个直径约3.5 Å的圆孔。同源蛋白CsoS4A和CsoS4B结构相似,CsoS4A、CsoS4B位于羧酶体二十面体的顶点。若敲除csoS4A基因,则会形成棒状羧酶体或不含顶点的正二十面体的羧酶体[5]。这些形态结构异常的羧酶体对CO2的固定效率很低[34]。通过现代生物信息学手段发现,在细菌编码羧酶体外壳蛋白的基因序列之间常包含编码Rubisco酶活化酶的基因[4]。这说明羧酶体外壳除了有作为骨架和控制物质进出的作用外,可能还具有酶活化功能。
2.2 羧酶体酶的结构和功能 2.2.1 碳酸酐酶(CA酶)的结构和功能由于进化上的差异,自然界中碳酸酐酶主要包括α、β、γ、ε四种类型[35]。其中α型羧酶体只含β型碳酸酐酶,即CsoSCA[36]。CsoSCA (碳酸酐酶)含有514个氨基酸,有3个主要的蛋白结构域:氨基端、中段和羧基端。CsoSCA的氨基端与羧酶体外壳紧密连接[31]。中段含有锌离子催化中心,可催化碳酸氢盐水解,它是执行碳酸酐酶催化功能的重要结构域。羧基端与中段的结构相似[37]。在羧酶体中,CsoSCA通常以二聚体形式存在[38]。催化过程中,HCO3-的氧原子首先与CsoSCA的锌离子结合,随后被分解为CO2和OH-,溶剂中的水分子结合了逸出的CO2位点,使整个催化过程得以循环。
CsoSCA与羧酶体外壳蛋白联系紧密。CsoSCA与2个CsoS1六聚体壳蛋白相互结合。尽管CsoSCA与CsoS1之间有紧密连接,但它并不对羧酶体的形成起决定作用。在敲除csoS3基因(编码CsoSCA的基因)后,羧酶体仍能形成。这是由于羧酶体内pH较细胞质更低[12],因此游离的H+仍可与HCO3-结合而释放CO2,使羧酶体继续发挥功能[36]。
进一步的研究表明,在羧酶体中,碳酸酐酶转化CO2的总速率远远超过Rubisco酶固定CO2的总速率,羧酶体的功能并非受限于碳酸酐酶[39];将碳酸酐酶量缩减到1/4,羧酶体对CO2的固定能力也不会明显减弱。既然如此,为何羧酶体中还含有大量碳酸酐酶?其原因至今不明。
2.2.2 核酮糖-1, 5-二磷酸羧化酶(Rubisco酶)的结构和功能自然界中存在4种形式的Rubisco酶,而迄今发现的羧酶体内只含Ⅰ型Rubisco酶,其中α-羧酶体包含IA Rubisco酶,β-羧酶体包含IB Rubisco酶[40]。Ⅰ型Rubisco酶由8个大亚基和8个小亚基组成。大亚基可分为氨基端和羧基端2个蛋白结构域,Rubisco酶的Mg2+催化活性位点位于大亚基的羧基端[41]。同时大亚基结构决定了Rubisco酶能否被封装在羧酶体内部。小亚基由123个氨基酸组成,不存在酶活性位点,与Rubisco酶的催化功能无关,但小亚基能结合并通过调节大亚基的位置暴露其活性位点,从而提高Rubisco酶催化活性[42],小亚基的突变会大大降低Rubisco酶的催化效率。相较于常见的Ⅰ型Rubisco酶,羧酶体内的Rubisco酶活性更高,约为前者的2-10倍[43]。
Rubisco酶对CO2的转化效率低(酶的转化数约为1-10 s–1)[44]、亲和力低、选择性低。而且,由于CO2与O2结构上的相似性,当有O2竞争时,Rubisco酶可与O2作用产生磷酸乙醇酸而非磷酸甘油酸,从而导致碳固定效率的降低[12]。一个循环中,Rubisco酶每固定5个CO2分子理论上可固定2个O2分子[45]。究其原因,可能是Rubisco酶出现时地球还处于厌氧环境。在3.5亿年漫长的进化中,随着地球上CO2浓度降低和O2浓度升高[46],生物进化形成了一套适应机制,即CO2浓缩机制,并由羧酶体承担。
2.3 羧酶体内辅助蛋白的结构和功能除了碳酸酐酶(CA酶)和核酮糖-1, 5-二磷酸羧化酶(Rubisco酶)外,羧酶体内还存在一些重要却又往往被忽视的辅助蛋白。
近年来的研究发现,在编码羧酶体的相关基因之中,存在许多以前被误认为是沉默基因的重要基因。在编码α-羧酶体的基因序列中发现了编码AAA+结构域的CsoCbbQ蛋白基因[47]。CsoCbbQ同样通过团聚形成六聚体,但其形成的六聚体边长(55 Å)远大于CsoS1六聚体的边长(36 Å)。因此,CsoCbbQ不能直接组成羧酶体的平面,然而一致的对称性可让CsoCbbQ附着在羧酶体外壳。免疫印迹法也证实了CsoCbbQ与羧酶体外壳蛋白之间的紧密联系。CsoCbbQ具有ATP酶的功能,又与外壳蛋白联系紧密,这一蛋白的发现深化了对现有羧酶体的理解。除此之外,羧酶体中独特的CPII蛋白可以感知环境中的碳酸氢盐[48],类似喋呤-4α-甲醇氨脱氢酶(被命名为acRAFs)的蛋白有助于Rubisco蛋白的装配[49]。
2.4 羧酶体的功能羧酶体存在于原核生物细胞内,用于加强细胞固定CO2的能力。细胞膜上存在的CO2和HCO3-转运蛋白(图 2)可以从细胞外单向转运CO2和HCO3-至细胞内,使细胞质中的无机碳(DIC)浓度提高为外界的40-1 000倍[50]。其中,细胞内大部分无机碳以HCO3-形式存在,这是由于HCO3-离子的质膜通透性高,便于积累[51]。小部分吸收的CO2水解为HCO3-,被羧酶体吸收[52]。羧酶体壳蛋白对碳酸盐有很好的渗透性,但可有效阻止CO2溢出。由于Rubisco酶的催化底物是CO2,而细胞中大部分无机碳为碳酸盐,不能被Rubisco酶利用,因此HCO3-被吸收后必需在羧酶体内由碳酸酐酶转化为CO2。随后通过卡尔文循环,将1分子CO2固定,加到1分子1, 5-二磷酸核酮糖(RUBP)上,产生2分子甘油酸-3-磷酸(3-PGA)[53],产物直接排出羧酶体供细胞使用。CO2浓缩机制提高了Rubisco酶的基质浓度,从而提高了CO2的固定效率,同时隔离了O2的竞争(图 2)。羧酶体具有团聚效应,细胞内的多个羧酶体往往自成团簇[54-55],形成集群效应。团簇结构可使羧酶体中泄漏的HCO3-被相邻的羧酶体快速利用[56],从而提高CO2固定效率。
3 羧酶体的检测 3.1 羧酶体的观察检测自养型脱氮细菌以CO2为唯一碳源合成细胞物质,拥有固定CO2的能力。CO2固定能力与细胞内羧酶体的形状、数目、结构和功能密切相关。以正常条件下羧酶体的性状作为参照,观察不同条件下羧酶体的形状、结构和数目的差别,可推测自养型脱氮细菌的生长代谢活性。
据文献报道,利用透射电镜、冷冻电镜对亚硝化单胞菌属(Nitrosomonas)、亚硝化螺菌属(Nitrosospira)、亚硝化球菌属(Nitrosococcus)等多个硝化菌属[57]以及硫氧化菌属(Thioalkalivibrio)等[58]多个反硝化菌属[55]进行检测,可观察到1-4个羧酶体。羧酶体的数目越多,细胞对CO2的固定能力越强。在蓝细菌中,检测到的羧酶体数目为2-15个,平均为7个[59],有时多达21个[52]。在化能自养菌Thiomicrospira crunogena细胞中,观测到的羧酶体数目甚至可达80个,数量众多的羧酶体保障了细胞在低无机碳浓度(20 μmol/L)下的快速生长[60]。在低无机碳胁迫条件下,自养型细菌会增加羧酶体数量,以维持自身生长的需要[61]。在化合物诱导下,羧酶体形状会发生异常变化[62],从正常的正二十面体转变为棒状或其他不规则结构使其固碳能力大大下降。因此观测细胞中羧酶体的数量和形状,对推断自养型脱氮细菌的生长状况具有重要的参考意义。
3.2 羧酶体活性的监测羧酶体的活性强弱是指固定利用CO2的能力强弱。以正常条件下羧酶体的无机碳固定能力为基准,检测羧酶体对无机碳的固定利用速率可判断羧酶体活性,从而推断脱氮细菌的生长代谢状态。
羧酶体原位活性测定需要对细胞进行透性化处理。处理后的细胞既保持了细胞内含物和结构的完整性,也增强了细胞膜的通透性[63]。在加入一定量透性化细胞的体系中,加入NaHCO3促使细胞活化,随后注入50 μmol/L NADH、0.25 μmol/L RUBP作为基质,利用分光光度计在波长340 nm处监测NADH的变化。参照每分子RUBP羧化需氧化2分子NADH的比例测算羧酶体活性。通过同位素标记法也可原位测定细胞的羧酶体活性[64]。在透性化细胞液中加入一定量NaH14CO3、RUBP作为反应物,反应30 min后加入盐酸终止,并加热驱赶CO2,通过液体闪烁计数仪计数,根据稳定的[14C]-3-PGA的产生量计算羧酶体活性。
羧酶体异位活性测定需要将羧酶体进行提纯。将细胞进行氯仿提取或超声破碎处理后,通过密度梯度离心,可从裂解液中提取完整的羧酶体[65],再经过凝胶层析或凝胶电泳进一步提纯。同理,以单位时间内基质中3-PGA[66]的增加量也可推算羧酶体活性。
4 厌氧氨氧化细菌中的羧酶体厌氧氨氧化菌是一种可在厌氧条件下将氨和亚硝酸盐转化为氮气的化能自养细菌[67]。虽然许多自养型细菌含有羧酶体,但迄今还未在厌氧氨氧化细菌中见到羧酶体。究其原因,一则可能是由于现已发现的厌氧氨氧化菌种类不多,只有5个属10个种[68],不能排除未发现的厌氧氨氧化菌具有羧酶体的可能;二则可能是因为现研究较多的厌氧氨氧化菌种Candidatus Brocadia anammoxidans、Candidatus Kuenenia stuttgartiensis等采用还原性乙酰辅酶A循环途径固定CO2[69-70],并不采用卡尔文循环固定CO2,无需羧酶体。对Candidatus Scalindua profunda进行宏基因组学分析发现,虽然这种厌氧氨氧化菌的固碳途径是还原性乙酰辅酶A循环[71],但在该菌中发现了编码Rubisco酶和CA酶的基因[72];转录组学分析也表明,Rubisco酶基因的转录水平很高。在海洋中新发现的厌氧氨氧化菌Candidatus Thioglobus autotrophicus中也发现了Rubisco酶、CA酶基因的编码区,且其具有卡尔文循环全部酶的编码基因,作者据此推测该菌的主要固碳途径是基于Rubisco酶的卡尔文循环[73]。以卡尔文循环作为主要固碳方式的厌氧氨氧化菌有可能含有羧酶体,对厌氧氨氧化菌中羧酶体的深入研究可促进厌氧氨氧化菌的生长,从而推动厌氧氨氧化工艺的探索和应用。
5 研究展望自养型细菌广泛存在于自然界,其中自养型脱氮细菌在氮素循环中起着重大作用。然而,由于其生长缓慢、细胞得率低、对环境条件变化敏感,导致相关研究进展缓慢。自养型脱氮细菌的生长与其CO2固定能力密切相关,而CO2固定能力又与羧酶体联系紧密。以先进的非培养技术开展针对典型脱氮细菌(硝化细菌、自养反硝化细菌、厌氧氨氧化细菌等)的基因组学、转录组学、蛋白组学以及代谢组学的探索,可从理论上推进脱氮细菌的研究。以羧酶体为研究对象探索自养型脱氮细菌的生长特性,有助于理解自养型脱氮菌的生长代谢并优化生物脱氮过程,推进生物脱氮工艺的开发。
随着研究范围和层次的不断扩展,对于羧酶体,仍存在以下应用方面的障碍和具有科学价值的问题值得今后深入研究:
(1) 羧酶体中不同组成成分的区别和各自是否具有未知的独特功能仍是困扰研究者们的问题。在羧酶体不同外壳蛋白之间结构和功能高度一致的表象下,是否各自有着不可替代的独特功能?羧酶体中碳酸酐酶数量为何远远超过需要?细胞中的羧酶体如何高效精准地完成自组装?
(2) 将羧酶体的众多基因在宿主细胞中高效地激活、表达,并使表达产物(羧酶体组分)有序组装,形成羧酶体结构并发挥应有功能仍然是一个技术难题。研究和应用自养脱氮菌中羧酶体无疑会加快自养型脱氮菌生长,加强其生物脱氮功能;但由于羧酶体组分和组装过程均较为复杂精密,致使在自养型脱氮菌及一些异源宿主中表达羧酶体并发挥功能依然极具挑战[74]。目前,已实现模式菌大肠杆菌作为宿主培育出具有羧化能力的大肠杆菌[75-77],但成功率不高;通过分析蛋白结构域的相互作用,人工构建嵌合蛋白,简化羧酶体结构,成功缩减了羧酶体形成所需的基因,降低了遗传调控与代谢压力[78],提高了转化的成功率[79]。要进一步提高羧酶体在自养型脱氮细菌及一些异源宿主内的转化成功率,其障碍不仅在于上文所述的简化羧酶体相关基因表达(包括羧酶体外壳相关基因的简化,内含酶Rubisco酶相关基因表达,细胞膜上无机碳转运酶基因表达的强化,游离碳酸酐酶基因表达的抑制),还在于促进羧酶体有序团聚等难题[45, 80]。
(3) 将羧酶体作为一种模式“类细胞器”,把该种结构拓展应用到其他领域是新的研究热点和发展方向。通过羧酶体,自养型脱氮细菌的碳同化过程可更为紧凑而高效。作为一种“类细胞器”,羧酶体在纳米微反应器和分子支架等一系列前沿科技研究中具有很高的应用价值[81]。以羧酶体为模式的“类细胞器”设计细胞内反应、构筑细胞微反应体系[82],除了应用于自养型脱氮细菌的同化生长上,也可应用于自养脱氮细菌的其他代谢生长和其他微生物的代谢和生长上。
(4) 通过研究羧酶体,探明生物演化的谜底、揭示进化本源的关系是新的尝试。伴随着微生物的进化,羧酶体呈现出稳定可遗传的变异。厌氧氨氧化菌、氨氧化古菌等自养脱氮细菌、古菌的系统发育学研究是目前的热点之一,对于这些微生物羧酶体的结构功能研究可在其中起到“管中窥豹”的作用。探明羧酶体的进化机制和羧酶体的分化[83],对于揭示自养型脱氮细菌系统发育的谜底至关重要[84]。
| [1] |
Schmidt I, Sliekers O, Schmid M, et al. New concepts of microbial treatment processes for the nitrogen removal in wastewater[J]. FEMS Microbiology Reviews, 2003, 27(4): 481-492. DOI:10.1016/S0168-6445(03)00039-1 |
| [2] |
Desmidt E, Monballiu A, de Clippeleir H, et al. Autotrophic nitrogen removal after ureolytic phosphate precipitation to remove both endogenous and exogenous nitrogen[J]. Water Science & Technology, 2013, 67(7): 1425-1433. |
| [3] |
Wang C, Liu ST, Xu XC, et al. Potential coupling effects of ammonia-oxidizing and anaerobic ammonium-oxidizing bacteria on completely autotrophic nitrogen removal over nitrite biofilm formation induced by the second messenger cyclic diguanylate[J]. Applied Microbiology and Biotechnology, 2017, 101(9): 3821-3828. DOI:10.1007/s00253-016-7981-y |
| [4] |
Kerfeld CA, Erbilgin O. Bacterial microcompartments and the modular construction of microbial metabolism[J]. Trends in Microbiology, 2015, 23(1): 22-34. DOI:10.1016/j.tim.2014.10.003 |
| [5] |
Tanaka S, Kerfeld CA, Sawaya MR, et al. Atomic-level models of the bacterial carboxysome shell[J]. Science, 2008, 319(5866): 1083-1086. DOI:10.1126/science.1151458 |
| [6] |
Rae BD, Long BM, Badger MR, et al. Functions, compositions, and evolution of the two types of carboxysomes: polyhedral microcompartments that facilitate CO2 fixation in cyanobacteria and some proteobacteria[J]. Microbiology and Molecular Biology Reviews, 2013, 77(3): 357-379. DOI:10.1128/MMBR.00061-12 |
| [7] |
Holthuijzen YA, Vonck J, Kuenen JG, et al. The carboxysome of Thiobacillus neapolitanus[J]. Antonie van Leeuwenhoek, 1984, 50(1): 90. |
| [8] |
Blanchard J, Abdul-Rahman F. Carboxysomes, Structure and Function[M]. Berlin: Springer, 2014, 1-3.
|
| [9] |
Beudeker RF, Kuenen JG. Carboxysomes: 'calvinosomes'?[J]. FEBS Letters, 1981, 131(2): 269-274. DOI:10.1016/0014-5793(81)80382-1 |
| [10] |
Holthuijzen YA, van Breemen JFL, Kuenen JG, et al. Protein composition of the carboxysomes of Thiobacillus neapolitanus[J]. Archives of Microbiology, 1986, 144(4): 398-404. DOI:10.1007/BF00409891 |
| [11] |
Vakeria D, Codd GA, Marsden WJN, et al. No evidence for DNA in cyanobacterial carboxysomes[J]. FEMS Microbiology Letters, 1984, 25(2/3): 149-152. |
| [12] |
Heinhorst S, Williams EB, Cai F, et al. Characterization of the carboxysomal carbonic anhydrase CsoSCA from Halothiobacillus neapolitanus[J]. Journal of Bacteriology, 2006, 188(23): 8087-8094. DOI:10.1128/JB.00990-06 |
| [13] |
Heinhorst S, Cannon GC, Shively JM. Carboxysomes and Carboxysome-Like Inclusions[M]. Berlin: Springer, 2006, 141-165.
|
| [14] |
Schmid MF, Paredes AM, Khant HA, et al. Structure of Halothiobacillus neapolitanus carboxysomes by cryo-electron tomography[J]. Journal of Molecular Biology, 2006, 364(3): 526-535. DOI:10.1016/j.jmb.2006.09.024 |
| [15] |
Yeates TO, Kerfeld CA, Heinhorst S, et al. Protein-based organelles in bacteria: carboxysomes and related microcompartments[J]. Nature Reviews Microbiology, 2008, 6(9): 681-691. DOI:10.1038/nrmicro1913 |
| [16] |
Roberts EW, Cai F, Kerfeld CA, et al. Isolation and characterization of the Prochlorococcus carboxysome reveal the presence of the novel shell protein CsoS1D[J]. Journal of Bacteriology, 2012, 194(4): 787-795. DOI:10.1128/JB.06444-11 |
| [17] |
Cannon GC, Baker SH, Soyer F, et al. Organization of carboxysome genes in the thiobacilli[J]. Current Microbiology, 2003, 46(2): 115-119. DOI:10.1007/s00284-002-3825-3 |
| [18] |
Baker SH, Lorbach SC, Rodriguez-Buey M, et al. The correlation of the gene csoS2 of the carboxysome operon with two polypeptides of the carboxysome in Thiobacillus neapolitanus[J]. Archives of Microbiology, 1999, 172(4): 233-239. DOI:10.1007/s002030050765 |
| [19] |
Espie GS, Kimber MS. Carboxysomes: cyanobacterial RubisCO comes in small packages[J]. Photosynthesis Research, 2011, 109(1/3): 7-20. |
| [20] |
Westphal K, Bock E, Cannon G, et al. Deoxyribonucleic acid in Nitrobacter carboxysomes[J]. Journal of Bacteriology, 1979, 140(1): 285-288. |
| [21] |
Holthuijzen YA, Maathuis FJM, Kuenen JG, et al. Carboxysomes of Thiobacillus neapolitanus do not contain extrachromosomal DNA[J]. FEMS Microbiology Letters, 1986, 35(2/3): 193-198. |
| [22] |
Kinney JN, Axen SD, Kerfeld CA. Comparative analysis of carboxysome shell proteins[J]. Photosynthesis Research, 2011, 109(1/3): 21-32. |
| [23] |
Barton LL, Bazylinski DA, Xu HF. Nanomicrobiology[M]. New York: Springer, 2014, 75-101.
|
| [24] |
Iancu CV, Ding HJ, Morris DM, et al. The structure of isolated Synechococcus strain WH8102 carboxysomes as revealed by electron cryotomography[J]. Journal of Molecular Biology, 2007, 372(3): 764-773. DOI:10.1016/j.jmb.2007.06.059 |
| [25] |
Tsai Y, Sawaya MR, Cannon GC, et al. Structural analysis of CsoS1A and the protein shell of the Halothiobacillus neapolitanus carboxysome[J]. PLoS Biology, 2007, 5(6): e144. DOI:10.1371/journal.pbio.0050144 |
| [26] |
Mahinthichaichan P, Morris D, Wang Y, et al. Bacterial carboxysome shell proteins are selectively permeable to carbon fixation substrates[J]. Biophysical Journal, 2017, 112(3): 239a. |
| [27] |
Sutter M, Faulkner M, Aussignargues C, et al. Visualization of bacterial microcompartment facet assembly using high-speed atomic force microscopy[J]. Nano Letters, 2016, 16(3): 1590-1595. DOI:10.1021/acs.nanolett.5b04259 |
| [28] |
Klein MG, Zwart P, Bagby SC, et al. Identification and structural analysis of a novel carboxysome shell protein with implications for metabolite transport[J]. Journal of Molecular Biology, 2009, 392(2): 319-333. DOI:10.1016/j.jmb.2009.03.056 |
| [29] |
Bonacci W, Teng PK, Afonso B, et al. Modularity of a carbon-fixing protein organelle[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(2): 478-483. DOI:10.1073/pnas.1108557109 |
| [30] |
Chaijarasphong T, Nichols RJ, Kortright KE, et al. Programmed ribosomal frameshifting mediates expression of the α-carboxysome[J]. Journal of Molecular Biology, 2016, 428(1): 153-164. DOI:10.1016/j.jmb.2015.11.017 |
| [31] |
Cai F, Dou ZC, Bernstein SL, et al. Advances in understanding carboxysome assembly in Prochlorococcus and Synechococcus implicate CsoS2 as a critical component[J]. Life, 2015, 5(2): 1141-1171. DOI:10.3390/life5021141 |
| [32] |
Cameron JC, Wilson SC, Bernstein SL, et al. Biogenesis of a bacterial organelle: the carboxysome assembly pathway[J]. Cell, 2013, 155(5): 1131-1140. DOI:10.1016/j.cell.2013.10.044 |
| [33] |
Kerfeld CA, Melnicki MR. Assembly, function and evolution of cyanobacterial carboxysomes[J]. Current Opinion in Plant Biology, 2016, 31: 66-75. DOI:10.1016/j.pbi.2016.03.009 |
| [34] |
Cai F, Menon BB, Cannon GC, et al. The pentameric vertex proteins are necessary for the icosahedral carboxysome shell to function as a CO2 leakage barrier[J]. PLoS One, 2009, 4(10): e7521. DOI:10.1371/journal.pone.0007521 |
| [35] |
Zhang X, Liu XD, Liang YL, et al. Metabolic diversity and adaptive mechanisms of iron- and/or sulfur-oxidizing autotrophic acidophiles in extremely acidic environments[J]. Environmental Microbiology Reports, 2016, 8(5): 738-751. DOI:10.1111/1758-2229.12435 |
| [36] |
Frost SC, McKenna R. Carbonic Anhydrase: Mechanism, Regulation, Links to Disease, and Industrial Applications[M]. Netherlands: Springer, 2014.
|
| [37] |
Sawaya MR, Cannon GC, Heinhorst S, et al. The structure of β-carbonic anhydrase from the carboxysomal shell reveals a distinct subclass with one active site for the price of two[J]. Journal of Biological Chemistry, 2006, 281(11): 7546-7555. DOI:10.1074/jbc.M510464200 |
| [38] |
Cannon GC, Heinhorst S, Kerfeld CA. Carboxysomal carbonic anhydrases: structure and role in microbial CO2 fixation[J]. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2010, 1804(2): 382-392. DOI:10.1016/j.bbapap.2009.09.026 |
| [39] |
Frey R, Mantri S, Rocca M, et al. Bottom-up construction of a primordial carboxysome mimic[J]. Journal of the American Chemical Society, 2016, 138(32): 10072-10075. DOI:10.1021/jacs.6b04744 |
| [40] |
Spreitzer RJ, Salvucci ME. Rubisco: structure, regulatory interactions, and possibilities for a better enzyme[J]. Annual Review of Plant Biology, 2002, 53: 449-475. DOI:10.1146/annurev.arplant.53.100301.135233 |
| [41] |
Brändén CI, Lindqvist Y, Schneider G. Protein engineering of rubisco[J]. Acta Crystallographica Section B: Structural Science, Crystal Engeering and Materials, 1991, 47(6): 824-835. DOI:10.1107/S0108768191007127 |
| [42] |
Chapman MS, Suh SW, Curmi PM, et al. Tertiary structure of plant RuBisCO: domains and their contacts[J]. Science, 1988, 241(4861): 71-74. DOI:10.1126/science.3133767 |
| [43] |
Menning KJ, Menon BB, Fox G, et al. Dissolved inorganic carbon uptake in Thiomicrospira crunogena XCL-2 is Δp- and ATP-sensitive and enhances RubisCO-mediated carbon fixation[J]. Archives of Microbiology, 2016, 198(2): 149-159. DOI:10.1007/s00203-015-1172-6 |
| [44] |
Giessen TW, Silver PA. Engineering carbon fixation with artificial protein organelles[J]. Current Opinion in Biotechnology, 2017, 46: 42-50. DOI:10.1016/j.copbio.2017.01.004 |
| [45] |
Walker BJ, VanLoocke A, Bernacchi CJ, et al. The costs of photorespiration to food production now and in the future[J]. Annual Review of Plant Biology, 2016, 67: 107-129. DOI:10.1146/annurev-arplant-043015-111709 |
| [46] |
Badger MR, Bek EJ. Multiple Rubisco forms in proteobacteria: their functional significance in relation to CO2 acquisition by the CBB cycle[J]. Journal of Experimental Botany, 2008, 59(7): 1525-1541. DOI:10.1093/jxb/erm297 |
| [47] |
Sutter M, Roberts EW, Gonzalez RC, et al. Structural characterization of a newly identified component of α-carboxysomes: the AAA+ domain protein CsoCbbQ[J]. Scientific Reports, 2015, 5: 16243. DOI:10.1038/srep16243 |
| [48] |
Wheatley NM, Eden KD, Ngo J, et al. A PⅡ-like protein regulated by bicarbonate: structural and biochemical studies of the carboxysome-associated CPⅡ protein[J]. Journal of Molecular Biology, 2016, 428(20): 4013-4030. DOI:10.1016/j.jmb.2016.07.015 |
| [49] |
Wheatley NM, Sundberg CD, Gidaniyan SD, et al. Structure and identification of a pterin dehydratase-like protein as a ribulose-bisphosphate carboxylase/oxygenase (RuBisCO) assembly factor in the α-carboxysome[J]. Journal of Biological Chemistry, 2014, 289(11): 7973-7981. DOI:10.1074/jbc.M113.531236 |
| [50] |
Badger MR, Kaplan A, Berry JA. Internal inorganic carbon pool of Chlamydomonas reinhardtii: evidence for a carbon dioxide-concentrating mechanism[J]. Plant Physiology, 1980, 66(3): 407-413. DOI:10.1104/pp.66.3.407 |
| [51] |
Price GD, Sültemeyer D, Klughammer B, et al. The functioning of the CO2 concentrating mechanism in several cyanobacterial strains: a review of general physiological characteristics, genes, proteins, and recent advances[J]. Canadian Journal of Botany, 1998, 76(6): 973-1002. DOI:10.1139/b98-081 |
| [52] |
Badger MR, Price GD. CO2 concentrating mechanisms in cyanobacteria: molecular components, their diversity and evolution[J]. Journal of Experimental Botany, 2003, 54(383): 609-622. DOI:10.1093/jxb/erg076 |
| [53] |
Prescott LM, Harley JP, Klein DA. Microbiology[M]. Shen P, Peng ZR, Trans. 5th ed. Beijing: Higher Education Press, 2003: 209-210 (in Chinese) Prescott LM, Harley JP, Klein DA. 微生物学[M]. 沈萍, 彭珍荣, 译. 5版. 北京: 高等教育出版社, 2003: 209-210 |
| [54] |
Ting CS, Hsieh C, Sundararaman S, et al. Cryo-electron tomography reveals the comparative three-dimensional architecture of Prochlorococcus, a globally important marine cyanobacterium[J]. Journal of Bacteriology, 2007, 189(12): 4485-4493. DOI:10.1128/JB.01948-06 |
| [55] |
Liberton M, Austin JR, Berg RH, et al. Unique thylakoid membrane architecture of a unicellular N2-fixing cyanobacterium revealed by electron tomography[J]. Plant Physiology, 2011, 155(4): 1656-1666. DOI:10.1104/pp.110.165332 |
| [56] |
Iancu CV, Morris DM, Dou ZC, et al. Organization, structure, and assembly of α-carboxysomes determined by electron cryotomography of intact cells[J]. Journal of Molecular Biology, 2010, 396(1): 105-117. DOI:10.1016/j.jmb.2009.11.019 |
| [57] |
Fiencke C, Bock E. Immunocytochemical localization of membrane-bound ammonia monooxygenase in cells of ammonia oxidizing bacteria[J]. Archives of Microbiology, 2006, 185(2): 99-106. DOI:10.1007/s00203-005-0074-4 |
| [58] |
Sorokin DY, Lysenko AM, Mityushina LL, et al. Thioalkalimicrobium aerophilum gen. nov., sp. nov. and Thioalkalimicrobium sibericum sp. nov., and Thioalkalivibrio versutus gen. nov., sp. nov., Thioalkalivibrio nitratis sp. nov., novel and Thioalkalivibrio denitrificancs sp. nov., novel obligately alkaliphilic and obligately chemolithoautotrophic sulfur-oxidizing bacteria from soda lakes[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(2): 565-580. DOI:10.1099/00207713-51-2-565 |
| [59] |
van de Meene AML, Hohmann-Marriott MF, Vermaas WFJ, et al. The three-dimensional structure of the cyanobacterium Synechocystis sp. PCC 6803[J]. Archives of Microbiology, 2006, 184(5): 259-270. DOI:10.1007/s00203-005-0027-y |
| [60] |
Dobrinski KP. Thiomicrospira crunogena: a chemoautotroph with a carbon concentrating mechanism[D]. Florida: Doctoral Dissertation of University of South Florida, 2009: 2-3 http://www.researchgate.net/publication/254705873_Thiomicrospira_crunogena_A_chemoautotroph_with_a_carbon_concentrating_mechanism
|
| [61] |
Orús MI, Rodríguez-Buey ML, Marco E, et al. Changes in carboxysome structure and grouping and in photosynthetic affinity for inorganic carbon in Anabaena strain PCC 7119 (Cyanophyta) in response to modification of CO2 and Na+ supply[J]. Plant & Cell Physiology, 2001, 42(1): 46-53. |
| [62] |
Wu TF, Liu YD, Song LR. Selection and ultrastructural observation of a high-CO2-requiring mutant of cyanobacterium Synechococcus sp. PCC7942[J]. Acta Botanica Sinica, 2000, 42(2): 116-121. |
| [63] |
Rasmussen RE, Erstad SM, Ramos-Martinez EM, et al. An easy and efficient permeabilization protocol for in vivo enzyme activity assays in cyanobacteria[J]. Microbial Cell Factories, 2016, 15: 186. DOI:10.1186/s12934-016-0587-3 |
| [64] |
Cannon GC, English RS, Shively JM. In situ assay of ribulose-1, 5-bisphosphate carboxylase/oxygenase in Thiobacillus neapolitanus[J]. Journal of Bacteriology, 1991, 173(4): 1565-1568. DOI:10.1128/jb.173.4.1565-1568.1991 |
| [65] |
Faulkner M, Rodriguez-Ramos J, Dykes GF, et al. Direct characterization of the native structure and mechanics of cyanobacterial carboxysomes[J]. Nanoscale, 2017, 9(30): 10662-10673. DOI:10.1039/C7NR02524F |
| [66] |
Di Marco G, Tricoli D. RuBP carboxylase determination by enzymic estimation of D-3-PGA formed[J]. Photosynthesis Research, 1983, 4(2): 145-149. DOI:10.1007/BF00052376 |
| [67] |
Kang D, Zheng P, Hu QY. Structure, morphology and function of Anammox granular sludge[J]. CIESC Journal, 2016, 67(10): 4040-4046. 康达, 郑平, 胡倩怡. 厌氧氨氧化结构体、形态与功能[J]. 化工学报, 2016, 67(10): 4040-4046. |
| [68] |
Zheng P, Zhang L. Characterization and classification of anaerobic ammonium oxidation (anammox) bacteria[J]. Journal of Zhejiang University (Agriculture & Life Science), 2009, 35(5): 473-481. 郑平, 张蕾. 厌氧氨氧化菌的特性与分类[J]. 浙江大学学报:农业与生命科学版, 2009, 35(5): 473-481. |
| [69] |
Schouten S, Strous M, Kuypers MMM, et al. Stable carbon isotopic fractionations associated with inorganic carbon fixation by anaerobic ammonium-oxidizing bacteria[J]. Applied and Environmental Microbiology, 2004, 70(6): 3785-3788. DOI:10.1128/AEM.70.6.3785-3788.2004 |
| [70] |
Ding S, Zheng P, Lu HF, et al. New understanding on metabolism of anaerobic ammonium oxidationbacteria based on metagenomics technology[J]. Chinese Journal of Appplied Environmental Biology, 2012, 18(4): 697-704. 丁爽, 郑平, 陆慧锋, 等. 基于宏基因组技术获得的对厌氧氨氧化菌代谢的新理解[J]. 应用与环境生物学报, 2012, 18(4): 697-704. |
| [71] |
Ragsdale SW. Enzymology of the acetyl-CoA pathway of CO2 fixation[J]. Critical Reviews in Biochemistry and Molecular Biology, 1991, 26(3/4): 261-300. |
| [72] |
van de Vossenberg J, Woebken D, Maalcke WJ, et al. The metagenome of the marine anammox bacterium 'Candidatus Scalindua profunda' illustrates the versatility of this globally important nitrogen cycle bacterium[J]. Environmental Microbiology, 2013, 15(5): 1275-1289. DOI:10.1111/emi.2013.15.issue-5 |
| [73] |
Shah V, Chang BX, Morris RM. Cultivation of a chemoautotroph from the SUP05 clade of marine bacteria that produces nitrite and consumes ammonium[J]. The ISME Journal, 2017, 11(1): 263-271. DOI:10.1038/ismej.2016.87 |
| [74] |
Baumgart M, Huber I, Abdollahzadeh I, et al. Heterologous expression of the Halothiobacillus neapolitanus carboxysomal gene cluster in Corynebacterium glutamicum[J]. Journal of Biotechnology, 2017, 258: 126-135. DOI:10.1016/j.jbiotec.2017.03.019 |
| [75] |
Parikh MR, Greene DN, Woods KK, et al. Directed evolution of RuBisCO hypermorphs through genetic selection in engineered E. coli[J]. Protein Engineering, Design and Selection, 2006, 19(3): 113-119. DOI:10.1093/protein/gzj010 |
| [76] |
Gonzalez-Esquer CR, Shubitowski TB, Kerfeld CA. Streamlined construction of the cyanobacterial CO2-fixing organelle via protein domain fusions for use in plant synthetic biology[J]. The Plant Cell, 2015, 27(9): 2637-2644. DOI:10.1105/tpc.15.00329 |
| [77] |
Kerfeld CA. Plug-and-play for improving primary productivity[J]. American Journal of Botany, 2015, 102(12): 1949-1950. DOI:10.3732/ajb.1500409 |
| [78] |
Long BM, Rae BD, Rolland V, et al. Cyanobacterial CO2-concentrating mechanism components: Function and prospects for plant metabolic engineering[J]. Current Opinion in Plant Biology, 2016, 31: 1-8. |
| [79] |
Gilbert N. Gates Foundation backs high-risk science for big wins[J]. Nature Plants, 2015, 1(3): 15022. DOI:10.1038/nplants.2015.22 |
| [80] |
Chessher A, Breitling R, Takano E. Bacterial microcompartments: biomaterials for synthetic biology-based compartmentalization strategies[J]. ACS Biomaterials Science & Engineering, 2015, 1(6): 345-351. |
| [81] |
Jordan PC, Patterson DP, Saboda KN, et al. Self-assembling biomolecular catalysts for hydrogen production[J]. Nature Chemistry, 2015, 8(2): 179-185. |
| [82] |
Cai F, Sutter M, Bernstein SL, et al. Engineering bacterial microcompartment shells: chimeric shell proteins and chimeric carboxysome shells[J]. ACS Synthetic Biology, 2015, 4(4): 444-453. DOI:10.1021/sb500226j |
| [83] |
Klanchui A, Cheevadhanarak S, Prommeenate P, et al. Exploring components of the CO2-concentrating mechanism in alkaliphilic cyanobacteria through genome-based analysis[J]. Computational and Structural Biotechnology Journal, 2017, 15: 340-350. DOI:10.1016/j.csbj.2017.05.001 |
| [84] |
Ting CS, Dusenbury KH, Pryzant RA, et al. The Prochlorococcus carbon dioxide-concentrating mechanism: evidence of carboxysome-associated heterogeneity[J]. Photosynthesis Research, 2015, 123(1): 45-60. DOI:10.1007/s11120-014-0038-0 |
 2018, Vol. 45
2018, Vol. 45