扩展功能
文章信息
- 陈东波, 柳成宾, 姜怡, 吕元林, 姜成林
- CHEN Dong-Bo, LIU Cheng-Bin, JIANG Yi, LYU Yuan-Lin, JIANG Cheng-Lin
- 来自西双版纳3种有毒植物的免培养与纯培养放线菌多样性及生物活性
- Diversity and bioactivity of un-cultured and cultivable actinomycetes from three toxic plants in Xishuangbanna, China
- 微生物学通报, 2018, 45(5): 1100-1111
- Microbiology China, 2018, 45(5): 1100-1111
- DOI: 10.13344/j.microbiol.china.170467
-
文章历史
- 收稿日期: 2017-06-29
- 接受日期: 2017-12-07
- 网络首发日期(www.cnki.net): 2018-01-09
2. 东北大学生命科学与健康学院 辽宁 沈阳 110819;
3. 中国科学院昆明植物研究所 云南 昆明 650204
2. College of Life and Health Sciences, Northeastern University, Shenyang, Liaoning 110819, China;
3. Kunming Institute of Botany, Chinese Academy of Sciences, Kunming, Yunnan 650204, China
目前通过纯培养方法从土壤放线菌分离新化合物变得日益困难,已知化合物的重复发现率越来越高[1]。由于抗生素的滥用使致病菌抗药性增加,迫使人们转向研究其他栖息地的微生物,以期从中开发更加有效的新抗生素。相对土壤放线菌来说,植物内生放线菌研究较少,发现新物种、新基因及新化合物的可能性更高[2]。在长期协同进化过程中,内生菌为了更好地适应植物微环境,内生菌的遗传物质开始发生变化比如整合宿主植物DNA[3],内生菌能够直接从植物体获得营养物质进行繁殖,同时产生抗生素、植物生长激素提高植物抗逆性、促进植物生长,因此内生菌获得了产生与宿主类似或相同化合物的能力[4]。
西双版纳热带雨林是北半球保存最完好的热带雨林,是我国植物种类最丰富的地区,特有植物种类非常丰富。该地区终年高温多雨,土壤微生物丰富,有机物和腐殖质代谢极为旺盛,植物光合作用强、蒸腾作用大,有利于内生放线菌的侵入、定殖。有毒植物是指对人和家畜等产生有害作用的植物。有毒植物的化学成分复杂、代谢产物种类丰富。目前,有毒植物的利用主要集中在植物次生代谢物的抗虫、抗菌活性,以及植物性生物农药的开发[5],对有毒植物内生放线菌的多样性及生物活性研究较少。吾甫尔·米吉提等[6]对新疆焮麻(Urtica cannabina L.)、新疆准葛尔乌头(Aaconitum soongaricum Stapf)、阿尔泰藜芦(Veratrum lobelianum Bernh)、软紫草[Arnebia euchroma (Fechhsch) G. L. Chu]和蓝麻黄(Ephedra glauca regel)等10多种新疆药用和有毒植物中分离获得50多种内生细菌及真菌,并从中筛选出部分对经济作物具有拮抗作用的菌株。郑毅等[7]从木本曼陀罗(Datura arborea L.)的根、茎、叶及花组织中分离出77株内生真菌,通过对77株内生真菌的发酵代谢产物的抗菌活性筛选研究发现木本曼陀罗植物内生真菌抗菌能力较强,抗菌谱较广。戴文君等[8]从见血封喉(Antiaris toxicaria Lesch.)根皮中分离获得6株内生真菌,通过抑菌活性研究发现内生真菌J6对耐甲氧西林金黄色葡萄球菌[Methicillin-resistant Staphylococcus aureus (MRSA)]有明显抑菌活性。本文选取箭毒木(Antiaris toxicaria Lesch.)、八角枫[Alangium chinense (Lour.) Harms]和马缨丹(Lantana camara L.) 3种有毒植物为实验材料[9],利用免培养测序和纯培养分离探究有毒植物放线菌的多样性,检测放线菌的抗菌活性和几类活性物质化合物合成基因簇,为开发新药提供新的菌种资源。
1 材料与方法 1.1 植物样品采集采用同一地点多株采样的方式,于2016年7月采自西双版纳傣族自治州景洪市勐腊县勐仑镇的热带雨林(21°88′N,101°26′E)地区,采集箭毒木、八角枫和马缨丹3种有毒植物的根、茎、叶样品。采集后立即用75%酒精擦拭切口部位,低温保存带回实验室。
1.2 样品表面消毒样品经过流水冲洗、超声波清洗后,依次使用有效氯为5%的次氯酸钠漂洗4 min,2.5% Na2S2O3漂洗5 min,75%乙醇漂洗5 min,无菌水清洗3–6次,置于无菌铺有滤纸的平皿中干燥。将最后一次清洗植物样品的无菌水吸取0.2 mL涂布在YIM 38培养基上,置于28 ℃培养3 w以上检测植物表面消毒效果。
1.3 主要试剂和仪器实验所需的溶菌酶、蛋白酶K、PCR所用的10×Buffer、Taq DNA聚合酶、dNTPs均购自昆明云科生物科技有限公司。PCR仪、凝胶成像系统、电泳仪等购自Bio-Rad公司。纯培养菌株16S rRNA基因通用引物序列分别为PA (5′-CAGAGTTTGATCC TGGCT-3′)和PB (5′-AGGAGGTGATCCAGCCGC A-3′),引物由上海捷瑞生物工程有限公司提供。PCR产物由上海捷瑞生物工程有限公司测序。
1.4 免培养测序及分析 1.4.1 植物样品DNA提取和高通量测序采用SDS方法提取植物样品基因组总DNA[10]。扩增16S rRNA基因V4区的引物是515F (5′-GTGYCAGCMGCCGCGGTA-3′)和806R (5′-GG ACTACHVGGGTWTCTAAT-3′),扩增后的PCR[10]产物用2%的琼脂糖凝胶电泳检测。
1.4.2 数据分析处理样品纯化检测后由北京诺禾致源科技股份有限公司使用Illumina HiSeq高通量测序。为方便测序及数据分析,分别用JDM1.1、BJF1.1、MYD1.1代替植物样品箭毒木、八角枫、马缨丹。测序数据下机后先截去BarCode和引物序列后使用FLASH[11]拼接得到原始Tags数据,参照QIIME (Version 1.7.0)[12]进行序列过滤。用UCHIME Algorithm[13]与Gold database数据库进行比对检测嵌合体序列,并最终去除其中的嵌合体序列得到有效数据[14]。利用UPARSE软件(Version 7.0.1001)[15]将相似性高于97%的序列聚类为一个分类单元,即一个OTU (Operational taxonomic unit)。采用Mothur方法与Silva[16]的核糖体数据库[17]进行物种注释分析、使用PyNAST软件(Version 1.2)[18]与GreenGene数据库中的“Core Set”数据信息进行快速多序列比对,得到所有OTU代表序列的系统发生关系。
1.5 纯培养放线菌分离 1.5.1 培养基分离培养基[19]:酵母浸出粉培养基(TWYE)、腐殖酸培养基(HV)、纤维素-脯氨酸培养基(CP)、木聚糖-天冬酰胺培养基(XA)、棉子糖-组氨酸培养基(RL)、琥珀酸钠培养基(SS)。培养基中加入抑制剂:制霉菌素50 mg/L,萘啶酸25 mg/L,重铬酸钾25 mg/L。菌株纯化、活化、保藏培养基:YIM 38[19],菌株发酵培养基:YIM 61[19]。指示菌培养基:LB培养基,马铃薯培养基(PDA)。
1.5.2 分离流程将消毒后的植物样品分别称取2 g根、茎、叶,用无菌刀具切成小块置于离心管中,加入18 mL 1%磷酸缓冲液无菌条件下匀浆处理5 min (8 000 r/min)。匀浆处理后的植物样品溶液200 r/min,振摇1 h,稀释10-2、10-3、10-4涂布在1.5.1中所述的分离培养基上,28 ℃培养3 w以上。
1.5.3 内生放线菌鉴定和保藏挑选单菌落,采取四区划线法接种于YIM 38平板纯化菌株,挑选单菌落接入YIM 38斜面培养基中,28 ℃培养7 d。根据菌落在YIM 38的形态特征以及显微镜下观察基内菌丝与气生菌丝的形态特征等,初步筛选菌株。收集菌体使用酶法小量[20]提取菌株DNA,与EzBioCloud数据库比对测序结果。从有毒植物中纯培养分离鉴定的菌株保藏在云南大学云南省微生物研究所。
1.6 抑菌活性检测 1.6.1 供试菌株和指示菌供试菌株为3种有毒植物纯培养分离、鉴定的内生放线菌,指示菌由中国典型培养物保藏中心提供:枯草芽孢杆菌(Bacillus subtilis CGMCC 1.1849)、金黄色葡萄球菌(Staphylococcus aureus CGMCC 1.2386)、结核分枝杆菌(Mycobacterium tuberculosis)、大肠杆菌(Escherichia coli CGMCC 1.2385)、白色念珠菌(Candida albicans CGMCC 2.2086)及黑曲霉(Aspergillus niger)。
1.6.2 供试菌株发酵和抑菌活性筛选采用纸片扩散法筛选抗菌活性。将供试菌株活化后转接YIM 61小量发酵,28 ℃、220 r/min培养7 d,每株菌发酵6 mL,以不接种菌株培养基当作空白组。吸取0.2 mL病原指示菌悬液涂布固体培养基表面,将直径8 mm小滤纸片浸透发酵液后接入指示菌平板,每株菌设置3次重复,并观测阴性对照组有无抑菌圈形成。37 ℃培养48 h,分别测量抑菌圈直径。
1.7 化合物合成基因检测 1.7.1 化合物合成基因引物7类化合物合成基因引物序列及目的条带大小如表 1所示[21-26]。7类化合物合成基因引物由上海捷瑞生物工程有限公司提供。
| 基因名称 Gene name |
基因长度 Gene length (bp) |
引物名称 Primer name |
引物序列 Primer sequence (5′→3′) |
参考文献 References |
| PKS-Ⅰ | 1 200-1 400 | K1F | TSAAGTCSAACATCGGBCA | [21] |
| M6R | CGCAGGTTSCSGTACCAG | |||
| PKS-Ⅱ | 600 | KSa | TSGCSTGCTTGGAYGCSATC | [22] |
| KSβ | TGGAANCCGCCGAABCCTCT | |||
| NRPS | 900 | A3F | GCSTACSYSATSTACACSTCSGG | [21] |
| A7R | SASGTCVCCSGTSCGGTAS | |||
| APH | 600 | STR-F | CGGCTGCTCGACCACGAC | [23] |
| STR-R | GTCCTCGATGTCCCACAG | |||
| HMG-CoA | 900 | HMGF | GGGCATCGCCGCGACCCTCGTCGACGAGCG | [24] |
| HMGR | GCGATGACGGCGAGGCGGCGGGCGTTCTC | |||
| CYP | 350 | PEH-1 | TGGATCGGCGACGACCGSVYCGT | [25] |
| PEH-2 | CCGWASAGSAYSCCGTCGTACTT | |||
| SAL | 750 | 5F | AACGCACGCCGTCGTTCTC | [26] |
| 5R | ACCAGCCACTTGCCGTCCTC |
25 μL PCR反应体系:10×Buffer 2.5 μL,2.5 μmol/L dNTPs 4 μL,10 μmol/L正、反向引物各0.5 μL,5 U/μL Taq DNA聚合酶1.5 μL,DMSO 1.5 μL,模板DNA 1 μL,加入ddH2O补充至25 μL。
PKS-Ⅰ、PKS-Ⅱ和NRPS基因PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,55 ℃ 1 min,72 ℃ 2 min (CYP基因延伸30 s),35个循环;72 ℃ 10 min;4 ℃保存。APH、HMG-CoA基因PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,62 ℃ 1 min (SAL基因退火温度为55 ℃),72 ℃ 1 min,35个循环;72 ℃ 10 min;4 ℃保存,PCR产物用1%的琼脂糖凝胶电泳检测。
2 结果与分析 2.1 免培养内生细菌多样性分析 2.1.1 测序深度分析如图 1所示,3种有毒植物样品的稀释曲线最终都归于平缓。说明测序趋于饱和,能够比较真实地反映3种植物样品的微生物群落。

|
| 图 1 3种有毒植物内生菌多样性稀释曲线 Figure 1 Rarefaction curves of endophyte diversity in three toxic plants 注:JDM1.1:箭毒木;BJF1.1:八角枫;MYD1.1:马缨丹(下同). Note: JDM1.1: Antiaris toxicaria Lesch.; BJF1.1: Alangium chinense (Lour.) Harms; MYD1.1: Lantana camara L. (The same as below). |
|
|
如表 2所示,测序覆盖率均超过99.8%,理论上测序数据已能覆盖样品中的全部序列。在97%相似性水平上进行聚类,3种有毒植物通过高通量测序获得204 352条有效数据,近1 795个OTU,其中箭毒木、八角枫和马缨丹的OTU数目分别为434、681和680。
| 样品 Sample |
有效数据 Taxon tags |
分类单元 OTUs |
覆盖率 Coverage |
Chao1指数 Chao1 index |
Simpson指数 Simpson index |
Shannon指数 Shannon index |
| 箭毒木A. toxicaria | 54 726 | 434 | 0.998 | 623.056 | 0.874 | 4.444 |
| 八角枫A. chinense | 74 425 | 681 | 0.998 | 686.186 | 0.225 | 1.430 |
| 马缨丹L. camara | 75 201 | 680 | 0.998 | 669.275 | 0.265 | 1.477 |
| 合计Total | 204 352 | 1 795 |
α多样性用Chao1指数和Simpson指数来评估样品内微生物群落多样性。其中,Chao1指数用来估计物种总量,Chao1指数越大,代表物种总量越多。从表 2可以看出,八角枫和马缨丹的Chao1指数明显高于箭毒木,可知八角枫和马缨丹的微生物菌群的总量多于箭毒木。Simpson指数和Shannon指数被用来反映样品中微生物多样性,其值越大,说明群落物种丰富度越大或均匀度越高[27]。箭毒木样品Simpson和Shannon指数较八角枫和马缨丹明显更高,可能是因为样品中物种的不均匀性较高,又因其所获得的有效序列较少,因此总的生物多样性较小。综合上述分析可知,八角枫和马缨丹的微生物群落结构多样性比箭毒木更丰富。
β多样性用于分析样品间微生物群落的相似性和差异性。如图 2所示,八角枫和马缨丹先聚到一支上,说明八角枫和马缨丹在属分类水平的内生放线菌群落结构多样性较为相似。而箭毒木则单独聚为一支,说明在属分类水平上,箭毒木的微生物群落结构与其他两种植物的放线菌群落结构差异较大。
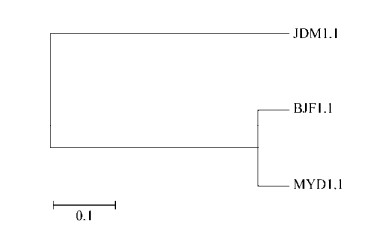
|
| 图 2 基于Weighted Unifrac距离的UPGMA聚类树 Figure 2 UPGMA clustering trees based on Weighted Unifrac distance |
|
|
对OTU的代表序列进行物种注释,3种有毒植物样品在门分类水平上的群落包括古菌域(Archaea)的广古菌门(Euryarchaeota)、奇古菌门(Thaumarchaeota),细菌域的18个门:酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿屈挠菌门(Chloroflexi)、蓝细菌门(Cyanobacteria)、迷踪菌门(Elusimicrobia)、丝状杆菌门(Fibrobacteres)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、硝化螺旋菌门(Nitrospirae)、变形杆菌门(Proteobacteria)、螺旋体门(Spirochaetes)、软壁菌门(Tenericutes)、热微菌门(Thermomicrobia)、热袍菌门(Thermotogae)、疣微菌门(Verrucomicrobia)、Latescibacteria、Saccharibacteria和暂定的RsaHF231、WD272门。箭毒木有11个门、八角枫有16个门、马缨丹有17个门。
在属分类水平上获得30个属的放线菌,箭毒木有15个属、八角枫和马缨丹均有29个属,其中八角枫和马缨丹共有28个相同的属:热酸菌属(Acidothermus)、放线异壁酸菌属(Actinoallomurus)、拟无枝酸菌属(Amycolatopsis)、节杆菌属(Arthrobacter)、双栖杆菌属(Bifidobacterium)、短状杆菌属(Brachybacterium)、细链孢菌属(Catenulispora)、纤维单胞菌属(Cellulomonas)、扣林氏菌属(Collinsella)、克洛斯氏菌属(Crossiella)、肠杆菌属(Enterorhabdus)、糖霉菌属(Glycomyces)、Iamia、Jatrophihabitans、生工菌属(Kribbella)、无色杆菌属(Leucobacter)、大理石雕菌属(Marmoricola)、微杆菌属(Microbacterium)、类诺卡氏菌属(Nocardiides)、厄氏菌属(Olsenella)、丙酸杆菌属(Propionibacterium)、假诺卡氏菌属(Pseudonocardia)、红球菌属(Rhodococcus)、红色杆菌属(Rubrobacter)、血杆菌属(Sanguibacter)、Senegalimassilia、鱼孢菌属(Sporichthya)、链霉菌属(Streptomyces),只有放线短链孢菌属(Actinocatenispora)和短杆菌属(Brevibacterium)不一样。
2.2 纯培养放线菌多样性分析 2.2.1 纯培养分离内生放线菌多样性通过1.5.1所述6种分离培养基从3种有毒植物分离、鉴定出11个属34株内生放线菌,经过BLAST比对分析结果显示,分别属于链霉菌目(Streptomycineae)、棒杆菌目(Corynebacterneae)、微球菌目(Micrococcineae)、小单孢菌目(Micromonosporineae) 4个目。其中链霉菌属(Streptomyces) 15株、迪茨菌属(Dietzia) 1株、分枝杆菌属(Mycobacterium) 1株、孢囊菌属(Verrucosispora) 1株、微杆菌属(Microbacterium) 6株、纤维微杆菌属(Cellulosimi- crobium) 4株、节杆菌属(Arthrobacter) 2株、短杆菌属(Brachybacterium) 1株、皮球菌属(Dermaco- ccus) 1株、微球菌属(Micrococcus) 1株、小单孢菌属(Micromonospora) 1株,可以看出在本试验研究中稀有放线菌多样性比较丰富。
如表 3所示,3种有毒植物纯培养分离内生放线菌的分布情况大致符合根 > 茎 > 叶,其中箭毒木获得菌株分布情况最为明显,而八角枫和马缨丹的某些器官中未能分离获得内生放线菌。依据16S rRNA基因序列相似性小于98%的菌株可能潜在新种的规则[28],分离自八角枫茎的YIM 140292-2与Microbacterium ginsengiterrae DCY37T (EU873314)的相似性为98.1%,该菌株有70%-80%的可能是潜在新种,说明有毒植物中稀有放线菌多样性非常丰富,可能含有大量新的放线菌资源。
| 编号 No. |
宿主植物 Host plant |
器官 Organ |
最相近的菌株(GenBank登录号) Closest cultivated species (GenBank accession No.) |
相似度 Similarity (%) |
| YIM 140329 | 箭毒木 | 根 | Streptomyces hydrogenans NBRC13475T (AB184868) | 99.9 |
| YIM 140330 | 箭毒木 | 根 | Cellulosimicrobium cellulans LMG16121T (CAOI01000359) | 99.9 |
| YIM 140331 | 箭毒木 | 根 | Streptomyces hydrogenans NBRC13475T (AB184868) | 100.0 |
| YIM 140334 | 箭毒木 | 根 | Brachybacterium paraconglomeratum LMG19861T (AJ415377) | 99.3 |
| YIM 140335 | 箭毒木 | 根 | Streptomyces hydrogenans NBRC13475T (AB184868) | 100.0 |
| YIM 140337 | 箭毒木 | 根 | Streptomyces malachitospinus NBRC101004T (AB249954) | 99.6 |
| YIM 140339 | 箭毒木 | 根 | Streptomyces tendae ATCC19812T (D63873) | 99.9 |
| YIM 140340 | 箭毒木 | 根 | Streptomyces malachitospinus NBRC101004T (AB249954) | 99.8 |
| YIM 140341-2 | 箭毒木 | 根 | Microbacterium maritypicum DSM12512T (AJ853910) | 99.9 |
| YIM 140350 | 箭毒木 | 根 | Streptomyces badius NRRLB-2567T (AY999783) | 99.9 |
| YIM 140351 | 箭毒木 | 根 | Streptomyces puniceus NBRC12811T (AB184163) | 100.0 |
| YIM 140352 | 箭毒木 | 根 | Streptomyces drozdowiczii NBRC101007T (AB249957) | 99.9 |
| YIM 140353 | 箭毒木 | 根 | Streptomyces badius NRRLB-2567T (AY999783) | 99.9 |
| YIM 140357 | 箭毒木 | 根 | Mycobacterium neworleansense ATCC49404T (AY457068) | 99.7 |
| YIM 140361 | 箭毒木 | 茎 | Microbacterium paraoxydans CF36T (AJ491806) | 99.1 |
| YIM 140363 | 箭毒木 | 茎 | Streptomyces roseolus NBRC12816T (AB184168) | 99.7 |
| YIM 140372-1 | 箭毒木 | 叶 | Arthrobacter agilis DSM20550T (X80748) | 98.7 |
| YIM 140372-2 | 箭毒木 | 叶 | Arthrobacter agilis DSM20550T (X80748) | 98.7 |
| YIM 140375 | 马缨丹 | 茎 | Micrococcus endophyticus YIM56238T (EU005372) | 99.6 |
| YIM 140378 | 马缨丹 | 茎 | Streptomyces anulatus NRRLB-2000T (DQ026637) | 99.9 |
| YIM 140379-1 | 马缨丹 | 茎 | Verrucosispora andamanensis SP03-05T (JX524154) | 100.0 |
| YIM 140386 | 马缨丹 | 茎 | Streptomyces coelescens DSM40421T (AF503496) | 100.0 |
| YIM 140387-1 | 马缨丹 | 茎 | Streptomyces coelescens DSM40421T (AF503496) | 99.9 |
| YIM 140387-2 | 马缨丹 | 茎 | Microbacterium resistens DMMZ1710T (Y14699) | 99.7 |
| YIM 140390 | 马缨丹 | 茎 | Dietzia cercidiphylli YIM65002T (EU375846) | 100.0 |
| YIM 140395 | 马缨丹 | 叶 | Cellulosimicrobium cellulans LMG16121T (CAOI01000359) | 99.8 |
| YIM 140396 | 马缨丹 | 叶 | Cellulosimicrobium cellulans LMG16121T (CAOI01000359) | 99.9 |
| YIM 140397 | 马缨丹 | 叶 | Microbacterium maritypicum DSM12512T (AJ853910) | 100.0 |
| YIM 140400 | 马缨丹 | 叶 | Streptomyces anulatus NRRLB-2000T (DQ026637) | 99.9 |
| YIM 140401 | 马缨丹 | 叶 | Cellulosimicrobium cellulans LMG16121T (CAOI01000359) | 99.8 |
| YIM 140403 | 马缨丹 | 叶 | Microbacterium murale 1-Gi-001T (HE585693) | 99.4 |
| YIM 140422 | 八角枫 | 根 | Micromonospora chersina DSM44151T (X92628) | 99.4 |
| YIM 140429-2 | 八角枫 | 茎 | Microbacterium ginsengiterrae DCY37T (EU873314) | 98.1 |
| YIM 140430 | 八角枫 | 茎 | Dermacoccus nishinomiyaensis DSM20448T (X87757) | 99.4 |
6种分离培养基获得内生放线菌数目表明:内生放线菌的分离对培养基的营养成分及比例有一定的偏好性,XA培养基分离获得菌株最多且多样性丰富,其次是TWYE > HV > CP,而棉子糖-组氨酸培养基、琥珀酸钠培养基获得菌株很少。寡营养的TWYE有利于内生放线菌的生长,但内生真菌和细菌则不能在营养贫乏的培养基上生长;HV培养基能有效抑制细菌的生长,对稀有放线菌的分离非常有效。稀有放线菌在HV培养基上形成大量孢子,有利于放线菌的形态鉴定[29]。添加纤维素、木聚糖及氨基酸的CP、XA分离效果突出,猜测内生放线菌进化出能利用植物组织纤维素等多聚物作为营养的酶,从而促进其定殖在植物内。
2.2.2 免培养测序与纯培养分离获得放线菌多样性比较如表 4所示,通过高通量测序与纯培养分离从3种有毒植物共获得37个属的内生放线菌,说明有毒植物内生放线菌多样性非常丰富,同时发现两种方法获得的菌属存在较大差别。
| 属 | 纯培养Cultivable | 免培养Non-culture | |||||
| Genus | JDM1.1 | BJF1.1 | MYD1.1 | JDM1.1 | BJF1.1 | MYD1.1 | |
| Acidothermus | + | + | |||||
| Actinoallomurus | + | + | |||||
| Actinocatenispora | + | + | |||||
| Amycolatopsis | + | + | |||||
| Arthrobacter | + | + | + | + | |||
| Bifidobacterium | + | + | + | ||||
| Brachybacterium | + | + | + | + | |||
| Brevibacterium | + | ||||||
| Catenulispora | + | + | |||||
| Cellulomonas | + | + | + | ||||
| Cellulosimicrobium | + | + | |||||
| Collinsella | + | + | + | ||||
| Crossiella | + | + | |||||
| Dermacoccus | + | ||||||
| Dietzia | + | ||||||
| Enterorhabdus | + | + | + | ||||
| Glycomyces | + | + | |||||
| Iamia | + | + | |||||
| Jatrophihabitans | + | + | |||||
| Kribbella | + | + | |||||
| Leucobacter | + | + | + | ||||
| Marmoricola | + | + | + | ||||
| Microbacterium | + | + | + | + | + | + | |
| Micrococcus | + | ||||||
| Mycobacterium | + | ||||||
| Micromonospora | + | ||||||
| Nocardiides | + | + | |||||
| Olsenella | + | + | + | ||||
| Propionibacterium | + | + | |||||
| Pseudonocardia | + | + | |||||
| Rhodococcus | + | + | + | ||||
| Rubrobacter | + | + | |||||
| Sanguibacter | + | + | + | ||||
| Senegalimassilia | + | + | + | ||||
| Sporichthya | + | + | |||||
| Streptomyces | + | +* | + | + | + | + | |
| Verrucosispora | + | ||||||
| Unidentified_Acidimicrobiales | + | + | |||||
| Unidentified_Coriobacteriaceae | + | + | |||||
| Unidentified_Gaiellales | + | + | |||||
| 合计Total | 6 | 4 | 6 | 15 | 29 | 29 | |
| 注:+:检测或分离到该菌属;空缺:未检测或分离到该菌属;*:八角枫中分离获得了链霉属但由于在后续实验受到污染,因此未在内生放线菌与相似菌株(表 3)和化合物合成基因检测(表 5)表格中列出.
Note: +: The genus had been detected or isolated; Blank: The genus had not been detected or isolated; *: The Streptomyces in Alangium chinense (Lour.) Harms had been isolated but polluted in the later exiperiment, which didn’t show in the table 3 and table 5. |
|||||||
高通量测序从3种植物共检测出内生放线菌30个属及3个未鉴定的属,其中箭毒木、八角枫、马缨丹分别检测到15、29、29个属;八角枫和马缨丹的高通量测序的放线菌组成极为相似,仅Actinocatenispora和Brevibacterium两个属之差。箭毒木检测到的放线菌仅15个属,放线菌多样性相对较低,因箭毒木汁液具有剧毒,推测对微生物也是一种“极端环境”。剧毒植物蕴含的放线菌数量、种类可能较少,但特殊菌种或新物种所占比重可能较大。
纯培养分离到的放线菌只有11个属,从箭毒木、八角枫、马缨丹分别获得6、4、6个属,分别占免培养检测放线菌的40%、13.8%、20.6%,可见大部分的放线菌仍然不能纯培养。而纯培养获得的Cellulosimicrobium、Dermacoccus、Dietzia、Micrococcus、Mycobacterium、Micromonospora和Verrucosispora等菌属用免培养技术却未能检测出,可能是16S rRNA基因V4区序列不足以分辨所有物种所致。
2.3 抑菌活性及化合物合成基因检测34株纯培养内生放线菌的抑菌活性筛选结果表明:14.7%内生放线菌具有抗1种及以上指示菌的活性,其中链霉菌属的广谱抗菌活性作用明显,比如从箭毒木根中分离的链霉菌YIM 140335、YIM 140340对枯草芽孢杆菌、金黄色葡萄球菌和白色念珠菌均有较强抑菌活性,其抑菌圈都在10 mm以上。八角枫茎中分离的稀有放线菌YIM 140430对枯草芽孢杆菌具有较强抑菌活性,马缨丹中分离的菌株为检测出具有抑菌活性的菌株。整体抗菌效果为抗革兰氏阳性细菌比抗革兰氏阴性细菌活性强,抗白色念珠菌比抗黑曲霉活性强。
如表 5所示,7类化合物合成基因检测结果:34株菌中有18株(52.9%)含有NRPS,有11株(32.4%)含有PKS-Ⅰ,有13株(38.2%)含有PKS-Ⅱ,有8株(23.5%)含有APH,有6株(17.6%)含有CYP。其中HMG-CoA和SAL这2类化合物合成基因未能检测出。链霉菌化合物合成基因检出率都比稀有放线菌高,尤其是NRPS和PKS的检出率,因为链霉菌的大部分次级代谢产物主要由PKS途径和NRPS途径产生[30]。至少有1个功能基因呈阳性的菌株占70.6%,其中稀有放线菌YIM 140372-2检测出含有APH和CYP这2类化合物合成基因。本研究中内生放线菌抑菌活性与化合物合成基因之间未见明显联系,有待进一步研究。
| 编号 No. |
聚酮合酶-Ⅰ PKS-Ⅰ |
聚酮合酶-Ⅱ PKS-Ⅱ |
非核糖体多肽合成酶 NRPS |
氨基糖苷磷酸转移酶 APH |
羟甲基戊二酸单酰辅酶A还原酶 HMG-CoA |
盐霉素 SAL |
多烯类 CYP |
| YIM 140329 | + | - | + | - | - | - | + |
| YIM 140330 | - | - | - | - | - | - | - |
| YIM 140331 | + | - | + | - | - | - | + |
| YIM 140334 | - | - | - | - | - | - | - |
| YIM 140335 | + | - | + | - | - | - | + |
| YIM 140337 | - | + | + | - | - | - | - |
| YIM 140339 | - | + | + | - | - | - | - |
| YIM 140340 | + | + | + | + | - | - | - |
| YIM 140341-2 | - | - | - | - | - | - | - |
| YIM 140350 | - | + | + | + | - | - | - |
| YIM 140351 | - | + | + | + | - | - | - |
| YIM 140352 | + | + | + | - | - | - | + |
| YIM 140353 | + | - | + | + | - | - | - |
| YIM 140357 | + | - | + | - | - | - | - |
| YIM 140361 | - | - | - | + | - | - | - |
| YIM 140363 | - | + | + | - | - | - | - |
| YIM 140372-1 | - | - | - | - | - | - | + |
| YIM 140372-2 | - | - | - | + | - | - | + |
| YIM 140375 | - | - | - | - | - | - | - |
| YIM 140378 | + | + | + | - | - | - | - |
| YIM 140379-1 | + | + | + | - | - | - | - |
| YIM 140386 | - | + | + | - | - | - | - |
| YIM 140387-1 | - | + | - | - | - | - | - |
| YIM 140387-2 | - | - | - | - | - | - | - |
| YIM 140390 | - | - | - | - | - | - | - |
| YIM 140395 | - | - | - | - | - | - | - |
| YIM 140396 | - | - | - | - | - | - | - |
| YIM 140397 | + | - | - | - | - | - | - |
| YIM 140400 | - | + | + | + | - | - | - |
| YIM 140401 | - | - | - | - | - | - | - |
| YIM 140403 | - | + | + | + | - | - | - |
| YIM 140422 | + | - | - | - | - | - | - |
| YIM 140429-2 | - | - | + | - | - | - | - |
| YIM 140430 | - | - | - | - | - | - | - |
| 注:+:阳性;-:阴性.
Note: +: Positive; -: Negative. |
|||||||
云南省西双版纳热带雨林因为独特的气候条件和地理环境,特有植物的物种多样性丰富。目前对有毒植物的研究主要集中在有毒植物的生物农药开发[5]、内生真菌的分离与抗菌活性筛选[6-8],对有毒植物中内生放线菌的研究较少。本研究通过高通量测序和纯培养分离从3种有毒植物中共获得37个属内生放线菌,说明有毒植物的内生放线菌多样性非常丰富。通过高通量测序从3种有毒植物分离获得的微生物包括门分类水平的古菌域的2个门、细菌域的18个门和暂定的RsaHF231、WD272门。其中箭毒木、八角枫和马缨丹分别为11、16和17个门。在属分类水平检测出30个属的内生放线菌,其中八角枫和马缨丹比箭毒木的微生物多样性更丰富。利用纯培养从3种有毒植物中获得11个属34株菌,其中箭毒木分离获得的菌株多样性为根 > 茎 > 叶,而未能从八角枫的叶中分离出内生放线菌。虽然八角枫和马缨丹中免培养检测的放线菌多样性比箭毒木更丰富,但大部分菌株未能通过纯培养分离获得。有毒植物对人和牲畜等真核生物具有毒害作用[9],本研究从3种有毒植物中获得了大量的放线菌资源,推测部分内生放线菌进化出适应有毒植物体内毒性环境的能力,但内生放线菌在有毒植物中的代谢机制及内生菌与宿主的相互作用有待进一步研究。本研究从西双版纳有毒植物中分离的内生放线菌多样性丰富,与秦盛等[31]对西双版纳热带雨林多种药用植物研究结果类似,大多具有抗菌活性,同时链霉菌菌株的NRPS和PKS基因阳性检出率相对较高。
综上所述,西双版纳热带雨林有毒植物的内生放线菌多样性丰富,抑菌活性突出。可以为生物农药及抗生素的开发提供潜在的菌种资源。
4 结论有毒植物的内生放线菌多样性非常丰富,免培养测序结合纯培养分离能够更好地揭示有毒植物内生放线菌的生物多样性,更加全面地发掘内生放线菌资源。通过抑菌活性和化合物合成基因检测,发现链霉菌属的生物活性作用明显,而且某些稀有放线菌同样具有较好的生物活性,表明西双版纳有毒植物内生放线菌资源具有开发潜力。
致谢: 本文部分实验得到云南大学微生物研究所蒋明星和郑松林同学的帮助,特此感谢!| [1] |
Liu CX. Challenges and thinking of current antibiotic development[J]. Chinese Journal of Antibiotics, 2017, 42(1): 1-12. 刘昌孝. 当代抗生素发展的挑战与思考[J]. 中国抗生素杂志, 2017, 42(1): 1-12. |
| [2] |
Matsumoto A, Takahashi Y. Endophytic actinomycetes: promising source of novel bioactive compounds[J]. The Journal of Antibiotics, 2017, 70(5): 514-519. DOI:10.1038/ja.2017.20 |
| [3] |
Germaine K, Keogh E, Garcia-Cabellos G, et al. Colonisation of poplar trees by gfp expressing bacterial endophytes[J]. FEMS Microbiology Ecology, 2004, 48(1): 109-118. DOI:10.1016/j.femsec.2003.12.009 |
| [4] |
Stierle A, Strobel G, Stierle D. Taxol and taxane production by Taxomyces andreanae, an endophytic fungus of Pacific yew[J]. Science, 1993, 260(5105): 214-216. DOI:10.1126/science.8097061 |
| [5] |
Wang H, Zhang HP, He JJ, et al. Poisonous plant, and their main route of the exploration and utilization[J]. Journal of Gansu Normal Colleges, 2010, 15(2): 29-31. 王瀚, 张和平, 何九军, 等. 有毒植物及其开发利用的主要途径[J]. 甘肃高师学报, 2010, 15(2): 29-31. |
| [6] |
Mijit G, Zhang HT, Abdulla H, et al. Isolation, identification and characterization of endophytic bacteria from medicinal and toxic plants in Xinjiang, China[A]//Abstreets of 2007 Annual Coference of Chinese Society of Microbiolegy[C]. Urumchi: Chinese Society of Microbiology, Xinjiang Autonomous Society for Microbiology, 2007: 94-96 (in Chinese) 吾甫尔·米吉提, 张洪涛, 艾山江·阿布都拉, 等.新疆药用和有毒植物内生菌的分离鉴定及其特性研究[A]//2007年中国微生物学会学术年会论文集[C].乌鲁木齐:中国微生物学会, 新疆自治区微生物学会, 2007: 94-96 |
| [7] |
Zheng Y, Chen YW, Zhang CH, et al. Screening of antibacterial activities of endophytic fungi in Datura arborea[J]. Journal of Fungal Research, 2007, 5(2): 101-106. 郑毅, 陈有为, 张传会, 等. 木本曼陀罗内生真菌抗菌活性的筛选研究[J]. 菌物研究, 2007, 5(2): 101-106. |
| [8] |
Dai WJ, Zhang XH, Huang GX, et al. Isolation, identification, and antimicrobial activity of endophytic fungi from Antiaris toxicaria[J]. Chinese Traditional and Herbal Drugs, 2009, 40(6): 955-957. 戴文君, 张秀环, 黄贵修, 等. 见血封喉内生真菌的分离鉴定及抑菌活性研究[J]. 中草药, 2009, 40(6): 955-957. |
| [9] |
Chen JS, Zheng S. Chinese Toxin Plants[M]. Beijing: Science Press, 1987, 59, 419-421, 612. 陈冀胜, 郑硕. 中国有毒植物[M]. 北京: 科学出版社, 1987, 59, 419-421, 612. |
| [10] |
Rang J, Li L, Tang Q, et al. Comparative study of bacterial DNA extraction methods for the third generation sequencing technology[J]. Journal of Natural Science of Hunan Normal University, 2015, 38(6): 14-20. 穰杰, 李莉, 唐琼, 等. 第三代测序细菌基因组DNA提取方法的比较[J]. 湖南师范大学自然科学学报, 2015, 38(6): 14-20. DOI:10.7612/j.issn.1000-2537.2015.06.003 |
| [11] |
Magoč T, Salzberg SL. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [12] |
Caporaso JG, Kuczynski J, Stombaugh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [13] |
Edgar RC, Haas BJ, Clemente JC, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [14] |
Haas BJ, Gevers D, Earl AM, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [15] |
Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [16] |
Wang Q, Garrity GM, Tiedje JM, et al. Na ve bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [17] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(Database issue): D590-D596. |
| [18] |
Yilmaz P, Parfrey LW, Yarza P, et al. The SILVA and "All-species Living Tree Project (LTP)" taxonomic frameworks[J]. Nucleic Acids Research, 2014, 42(Database issue): D643-D648. |
| [19] |
Qin S, Li J, Chen HH, et al. Isolation, diversity, and antimicrobial activity of rare actinobacteria from medicinal plants of tropical rain forests in Xishuangbanna, China[J]. Applied and Environmental Microbiology, 2009, 75(19): 6176-6186. DOI:10.1128/AEM.01034-09 |
| [20] |
Cao YR. Study on the diversity of culturable actinomycetes in forest soils in southwest China[D]. Kunming: Master's Thesis of Yunnan University, 2009 (in Chinese) 曹艳茹. 西南部分地区森林土壤可培养放线菌多样性研究[D]. 昆明: 云南大学硕士学位论文, 2009 http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y1586291 |
| [21] |
Ayuso-Sacido A, Genilloud O. New PCR primers for the screening of NRPS and PKS-Ⅰ systems in actinomycetes: detection and distribution of these biosynthetic gene sequences in major taxonomic groups[J]. Microbial Ecology, 2005, 49(1): 10-24. DOI:10.1007/s00248-004-0249-6 |
| [22] |
Metsä-Ketelä M, Salo V, Halo L, et al. An efficient approach for screening minimal PKS genes from Streptomyces[J]. FEMS Microbiology Letters, 1999, 180(1): 1-6. DOI:10.1111/fml.1999.180.issue-1 |
| [23] |
Anderson AS, Clark DJ, Gibbons PH, et al. The detection of diverse aminoglycoside phosphotransferases within natural populations of actinomycetes[J]. Journal of Industrial Microbiology and Biotechnology, 2002, 29(2): 60-69. DOI:10.1038/sj.jim.7000260 |
| [24] |
Sigmund JM, Clark DC, RaineyFA, et al. Detection of eubacterial 3-hydroxy-3-methylglutaryl coenzyme a reductases from natural populations of actinomycetes[J]. Microbial Ecology, 2003, 46(1): 106-112. DOI:10.1007/s00248-002-2029-5 |
| [25] |
Hwang YB, Lee MY, Park HJ, et al. Isolation of putative polyene-producing actinomycetes strains via PCR-based genome screening for polyene-specific hydroxylase genes[J]. Process Biochemistry, 2007, 42(1): 102-107. DOI:10.1016/j.procbio.2006.06.031 |
| [26] |
Knirschová R, Nováková R, Fecková L, et al. Multiple regulatory genes in the salinomycin biosynthetic gene cluster of Streptomyces albus CCM 4719[J]. Folia Microbiologica, 2007, 52(4): 359-365. DOI:10.1007/BF02932090 |
| [27] |
Fan XB, Liang QY, Niu MY, et al. The diversity and richness of archaea in the northern continental slope of South China Sea[J]. Microbiology China, 2017, 44(7): 1589-1601. 范习贝, 梁前勇, 牛明扬, 等. 中国南海北部陆坡沉积物古菌多样性及丰度分析[J]. 微生物学通报, 2017, 44(7): 1589-1601. |
| [28] |
Xu LH, Li WJ, Liu ZH. Actinomycetes Systematics—Principle, Methods and Practice[M]. Beijing: Science Press, 2007, 202. 徐丽华, 李文均, 刘志恒. 放线菌系统学——原理、方法及实践[M]. 北京: 科学出版社, 2007, 202. |
| [29] |
Cai AQ, Tian XL, Zhou SN. Analysis of degrading enzyme activities of endophytic actinomycete from rice[J]. Journal of Shaoguan University, 2007, 28(3): 107-109. 蔡爱群, 田新莉, 周世宁. 水稻内生放线菌降解酶活性的分析[J]. 韶关学院学报, 2007, 28(3): 107-109. |
| [30] |
Jenke-Kodama H, Sandmann A, Müller R, et al. Evolutionary implications of bacterial polyketide synthases[J]. Molecular Biology and Evolution, 2005, 22(10): 2027-2039. DOI:10.1093/molbev/msi193 |
| [31] |
Qin S, Zhao LX, Chen Y, et al. Endophytic actinomycetes associated with medicinal plants: isolation, antimicribial activity and identification[J]. Microbiology China, 2009, 36(11): 1693-1699. 秦盛, 赵立兴, 陈云, 等. 药用植物内生放线菌的分离、筛选及活性菌株YIM61470鉴定[J]. 微生物学通报, 2009, 36(11): 1693-1699. |
 2018, Vol. 45
2018, Vol. 45




