扩展功能
文章信息
- 余丹, 毛娉, 宋颀, 宋慧林, 陈静, 梁运祥, 胡咏梅
- YU Dan, MAO Ping, SONG Qi, SONG Hui-Lin, CHEN Jing, LIANG Yun-Xiang, HU Yong-Mei
- 基于高通量测序的传统甜面酱自然发酵过程中的微生物群落结构及其动态演替
- Microbial community and dynamic changes during traditional sweet paste fermentation analyzed by high-throughput sequencing
- 微生物学通报, 2018, 45(5): 1061-1072
- Microbiology China, 2018, 45(5): 1061-1072
- DOI: 10.13344/j.microbiol.china.170744
-
文章历史
- 收稿日期: 2017-09-19
- 接受日期: 2017-12-28
- 网络首发日期(www.cnki.net): 2018-03-19
甜面酱,是以面粉为主要原料,经微生物发酵酿制而成的甜咸适口、具有特殊滋味和体态的中国传统调味品,已成为北京烤鸭等名菜所必不可少的调味酱。尤其是传统曲法自然发酵的甜面酱,经长时间日晒夜露,滋味鲜美、风味独特,拥有浓郁的酱香和酯香,深受消费者喜爱。
目前采用传统可培养方法从甜面酱中已分离鉴定出的微生物主要有霉菌、酵母菌、乳酸菌和芽胞杆菌等[1-4]。由于涉及到的微生物种类繁多,而且发酵过程中菌种间变化较为复杂,因此运用传统可培养方法很难了解到参与甜面酱发酵过程的微生物组成及全貌。运用聚合酶链式反应-变性梯度凝胶电泳(Polymerase chain reaction denaturing gradient gel electrophoresis,PCR-DGGE)技术分析传统甜面酱自然发酵过程中微生物群落结构的变化,鉴定到的优势物种也不够全面[5]。高通量测序技术具有一般读长较短、能细致地分析样品全貌且可最大限度地展示样品中的微生物种类和丰度等特点,所以又称为深度测序[6]。近年来,食品科学界已广泛应用高通量测序技术来动态监测各种食品(如酱油、豆瓣酱、宜宾芽菜和白酒等)发酵过程中微生物的多样性[7-12],但目前尚没有采用该技术分析传统甜面酱自然发酵过程中的微生物群落结构及其动态演替的报道。
本研究采用高通量测序技术系统全面地研究传统甜面酱自然发酵过程中的微生物群落及其动态变化规律,旨在确定其中的优势菌,为甜面酱的生产与质量控制提供理论依据。
1 材料与方法 1.1 材料实验材料取自重庆某公司的传统甜面酱自然发酵过程中的曲样和酱醪(2014年9月5日投料)。
1.2 主要试剂和仪器土壤基因组提取试剂盒,MP Biomedicals公司;DNA聚合酶,北京全式金生物技术有限公司。NanoDrop 2000紫外可见分光光度计、高速冷冻离心机、小型台式离心机、PCR仪,赛默飞世尔科技公司;DNA定量仪和MiSeq测序仪,Illumina公司。
1.3 甜面酱制备和样品采集6 000 kg的面粉加水蒸煮后冷至40–45 ℃,按每kg原料接种米曲霉(Aspergillus oryzae) 108−109个孢子(预混在25 kg干面粉中),在通风曲床上30–35 ℃培养43 h,随后加入盐水(盐的终浓度为7.5%,水的终浓度为55%),在发酵池中进行180 d的后发酵(从2014年9月至2015年2月,当地月平均气温分别为23.7、19.1、13.0、8.0、8.4和9.8 ℃)。本实验取前发酵阶段培养0、18、27和43 h的曲样以及后发酵阶段0、7、14、21、30、60、90、120、150和180 d的酱醪样品。取样方法为5点取样法,选取曲床(取样深度20 cm左右)或酱池(取样深度50 cm左右)四角的样品及中心处样品,在无菌条件下粉碎混匀后,−80 ℃保存备用。
1.4 样品总DNA提取、PCR扩增、产物纯化及定量将曲样或酱醪样品送至上海美吉生物医药科技有限公司,采用土壤基因组提取试剂盒提取总DNA,具体操作方法按照试剂盒内说明书操作。随后进行细菌和真菌特异区域的PCR扩增。16S rRNA基因V3−V4区的测序引物为338F (5′-ACTCCTAC GGGAGGCAGCAG-3′)和806R (5′-GGACTACHVG GGTWTCTAAT-3′)[13-14];ITS1区的引物为ITS1F (5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2 (5′-GCTGCGTTCTTCATCGATGC-3′)[15]。PCR全部实验样本进行3个重复。PCR正式试验采用TransStart FastPfu DNA Polymerase,20 μL反应体系为:5×FastPfu Buffer 4 μL,dNTPs (2.5 mmol/L) 2 μL,正、反向引物(5 μmol/L)各0.8 μL,FastPfu Polymerase (2.5 U/μL) 0.4 μL,BSA (0.8 μg/μL) 0.2 μL,DNA 10 ng,ddH2O补至20 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 45 s,共34个循环;72 ℃ 10 min。采用凝胶回收试剂盒回收PCR产物,用Tris-HCl洗脱,并将PCR产物用DNA荧光定量系统进行检测定量。
1.5 MiSeq文库构建、测序与拼接构建MiSeq文库时,首先通过PCR将Illumina官方接头序列添加至目标区域外端;使用凝胶回收试剂盒切胶回收PCR产物;然后采用Tris-HCl缓冲液洗脱产物,2%琼脂糖电泳检测;最后采用氢氧化钠对PCR产物变性,产生单链DNA片段,用Illumina MiSeq平台进行测序。将MiSeq测序得到的PE reads根据Overlap关系进行拼接,同时对序列质量进行质控和过滤,根据序列首尾两端的Barcode和引物序列区分样品得到有效的序列,并且校正序列方向,使用软件FLASH和Trimmomatic得到优化序列。
1.6 数据分析将测序结果在美吉公司的I-sanger在线生物信息分析平台上进行分析。区分样本后采用Usearch软件进行OTU (Operational taxonomic unit)聚类分析。对于≥99%相似水平下的OTU进行生物信息统计分析,每个OTU代表一个物种[16]。基于OTU聚类分析及各个分类水平下的物种比对结果,利用QIIME软件对OTU进行多种多样性指数分析,以及对测序深度的检测[17];基于分类学信息,在各个分类水平上进行群落结构的统计分析。在各个分类水平统计各样品组成。细菌16S rRNA基因V3−V4区采用Sliva细菌数据库比对;真菌ITS1区采用Unite真菌数据库比对。采用QIIME平台软件和RDP classifier算法,选择置信度阈值≥0.9进行物种注释和丰度分析[18]。
2 结果与分析 2.1 测序数据统计及Alpha多样性统计对14个曲样或酱醪样品的真菌群落进行测序分析,共得到540 914条有效序列。14个样本的Coverage测序深度指数、Chao1指数和Shannon指数统计分析结果见表 1。Coverage指数是指各样本文库的覆盖率,其数值越高则样本中序列被测出的概率越高,该指数反映测序结果是否代表了样本中微生物的真实情况。Chao1指数是用Chao1算法估计样本中所含OTU数目的指数,Chao1在生态学中常用来估计物种总数。Shannon指数是用来估算样本中微生物多样性的指数之一,在生态学中常用来定量描述一个区域的生物多样性,Shannon指数越大说明群落多样性越高。
| 样品名称 Sample |
平均长度 Average length (bp) |
Chao1指数 Chao1 index |
Shannon指数 Shannon index |
覆盖率 Coverage (%) |
| 0 h | 276.9 | 37.75 | 0.139 4 | 99.99 |
| 18 h | 277.0 | 7.50 | 0.008 0 | 100.00 |
| 27 h | 277.0 | 4.00 | 0.004 9 | 100.00 |
| 43 h | 277.0 | 3.00 | 0.002 7 | 100.00 |
| 0 d | 277.0 | 2.00 | 0.004 6 | 100.00 |
| 7 d | 277.0 | 13.00 | 0.023 7 | 99.99 |
| 14 d | 276.8 | 40.20 | 0.111 8 | 99.98 |
| 21 d | 276.9 | 40.17 | 0.030 5 | 99.97 |
| 30 d | 275.6 | 81.50 | 0.469 6 | 99.99 |
| 60 d | 276.7 | 50.75 | 0.068 7 | 99.97 |
| 90 d | 270.9 | 85.20 | 1.160 1 | 99.99 |
| 120 d | 312.7 | 33.00 | 0.316 4 | 99.98 |
| 150 d | 310.7 | 43.11 | 0.589 9 | 99.98 |
| 180 d | 319.6 | 6.00 | 0.020 7 | 99.99 |
由表 1可知,Coverage测序深度指数均大于99.9%,表明测序深度已覆盖到测试样品中的大部分物种,可以真实展示曲样或酱醪中的绝大多数真菌。14个样本的Chao1指数和Shannon指数均较低,可知甜面酱自然发酵过程中真菌物种的多样性不高。在前发酵(制曲)阶段,Chao1指数和Shannon指数均逐渐减小,43 h酱醪不及0 h曲样的1/10,说明制曲过程中真菌物种总数和种类呈减少趋势,制曲阶段真菌物种趋向单一化。盐水的加入可能带入了新的真菌物种,不过也有一定的稀释作用。后发酵阶段样品的Chao1指数和Shannon指数呈先上升后下降的趋势,在90 d样本中达到最大值,分别为85.20和1.160 1,说明甜面酱后发酵90 d时真菌物种最为丰富。随后,真菌物种总量相对减少,变化较大,物种丰度下降明显;至后发酵180 d时,酱醪样品中的Chao1指数和Shannon指数仅分别为6.00和0.020 7。
对14个不同时间段的甜面酱曲样或酱醪样品进行细菌群落测序分析,得到有效序列数为514 079条。多样性指数统计分析结果表明,Coverage测序深度指数均大于99%,表明测序深度已经覆盖到测试样品中大部分物种,可以真实展示样品中的绝大多数细菌(表 2)。样品中细菌的Chao1指数相对真菌而言普遍较大,说明在甜面酱制作过程中细菌高于真菌的物种总数。0 h曲样的Chao1指数较大,为96.57,制曲43 h时降低至10.00,说明前发酵阶段细菌的物种总数不断减少,这可能与曲料中曲霉的旺盛生长繁殖有关。进入后发酵阶段,样品中细菌的Chao1指数逐步上升,14 d达到最大值172.00,在随后的21 d−150 d的样本中略有起伏。180 d样本的细菌Chao1指数降低至后发酵阶段的最低值(79.80),与150 d样本的Chao1指数(170.48)有较大差别。
| 样品名称 Sample |
平均长度 Average length (bp) |
Chao1指数 Chao1 index |
Shannon指数 Shannon index |
覆盖率 Coverage (%) |
| 0 h | 431.9 | 96.57 | 2.135 0 | 99.80 |
| 18 h | 437.4 | 51.17 | 1.290 0 | 99.83 |
| 27 h | 438.4 | 57.20 | 0.940 4 | 99.79 |
| 43 h | 449.6 | 10.00 | 0.110 2 | 99.95 |
| 0 d | 443.6 | 81.00 | 1.421 2 | 99.70 |
| 7 d | 437.4 | 96.00 | 1.186 9 | 99.65 |
| 14 d | 443.1 | 172.00 | 3.076 5 | 99.79 |
| 21 d | 449.0 | 108.88 | 1.222 0 | 99.65 |
| 30 d | 445.9 | 142.16 | 2.099 8 | 99.74 |
| 60 d | 448.7 | 123.25 | 1.868 9 | 99.82 |
| 90 d | 449.1 | 154.36 | 1.824 1 | 99.74 |
| 120 d | 449.0 | 122.67 | 1.215 8 | 99.62 |
| 150 d | 449.7 | 170.48 | 0.880 0 | 99.29 |
| 180 d | 449.6 | 79.80 | 0.814 9 | 99.74 |
在整个发酵时期,细菌样本的Shannon指数波动较大,变化趋势不如Chao1指数明显。43 h样品的Shannon指数从制曲0 h的2.135 0降低到最小值0.110 2,表明制曲结束时样本细菌群落组成最为简单。14 d样本的Shannon指数达最大值3.076 5,说明后发酵14 d样本中细菌群落组成最为复杂。总体来讲,样本细菌的Shannon指数远大于真菌的Shannon指数,说明在传统甜面酱自然发酵过程中细菌物种丰富度远高于真菌。
2.2 样品间Beta多样性分析对传统甜面酱自然发酵过程中的14个曲样或酱醪样本进行种水平上的主成分(Principal component analysis,PCA)分析,如图 1所示。主成分1 (PC1)和主成分2 (PC2)分别占所有差异的97.95%和1.89%。根据距离远近进行归类,将14个样本分为3大类。PCA分析结果显示,120 d、150 d和180 d酱醪样品的距离最近,样本最相似性归为一类;样品间Beta多样性分析发现,后发酵90 d的酱醪中甜面酱真菌的多样性最高,具有最高的Chao1指数和Shannon指数。90 d酱醪样品和其他样品距离较远,物种总量和丰度具有特异性,单独归为一类;其余样品距离较近,在物种组成和比例上类似,因此也归为一类。

|
| 图 1 属水平上真菌样本的PCA分析 Figure 1 The principal component analysis of fungal genes at the species level |
|
|
对传统甜面酱自然发酵过程中的14个曲样或酱醪样本进行属水平上的PCA分析(图 2),研究细菌样本的相似性和差异性。主成分1 (PC1)和主成分2 (PC2)分别占所有差异的73.26%和22.71%,根据距离远近进行归类,将14个样本共分为5大类。由图 2可见,14 d酱醪样本和其他样本距离较远,说明后发酵14 d的酱醪中细菌的多样性最高,具有最高的Chao1和Shannon指数,因此归为单独的一类。43 h曲样的细菌组成最为简单,这个样本与其他样本相隔距离也较远,也归为单独的一类。27 h曲样和7 d、21 d、120 d、150 d以及180 d的酱醪样本距离最近,归为一类。0 h曲样和30 d、60 d以及90 d的酱醪样本具有相似的细菌组成,归为一类。还有一类是18 h曲样和0 d酱醪的样本,距离最近。

|
| 图 2 属水平上细菌样本的PCA分析 Figure 2 The principal component analysis of bacteria genes at the genus level |
|
|
采用RDP classifier对各样品中的OTU依次进行门(Phylum)纲(Class)目(Order)科(Family)属(Genus)等分类水平的信息分析,一共获得6个门水平真菌,由子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)和壶菌门(Chytridiomycota)以及无法归类的真菌(Unclassified和Norank)组成;还有29个门水平细菌,由厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia)以及一些无法归类的细菌(Unclassified)和其他(Others)相对丰度较小的门细菌组成。
由图 3可知,传统甜面酱自然发酵过程中在门水平上的优势真菌为子囊菌门(Ascomycota)。子囊菌门在各个样品中的相对丰度均较高,除了90 d酱醪样品,在其他样品中含量接近100%。90 d的酱醪样品在门水平多样性较高,含有82.84%子囊菌门(Ascomycota)、13.07%担子菌门(Basidiomycota)、3.77%无法归类的真菌和其他门类。

|
| 图 3 门水平上真菌样本的群落组成分析 Figure 3 The community abundance analysis of fungal genes at the phylum level |
|
|
门水平上的细菌群落结构如图 4所示,结果表明整个发酵过程优势菌门为厚壁菌门(Firmicutes),次优势菌门为变形菌门(Proteobacteria)。14 d酱醪样品在门水平上的多样性较高,含有48.97%厚壁菌门(Firmicutes)、21.16%变形菌门(Proteobacteria)、20.44%拟杆菌门(Bacteroidetes)、4.91%疣微菌门(Verrucomicrobia)、2.01%放线菌门(Actinobacteria)和2.52%其他菌门。
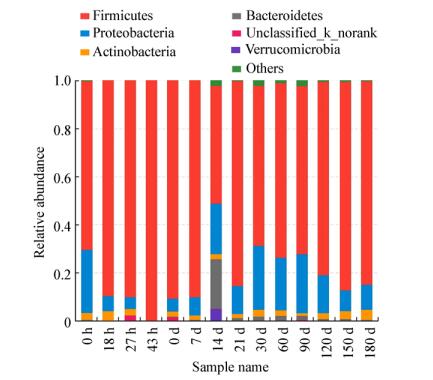
|
| 图 4 门水平上细菌样本的群落组成分析 Figure 4 The community abundance analysis of bacteria genes at the phylum level |
|
|
传统甜面酱自然发酵过程中在属水平上共检出100个真菌菌属,主要有曲霉属(Aspergillus)、接合酵母属(Zygosaccharomyces)、梗孢酵母属(Sterigmatomyces)、假丝酵母属(Candida)、Wallemia、其他菌属和无法归类的真菌。如图 5所示,在前发酵阶段和后发酵90 d前,样品中真菌属水平上优势菌属为曲霉属(Aspergillus),后发酵120 d、150 d、180 d样品中大量含有接合酵母属(Zygosaccharomyces),并且相对丰度含量较高,分别为91.06%、85.42%、99.72%。90 d酱醪样品在属水平上的多样性较高,含有76.96%曲霉属(Aspergillus)、9.04%梗孢酵母属(Sterigmatomyces)、2.04%假丝酵母属(Candida)、1.80% Wallemia、6.38%其他菌属和3.77%无法归类的真菌。
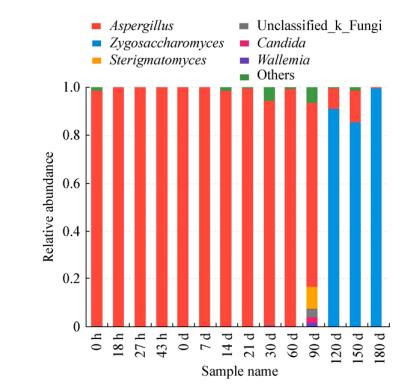
|
| 图 5 属水平上真菌样本的群落组成分析 Figure 5 The community abundance analysis of fungal genes at the genus level |
|
|
传统甜面酱自然发酵过程中在属水平上共检出432个细菌菌属,主要有芽胞杆菌属(Bacillus)、葡萄球菌属(Staphylococcus)、雷尔氏菌属(Raistonia)和泛菌属(Pantoea)等。
由图 6可见,芽胞杆菌属(Bacillus)在整个发酵阶段一直是稳定的优势细菌菌属。样本丰度较大的另一细菌菌属为葡萄球菌属(Staphylococcus)。通过高通量测序分析发现,18 h曲样中的Staphylococcus含量较多,后期占样本的比例逐步减少。0 d酱醪的样本中也检测到大量的葡萄球菌属,推测与酱池环境或盐水的加入有关。随着后发酵的进行,葡萄球菌属所含比例又逐步减少。此外,样本中被检出的丰度位于前10的细菌菌属还有雷尔氏菌属(Raistonia)、泛菌属(Pantoea)、伯克霍尔德氏菌属(Burkholderia)、片球菌属(Pediococcus)、鞘氨醇单胞菌属(Sphingomonas)、双歧杆菌属(Bifidobacterium)、霍氏真杆菌属(Faecalibacterium)和考克式菌属(Kocuria)等。
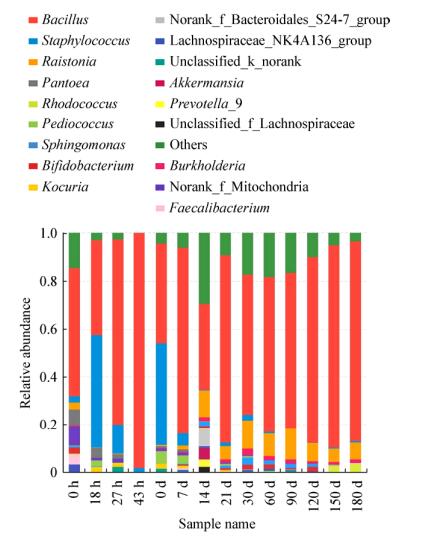
|
| 图 6 属水平上细菌样本的群落组成分析 Figure 6 The community abundance analysis of bacteria genes at the genus level |
|
|
群落Heatmap图是通过颜色区别来展示和表现二维矩阵或者表格中的数据大小,并呈现群落物种的组成信息。采用群落Heatmap图分析传统甜面酱自然发酵过程中所有真菌物种中含量前10个菌属和甜面酱发酵过程的14个样品之间的交互关系,见图 7。
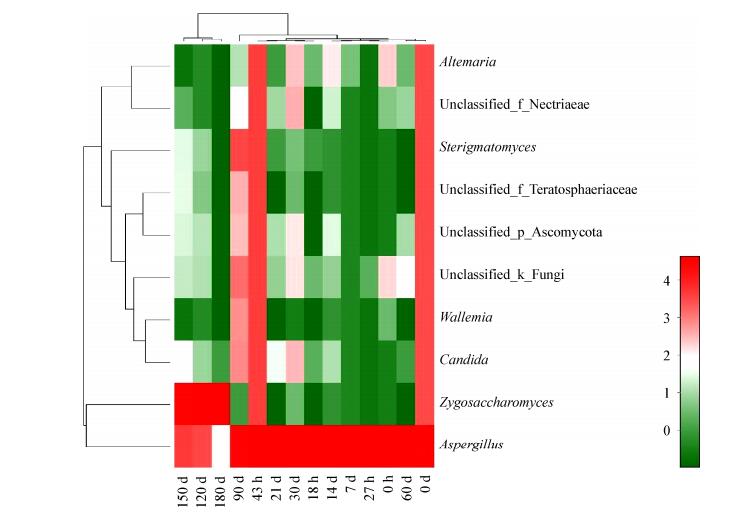
|
| 图 7 属水平上真菌样本的群落Heatmap图 Figure 7 The community heatmap of fungal genes at the genus level 注:热图下侧和右侧分别为样品名称和物种名称,左侧和上侧分别为物种聚类树和样本聚类树.热图中颜色的深浅分别代表物种丰度的高低. Note: The bottom and the right side of the heat map are the sample name and species name respectively, the left and the upper side are the species clustering tree and the sample clustering tree respectively. The shade of color represents the abundance of species. |
|
|
如图 7所示,在14个样品中Aspergillus和Zygosaccharomyces菌属的距离比较接近,归属同一分支,作为优势菌属在传统甜面酱自然发酵的过程中此消彼长。梗孢酵母菌属(Sterigmatomyces)、假丝酵母菌属(Candida)、Wallemia和一个无法归类的真菌菌属在90 d酱醪样本中含量较大,但在120 d、150 d和180 d的酱醪样本中含量很少。
图 8是传统甜面酱曲样或酱醪样品属水平上的细菌群落Heatmap图,反映了所有样品中细菌中含量前10个菌属和甜面酱发酵过程中的14个样品之间交互关系。由图 8可知,芽胞杆菌属(Bacillus)和葡萄球菌属(Staphylococcus)在各个样本中含量均较为丰富。罗尔斯通菌属(Ralstonia)从酱醪发酵14 d后丰度变大,伯克霍尔德氏菌属(Burkholderia)、鞘氨醇单胞菌属(Sphingomonas)和双歧杆菌属(Bifidobacterium)在发酵后期含量增多,推测这些菌属可能与甜面酱成熟有一定联系。泛菌属(Pantoea)、片球菌属(Pediococcus)和考克式菌属(Kocuria)在制曲阶段含量较多,发酵后期减少,推测这些细菌菌属能较好地适应制曲阶段的环境。
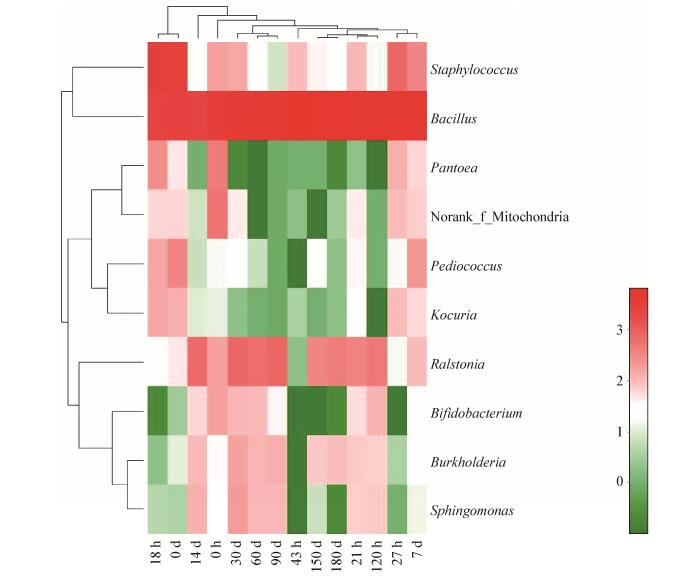
|
| 图 8 属水平上细菌样本的群落Heatmap图 Figure 8 The community heatmap of bacteria genes at the genus level 注:热图下侧和右侧分别为样品名称和物种名称,左侧和上侧分别为物种聚类树和样本聚类树.热图中颜色的深浅分别代表物种丰度的高低. Note: The bottom and the right side of the heat map are the sample name and species name respectively, the left and the upper side are the species clustering tree and the sample clustering tree respectively. The shade of color represents the abundance of species. |
|
|
本文采用高通量测序技术从传统甜面酱曲样和酱醪中共鉴定到100个真菌菌属和432个细菌菌属。对比Chao1和Shannon指数以及样品间Beta多样性分析发现,后发酵90 d的酱醪中甜面酱真菌的多样性最高,而细菌的多样性是在后发酵14 d时最高。推测在后发酵14 d许多细菌和丝状真菌因无法适应酱醪的高盐度或高水分而相继死亡,而代之以一些耐盐的酵母菌,且在后发酵90 d时达到峰值。前发酵阶段和后发酵0 d–90 d的优势真菌为曲霉属(Aspergillus,≥76.96%)。后发酵120 d–180 d的优势真菌为接合酵母菌属(Zygosaccharomyces,≥85.42%)。此外还有少量假丝酵母菌属(Candida)和梗孢酵母菌属(Sterigmatomyces)等菌属。芽胞杆菌属(Bacillus)在整个发酵阶段一直是最主要的优势细菌(其中前发酵43 h时占98.07%)。曲样和酱醪中的优势细菌菌属还有葡萄球菌属(Staphylococcus)、雷尔氏菌属(Raistonia)、泛菌属(Pantoea)、伯克霍尔德氏菌属(Burkholderia)、片球菌属(Pediococcus)、鞘氨醇单胞菌属(Sphingomonas)、双歧杆菌属(Bifidobacterium)、霍氏真杆菌属(Faecalibacterium)、考克式菌属(Kocuria)和乳杆菌属(Lactobacillus)等。
高通量测序结果表明,前发酵阶段和后发酵阶段前期(后发酵0–90 d)的主要优势真菌菌属是曲霉属。结合甜面酱生产工艺,在制曲阶段接入的起始菌株是米曲霉(Aspergillus oryzae)沪酿3.042,因此可推断大量存在的是米曲霉。米曲霉不仅产生丰富的淀粉酶类和蛋白酶,将物料中的淀粉和蛋白质等大分子物质降解为小分子的糖和氨基酸等,直接影响甜面酱的理化指标(如还原糖和氨基酸态氮等),而且有较强的产生醇类和酯类物质的能力,对发酵产物中的风味物质形成也有一定的影响[19-21]。
最近叶碧霞等采用传统可培养技术发现,保温发酵0–60 d的甜面酱中的优势真菌菌株均为米曲霉,没有检测到酵母菌[22]。与之相比,自然发酵的甜面酱中检测到较多的酵母菌。酵母菌在发酵繁殖阶段,通过酯化酶利用乙醇和细菌产生的有机酸生成酯类物质,形成甜面酱的风味主体[23]。本研究发现,除了米曲霉外,大量的酵母菌也参与了传统甜面酱的发酵;米曲霉和酵母菌在传统甜面酱自然发酵中此消彼长。在后发酵阶段,作为丝状真菌的米曲霉因无法适应酱醪的高盐度和高水分而逐渐消亡,代之以耐盐的酵母菌,尤其是结合酵母菌属(Zygosaccharomyces)。传统自然发酵之所以滋味鲜美、风味独特,拥有浓郁的酱香和酯香,推测主要有两个方面的原因:(1)传统甜面酱自然发酵的时间较长,一般为180 d左右,为微生物代谢积累风味物质提供了时间条件;(2)自然发酵会从自然环境中接入各种风味酵母菌,酵母菌代谢产生的醇类以及酯类(与细菌产生的酸类形成)为甜面酱提供了更丰富的风味物质。
据文献报道,芽胞杆菌(Bacillus)在酱油和非洲槐豆发酵中所产生的无机酸、氨基酸和酯类对复杂风味的形成具有重要作用[24-25]。芽胞杆菌分泌的淀粉酶、杀真菌素和纤溶蛋白酶会促进豆豉发酵后熟,并且有益于人类身体健康[25-27]。沈芳等发现在保温发酵和自然发酵的甜面酱中均可筛选到芽胞杆菌[3];吉志伟等也从自然发酵的甜面酱中筛选到芽胞杆菌[4];在郫县豆瓣的研究中,运用可培养方法确定芽胞杆菌也是优势细菌[28-29],说明芽胞杆菌在甜面酱和豆瓣酿造过程中广泛存在,这可能与芽胞杆菌耐热及抗逆性有关。本研究基于高通量测序技术发现,传统甜面酱自然发酵中芽胞杆菌作为优势细菌在整个发酵阶段一直稳定存在。这与Mao等PCR-DGGE的检测结果较为一致[5]。在酱醪和曲样中均检测到芽胞杆菌的存在,说明芽胞杆菌能够适应甜面酱发酵过程中的各种环境条件变化(例如盐水的加入、酵母菌的繁殖和户外发酵等),与甜面酱的风味形成也有一定的关联。从传统食品中分离和筛选优势微生物可能是获得功能发酵微生物的一种重要研究手段。直接添加筛选的真菌和细菌到原料中作为发酵起始菌株去制作发酵食品也已经形成一种突破,可以达到高度控制发酵过程和得到标准化产品的目的[30]。例如,目前从传统调味品中分离筛选的风味酵母菌(湖北安琪公司生产的鲁氏酵母JS和球拟酵母R)已被商业化生产,已成功用于酱油和酱类的酿造中[31-33]。微生物的种类和数量比例对传统酿造食品风味的形成起到了至关重要的作用。结合理化指标的动态变化规律,还可以深入探讨甜面酱质量和风味与微生物活动之间的关联[5]。例如产香酵母代谢产生的醇类或酯类等物质决定了酱类的风味和香气,这些风味菌对甜面酱的风味提升具有重要的意义[34]。因此,本研究运用高通量测序技术手段,不仅能全面地揭示参与传统甜面酱自然发酵过程中的微生物群落及其动态变化规律,为研究微生物对传统甜面酱风味形成的影响提供理论依据,而且为后续优势功能菌的分离筛选以及人工接种风味菌以提高甜面酱质量奠定了坚实的基础。
| [1] |
Liu CX, Meng Y, Li DS, et al. Isolation and initiative identification of dominant bacteria from the traditional sweet flour paste koji[J]. China Condiment, 2011, 36(5): 84-87. 刘彩香, 孟鸳, 李冬生, 等. 传统甜面酱种曲中优势菌种的分离及初步鉴定[J]. 中国调味品, 2011, 36(5): 84-87. |
| [2] |
Shi JJ, Zhang JJ, Deng J, et al. Identification of aroma-producing and thermostable yeast strains from the Spontaneous fermented sweet sauce and detection of their volatile aroma components[J]. Food and Fermentation Industries, 2014, 40(9): 167-171. 石娇娇, 张建军, 邓静, 等. 自然发酵甜面酱中耐高温生香酵母的鉴定与挥发性香气成分分析[J]. 食品与发酵工业, 2014, 40(9): 167-171. |
| [3] |
Shen F, Wu HC, Deng J, et al. Analysis on microorganism in the course of different fermentation techniques of sweet flour paste in Sichuan[J]. China Condiment, 2013, 38(6): 41-45, 54. 沈芳, 吴华昌, 邓静, 等. 四川甜面酱不同发酵工艺中的菌相分析[J]. 中国调味品, 2013, 38(6): 41-45, 54. |
| [4] |
Ji ZW, Liu Y, Deng J, et al. Isolation and identification of a Bacillus strain from the spontaneous fermented sweet bean sauce and preliminary study on its probiotic properties[J]. China Condiment, 2015, 40(12): 26-30. 吉志伟, 刘阳, 邓静, 等. 自然发酵甜面酱中一株芽孢杆菌的分离鉴定及益生特性初探[J]. 中国调味品, 2015, 40(12): 26-30. DOI:10.3969/j.issn.1000-9973.2015.12.007 |
| [5] |
Mao P, Hu YL, Liao TT, et al. Microbial diversity during fermentation of sweet paste, a Chinese traditional seasoning, using PCR-denaturing gradient gel electrophoresis[J]. Journal of Microbiology and Biotechnology, 2017, 27(4): 678-684. DOI:10.4014/jmb.1605.05011 |
| [6] |
Sultan M, Schulz MH, Richard H, et al. A global view of gene activity and alternative splicing by deep sequencing of the human transcriptome[J]. Science, 2008, 321(5891): 956-960. DOI:10.1126/science.1160342 |
| [7] |
Sulaiman J, Gan HM, Yin WF, et al. Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine[J]. Frontiers in Microbiology, 2014, 5: 556. |
| [8] |
Wu LH, Lu ZM, Gong JS, et al. Application of next generation sequencing in studying food microorganisms—a review[J]. Chinese Journal of Biotechnology, 2016, 32(9): 1164-1174. 吴林寰, 陆震鸣, 龚劲松, 等. 高通量测序技术在食品微生物研究中的应用[J]. 生物工程学报, 2016, 32(9): 1164-1174. |
| [9] |
Mi QL, Li XM, Guan Y, et al. Application of high-throughput sequencing in food microbial ecology: a review[J]. Food Science, 2016, 37(23): 302-308. 米其利, 李雪梅, 管莹, 等. 高通量测序在食品微生物生态学研究中的应用[J]. 食品科学, 2016, 37(23): 302-308. DOI:10.7506/spkx1002-6630-201623049 |
| [10] |
Zhou HL, Li S, Zhang L, et al. Bacterial diversity during natural fermentation of soybean paste under variable temperature conditions[J]. Food Science, 2017, 38(14): 120-126. 周红丽, 李莎, 张灵, 等. 变温发酵模式下豆瓣酱自然发酵过程中细菌多样性研究[J]. 食品科学, 2017, 38(14): 120-126. DOI:10.7506/spkx1002-6630-201714018 |
| [11] |
Zuo Y, Wang XL, Ye BX, et al. Based on high-throughput sequencing analysis of bacterial community structure in Yibin sprouts[J]. Science and Technology of Food Industry, 2016, 37(10): 242-245, 250. 左勇, 王小龙, 叶碧霞, 等. 基于高通量测序对宜宾芽菜中细菌群落结构分析[J]. 食品工业科技, 2016, 37(10): 242-245, 250. |
| [12] |
Huang YN, Xiong XM, Hu YL, et al. Bacterial community and diversity in pit mud of Baiyunbian liquor analyzed by PCR-DGGE and high-throughput sequencing[J]. Microbiology China, 2017, 44(2): 375-383. 黄莹娜, 熊小毛, 胡远亮, 等. 基于PCR-DGGE和高通量测序分析白云边酒窖泥细菌群落结构与多样性[J]. 微生物学通报, 2017, 44(2): 375-383. |
| [13] |
Walter J, Tannock GW, Tilsala-Timisjarvi A, et al. Detection and identification of gastrointestinal Lactobacillus species by using denaturing gradient gel electrophoresis and species-specific PCR primers[J]. Applied and Environmental Microbiology, 2000, 66(1): 297-303. DOI:10.1128/AEM.66.1.297-303.2000 |
| [14] |
Yu Y, Lee C, Kim J, et al. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J]. Biotechnology and Bioengineering, 2005, 89(6): 670-679. DOI:10.1002/(ISSN)1097-0290 |
| [15] |
Gardes M, Bruns TD. ITS primers with enhanced specificity for basidiomycetes-application to the identification of mycorrhizae and rusts[J]. Molecular Ecology, 1993, 2(2): 113-118. DOI:10.1111/j.1365-294X.1993.tb00005.x |
| [16] |
Edgar RC. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [17] |
Kemp PF, Aller JY. Bacterial diversity in aquatic and other environments: what 16S rDNA libraries can tell us[J]. FEMS Microbiology Ecology, 2004, 47(2): 161-177. DOI:10.1016/S0168-6496(03)00257-5 |
| [18] |
Wang Q, Garrity GM, Tiedje JM, et al. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [19] |
Meng Y, Qiao Y, Kang X, et al. The analysis of volatile components in sweet flour paste strain[J]. Science and Technology of Food Industry, 2011, 32(12): 229-232. 孟鸳, 乔宇, 康旭, 等. 甜面酱成曲的挥发性成分分析[J]. 食品工业科技, 2011, 32(12): 229-232. |
| [20] |
Feng YZ, Cui C, Zhao HF, et al. Effect of koji fermentation on generation of volatile compounds in soy sauce production[J]. International Journal of Food Science and Technology, 2013, 48(3): 609-619. DOI:10.1111/ijfs.12006 |
| [21] |
Zhao GZ, Yao YP, Wang CL, et al. Comparative genomic analysis of Aspergillus oryzae strains 3.042 and RIB40 for soy sauce fermentation[J]. International Journal of Food Microbiology, 2013, 164(2/3): 148-154. |
| [22] |
Ye BX, Zuo Y, Lai L, et al. Research on microorganism in sweet flour paste during the insulation fermentation[J]. China Condiment, 2017, 42(4): 80-84. 叶碧霞, 左勇, 赖琳, 等. 甜面酱保温发酵过程中微生物的研究[J]. 中国调味品, 2017, 42(4): 80-84. |
| [23] |
Zeng CW. Analysis on microorganism and flavor substance in the course of the sweet flour paste brewing[D]. Wuhan: Master's Thesis of Hubei University of Technology, 2009 (in Chinese) 曾灿伟. 甜面酱酿制过程中的菌相分析及风味成分研究[D]. 武汉: 湖北工业大学硕士学位论文, 2009 |
| [24] |
Hui YH, Meunier-Goddik L, Hansen ÅS, et al. Handbook of Food and Beverage Fermentation Technology[M]. New York: Marcel Dekker, 2004, 514-548.
|
| [25] |
Ouoba LⅡ, Diawara B, Annan NT, et al. Volatile compounds of Soumbala, a fermented African locust bean (Parkia biglobosa) food condiment[J]. Journal of Applied Microbiology, 2005, 99(6): 1413-1421. DOI:10.1111/jam.2005.99.issue-6 |
| [26] |
Jeyaram K, Singh WM, Premarani T, et al. Molecular identification of dominant microflora associated with 'Hawaijar'-a traditional fermented soybean (Glycine max (L.)) food of Manipur, India[J]. International Journal of Food Microbiology, 2008, 122(3): 259-268. DOI:10.1016/j.ijfoodmicro.2007.12.026 |
| [27] |
Gilardi G, Baudino M, Garibaldi A, et al. Efficacy of biocontrol agents and natural compounds against powdery mildew of zucchini[J]. Phytoparasitica, 2012, 40(2): 147-155. DOI:10.1007/s12600-011-0206-0 |
| [28] |
Dong D, Guan TW, Zhao HP, et al. Differences in microbial diversity at two different fermentation stages of soybean paste[J]. China Brewing, 2014, 33(11): 55-58. 董丹, 关统伟, 赵辉平, 等. 两个不同发酵时期豆瓣中微生物多样性的差异对比[J]. 中国酿造, 2014, 33(11): 55-58. DOI:10.11882/j.issn.0254-5071.2014.11.012 |
| [29] |
Zhao HP, Guan TW, Dong D, et al. Isolation of culturable microorganisms in Pixian bean paste and its phylogenetic diversity analysis and enzyme activity screening[J]. China Condiment, 2015, 40(8): 5-9. 赵辉平, 关统伟, 董丹, 等. 郫县豆瓣中可培养细菌多样性分析及酶活性初筛[J]. 中国调味品, 2015, 40(8): 5-9. |
| [30] |
Leroy F, de Vuyst L. Lactic acid bacteria as functional starter cultures for the food fermentation industry[J]. Trends in Food Science and Technology, 2004, 15(2): 67-78. DOI:10.1016/j.tifs.2003.09.004 |
| [31] |
Gong HK. Tendency analysis on natural ferment of soy-bean paste and research on fermentation using multi-bacteria via artificial inoculation[D]. Wuxi: Master's Thesis of Jiangnan University, 2004 (in Chinese) 贡汉坤. 传统豆酱自然发酵的动态分析及人工接种多菌种发酵研究[D]. 无锡: 江南大学硕士学位论文, 2004 |
| [32] |
Zhao JX, Dai XJ, Liu XM, et al. Changes in microbial community during Chinese traditional soybean paste fermentation[J]. International Journal of Food Science and Technology, 2009, 44(12): 2526-2530. DOI:10.1111/ifs.2009.44.issue-12 |
| [33] |
Lei JC, Li ZJ, Zhou J, et al. The application of active dry yeast in soy sauce brewing[J]. China Brewing, 2012, 31(6): 162-165. 雷锦成, 李志军, 周俊, 等. 活性干酵母在酱油中的应用[J]. 中国酿造, 2012, 31(6): 162-165. |
| [34] |
Lee SJ, Ahn B. Comparison of volatile components in fermented soybean pastes using simultaneous distillation and extraction (SDE) with sensory characterisation[J]. Food Chemistry, 2009, 114(2): 600-609. DOI:10.1016/j.foodchem.2008.09.091 |
 2018, Vol. 45
2018, Vol. 45




