扩展功能
文章信息
- 张博阳, 朱天辉, 韩珊, 王莹, 李姝江, 谯天敏
- ZHANG Bo-Yang, ZHU Tian-Hui, HAN Shan, WANG Ying, LI Shu-Jiang, QIAO Tian-Min
- 桑氏链霉菌几丁质酶ChiKJ40基因的克隆表达及其抑菌作用
- Cloning, expression and antibacterial functions of ChiKJ40, a chitinase gene from Streptomyces sampsonii
- 微生物学通报, 2018, 45(5): 1016-1026
- Microbiology China, 2018, 45(5): 1016-1026
- DOI: 10.13344/j.microbiol.china.170572
-
文章历史
- 收稿日期: 2017-07-22
- 接受日期: 2017-09-20
- 网络首发日期(www.cnki.net): 2017-10-16
几丁质又称甲壳素,是由N-乙酰基-D-氨基葡萄糖胺通过β-1, 4糖苷键连接而成的线性多糖[1]。几丁质是地球上仅次于纤维素的第二丰富的生物聚合物,广泛存在于甲壳类如螃蟹和虾的壳、昆虫的外骨骼、真菌、酵母、藻类和其他无脊椎动物的内部结构中[2]。几丁质酶(EC 3.2.1.14)是一类可水解几丁质的糖基水解酶,几丁质酶能够降解几丁质成低分子量壳寡糖的功能广泛应用于工业、农业和医疗[3],如诱导、抗肿瘤、生物防治植物病原真菌和有害昆虫中的作用[4]。几丁质酶具有可诱导性、安全无毒副作用等特点,几丁质酶和产生几丁质酶的菌株除了可以单独用于植物病虫害防治以外,也可以通过和其他抗生素与杀虫剂共同使用来减少其他化学试剂的用量,进而减少对环境和人类健康的危害,因此其在农林业上的应用范围正在不断扩大。
根据几丁质酶的氨基酸序列同源性,将几丁质酶划分成5种类型,归类为2个家族,即18家族和19家族糖基水解酶[5]。但是,近年来确定了一些几丁质酶属于20、23家族和48家族的糖基水解酶[6]。18家族几丁质酶在生物体中广泛分布,包括细菌、真菌、植物、哺乳动物、昆虫和病毒等[7],19家族几丁质酶主要存在于植物中,在一些放线菌中也被发现。桑氏链霉菌(Streptomyces sampsonii)能够产生多种胞外水解酶[8-10],几丁质酶是其中之一。S. sampsonii KJ40是本实验室从健康杨树的根际土壤分离到的一株产几丁质酶且对紫丝核菌(Rhizoctonia violacea)生长有明显抑制作用的生防链霉菌,可以预防杨树紫纹羽病的发生,显著提高杨树生长能力[11]。目前,虽然国内外研究人员从土壤[8-9, 11]和海沉积物[10]中分离到多株产几丁质酶的S. sampsonii,但对S. sampsonii几丁质酶的研究主要局限在产几丁质酶菌株发酵粗提液的生物活性上。虽然雷美艳[12]从S. sampsonii基因组中扩增并克隆得到411 bp和465 bp大小的2个几丁质酶基因片段,但S. sampsonii单个几丁质酶完整基因的克隆及其在大肠杆菌(Escherichia coli)中的表达未见报道。本研究克隆S. sampsonii的几丁质酶基因,构建其pET系列的原核表达载体,表达纯化重组蛋白,观察重组蛋白抑菌活性,为链霉菌几丁质酶后续理论研究和大规模生产奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株、载体及培养基S. sampsonii KJ40由四川农业大学森林保护林木病理实验室保存提供,该菌株已于2012年4月13日保藏在中国微生物菌种保藏管理委员会普通微生物中心,保藏编号为CGMCC 5996;桉树焦枯病菌(Cylindrocladium scoparium)、栗疫病菌(Cryphonectria parasitica)、链格孢菌(Alternaria alternata)、紫丝核菌(Rhizoctonia violacea)由四川农业大学森林保护林木病理实验室保存提供。E. coli DH5α和E. coli BL21(DE3)购于北京全式金生物技术有限公司。
pMD19-T载体用于构建克隆载体,购于宝生物工程(大连)有限公司;pET-32a载体用于构建表达载体,由四川农业大学森林保护林木病理实验室保存提供;引物合成和基因测序由成都擎科梓熙公司完成。
营养肉汤培养基(NB,g/L):蛋白胨10.0,牛肉膏5.0,氯化钠5.0,pH 7.2-7.4。LB液体培养基(g/L):胰蛋白胨10.0,酵母膏5.0,氯化钠10.0,pH 7.0。固体培养基则在每升液体培养基中加琼脂15 g。
1.1.2 主要试剂和仪器溶菌酶(Lysozyme)、氨苄青霉素(Ampicillin)、5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-Gal)、1×磷酸盐缓冲液(PBS,pH 7.2-7.4,0.01 mol/L)和Bradford蛋白浓度测定试剂盒购于北京索莱宝科技有限公司;异丙基硫代半乳糖苷(IPTG)、细菌基因组DNA提取试剂盒、通用型DNA纯化回收试剂盒、质粒小提试剂盒购于天根生化科技(北京)有限公司;SolutionⅠ、DL2000 DNA marker、DL5000 DNA marker购于宝生物工程(大连)有限公司;2×TransTaq High Fidelity (HiFi) PCR SuperMix Ⅰ、2×TransTaq High Fidelity (HiFi) PCR SuperMix Ⅱ购于北京全式金生物技术有限公司;DNA限制性内切酶Hind Ⅲ、BamHⅠ、XhoⅠ、T4 DNA连接酶购于NEB (北京)有限公司;His标记蛋白质微量纯化试剂盒购于天恩泽公司。
梯度PCR仪,德国艾本德公司;振荡培养箱,哈尔滨市东联电子技术开发有限公司;电泳仪,北京市六一仪器厂;智能人工气候箱,韶关市广智科技设备有限公司;Gel Doc XR凝胶成像系统,美国伯乐公司。
1.2 方法 1.2.1 几丁质酶基因克隆载体的构建通过对S. sampsonii KJ40全基因组序列进行测序及功能预测分析(基因组GenBank登录号为LORI00000000),获得一个编码几丁质酶的基因。利用软件Primer Premier 5.0设计引物,上游引物F1:5′-ATGCGTACCCGTCTGATCG-3′;下游引物R1:5′-TCAGCAGCTGAGGTTGTCG-3′。以基因组DNA为模板进行PCR扩增。25 μL PCR反应体系:ddH2O 10 μL,2×TransTaq High Fidelity (HiFi) PCR SuperMix Ⅰ 12.5 μL,DNA模板0.5 μL,上、下游引物(10 μmol/L)各1 μL。PCR反应条件:94 ℃ 3 min;94 ℃ 30 s,57.8 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 5 min;4 ℃保存。
纯化回收的PCR扩增目的片段与pMD19-T载体16 ℃连接,之后转化至E. coli DH5α感受态细胞中。通过LB/X-Gal/Amp平板对重组子进行筛选,阳性的菌液送往成都擎科梓熙公司进行DNA测序。构建好的克隆载体质粒pMD19-T-ChiKJ40-1用限制性内切酶Hind Ⅲ和BamHⅠ进行双酶切检测。
1.2.2 基因序列和蛋白质生物信息学预测分析DNAMAN软件推测ORF及氨基酸序列,利用NCBI数据库BLAST程序将得到的核苷酸序列和氨基酸序列进行比对;利用在线ExPasy系统中的ProtParam、Compute pI/Mw工具预测蛋白的理论分子量和等电点等;ProtScale预测蛋白质疏水性;TMHMM Server Version 2.0预测跨膜区域;在线软件SignalP 4.1 Server预测蛋白质信号肽;在线网站NPS@SOPMA预测蛋白的二级结构;在线软件SWISS-MODEL预测蛋白质三级结构。
1.2.3 几丁质酶基因表达载体的构建以重组质粒pMD19-T-ChiKJ40-1为模板,通过上游引物F2 (5′-CGCGGATCCGACACCCGCGC CGCCGCCG-3′)和下游引物R2 (5′-CCGCTCGAGT CAGCAGCTGAGGTTGTCG-3′)扩增含BamHⅠ和XhoⅠ酶切位点而不含信号肽的ChiKJ40基因序列,并连接到pMD19-T载体,构建pMD19-T-ChiKJ40-2质粒转化至E. coli DH5α感受态细胞中,得到阳性转化子。以pMD19-T-ChiKJ40-2质粒为模板,再次扩增几丁质酶基因,用限制性内切酶BamHⅠ和XhoⅠ双酶切目的片段和pET-32a(+),用T4 DNA连接酶连接,转化至E. coli DH5α感受态细胞中,构建好的表达载体用限制性内切酶BamHⅠ和XhoⅠ进行双酶切检测。
1.2.4 重组几丁质酶的诱导表达将构建好的表达载体质粒转化至BL21(DE3)感受态细胞中,挑取单菌落于10 mL的LB (100 mg/L Amp)培养基,37 ℃、200 r/min振荡培养过夜。将300 μL过夜培养液加入30 mL的LB (100 mg/L Amp)液体培养基中37 ℃、200 r/min振荡培养3 h,取3 mL作为不加IPTG的空白对照,剩余培养液分装于6只灭菌干燥的试管中,每只试管3 mL,分别加入IPTG (IPTG终浓度为0.2、0.4、0.6、0.8、1.0、1.2 mmol/L),以IPTG诱导空载体菌液和不加IPTG诱导的空载体作对照。
诱导3 h,分别取1 mL上述培养液,12 000 r/min离心1 min,弃去上清。取1 mL PBS重悬菌体,12 000 r/min离心1 min,弃去上清。60 μL PBS重悬菌体,加入20 μL 4×Protein SDS-PAGE loading buffer,沸水浴10 min使之变性,12 000 r/min离心10 min,取上清液进行SDS-PAGE电泳分析。
1.2.5 重组几丁质酶可溶性检测和纯化取1 mL 0.2 mmol/L IPTG 16 ℃诱导过夜的菌液,PBS洗涤重悬,液氮反复冻融菌液10次破细胞壁,12 000 r/min离心10 min收集上清液与沉淀,沉淀用60 μL 8 mol/L尿素重悬,4 ℃静置30 min,加4×Protein SDS-PAGE loading buffer,沸水浴10 min使之变性,12 000 r/min离心10 min取上清液进行SDS-PAGE电泳分析,观察上清与沉淀的表达产物含量,确定表达蛋白主要是以可溶性形式存在还是包涵体形式存在。使用天恩泽一站式His标记蛋白质微量纯化试剂盒对重组几丁质酶进行纯化。
1.2.6 重组几丁质酶的浓度与活性测定使用Bradford蛋白浓度测定试剂盒,测定粗酶液和纯化酶液的浓度。使用几丁质酶试剂盒测定粗酶液和纯化酶液的几丁质酶活性。
1.2.7 重组几丁质酶对几种致病真菌菌丝形态的影响C. scoparium、C. parasitica、A. alternata、R. violacea在PDA平板培养基中28 ℃培养7 d,直到整个平板覆盖满菌丝。用打孔器打取病原菌菌饼(d=5 mm),去除菌饼携带的固体培养基,将菌丝置于0.40 mg/mL纯化酶液中反应24 h,取1滴含病原菌菌丝的酶液于玻璃载玻片上,用显微镜对菌丝形态进行观察,以未用重组酶液处理的正常菌丝作对照。
2 结果与分析 2.1 几丁质酶基因克隆与鉴定几丁质酶基因PCR产物电泳结果见图 1的1号泳道,得到亮度较大、片段大小约726 bp的单一条带。使用限制性内切酶Hind Ⅲ和BamHⅠ双酶切检测pMD19-T-ChiKJ40-1载体质粒见图 1中的2号泳道,双酶切得到约760 bp的条带和约2 690 bp的条带,证明克隆载体构建成功,将此基因命名为ChiKJ40,提交NCBI数据库,登录号为MF434484。

|
| 图 1 DNA电泳检测 Figure 1 The electrophoresis results of the DNA 注:M1:DL2000 DNA marker;1:ChiKJ40基因PCR产物;M2:DL5000 DNA marker;2:载体pMD19-T-ChiKJ40-1双酶切产物. Note: M1: DL2000 DNA marker; 1: The PCR products of the ChiKJ40 gene; M2: DL5000 DNA marker; 2: The products of pMD19-T-ChiKJ40-1 vector by restriction enzyme digestion. |
|
|
通过NCBI网站BLAST比对,ChiKJ40基因核苷酸序列与Streptomyces albus SM254和Streptomyces sp. FR-008的一段编码几丁质酶726 bp大小的核苷酸序列一致性达99%。利用DNAMAN软件分析,ChiKJ40基因开放阅读框(Open reading frame,ORF)大小为726 bp,编码241个氨基酸,核苷酸序列及其编码的氨基酸见图 2。ChiKJ40基因编码氨基酸的保守结构域属糖苷水解酶19家族,与Streptomyces wadayamensis、Streptomyces sp. FR-008、Streptomyces sp. ScaeMP-6W和Streptomyces sp. BvitLS-983编码的几丁质酶基因的氨基酸序列一致性为100%。预测蛋白质的理论相对分子质量为25 677.28 Da,理论等电点为4.55,正电荷残基总数(Arg+Lys)为13,负电荷残基总数(Asp+Glu)为25,分子式为C1128H1711N311O364S7,脂肪系数为72.57,总平均亲水性为-0.261,是稳定蛋白,亲水性氨基酸总值较大,表明蛋白为亲水性蛋白。蛋白氨基酸组成见表 1,共由20种氨基酸组成,其中甘氨酸(Gly)和丙氨酸(Ala)所占比例最大,各占11.2%。预测蛋白存在一个跨膜区域,位于第5-27位氨基酸,第1-4位氨基酸在膜内部,第28-241位氨基酸在膜外部,存在信号肽,剪切位点位于第29-30位氨基酸,成熟肽始于第30位氨基酸。蛋白二级结构:α螺旋氨基酸78个,占32.37%;延伸链氨基酸47个,占19.50%;无规则卷曲氨基酸81个,占33.61%;β转角氨基酸有35个,占14.52%;整个结构以α螺旋和无规则卷曲为主。蛋白三级结构预测见图 3。

|
| 图 2 ChiKJ40基因核苷酸序列及其编码的氨基酸 Figure 2 The sequence of ChiKJ40 gene and its encoded amino acid sequence |
|
|
| 氨基酸名称 Amino acid |
数量 Number of amino acid |
所占百分比 Percentage (%) |
| Ala (A) | 27 | 11.2 |
| Arg (R) | 9 | 3.7 |
| Asn (N) | 17 | 7.1 |
| Asp (D) | 15 | 6.2 |
| Cys (C) | 4 | 1.7 |
| Gln (Q) | 10 | 4.1 |
| Glu (E) | 10 | 4.1 |
| Gly (G) | 27 | 11.2 |
| His (H) | 4 | 1.7 |
| Ile (I) | 9 | 3.7 |
| Leu (L) | 20 | 8.3 |
| Lys (K) | 4 | 1.7 |
| Met (M) | 3 | 1.2 |
| Phe (F) | 10 | 4.1 |
| Pro (P) | 11 | 4.6 |
| Ser (S) | 17 | 7.1 |
| Thr (T) | 18 | 7.5 |
| Trp (W) | 4 | 1.7 |
| Tyr (Y) | 10 | 4.1 |
| Val (V) | 12 | 5.0 |

|
| 图 3 蛋白三级结构预测 Figure 3 Prediction of protein tertiary structure |
|
|
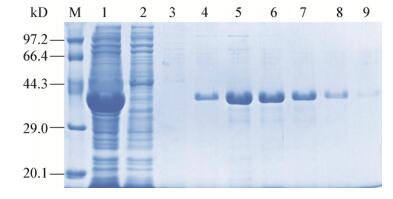
表达载体质粒用BamHⅠ和XhoⅠ双酶切检测见图 4,有约650 bp大小的条带,与ChiKJ40基因去除信号肽后的片段大小一致,证明重组表达载体构建成功。重组质粒转化至BL21(DE3)感受态细胞中进行诱导表达,SDS-PAGE凝胶鉴定结果见图 5。空载体经诱导表达约20.4 kD的标签蛋白,未诱导的空载体和重组质粒菌液表达蛋白中无突出条带,而经IPTG诱导后,pET-32a(+)- ChiKJ40菌液在约42 kD处出现明显条带,条带大小是去除信号肽后的目的蛋白与标签蛋白大小之和,0.2、0.4、0.6、0.8、1.0、1.2 mmol/L的IPTG诱导重组质粒菌液表达,重组蛋白的产量无显著变化。

|
| 图 4 表达载体质粒BamH I和Xho I双酶切结果 Figure 4 The identification of expression vector by BamH I and Xho I restriction enzymes digestion 注:M:DL5000 DNA marker;1:载体pET-32a(+)-ChiKJ40双酶切产物. Note: M: DL5000 DNA marker; 1: The products of pET-32a(+)-ChiKJ40 vector by restriction enzyme digestion. |
|
|

|
| 图 5 pET-32a(+)-ChiKJ40菌液IPTG诱导聚丙酰胺凝胶电泳分析 Figure 5 SDS-PAGE analysis of pET-32a(+)-ChiKJ40 induced by IPTG 注:M:蛋白质Marker;1:pET-32a(+)未诱导;2:pET-32a(+)诱导(IPTG浓度为1.0 mmol/L);3−9: 0、0.2、0.4、0.6、0.8、1.0、1.2 mmol/L IPTG诱导pET-32a(+)-ChiKJ40 (样品为菌液全菌细胞破碎液). Note: M: Protein marker; 1: pET-32a(+) not induced; 2: pET-32a(+) induced by IPTG (1.0 mmol/L); 3−9: pET-32a(+)-ChiKJ40 induced by IPTG, concentration of IPTG was 0, 0.2, 0.4, 0.6, 0.8, 1.0, 1.2 mmol/L (the samples were cell homogenates). |
|
|
由图 6可见,16 ℃诱导过夜菌液的重组蛋白主要以可溶性形式存在于上清,小部分以包涵体存在于沉淀中。由图 7可发现,50、100、150、200、250和300 mmol/L浓度的咪唑溶液均可以洗脱重组蛋白,其中100 mmol/L浓度的咪唑溶液洗脱效果最好。

|
| 图 6 重组蛋白可溶性检测 Figure 6 The soluble detection of recombinant proteins 注:M:蛋白质Marker;1、2:空载体细胞裂解物上清;3:空载体细胞裂解物沉淀;4、5:重组载体细胞裂解物上清;6:重组载体细胞裂解物沉淀. Note: M: Protein marker; 1, 2: Supernatant of cell lysate from pET-32a(+); 3: Sediment of cell lysate from pET-32a(+); 4, 5: Supernatant of cell lysate from recombinant vector; 6: Sediment of cell lysate from recombinant vector. |
|
|
|
| 图 7 重组蛋白纯化结果 Figure 7 Purification of recombinant protein 注:M:蛋白质Marker;1:重组载体细胞裂解物上清;2−9:0、10、50、100、150、200、250和300 mmol/L咪唑洗脱液. Note: M: Protein marker; 1: Supernatant of cell lysate from recombinant vector; 2−9: Liquid were eluted with 0, 10, 50, 100, 150, 200, 250 and 300 mmol/L imidazole. |
|
|
使用Bradford蛋白浓度测定试剂盒测定粗酶液和纯化酶液的浓度,几丁质酶试剂盒测定粗酶液和纯化酶液的几丁质酶活性,结果见表 2。纯化酶液酶比活力为0.115 U/mg,纯化倍数为2.8,酶活回收率为57.5%。
| 项目 Items |
粗酶液 Crude enzyme solution |
Ni-Agarose柱 Column of Ni-Agarose |
| 蛋白浓度 Protein concentration (g/L) |
1.94 | 0.40 |
| 几丁质酶活性 Chitinase activity (U/mL) |
0.080 | 0.046 |
| 比活力 Specific activity (U/mg) |
0.041 | 0.115 |
| 纯化倍数 Purification fold |
1.00 | 2.80 |
| 酶活回收率 Recovery rate of enzyme activity (%) |
100.0 | 57.5 |
纯化后的酶液浸泡处理病原菌菌丝后,C. scoparium、C. parasitica、A. alternata、R. violacea菌丝形态与对照相比发生了不同程度的变化,见图 8。重组几丁质酶处理后,C. scoparium、C. parasitica和A. alternata菌丝细胞出现分节、膨胀,R. violacea菌丝溶解且部分被破坏成碎片。

|
| 图 8 病原真菌菌丝形态 Figure 8 Hyphae morphology of pathogenic fungal 注:A−D:C. scoparium、C. parasitica、A. alternata、R. violacea正常菌丝形态(400×);a−d:重组几丁质酶处理后C. scoparium、C. parasitica、A. alternata、R. violacea菌丝形态(400×). Note: A−D: The normal morphology of mycelia from C. scoparium, C. parasitica, A. alternate, R. violacea (400×); a−d: The morphology of mycelia from C. scoparium, C. parasitica, A. alternate, R. violacea treated by the recombinant chitinase (400×). |
|
|
几丁质酶多作为生物杀菌或杀虫剂应用在农林业上,以提高植物的抗病能力。产几丁质酶微生物能够分解植物病原真菌细胞壁的重要成分几丁质,依据此特性,传统方式是直接利用几丁质酶产生菌防治植物真菌病害。孙辉等[13]研究发现毛壳菌(Chaetomium sp. ymf1. 00843)所产胞外几丁质酶有效抑制立枯丝核菌(Rhizoctonia solani)和烟草赤星病菌(Alternaria alternata)菌丝的生长。苏明慧等[14]从短短芽孢杆菌FM4B发酵液分离到几丁质酶,发现酶液对西瓜枯萎病菌(Fusarium oxysporum)、棘孢青霉(Penicillium aculeatum)、哈茨木霉(Trichoderma harzianum)、根霉(Rhizopus)和灰霉(Botrytis cinerea)有明显的抑菌作用,且随着酶液质量浓度的增加抑菌效果增大。几丁质酶虽然在生物防治中具有很大的应用潜力,但分离出的几丁质酶活性不高。为了几丁质酶的商业化生产,研究者们不断尝试分离高产菌株或通过改变发酵条件来提高其产量[15]。由于其调控机理较为复杂以及发酵分离、提取工艺的不成熟,能够成功应用于工业化生产的几丁质酶并不多见。分子生物学技术的发展为解决这一难题提供了一种新的思路:将几丁质酶基因克隆并转入适当载体进行高效表达,以满足大规模生产和研究应用的需要。
S. sampsonii的ChiKJ40基因编码氨基酸的保守结构域属糖苷水解酶19家族,链霉菌属几丁质酶多属于19家族[7]。19家族几丁质酶起初在高等植物中被发现,在微生物中只发现了18家族几丁质酶。Ohno等[16]第一次从高等植物之外的Streptomyces griseus HUT6037发现编码19家族几丁质酶基因,之后19家族几丁质酶陆续在其他放线菌中被发现[17],19家族几丁质酶三级结构类似于溶菌酶。不同生物来源的几丁质酶分子质量差异很大,一般是20-90 kD。细菌几丁质酶多为20-60 kD,与植物几丁质酶(25-40 kD)大小接近,比昆虫几丁质酶(40-85 kD)小[18]。本研究在E. coli中表达的重组ChiKJ40基因分子量约为25.6 kD,但是在SDS-PAGE蛋白质条带分析中,发现表达蛋白条带大小接近42 kD,该分子量大小和实际的ChiKJ40基因分子量大小看起来有一定的差距。这主要是因为在pET-32a(+)载体自身表达的蛋白质总分子量大小为20.4 kD,其中包含大小为12 kD的Trx-tag,2个大小为0.8 kD的His-tag和大小为1.7 kD的S-tag,剩下的5.1 kD来源于介于这些标签和终止密码子之间的54个氨基酸。
E. coli表达系统的优点在于遗传背景研究深入清楚、繁殖快、成本低、表达量高以及有大量可利用的表达载体、宿主和纯化系统等特点,成为目前应用最广的表达体系[19]。外源蛋白往往在获得高水平表达的同时,容易被宿主蛋白酶降解或者形成包涵体,目前国内外对蛋白质体外复性研究较多,但其过程往往费时、费力且不经济,因此,探索外源蛋白在E. coli中的可溶性表达具有广泛的应用前景[20]。通过降低培养温度来降低蛋白合成速度,降低有聚合倾向的中间体的浓度,避免形成包涵体[21]。ChiKJ40基因通过pET-32a载体高效表达,16 ℃诱导过夜,重组蛋白主要以可溶性形式存在于上清,易于纯化。pET表达质粒含有一段组氨酸标签的基因序列,便于表达蛋白的鉴定和纯化。本研究通过不同浓度咪唑溶液洗脱纯化,100 mmol/L浓度的咪唑溶液洗脱效果最佳,纯化酶液酶比活力为0.115 U/mg,纯化倍数为2.8,酶活回收率为57.5%。García-Fraga等[22]克隆表达海洋来源的被囊假交替单胞菌(Pseudoalteromonas tunicata) 19家族的几丁质酶Ptchi19p,重组蛋白Ptchi19p粗酶液活性为0.257 U,纯化酶液活性为0.228 U,几丁质酶Ptchi19p对F. oxysporum和黑曲霉(Aspergillus niger)有明显的抑菌作用。Hjort等[23]克隆表达苏云金芽孢杆菌BUPM255的几丁质酶Chi18H8,纯化后的酶液能够抑制胶孢炭疽菌(Colletotrichum gloeosporioides)、禾谷镰刀菌(Fusarium graminearum)、F. oxysporum和A. alternata的菌丝生长。
几丁质酶能够降解真菌细胞壁的主要组成成分几丁质[24],目前,植物酶类中发现多种有抑菌活性的19家族几丁质酶,而在细菌酶类中发现较少[25-28]。国内外研究人员[8, 11, 29-30]发现S. sampsonii对A. alternata、F. oxysporum等多种病原真菌菌丝生长有抑制作用。本研究关于S. sampsonii几丁质酶基因原核表达产物的抑菌活性属首次报道,重组几丁质酶对多种病原菌的细胞结构具有破坏作用,推测S. sampsonii KJ40产几丁质酶是其具有真菌拮抗能力的重要原因之一。目前,关于S. sampsonii生防基因的研究不多,雷美艳[12]仅从S. sampsonii基因组中扩增出2个几丁质酶基因片段,未能克隆到一个完整的几丁质酶基因。ChiKJ40 ORF大小为726 bp,编码241个氨基酸,含编码一个几丁质酶的完整序列,因此本研究对S. sampsonii几丁质酶基因ChiKJ40的研究,完善和补充了S. sampsonii的生防背景,为几丁质酶基因找到了新的来源,并为其应用奠定了理论基础。
| [1] |
Lin YW, Xu C, Lu CH. Medical application of chitin and its derivatives[J]. Journal of Fujian Medical University, 1999, 33(2): 226-228. 林友文, 许晨, 卢灿辉. 甲壳素及其衍生物的医学应用[J]. 福建医科大学学报, 1999, 33(2): 226-228. |
| [2] |
Kurita K. Controlled functionalization of the polysaccharide chitin[J]. Progress in Polymer Science, 2001, 26(9): 1921-1971. DOI:10.1016/S0079-6700(01)00007-7 |
| [3] |
Yuli PE, Suhartono MT, Rukayadi Y, et al. Characteristics of thermostable chitinase enzymes from the indonesian Bacillus sp. 13.26[J]. Enzyme and Microbial Technology, 2004, 35(2/3): 147-153. |
| [4] |
Mathivanan N, Kabilan V, Murugesan K. Purification, characterization, and antifungal activity of chitinase from Fusarium chlamydosporum, a mycoparasite to groundnut rust, Puccinia arachidis[J]. Canadian Journal of Microbiology, 1998, 44(7): 646-651. DOI:10.1139/w98-043 |
| [5] |
Henrissat B, Bairoch A. New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J]. Biochemical Journal, 1993, 293(3): 781-788. DOI:10.1042/bj2930781 |
| [6] |
Arimori T, Kawamoto N, Shinya S, et al. Crystal structures of the catalytic domain of a novel glycohydrolase family 23 chitinase from Ralstonia sp. A-471 reveals a unique arrangement of the catalytic residues for inverting chitin hydrolysis[J]. Journal of Biological Chemistry, 2013, 288(26): 18696-18706. DOI:10.1074/jbc.M113.462135 |
| [7] |
Hamid R, Khan MA, Ahmad M, et al. Chitinases: an update[J]. Journal of Pharmacy & BioAllied Sciences, 2013, 5(1): 21-29. |
| [8] |
Malviya MK, Pandey A, Trivedi P, et al. Chitinolytic activity of cold tolerant antagonistic species of Streptomyces isolated from glacial sites of Indian Himalaya[J]. Current Microbiology, 2009, 59(5): 502-508. DOI:10.1007/s00284-009-9466-z |
| [9] |
Kim SS, Kang SI, Kim JS, et al. Biological control of root-knot nematode by Streptomyces sampsonii KK1024[J]. Korean Journal of Soil Science and Fertilizer, 2011, 44(6): 1150-1157. DOI:10.7745/KJSSF.2011.44.6.1150 |
| [10] |
Augustine D. Actinomycete isolates from Arabian Sea and Bay of Bengal: biochemical, molecular and functional characterization[D]. Cochin: Doctoral Dissertation of Cochin University of Science and Technology, 2014
|
| [11] |
Li SJ, Zhu TH, Peng Y, et al. Characteristics of chitinase-produced by Streptomycete sampsonii with antimicrobial activity and its biocontrol to Rhizoctonia violacea[J]. Journal of Northeast Forestry University, 2014, 42(3): 116-121. 李姝江, 朱天辉, 彭艳, 等. 桑氏链霉菌产几丁质酶特性及对杨树紫纹病的生防作用[J]. 东北林业大学学报, 2014, 42(3): 116-121. |
| [12] |
Lei MY. The features of chitinase and gene cloning of Chitinase gene in Streptomyces sampsonii KJ42[D]. Yaʼan: Master's Thesis of Sichuan Agricultural University, 2012 (in Chinese) 雷美艳. Streptomyces sampsonii KJ42几丁质酶特性及其基因克隆[D]. 雅安: 四川农业大学硕士学位论文, 2012 |
| [13] |
Sun H, Yang JK, Zhang KQ. Enzymatic properties and antibacterial activity of chitinase from Chaetomium sp. YMF1.00843[J]. Journal of Northwest A & F University (Natural Science Edition), 2015, 43(5): 168-173. 孙辉, 杨金奎, 张克勤. 毛壳菌几丁质酶的酶学性质及抑菌活性研究[J]. 西北农林科技大学学报:自然科学版, 2015, 43(5): 168-173. |
| [14] |
Su MH, Hu XQ, Gu DH, et al. Purification and characterization of chitinase from the fermentation broth of Brevibacillus brevis FM4B[J]. Food Science, 2015, 36(19): 176-179. 苏明慧, 胡雪芹, 顾东华, 等. 短短芽孢杆菌几丁质酶的分离纯化及酶学性质[J]. 食品科学, 2015, 36(19): 176-179. DOI:10.7506/spkx1002-6630-201519031 |
| [15] |
Xie CC, Jia HY, Chen YH. Regulation of chitinase genes expression in bacteria[J]. Hereditas (Beijing), 2011, 33(10): 1029-1038. 谢池楚, 贾海云, 陈月华. 细菌几丁质酶基因的表达调控[J]. 遗传, 2011, 33(10): 1029-1038. |
| [16] |
Ohno T, Armand S, Hata T, et al. A modular family 19 chitinase found in the prokaryotic organism Streptomyces griseus HUT 6037[J]. Journal of Bacteriology, 1996, 178(17): 5065-5070. DOI:10.1128/jb.178.17.5065-5070.1996 |
| [17] |
Kawase T, Saito A, Sato T, et al. Distribution and phylogenetic analysis of family 19 chitinases in Actinobacteria[J]. Applied and Environmental Microbiology, 2004, 70(2): 1135-1144. DOI:10.1128/AEM.70.2.1135-1144.2004 |
| [18] |
Bhattacharya D, Nagpure A, Gupta RK. Bacterial chitinases: properties and potential[J]. Critical Reviews in Biotechnology, 2007, 27(1): 21-28. DOI:10.1080/07388550601168223 |
| [19] |
Terpe K. Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems[J]. Applied Microbiology and Biotechnology, 2006, 72(2): 211-222. DOI:10.1007/s00253-006-0465-8 |
| [20] |
Zhu HY, Li Q. Strategies for expression of soluble heterologous proteins in Escherichia coli[J]. The Chinese Journal of Process Engineering, 2006, 6(1): 150-155. 朱红裕, 李强. 外源蛋白在大肠杆菌中的可溶性表达策略[J]. 过程工程学报, 2006, 6(1): 150-155. |
| [21] |
Urban A, Ansmant I, Motorin Y. Optimisation of expression and purification of the recombinant Yol066 (Rib2) protein from Saccharomyces cerevisiae[J]. Journal of Chromatography B, 2003, 786(1/2): 187-195. |
| [22] |
García-Fraga B, da Silva AF, López-Seijas J, et al. A novel family 19 chitinase from the marine-derived Pseudoalteromonas tunicata CCUG 44952T: heterologous expression, characterization and antifungal activity[J]. Biochemical Engineering Journal, 2015, 93: 84-93. DOI:10.1016/j.bej.2014.09.014 |
| [23] |
Hjort K, Presti I, Elv ng A, et al. Bacterial chitinase with phytopathogen control capacity from suppressive soil revealed by functional metagenomics[J]. Applied Microbiology and Biotechnology, 2014, 98(6): 2819-2828. DOI:10.1007/s00253-013-5287-x |
| [24] |
Adrangi S, Faramarzi MA. From bacteria to human: a journey into the world of chitinases[J]. Biotechnology Advances, 2013, 31(8): 1786-1795. DOI:10.1016/j.biotechadv.2013.09.012 |
| [25] |
Huang LX, Garbulewska E, Sato K, et al. Isolation of genes coding for chitin-degrading enzymes in the novel chitinolytic bacterium, Chitiniphilus shinanonensis, and characterization of a gene coding for a family 19 chitinase[J]. Journal of Bioscience and Bioengineering, 2012, 113(3): 293-299. DOI:10.1016/j.jbiosc.2011.10.018 |
| [26] |
Kawase T, Yokokawa S, Saito A, et al. Comparison of enzymatic and antifungal properties between family 18 and 19 chitinases from S. coelicolor A3(2)[J]. Bioscience, Biotechnology, and Biochemistry, 2006, 70(4): 988-998. DOI:10.1271/bbb.70.988 |
| [27] |
Kaomek M, Mizuno K, Fujimura T, et al. Cloning, expression, and characterization of an antifungal chitinase from Leucaena leucocephala de Wit[J]. Bioscience, Biotechnology, and Biochemistry, 2003, 67(4): 667-676. DOI:10.1271/bbb.67.667 |
| [28] |
Wong JH, Ng TB, Cheung RCF, et al. Proteins with antifungal properties and other medicinal applications from plants and mushrooms[J]. Applied Microbiology and Biotechnology, 2010, 87(4): 1221-1235. DOI:10.1007/s00253-010-2690-4 |
| [29] |
Peng Y, Zhu TH, Zhang BY, et al. Isolation, purification and partial characterization of an antifungal protein from Streptomyces sampsonii KJ07[J]. Microbiology China, 2016, 43(9): 1980-1987. 彭艳, 朱天辉, 张博阳, 等. 桑氏链霉菌KJ07抗菌蛋白的分离纯化及部分特性[J]. 微生物学通报, 2016, 43(9): 1980-1987. |
| [30] |
Jain PK, Jain PC. Isolation, characterization and antifungal activity of Streptomyces sampsonii GS 1322[J]. Indian Journal of Experimental Biology, 2007, 45(2): 203-206. |
 2018, Vol. 45
2018, Vol. 45




