扩展功能
文章信息
- 杨秀清, 陈彦梅, 魏国琴, 陈林勇
- YANG Xiu-Qing, CHEN Yan-Mei, WEI Guo-Qin, CHEN Lin-Yong
- 寺河矿煤地质产甲烷微生物菌群的保藏和产甲烷性能
- Preservation and methane production of methanogenic microbial consortia from coal geological environment in Sihe Mine
- 微生物学通报, 2018, 45(5): 951-961
- Microbiology China, 2018, 45(5): 951-961
- DOI: 10.13344/j.microbiol.china.171053
-
文章历史
- 收稿日期: 2017-12-14
- 接受日期: 2018-02-27
- 网络首发日期(www.cnki.net): 2018-03-23
2. 煤与煤层气共采国家重点实验室 山西 晋城 048000;
3. 易安蓝焰煤与煤层气共采技术有限责任公司 山西 晋城 048000
2. State Key Laboratory of Coal and CBM Co-mining, Jincheng, Shanxi 048000, China;
3. Yi'an Lanyan Coal and Coal-bed Methane Simultaneous Extraction Technology Co. Ltd., Jincheng, Shanxi 048000, China
煤层气(Coalbed methane,CBM)是一种在煤层中自生自储存在的非常规天然气,是天然气的重要补充来源。目前,煤层气的开发及利用受到了世界各国的重视,我国也大力提倡开发及利用煤层气。近年来,我国井下煤层气抽采利用活跃,煤层气抽采量逐年增大[1],造成了部分煤层气井产量持续走低或接近衰退期的现状,如何实现煤层气的再生与增产已成为当今煤层气开发的重要研究内容。
根据成因来分,煤层气可以分为热成因和生物成因煤层气[2-3]。其中,生物成因煤层气是在混合微生物菌群的作用下通过降解煤来产生。据报道,世界上大约有20%的天然气属于微生物成气[4]。然而到目前为止,微生物是如何在厌氧条件下降解煤,有哪些功能微生物参与其中还并不是很清楚。近年来,国内外有许多研究人员在实验室条件下通过利用微生物作用于煤成功地模拟了煤层气的生成[5-8],这对于实现煤层气的大规模生产及探究生物成气的机理都具有重要的指导作用。产甲烷菌群作为实验室生物模拟产气的接种菌源,其菌群的稳定性和产甲烷功能性对产气而言非常重要。因此,在对产甲烷菌群的研究和应用中,探索产甲烷菌群的保藏条件就显得非常必要。目前,有关微生物菌种的保藏多为单一菌种的保藏。其保藏方法主要有:甘油冷冻保藏法、液体石蜡保藏法、真空冷冻干燥保藏法、冷冻保藏法及载体保藏法等。生物煤层气是由多种厌氧混合菌共同作用代谢煤基质产生,有关煤层气产甲烷菌群的保藏涉及多种厌氧混合菌种的保藏。在保藏过程中,保持菌群结构的稳定,避免关键菌种的丢失,选择合适的环境防止保藏菌种活性的过快衰减,这些对于应用其进行产甲烷的生产及研究尤为重要。而截至目前,关于该混合产甲烷菌群的大量长期保藏还鲜有报道。产甲烷菌群一般可以分为外源产甲烷菌群和煤层本源产甲烷菌群。虽然外源产甲烷菌群容易获得,但存在与煤层环境适应的问题,且难以了解煤层中产甲烷菌群的真实结构特征,容易造成对煤层生物成气机理的模糊认识。而原沉积煤层中的本源产甲烷菌群对煤环境的适应性更强,能够在模拟产气实验中保持活力并迅速繁殖,有利于煤的生物气化及微生物成气机理的研究。
本文以从山西寺河矿煤层气井收集到的含产甲烷菌群的产出水作为待保藏出发菌源,通过产甲烷菌群的2次富集驯化培养后,再借鉴以上提到的多种菌种保藏方法,设计了3种驯化后产甲烷菌群的保藏方案,并分别于设定的温度条件下进行了菌种的密封厌氧保藏。经过基于高通量测序监测菌种保藏过程中菌群结构的特征及其动态变化和以各保藏试验保藏的菌种进行生物气模拟实验,以验证保藏方法的可行性,从而为大量高效地保藏煤层产甲烷菌群提供依据。
1 材料与方法 1.1 样品采集 1.1.1 煤样实验用无烟煤采自山西沁水盆地寺河煤矿,采样后大块无烟煤经粉碎机粉粹至5-10 cm3小煤块,于空气中堆放半年以上,用作产甲烷菌群基础盐保藏的基质。小块无烟煤进一步粉碎至粉状,用于产甲烷菌群富集驯化培养及产气实验。
实验用褐煤采自云南昭通煤矿,采样后褐煤经粉碎机粉碎至粉状,用于产气实验。
1.1.2 产甲烷菌群样品实验保藏用产甲烷菌群的出发菌源为山西寺河矿区121煤层气井中的产出水样,该水样含有煤层本源产甲烷菌群。产出水在煤层气产气井排水口进行收集,收集好的水样置于冰上,快速运回实验室备用或置于4 ℃短时间保存。
1.2 主要试剂和仪器EasyTaq polymerase,北京全式金生物技术有限公司;DNA胶回收试剂盒、甘油、L-半胱氨酸、Proteinase K,生工生物工程(上海)股份有限公司;SDS (十二烷基磺酸钠),北京索莱宝科技有限公司;琼脂糖凝胶DNA回收试剂盒,中科瑞泰(北京)生物科技有限公司。
YQX-П型厌氧手套箱,上海跃进医疗器械厂;PCR仪,美国Bio-Rad公司;气相色谱仪,日本岛津公司。
1.3 产甲烷菌群保藏 1.3.1 营养液的制备基础盐液配方(g/L):酵母粉2.0,KH2PO4 1.5,K2HPO4 2.9,NH4Cl 1.8,MgCl2 0.4。
LB培养液配方(g/L):胰蛋白胨10.0,氯化钠5.0,酵母提取物5.0。
1.3.2 产甲烷菌群的富集驯化培养在厌氧手套箱中,将煤层产出水样加入到500 mL厌氧瓶中,加入等体积的灭菌基础盐液,再加入终浓度为0.04%的除氧L-半胱氨酸及适量无烟煤粉,轻轻混匀,迅速密封厌氧瓶,并于25 ℃条件下静置培养。期间每周用无菌注射器从厌氧瓶瓶顶插入进行气体的自动收集,收集到的气体取0.5 mL进行气相色谱分析,并计算甲烷生成的相对含量。待产气稳定后,取100 mL的富集培养液加入到新的无菌厌氧瓶中,同样再加入等体积的灭菌基础盐液、除氧L-半胱氨酸及适量无烟煤粉,密封厌氧瓶进行菌群的2次富集驯化培养。在2次富集驯化培养期间,用上述同样方法进行气体的收集及甲烷气含量的测定。其中,产甲烷菌群2次富集驯化后的培养液拟作为以下菌种保藏试验中的待保藏菌源。
1.3.3 菌种保藏试验(1) 甘油/L-半胱氨酸保藏
在厌氧手套箱中,将1.3.2部分驯化后的产甲烷菌群培养液加入到500 mL的厌氧瓶中,然后再加入等体积的灭菌甘油和除氧L-半胱氨酸溶液。其中,甘油的最终质量分数为15%,L-半胱氨酸的终浓度为0.04%。将瓶中液体混匀后分别置于-80、4、25 ℃条件下密封静置保存。
(2) 富营养保藏
在厌氧手套箱中,将1.3.2部分驯化后的产甲烷菌群培养液加入到500 mL的厌氧瓶中,然后再加入等体积的灭菌LB培养液,再加入终浓度为0.04%的除氧L-半胱氨酸混匀,密封厌氧瓶分别置于-80、4、25 ℃条件下静置保存。
(3) 煤基-基础盐保藏
于自制发酵罐中加入1/4体积的1.3.2部分驯化后的产甲烷菌群培养液,然后加入等体积的灭菌基础盐液,再加入终浓度为0.04%的除氧L-半胱氨酸及60 g/L的小块无烟煤,从罐底通入高纯氮气30 min以维持罐内厌氧环境后,密封各自制发酵罐,分别静置于-80、4、25 ℃条件下保存。在保存的过程中,每隔半年时间从发酵罐内取出一半体积的培养液,加入等体积的灭菌基础盐液(含终浓度为0.04%的除氧L-半胱氨酸),接着密封保藏。
1.4 产气实验向1 L厌氧瓶中加入250 g煤粉(褐煤或无烟煤)、350 mL基础盐液及0.408 mg刃天青,1×105 Pa灭菌30 min。在厌氧手套箱内向1 L厌氧瓶中加入800 μL无菌的20% L-半胱氨酸,然后向厌氧瓶中持续通入高纯氮气,至培养基颜色近无色,密封厌氧瓶待用。
在厌氧手套箱中,在上述1 L厌氧瓶中(含无烟煤)接种各保藏试验在菌种保藏35 d后的保藏液进行产气实验,菌种的接种量为50 mL。密封各产气实验用厌氧瓶,于厌氧箱中静置培养1-3个月,期间每周从厌氧瓶瓶顶插入无菌注射器进行气体的自动收集,收集到的气体取0.5 mL进行气相色谱分析,并计算甲烷生成的相对含量。
在厌氧手套箱中,在上述1 L厌氧瓶中(含无烟煤)分别接种25 ℃条件下基础盐保藏试验在菌种保藏42、70、105、209、369、459 d后的保藏液,接种量为50 mL。密封厌氧瓶,于厌氧箱中静置培养1-3个月,期间每周从厌氧瓶瓶顶插入无菌注射器进行气体的自动收集,收集到的气体取0.5 mL进行气相色谱分析,并计算甲烷生成的相对含量。
在厌氧手套箱中,在上述1 L厌氧瓶中(含褐煤)分别接种25 ℃条件下基础盐保藏试验在菌种保藏687、710、742、758、771、804、839 d后的保藏液,接种量为50 mL。密封厌氧瓶,于厌氧箱中静置培养1-3个月,期间每周从厌氧瓶瓶顶插入无菌注射器进行气体的自动收集,收集到的气体取0.5 mL进行气相色谱分析,并计算甲烷生成的相对含量。
1.5 煤基-基础盐保藏过程中微生物菌群结构的动态变化 1.5.1 微生物总基因组DNA的提取25 ℃条件下产甲烷菌群煤基-基础盐保藏过程中分别于35、42、70及105 d进行样品采集。将采集的各样品于12 000 r/min条件下离心1 min,收集菌体细胞用于微生物总基因组DNA的提取。基因组DNA的提取参照杨秀清等[9]的报道。简言之,收集的菌体细胞用小型珠磨式研磨器破碎,经SDS与Proteinase K消化、酚-氯仿抽提、异丙醇沉淀、75%乙醇洗涤沉淀后,加入灭菌双蒸水溶解沉淀即得微生物总基因组DNA。0.7%的琼脂糖凝胶电泳检测提取基因组DNA的长度及完整性。
1.5.2 16S rRNA基因片段的扩增用提取的基因组DNA作模板,进行细菌16S rRNA基因V1-V3区及古菌16S rRNA基因V3-V4区片段的扩增。细菌16S rRNA基因V1-V3区PCR扩增引物采用27F (5′-AGAGTTTGATCCTGGCTCA G-3′)[10]和518R (5′-ATTACCGCGGCTGCTGG-3′)[11]。PCR反应体系为:10×EasyTaq buffer (含Mg2+) 5 μL,dNTPs (2.5 mmol/L) 4 μL,上、下游引物(10 μmol/L)各2 μL,EasyTaq polymerase (5 U/μL) 1 μL,基因组DNA 0.5 μL,加无菌双蒸水至50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,55 ℃ 45 s,72 ℃ 45 s,35个循环;72 ℃ 10 min。古菌16S rRNA基因V3-V4区PCR扩增引物采用344F (5′-ACGGGGY GCAGCAGGCGCGA-3′)[12]和0691R (5′-GGATTAC ARGATTTCAC-3′)[13]。PCR反应体系同细菌16S rRNA基因V1-V3区。PCR反应条件:95 ℃ 5 min;95 ℃ 45 s,55 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min。细菌和古菌的PCR反应产物均用1.5%琼脂糖凝胶电泳检测,以验证PCR扩增片段长度的正确性,随后用胶回收试剂盒对目的产物进行纯化,具体步骤参照说明书。最后将纯化后的产物进行Roche 454 FLX高通量测序。高通量测序的原始数据提交至NCBI (National center for biotechnology information)的SRA (Short read archive)数据库中。
1.5.3 高通量测序数据处理样品测序完成后,用开头的标签序列进行区别,每条序列读长(Read)均进行质量检测。使用Mothur软件去除接头序列、引物二聚体和低复杂性序列[14];在去除预处理的靶序列后,使用Mothur的聚类软件校正序列的错误;最后使用Chimeras. uchime去除嵌合体序列。通过使用Uclust将类似序列聚类为操作分类单位(Operational taxonomic unit,OTU,97%相似性),处理后的序列通过软件RDP分类器进行物种分类。同时基于OTU数量的结果,评估样品的α多样性,指标包含Chao1指数、辛普森指数(Simpson index)和香农指数(Shannon index),并且通过稀释曲线分析来确定每个样品中序列的相对丰度。
2 结果与分析 2.1 产甲烷菌群的富集驯化及产气在进行产甲烷菌群的保藏试验之前,首先对原始煤层产出水样中的产甲烷菌群以无烟煤为碳源进行了菌群的2次富集驯化培养。在第2次富集驯化培养期间以收集到的气体体积量对培养时间进行绘图分析,其产气曲线如图 1所示。在接种后34 d产甲烷菌群处于潜伏期,无气体产生;40-56 d,产甲烷菌群进入了快速产气阶段,产气总量达1.056 L;其中甲烷气含量为624.89 mL;随后,在经历一小段产气停止期后,在培养的第69天,产甲烷菌群进入了2次产气阶段,此时甲烷气的含量最高达到了50.70%,随即停止富集驯化培养,厌氧瓶中的培养液立即用作产甲烷菌种保藏试验中的保藏菌种。
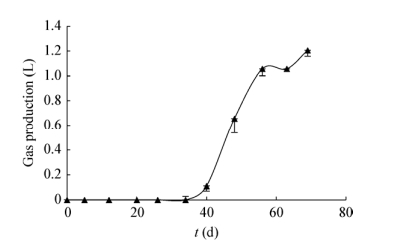
|
| 图 1 煤层产出水样2次富集产气曲线 Figure 1 Gas production curve for the second enrichment of coalbed formation water |
|
|
煤层产出水样经2次富集驯化培养后得到的含产甲烷菌群的培养液进行了3种方案的菌种保藏试验。其中,为了检测各菌种保藏试验中产甲烷菌群的活力,在各菌种保藏试验的第35天,分别以各保藏液为接种剂、以无烟煤为碳源进行了产气实验。在产气培养的3个月内,间断进行甲烷气含量的测定,以最高产甲烷含量为代表进行分析。各组产气实验结果如表 1所示。结果表明,4 ℃及25 ℃条件下进行的甘油/L-半胱氨酸保藏试验保藏的产甲烷菌群活性较低,在产气实验中有微量的甲烷气生成。-80 ℃条件下甘油/L-半胱氨酸保藏的菌种不利于后续的产气实验,几乎没有甲烷气生成。25 ℃条件下进行的富营养保藏试验,其保藏的菌种在产气实验中没有甲烷生成,但生成氢气,而-80 ℃及4 ℃条件下的富营养保藏菌种在产气实验中没有气体产生。煤基-基础盐菌种保藏试验中,25 ℃条件下保藏的菌种在产气实验中产生较高含量的甲烷气,-80 ℃及4 ℃条件下的菌种在产气实验中要么不产气,要么只产氢气。综合以上分析结果,25 ℃条件下的煤基-基础盐菌种保藏方法相对而言是较为适合厌氧产甲烷菌群的一种菌种保藏方法。
| 保藏方案 Preservation method |
保藏温度 Preservation temperature (℃) |
甲烷含量 Methane content (%) |
氢气含量 Hydrogen content (%) |
| 甘油/L-半胱氨酸保藏 Glycerol/L-cysteine preservation |
-80 | 0.00 | 0.00 |
| 4 | 11.00 | 0.00 | |
| 25 | 9.81 | 0.00 | |
| 富营养保藏 Eutrophication preservation |
-80 | 0.00 | 0.00 |
| 4 | 0.00 | 0.00 | |
| 25 | 0.00 | 5.46 | |
| 煤基-基础盐保藏 Coal-basic salt preservation |
-80 | 0.00 | 0.00 |
| 4 | 0.00 | 12.31 | |
| 25 | 35.25 | 3.23 |
以25 ℃条件下煤基-基础盐保存的第42、70、105、209、369和459天的产甲烷菌群分别为接种剂进行产气实验。各实验产甲烷结果如图 2所示。结果表明,以无烟煤为碳源进行产气培养,各实验的产甲烷量各不相同,在12%-25%之间。

|
| 图 2 无烟煤培养条件下的产气结果 Figure 2 The result of gas production from cultivate with anthracite 注: 1-6:以25 ℃条件下煤基-基础盐保存的第42、70、105、209、369和459天产甲烷菌群进行的产气实验. Note: 1-6: Gas production experiments inoculated with methanogenic microbial consortia from coal and basic salt preservation test under 25 ℃ at 42, 70, 105, 209, 369, and 459 days, respectively. |
|
|
以25 ℃条件下煤基-基础盐保存的第687、710、742、758、771、804和839天的产甲烷菌群分别为接种剂进行产气实验。各实验产甲烷结果如图 3所示。结果显示,以褐煤为碳源进行产气培养,各实验中甲烷的生成量普遍较高,介于24%-73%。综合以上结果,表明25 ℃条件下煤基-基础盐保藏的产甲烷菌群在3年内都具有较高的产甲烷活性,进而也证明了25 ℃条件下煤基-基础盐保藏煤层产甲烷菌群的可行性及可靠性。

|
| 图 3 褐煤培养条件下的产气结果 Figure 3 The result of gas production from cultivate with lignite 注: 1-7:以25 ℃条件下煤基-基础盐保存的第687、710、742、758、771、804和839天产甲烷菌群进行的产气实验. Note: 1-7: Gas production experiments inoculated with methanogenic microbial consortia from coal and basic salt preservation test under 25 ℃ at 687, 710, 742, 758, 771, 804, and 839 days, respectively. |
|
|
提取25 ℃条件下煤基-基础盐产甲烷菌群保藏过程中各时期菌群的基因组DNA,经琼脂糖凝胶电泳检测,提取的基因组DNA主带均较完整,且条带大小均在23 kb左右,满足后续的实验要求。用细菌16S rRNA基因V1-V3区通用引物及古菌16S rRNA基因V3-V4区通用引物分别进行PCR扩增,扩增产物经琼脂糖凝胶DNA回收试剂盒回收特异性片段后进行电泳检测,细菌16S rRNA基因V1-V3区条带大小均在500 bp左右,无杂带污染,扩增产物可用于Roche 454 FLX高通量测序。古菌16S rRNA基因V3-V4区条带大小均在350 bp左右,无杂带污染,扩增产物可用于Roche 454 FLX高通量测序。
2.4 高通量测序的序列分析如表 2所示,所有样品测序完成后,细菌共得到97 403条优质序列可用于后续分析,优质序列读长数在18 381-27 408,包含的OTU数目在3 132-5 794;古菌共得到118 045条优质序列可用于后续分析,优质序列读长数在25 694-34 384,包含的OTU数目在144-495;同时对每个样品的优质序列按序列相似度97%进行OTU聚类和后续分析,用Chao1、Shannon、Simpson指数来衡量所检测样品中细菌和古细菌多样性和丰度。Chao1指数在生态学中常用来估计物种总数,表征群落的丰富程度,Chao1指数值越大,说明该群落的丰富度越高。Shannon指数和Simpson指数用来反映群落的多样性,Shannon值越大,说明该群落的多样性越高;而Simpson指数值越大,则说明该群落的多样性越低。由表 2可以看出,从25 ℃条件下基础盐保藏试验中采集的样品,随着采样时间的推迟,细菌的Shannon指数呈增长趋势,Simpson指数呈降低趋势,说明驯化的产甲烷菌群在煤基-基础盐菌种保藏的过程中,细菌的多样性逐渐增加。所有样品的丰度稀释曲线不能达到稳定期,而Shannon多样性稀疏曲线几乎都达到饱和状态(数据未显示),表明尽管进一步的测序可以产生新的物种类型,但样品中的大部分微生物多样性已经被揭示。
| Strains | Items | Samples ID | |||
| 1 | 2 | 3 | 4 | ||
| Bacteria | Reads | 27 408 | 18 381 | 25 800 | 25 814 |
| OTUs | 3 995 | 3 132 | 5 221 | 5 794 | |
| Shannon index | 5.569 | 5.414 | 6.731 | 7.043 | |
| Simpson index | 0.053 | 0.045 | 0.005 | 0.004 | |
| Chao1 index | 11 838 | 9 704 | 16 339 | 16 791 | |
| Archaea | Reads | 25 694 | 34 384 | 30 292 | 27 675 |
| OTUs | 144 | 186 | 183 | 495 | |
| Shannon index | 1.250 | 0.498 | 0.816 | 1.672 | |
| Simpson index | 0.455 | 0.873 | 0.679 | 0.556 | |
| Chao1 index | 399 | 473 | 504 | 841 | |
| 注: 1-4: 25 ℃条件下煤基-基础盐保存的第35、42、70和105天产甲烷菌群样品. Note: 1-4: Samples of methanogenic consortia with different sampling time in coal and basic salt preservation test under 25 ℃ at 35, 42, 70, and 105 days, respectively. | |||||
研究获得的Roche 454 FLX高通量测序结果全部提交至NCBI SRA (Sequence read archive)数据库,获取登录号为SRA485238。
2.6 Roche 454 FLX高通量测序分析产甲烷菌群保藏过程中微生物菌群结构及动态变化为研究产甲烷菌群在25 ℃条件下煤基-基础盐保藏过程中微生物群落结构的特征及其动态变化,以对煤基-基础盐保藏过程的前105天为代表在菌种门水平(图 4)和属水平(图 5、6)上进行了统计分析。结果表明,煤基-基础盐菌种保藏过程中不论在门水平上还是属水平上,菌群结构均发生了显著变化。在菌种保藏的第35天样品中,变形菌门(Proteobacteria)在检测到的细菌门类中占据主导地位,所占百分比为92.06%,检测到的其他菌门还包括厚壁菌门(Firmicutes,7.66%)、放线菌门(Actinobacteria,0.22%)和拟杆菌门(Bacteroidetes,0.04%)等(图 4)。这一发现与先前在煤层气田产出水中的报道类似[10, 15]。细菌属类水平的鉴定主要为假单胞菌属(Pseudomonas,85.17%)(图 5)。但随着菌种保藏时间的延续(35-105 d),微生物的菌群结构发生了很大变化,优势菌种所占比例明显下降,在细菌门类水平上,Proteobacteria逐渐减少,Firmicutes显著增加,Bacteroidetes、Synergistetes、Chloroflexi也有所增加(图 4)。在细菌属类水平上,最初的主要优势菌——假单胞菌属(Pseudomonas)随着时间变化,其所占比例也逐渐减少,在保藏的105 d后降为2.77% (图 5),而梭菌属(Clostridium)、气单胞菌属(Aeromonas)、Ornatilinea等所占比例却明显增大(图 5)。相对于细菌的菌群结构,古菌菌属结构动态相对处于稳定的状态。在菌种保藏的初期(35 d),甲烷八叠球菌目(Methanosarcinales)占主导地位,包括甲烷八叠球菌属(Methanosarcina,69.67%)和甲烷鬃毛菌属(Methanosaeta,28.65%) (图 6),其中Methanosarcina为主要的优势菌属。随着保藏时间的延长,Methanosarcina所占比例明显增加,仍为主要优势菌属。

|
| 图 4 不同保藏时间段门类水平上细菌群落丰度分布 Figure 4 Distribution of bacterial community abundance sampling in different preservation time at the phylum level 注: 1-4: 25 ℃条件下煤基-基础盐保存的第35、42、70和105天产甲烷菌群样品; 丰度小于0.1%的序列被归类为"其他". Note: 1-4: Samples of methanogenic consortia with different sampling time in coal and basic salt preservation test under 25 ℃ at 35, 42, 70, and 105 days, respectively; Abundance of sequences of less than 0.1% was classified as 'Others'. |
|
|

|
| 图 5 不同保藏时间段属类水平上细菌群落丰度分布 Figure 5 Distribution of bacterial community abundance sampling in different preservation time at the genus level 注: 1-4: 25 ℃条件下煤基-基础盐保存的第35、42、70和105天产甲烷菌群样品; 丰度小于0.1%的序列被归类为"其他". Note: 1-4: Samples of methanogenic consortia with different sampling time in coal and basic salt preservation test under 25℃ at 35, 42, 70, and 105 days, respectively; Abundance of sequences of less than 0.1% was classified as 'Others'. |
|
|
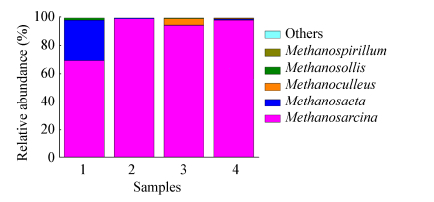
|
| 图 6 不同保藏时间段属类水平上古菌群落丰度分布 Figure 6 Distribution of archaeal community abundance sampling in different preservation time at the genus level 注: 1-4: 25 ℃条件下煤基-基础盐保存的第35、42、70和105天产甲烷菌群样品; 丰度小于0.1%的序列被归类为"其他". Note: 1-4: Samples of methanogenic consortia with different sampling time in coal and basic salt preservation test under 25 ℃ at 35, 42, 70, and 105 days, respectively; Abundance of sequences of less than 0.1% was classified as 'Others'. |
|
|
在科研和生产实践中,关于菌种的保藏多涉及单一菌种的保藏,都主要是通过借鉴低温、缺氧、干燥或饥饿等手段以降低保藏菌种的生理代谢活性,从而延长菌种的保藏时间[16-18]。而有关混合菌群的保藏一般比较困难,相关的研究报道也非常少[19]。混合菌群的保藏,不仅需要保证菌群结构的完整性,而且也需要选择合适的条件以确保保藏混合菌群的功能活性。在微生物降解煤生产甲烷的生物成气过程中,涉及到的功能微生物可能多达上百种,关于产甲烷菌群的保藏就会更加困难。在本研究中,我们参考了几种纯种微生物菌种的保藏方法,比如:甘油/L-半胱氨酸法参考了甘油冷冻保藏法,通过添加甘油对煤地质微生物进行保护,同时添加L-半胱氨酸以维持厌氧环境;富营养保藏法本意是想尽可能为煤层微生物提供充足的养分,让其最大限度地保持结构和功能的稳定;煤基-基础盐法主要参考了土壤微生物常用的保藏方法——沙土管保藏法,通过添加块状无烟煤及L-半胱氨酸来模拟煤层原位产甲烷菌群的厌氧生存环境。3种菌种保藏方法分别在不同设定温度条件下对煤层产甲烷菌群进行了厌氧保存,结果表明,25 ℃条件下煤基-基础盐保藏的混合菌群其产甲烷活性最高,且产甲烷功能活性也较稳定。区别于传统菌种保藏方法抑制微生物的活性,25 ℃条件下的煤基-基础盐菌种保藏在本质上既是产甲烷菌群富集驯化的过程,又是煤层微生物降解煤产甲烷的过程。该方法通过提供合适的环境促使保藏微生物菌群始终维持在较高的活性状态,明显有别于对传统菌种保藏原理的认知。在25 ℃煤基-基础盐产甲烷混合菌群的保藏试验中,采用了块状无烟煤作为菌种保藏的基质,这不仅可以为所要保藏的菌种提供生长所必需的碳源,同时也可以作为菌种粘附介质以保持菌群的稳定性;25 ℃煤基-基础盐保藏罐内L-半胱氨酸的添加可以为菌种提供厌氧环境,进而有利于产甲烷菌群的生长繁殖;而保藏基础盐液的间断加入,能够使菌群的活性长时间维持在较高水平。在此保藏体系中,产甲烷菌群在煤基质的促使下得到了很好的驯化,有利于保藏菌种在后续产气实验中迅速增殖并稳定发挥产甲烷的活力。此外,该保藏方法是在25 ℃条件下进行的,不需要超低温设备,也不需要特殊设备,方便快捷且适合不同量菌种的长期保藏。
在以16S rRNA基因高通量测序为基础进行煤基-基础盐保藏过程中产甲烷菌群结构的动态变化分析中,保藏体系中代表性产甲烷菌群的获取尤为重要。有研究发现,在微生物降解煤有机质的固液体系中,一部分微生物趋向于吸附在煤基质表面以直接获得能源物质,从而导致了体系中微生物在固、液相中出现分布不均匀的情况[20-21]。然而,由于煤基质的理化性质复杂,直接从煤基质中提取微生物的总基因组DNA会造成多种酶促反应抑制因子的污染[21],从而无法对其中微生物的遗传信息进行解析。本文在参考学者们前期工作的基础上[10, 22],对保藏液相体系中的微生物进行了基于16S rRNA基因的高通量测序分析,以此来代表反映煤层气产甲烷菌群在整个保藏体系中的结构特征。
微生物降解煤生成甲烷是一个复杂的过程,它需要发酵细菌、产氢产乙酸菌及产甲烷古菌共同协作完成[23]。不同的功能菌可以降解复杂的大分子物质产生代谢产物,这些中间代谢产物又被降解成一些简单的化合物供产甲烷古菌利用来产生甲烷。在25 ℃条件下的煤基-基础盐产甲烷菌群保藏过程中,随着菌种保藏时间的延长,细菌门类Proteobacteria逐渐减少,而Firmicutes显著增加,这与先前的研究结果类似[24]。Firmicutes在煤层气盆地的菌群结构中往往占有很少的部分,而其却在实验室产气培养中发挥着重要的作用。在细菌属类水平上,Pseudomonas在菌种保藏初期占据主导地位,随着时间的延长,其所占百分比急剧下降但仍然存在。同时,保藏体系中相应的Clostridium、Citrobacter、Aeromonas、Proteocatella、Proteiniphilum等菌属的丰度都有所增加,这些菌属都属于发酵细菌或是产氢产乙酸菌,经共同协作参与煤的降解过程。例如:Pseudomonas是一种严格需氧的革兰氏阴性细菌,被广泛发现于土壤、水、植物和别的环境中,能够降解烷烃、烯烃和多环芳烃[25];Clostridium是一种特殊的厌氧菌,是pH中性溶剂、混合酸及乙醇的生产者,也是同型乙酸发酵者[24];Citrobacter属于兼性厌氧菌,可利用柠檬酸盐作为唯一的碳源通过发酵葡萄糖产生酸和H2/CO2;Proteiniphilum属于专性厌氧菌,可以水解一些含氮化合物产生乙酸和丙酸[26]。Pseudomonas的检出表明在进行菌种保藏试验时,单纯地从罐底通入高纯氮气并不能保证罐内处于严格的厌氧状态。培养液或煤基质中可能会残留微量的氧气,随着保藏时间的延长,罐内氧气含量急剧降低,致使优势菌属Pseudomonas的丰度逐渐下降。从保藏体系中进行取样时也会带入微量的氧气,这可以解释Pseudomonas在保藏后期依然被检出微量的情况。各时期保藏体系中检出的主要优势古菌属均为Methanosarcina,它具有广泛的可利用底物,除了能够还原H2/CO2及分解甲基化合物,也能够分解乙酸[27]。但在有关煤开采的研究中,降解乙酸途径是主要的一种产甲烷途径,因为乙酸盐比氢气更易获得[28]。同时,在菌种的同期保藏体系中检出的细菌属都主要以产酸细菌为主。因此,保藏体系中检测到的Methanosarcina产甲烷途径应属于乙酸型产甲烷途径。此外,Methanosarcina可以在间歇氧气存在的条件下存活[24],这可能也是促使Methanosarcina在保藏体系中生长繁殖占优势的原因之一。
| [1] |
Zhai C, Lin BQ, Wang L. Status and problems of drainage and utilization of downhole coalbed methane in coal mines in China[J]. Natural Gas Industry, 2008, 28(7): 23-26. 翟成, 林柏泉, 王力. 我国煤矿井下煤层气抽采利用现状及问题[J]. 天然气工业, 2008, 28(7): 23-26. |
| [2] |
Scott AR, Kaiser WR, Ayers WB. Thermogenic and secondary biogenic gases, San Juan Basin, Colorado and New Mexico-implications for coalbed gas producibility[J]. AAPG Bulletin, 1994, 78(8): 1186-1209. |
| [3] |
Faiz M, Hendry P. Significance of microbial activity in Australian coal bed methane reservoirs ― a review[J]. Bulletin of Canadian Petroleum Geology, 2006, 54(3): 261-272. DOI:10.2113/gscpgbull.54.3.261 |
| [4] |
Rice DD, Claypool GE. Generation, accumulation, and resource potential of biogenic gas[J]. AAPG Bulletin, 1981, 65(1): 5-25. |
| [5] |
Fallgren PH, Zeng CP, Ren ZY, et al. Feasibility of microbial production of new natural gas from non-gas-producing lignite[J]. International Journal of Coal Geology, 2013, 115: 79-84. DOI:10.1016/j.coal.2013.03.003 |
| [6] |
Wang H, Lin H, Dong YB, et al. Experiments on the gas production of brown coal degraded by exogenous methanogens[J]. Petroleum Exploration and Development, 2012, 39(6): 764-768. 汪涵, 林海, 董颖博, 等. 外源产甲烷菌降解褐煤产气实验[J]. 石油勘探与开发, 2012, 39(6): 764-768. |
| [7] |
Wang AK, Qin Y, Lan FJ. Processes and possible pathways of biogenic coalbed methane generation from lignites based on parent methanogen[J]. Geological Journal of China Universities, 2012, 18(3): 485-489. 王爱宽, 秦勇, 兰凤娟. 基于本源菌的褐煤生物气生成过程与可能途径[J]. 高校地质学报, 2012, 18(3): 485-489. |
| [8] |
Wang BY, Tai C, Wu L, et al. Methane production from lignite through the combined effects of exogenous aerobic and anaerobic microflora[J]. International Journal of Coal Geology, 2017, 173: 84-93. DOI:10.1016/j.coal.2017.02.012 |
| [9] |
Yang XQ, Wu RW, Han ZY, et al. Analysis of methanogenic community and pathway of coalbed methane fields in the Qinshui Basin based on mcrA gene[J]. Microbiology China, 2017, 44(4): 795-806. 杨秀清, 吴瑞薇, 韩作颖, 等. 基于mcrA基因的沁水盆地煤层气田产甲烷菌群与途径分析[J]. 微生物学通报, 2017, 44(4): 795-806. |
| [10] |
Guo HG, Yu ZS, Liu RY, et al. Methylotrophic methanogenesis governs the biogenic coal bed methane formation in Eastern Ordos Basin, China[J]. Applied Microbiology and Biotechnology, 2012, 96(6): 1587-1597. DOI:10.1007/s00253-012-3889-3 |
| [11] |
Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [12] |
Raskin L, Stromley JM, Rittmann BE, et al. Group-specific 16S rRNA hybridization probes to describe natural communities of methanogens[J]. Applied and Environmental Microbiology, 1994, 60(4): 1232-1240. |
| [13] |
Watanabe T, Asakawa S, Nakamura A, et al. DGGE method for analyzing 16S rDNA of methanogenic archaeal community in paddy field soil[J]. FEMS Microbiology Letters, 2004, 232(2): 153-163. DOI:10.1016/S0378-1097(04)00045-X |
| [14] |
Schloss PD, Westcott SL, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [15] |
Midgley DJ, Hendry P, Pinetown KL, et al. Characterisation of a microbial community associated with a deep, coal seam methane reservoir in the Gippsland Basin, Australia[J]. International Journal of Coal Geology, 2010, 82(3/4): 232-239. |
| [16] |
Su LM, Li S. Preservation method for L-arginine-producing strain of Brevibacterium flavum[J]. Industrial Microbiology, 2009, 39(1): 60-62. 苏令鸣, 李爽. 产精氨酸的黄色短杆菌菌种保藏方法的研究[J]. 工业微生物, 2009, 39(1): 60-62. |
| [17] |
Zhao WJ, Xu SY. A simple and effective method for preserving Xanthomonas canpestris[J]. Science and Technology of Food Industry, 2005, 26(1): 131. 赵文娟, 徐升运. 一种简单有效的黄原胶菌种保藏方法[J]. 食品工业科技, 2005, 26(1): 131, 142. |
| [18] |
Satoh K, Tanaka T, Oguro Y, et al. Improvement of preservation method for ammonia-oxidizing bacteria by freeze-drying[J]. Soil Science and Plant Nutrition, 2004, 50(5): 777-781. DOI:10.1080/00380768.2004.10408535 |
| [19] |
Jiang H, Gou ZX, Han B, et al. Study on preservation methods of mixed methane-oxidizing bacteria[J]. Microbiology China, 2014, 41(7): 1463-1469. 江皓, 缑仲轩, 韩冰, 等. 甲烷氧化混合菌的保藏方法研究[J]. 微生物学通报, 2014, 41(7): 1463-1469. |
| [20] |
Klein DA, Flores RM, Venot C, et al. Molecular sequences derived from paleocene fort union formation coals vs. associated produced waters: implications for CBM regeneration[J]. International Journal of Coal Geology, 2008, 76(1/2): 3-13. |
| [21] |
Li DM, Hendry P, Faiz M. A survey of the microbial populations in some Australian coalbed methane reservoirs[J]. International Journal of Coal Geology, 2008, 76(1/2): 14-24. |
| [22] |
Strąpoć D, Picardal FW, Turich C, et al. Methane-producing microbial community in a coal bed of the Illinois Basin[J]. Applied and Environmental Microbiology, 2008, 74(8): 2424-2432. DOI:10.1128/AEM.02341-07 |
| [23] |
Springer E, Sachs MS, Woese CR, et al. Partial gene sequences for the a subunit of methyl-coenzyme M reductase (mcrI) as a phylogenetic tool for the family Methanosarcinaceae[J]. International Journal of Systematic Bacteriology, 1995, 45(3): 554-559. DOI:10.1099/00207713-45-3-554 |
| [24] |
Colosimo F, Thomas R, Lloyd JR, et al. Biogenic methane in shale gas and coal bed methane: a review of current knowledge and gaps[J]. International Journal of Coal Geology, 2016, 165: 106-120. DOI:10.1016/j.coal.2016.08.011 |
| [25] |
Rockne KJ, Chee-Sanford JC, Sanford RA, et al. Anaerobic naphthalene degradation by microbial pure cultures under nitrate-reducing conditions[J]. Applied and Environmental Microbiology, 2000, 66(4): 1595-1601. DOI:10.1128/AEM.66.4.1595-1601.2000 |
| [26] |
Chen SY, Dong XZ. Proteiniphilum acetatigenes gen. nov., sp. nov., from a UASB reactor treating brewery wastewater[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(6): 2257-2261. DOI:10.1099/ijs.0.63807-0 |
| [27] |
Park SY, Liang YN. Biogenic methane production from coal: a review on recent research and development on microbially enhanced coalbed methane (MECBM)[J]. Fuel, 2016, 166: 258-267. DOI:10.1016/j.fuel.2015.10.121 |
| [28] |
Beckmann S, Lueders T, Krüger M, et al. Acetogens and acetoclastic Methanosarcinales govern methane formation in abandoned coal mines[J]. Applied and Environmental Microbiology, 2011, 77(11): 3749-3756. DOI:10.1128/AEM.02818-10 |
 2018, Vol. 45
2018, Vol. 45




