扩展功能
文章信息
- 方晓梅, 柏菁璘, 游雪甫, 马百平, 张玉琴, 余利岩
- FANG Xiao-Mei, BAI Jing-Lin, YOU Xue-Fu, MA Bai-Ping, ZHANG Yu-Qin, YU Li-Yan
- 蜘蛛抱蛋属植物内生细菌的多样性及其抗菌活性筛选
- Endophytic bacteria diversity of Aspidistra Ker-Gawl. and their antimicrobial activities
- 微生物学通报, 2018, 45(4): 836-847
- Microbiology China, 2018, 45(4): 836-847
- DOI: 10.13344/j.microbiol.china.170286
-
文章历史
- 收稿日期: 2017-04-08
- 接受日期: 2017-11-23
- 网络首发日期(www.cnki.net): 2017-12-27
2. 军事医学科学院放射与辐射医学研究所 北京 100850
2. Institute of Radiation Medicine, Academy of Military Medical Sciences, Beijing 100850, China
现阶段的内生菌研究中,Petrini提出的内生菌概念被广泛接受[1]。他将内生菌定义为那些在其生活史中的某一段时期生活在植物组织内,对植物组织没有引起明显病害症状的微生物[2]。植物内生菌与宿主植物长期共处存在复杂的微生态关系:一方面,植物内生菌侵入宿主植物后,不易受外界环境条件的影响,具有相对稳定的生存空间,可以长期在其中定殖和分布[3];另一方面,内生菌次级代谢产物具有刺激植物生长发育或提高宿主植物胁迫抗性能力的作用[4]。Schulz和Boyle将内生菌与其宿主的关系假定为一种“对抗的平衡”[5]。药用植物内生菌不仅能够参与植物次生代谢及成分的转化合成,而且还可独立产生丰富的次级代谢产物,其中不乏有应用前景广阔的新型生物活性物质[6-8],在制药领域具有巨大的潜力[9]。以临床公认的抗肿瘤药物—紫杉醇为代表,学者们对植物内生真菌进行了广泛、深入的研究,然而有关植物内生细菌的生物学作用及其活性物质的研究却相对滞后[3]。随着各生物学领域的不断发展,学者们认为植物内生细菌具备更多独特的生物学特性,将会成为新兴的微生物资源,对人类生活发挥更大的作用[4, 10-11]。目前学者们已经从内生细菌获得了潜在的具有抗菌、抗氧化、抗神经变性疾病和酶抑制剂属性的药物先导化合物[8-16]。蜘蛛抱蛋是百合科蜘蛛抱蛋属的多年生草本植物,其根状茎有祛风解毒、散瘀止痛、健胃生肌等功效。现代药理研究表明,蜘蛛抱蛋的挥发性分泌物对木霉、黑曲霉和金黄色葡萄球菌的生长抑制较强[17];从卵叶蜘蛛抱蛋中分离到的卵叶酮A对革兰氏阴性菌的抑菌效果优于盐酸小檗碱[18]。还有研究表明蜘蛛抱蛋煎剂对结肠炎耶尔森菌和摩根变形杆菌有抑制作用,而且能够增加胃蛋白酶活性,具有较好的抗溃疡作用[19]。该药在民间使用量日益增长,然而蜘蛛抱蛋分布区域狭窄,种群较稀少,加之人为大量采挖严重,资源急剧减少[19]。本文拟对采集自贵州植物园的9种13株蜘蛛抱蛋植物进行内生菌的多样性研究,并对分离到的菌株进行多种抗菌活性筛选,为寻找能够产生新颖化学结构和作用机理次级代谢产物的生物活性菌株奠定基础。
1 材料与方法 1.1 植物样品蜘蛛抱蛋属9个种的13株植物采集于贵阳药用植物园。植物名称详见表 1。
| 植物编号 Plant No. |
植物名称 Species of Aspidistra |
植物组织部位 Plant tissue |
各植物组织中分离得到的细菌数量(株) Strain numbers isolated from different tissues |
从不同培养基上分离得到的细菌数量(株) Strain numbers isolated from different media |
合计(株) Total |
||||||
| 根 Root | 叶 Leaf | M1 | M3 | M4 | M5 | M6 | |||||
| 1 | 广西蜘蛛抱蛋A. retusa | 根 | 18 | 0 | 1 | 0 | 0 | 14 | 3 | 18 | |
| 叶 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | |||
| 2 | 贵州蜘蛛抱蛋A. guizhouensis | 根 | 13 | 0 | 1 | 2 | 3 | 5 | 2 | 13 | |
| 叶 | 0 | 3 | 0 | 1 | 2 | 0 | 0 | 3 | |||
| 3 | 荔波蜘蛛抱蛋 | 根 | 18 | 0 | 0 | 4 | 7 | 4 | 3 | 18 | |
| A. liboensis | 叶 | 0 | 10 | 2 | 4 | 0 | 1 | 3 | 10 | ||
| 4 | 平塘蜘蛛抱蛋 | 根 | 25 | 0 | 3 | 9 | 2 | 8 | 3 | 25 | |
| A. pingtangensis | 叶 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 5 | 伞柱蜘蛛抱蛋 | 根 | 9 | 0 | 0 | 4 | 0 | 1 | 4 | 9 | |
| A. fungilliformis | 叶 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 6 | 伞柱蜘蛛抱蛋 | 根 | 8 | 0 | 5 | 0 | 1 | 1 | 1 | 8 | |
| A. fungilliformis | 叶 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 1 | ||
| 7 | 四川蜘蛛抱蛋 | 根 | 16 | 0 | 3 | 1 | 2 | 2 | 8 | 16 | |
| A. sichuanensis | 叶 | 0 | 11 | 4 | 2 | 0 | 0 | 5 | 11 | ||
| 8 | 四川蜘蛛抱蛋 | 根 | 15 | 0 | 6 | 0 | 0 | 2 | 7 | 15 | |
| A. sichuanensis | 叶 | 0 | 3 | 1 | 0 | 0 | 2 | 0 | 3 | ||
| 9 | 四川蜘蛛抱蛋 | 根 | 7 | 0 | 1 | 0 | 1 | 1 | 4 | 7 | |
| A. sichuanensis | 叶 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | ||
| 10 | 线叶蜘蛛抱蛋 | 根 | 16 | 0 | 3 | 4 | 5 | 0 | 4 | 16 | |
| A. linearifolia | 叶 | 0 | 3 | 1 | 0 | 0 | 2 | 0 | 3 | ||
| 11 | 线叶蜘蛛抱蛋 | 根 | 15 | 0 | 12 | 1 | 2 | 0 | 0 | 15 | |
| A. linearifolia | 叶 | 0 | 5 | 0 | 3 | 2 | 0 | 0 | 5 | ||
| 12 | 长瓣蜘蛛抱蛋 | 根 | 5 | 0 | 0 | 0 | 3 | 0 | 2 | 5 | |
| A. longipetala | 叶 | 0 | 3 | 0 | 1 | 1 | 1 | 0 | 3 | ||
| 13 | 蜘蛛抱蛋 | 根 | 23 | 0 | 3 | 4 | 6 | 5 | 5 | 23 | |
| A. elatior | 叶 | 0 | 7 | 1 | 1 | 2 | 1 | 2 | 7 | ||
| 小计 Subtotal |
13株植物26个植物组织 13 Species, 26 Tissues |
188 | 46 | 47 | 41 | 39 | 51 | 56 | 234 | ||
抑制剂(mg/L):萘啶酮酸25.0,氨曲南25.0,制霉菌素50.0,重铬酸钾25.0。抑制剂均购自美国Sigma公司。
1.3 培养基 1.3.1 分离培养基M1 (g/L):酵母粉0.25,磷酸氢二钾0.50,琼脂15.00,pH 7.2。
M3 (g/L):丙酸钠2.00,硝酸铵0.10,氯化钾0.10,七水硫酸镁0.05,七水硫酸亚铁0.05,琼脂15.00,pH 7.2。
M4 (g/L):淀粉2.00,磷酸氢二钾0.50,硫酸镁0.50,硝酸钾1.00,氯化钠0.40,FeSO4·7H2O 0.01,琼脂15.00,pH 7.2。
M5 (g/L):琥珀酸钠0.90,磷酸二氢铵0.50,七水硫酸镁0.10,七水硫酸亚铁0.01,琼脂15.00,pH 7.2。
M6 (g/L):复合维生素0.01,琼脂15.00,pH 7.2。
以上培养基均1×105 Pa灭菌15 min。
1.3.2 菌株纯化及继代培养基PYG培养基(g/L):蛋白胨3.0,酵母粉5.0,丙三醇10 mL,pH 7.2。1×105 Pa灭菌15 min。
1.3.3 发酵培养基放线菌A1培养基(g/L):黄豆粉15.0,可溶性淀粉30.0,磷酸氢二钾0.5,硫酸亚铁0.5,硫代硫酸钠20.0 μg,氯化钾0.3,pH 7.2。1×105 Pa灭菌15 min。
放线菌A2培养基(g/L):葡萄糖10.0,可溶性淀粉30.0,棉籽饼粉20.0,酵母膏3.0,硫酸铵3.0,硫酸镁1.0,磷酸氢二钾1.0,氯化钠1.0,碳酸钙5.0,pH 7.2。1×105 Pa灭菌15 min。
细菌B1培养基(g/L):葡萄糖20.0,干酵母3.0,硫酸镁0.5,氯化钠0.5,黄豆饼粉20.0,磷酸氢二钾1.0,氯化钾0.5,维生素B 0.5 μg。1×105 Pa灭菌15 min。
细菌B2培养基(g/L):大豆蛋白胨20.0,葡萄糖2.5,氯化钠5.0,磷酸氢二钾2.5,维生素B 0.5 μg。1×105 Pa灭菌15 min。
1.4 检定菌耻垢分枝杆菌(Mycobacterium smegmatis ATCC 700044)、水稻白叶枯菌(Xanthomonas oryzae PXO99A)、白色念珠菌(Candida albicans ATCC 10231)、肺炎雷伯菌(Klebsiella pneumoniae ATCC 700603)、粪肠球菌HH22 (Enterococcus faecalis HH22,临床分离株,对庆大霉素、青霉素、红霉素和四环素等多重耐药),由中国药学微生物菌种保藏管理中心和本所药理室保藏。
1.5 植物表面消毒及菌株分离、保藏植物表面消毒、内生菌分离及菌株保藏参考魏玉珍等[20]的方法。
1.6 菌种初步鉴定和多样性分析菌种初步鉴定参照徐丽华等主编的《放线菌系统学》相关方法操作[21]。将同一植物组织来源的内生菌根据菌落和菌体形态初步观察排除重复菌株,并统计分离菌株的组织来源及培养基。将同一宿主植物来源的分离菌株进一步依据形态特征排重。测定内生细菌的16S rRNA基因序列,并将结果提交EzTaxon网站(http://www.eztaxon.org)[22],与相关属种中有效发表菌株的16S rRNA基因序列比对,初步判定菌株的所属类群。利用软件(http://bioinfogp.cnb.csic.es/tools/venny)绘制Venn图,比较内生菌种群在不同植物组织(根部组织和叶片组织)中的生态分布情况;根据Eztaxon中序列对比结果,从GenBank数据库中调集相关序列,运用ClustalX 1.8软件进行比对并计算供试菌株与参比菌株之间的序列相似性[23];应用MEGA 5.0软件[24]采用邻接法(Neighbour-Joining)构建系统进化树,分析内生细菌的系统发育地位及物种多样性特点。
1.7 发酵液抗菌活性测定将内生细菌依据菌株类型分别接种于2种发酵培养基中(典型放线菌形态菌株接种于放线菌A1、A2培养基,细菌形态菌株接种于细菌B1、B2培养基),28 ℃、200 r/min培养6 d。取7.5 mL发酵液于4 ℃、6 000 r/min离心10 min得到上清;菌体部分用7.5 mL丙酮抽提,4 ℃低温真空浓缩至干燥后溶于1 mL的DMSO中得到菌体粗提样品。用直径为6 mm的圆形无菌滤纸片分别吸取40 μL发酵液上清和菌体粗提样品,置于含有相应检测菌的检定平板上,培养18 h后测定抑菌圈直径大小。
2 结果与分析 2.1 蜘蛛抱蛋植株内生细菌分离结果内生细菌在植物体中的分布具有普遍性、多样性的特点,在目前研究过的所有植物中均发现有内生细菌[8]。本研究从9个种13株26份蜘蛛抱蛋植物组织中初步分离得到234株纯培养物(表 1)。由表 1可知,内生细菌普遍存在于不同种的蜘蛛抱蛋植物中;根部组织来源的菌株数量明显高于叶片组织;M5和M6培养基的分离效果较好,分别得到51和56株纯培养物。
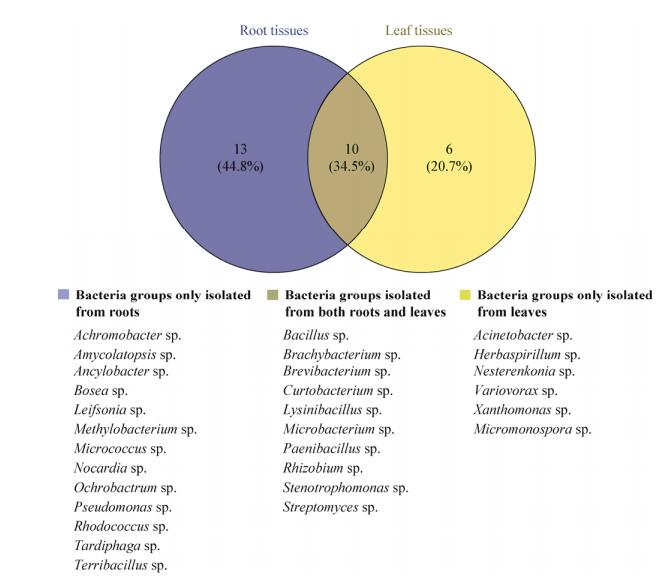
2.2 蜘蛛抱蛋内生细菌在宿主植株内的生态分布将分离得到的内生细菌按照宿主植株来源不同,经过形态排重后最终得到156株纯培养物,测定其16S rRNA基因序列。将结果提交EzTaxon网站,初步判定这些内生细菌分属于29个属。Venn图(图 1)显示芽孢杆菌属(Bacillus)、链霉菌属(Streptomyces)等10个属的菌株在根部和叶片组织均有分布,占内生菌种属总数的34.5%;13个菌属的菌株(如Achromobacter sp.、Amycolatopsis sp.、Ancylobacter sp.等)仅在根部组织中分离得到,占44.8%,6个菌属的菌株仅在叶片组织中分离得到(如Acinetobacter sp.、Herbaspirillum sp.和Nesterenkonia sp.等),占20.7%。
|
| 图 1 蜘蛛抱蛋植株根部和茎部组织分离得到的微生物类群比较Venn图 Figure 1 Venn map of endophytic bacteria groups isolated from different plant tissues of Aspidistra spp. |
|
|
16S rRNA基因序列比对结果显示蜘蛛抱蛋内生菌呈现出较丰富的物种多样性:(1)基于16S rRNA基因序列构建的系统进化树(图 2)显示156个菌株分属于放线菌门、变形菌门和厚壁菌门3个门,10个目22个科的29个属。其中放线菌门86株,占55.1%,归属于5个目8个科12个属;厚壁菌门42株,占26.9%,归属于Bacillales目3个科的4个属;变形菌门28株,占17.9%,归属于4个目11个科的13个属。(2)内生菌中链霉菌属(Streptomyces)、芽孢杆菌属(Bacillus)、微杆菌属(Microbacterium)、类芽孢杆菌属(Paenibacillus)占据一定的优势,分别分离到41、26、20和11株。(3)分离得到了一些稀有的物种,如Ochrobactrum、Stenotrophomonas、Variovorax、Herbaspirillum、Achromobacter、Tardiphaga等属的菌株。此外,分离得到的7个菌株与其近源菌株的16S rRNA基因序列相似性低于98.5%,可能代表了6个潜在的新分类单元(新种),它们分别属于微杆菌属(Microbacterium)、涅斯捷连科氏菌属(Nesterenkonia)、类芽孢杆菌属(Paenibacillus)、屈曲杆菌属(Ancylobacter)、短杆菌属(Brevibacterium)和嗜麦芽窄食单胞菌属(Stenotrophomonas),其确切的分类地位需要综合多相分类的各方面数据来进一步确定[25]。

|
| 图 2 156株蜘蛛抱蛋内生细菌基于16S rRNA基因序列构建的系统发育树 Figure 2 Neighbour-Joining phylogenetic tree based on the 16S rRNA genes sequence of 156 endophytic bacteria isolated from Aspidistra spp. 注:菌种名称后括号内的数字代表该属内分离得到的菌株数目;菌株名称后括号内的序号为NCBI序列编号;分支点上数字为重复1 000次的自展值;标尺0.02为核苷酸替换率;加粗字体的菌株为潜在的新的分类单元或稀有种属. Note: Numbers after the spp. indicate the number of isolated strains; NCBI accession numbers were listed behind strain numbers. Numbers at nodes indicate percentage levels of bootstrap support based on a Neighbour-Joining analysis of 1 000 resampled datasets. The scale bar indicates 0.02 substitutions per nucleotide position. The strains marked bold font indicate the potential novel or rare species. |
|
|
以耻垢分枝杆菌(Mycobacterium smegmatis ATCC 700044)、水稻白叶枯菌(Xanthomonas oryzae PXO99A)、白色念珠菌(Candida albicans ATCC 10231)、肺炎雷伯菌(Klebsiella pneumoniae ATCC 700603)和粪肠球菌HH22 (Enterococcus faecalis HH22)为检定菌,对分离到的156株内生菌发酵液离心、粗提得到的624份上清和菌体粗提样品进行了抗菌(包括抗耐药菌)活性的初步检测,发现38株内生菌的48份发酵液粗提品显示了对检定菌的抗菌活性,初筛菌株阳性率为23.7% (表 2)。阳性发酵液中有31份发酵液粗提品对一种或多种检定菌表现出较强的抑制活性(抑菌直径 > 10 mm);25份发酵液具有广谱抗菌特性,同时对2种以上检定菌有抑制作用,而其余23个发酵液则表现出相对特异的抗菌活性。
| 发酵编号 Fermentation No. |
菌株号 Strain No. |
菌株来源 Isolation source |
近源菌株 Top-hit type strain |
发酵液上清抗菌活性(抑菌圈直径,mm) Antibiotic activities of fermentation supernatant (inhibition diameter, mm) |
菌体粗提样品抗菌活性(抑菌圈直径,mm) Antibiotic activities of bacterial extracts (inhibition diameter, mm) |
|||||||||
| PXO99A | HH22 | ATCC 700603 | ATCC 700044 | ATCC 10231 | PXO99A | HH22 | ATCC 700603 | ATCC 700044 | ATCC 10231 | |||||
| I15AB-00404 | I15A-00404 | 线叶蜘蛛抱蛋根部 | S. somaliensis NBRC 12916T | - | - | - | - | - | - | - | - | - | + | |
| I15AA-00409 | I15A-00409 | 线叶蜘蛛抱蛋根部 | S. fulvissimus DSM 40593T | - | - | - | - | - | - | - | - | - | + | |
| I15AB-00409 | I15A-00409 | 线叶蜘蛛抱蛋根部 | S. fulvissimus DSM 40593T | - | - | - | - | - | - | - | - | - | + | |
| I15AB-00411 | I15A-00411 | 荔波蜘蛛抱蛋根部 | S. badius NRRL B-2567T | - | - | - | 10.0 | - | - | - | - | - | - | |
| I15AA-00412 | I15A-00412 | 荔波蜘蛛抱蛋根部 | S. pluricolorescens NBRC 12808T | - | 11.0 | - | - | - | - | - | - | + | - | |
| I15AB-00412 | I15A-00412 | 荔波蜘蛛抱蛋根部 | S. pluricolorescens NBRC 12808T | + | + | 14.0 | - | + | - | - | - | 6.5 | - | |
| I15AA-00413 | I15A-00413 | 荔波蜘蛛抱蛋叶片 | S. tendae ATCC 19812T | 11.0 | - | - | - | 14.0 | + | - | - | + | - | |
| I15AB-00413 | I15A-00413 | 荔波蜘蛛抱蛋叶片 | S. tendae ATCC 19812T | - | 14.0 | - | - | - | - | - | - | - | - | |
| I15AB-00415 | I15A-00415 | 罗甸蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | - | - | - | - | - | - | - | - | - | 7.0 | |
| I15AB-00416 | I15A-00416 | 荔波蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | - | - | - | - | - | - | - | - | + | 6.0 | |
| I15AA-00418 | I15A-00418 | 荔波蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | - | + | - | - | - | - | - | - | + | + | |
| I15AB-00418 | I15A-00418 | 荔波蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | - | - | - | - | - | - | - | - | - | 6.0 | |
| I15AA-00420 | I15A-00420 | 荔波蜘蛛抱蛋根部 | S. sindenensis NBRC 13475T | + | - | - | - | - | - | - | - | - | + | |
| I15AB-00420 | I15A-00420 | 荔波蜘蛛抱蛋根部 | S. sindenensis NBRC 13475T | - | - | - | - | - | - | - | - | - | + | |
| I15AB-00423 | I15A-00423 | 伞柱蜘蛛抱蛋根部 | S. hydrogenans NBRC 13476T | - | - | - | - | - | - | - | - | + | 7.0 | |
| I15AA-00424 | I15A-00424 | 伞柱蜘蛛抱蛋根部 | S. badius NRRL B-2567T | + | + | - | - | - | - | - | - | - | - | |
| I15AA-00432 | I15A-00432 | 四川蜘蛛抱蛋叶片 | S. vinaceusdrappus NRRL 2363T | - | - | 13.0 | - | - | - | - | - | - | - | |
| I15AB-00432 | I15A-00432 | 四川蜘蛛抱蛋叶片 | S. vinaceusdrappus NRRL 2363T | 13.0 | - | 21.0 | - | 14.0 | 16.0 | 21.0 | 16.0 | 10.0 | ||
| I15AB-00435 | I15A-00435 | 四川蜘蛛抱蛋叶片 | S. rochei NBRC 12908T | 15.0 | - | 18.0 | - | 13.0 | 13.5 | 12.0 | 8.0 | - | - | |
| I15AA-00442 | I15A-00442 | 线叶蜘蛛抱蛋根部 | St. chelatiphaga LPM-5T | 9.8 | - | - | - | - | - | - | - | - | - | |
| I15AB-00452 | I15A-00452 | 线叶蜘蛛抱蛋叶片 | St. plicatus NBRC 13071T | - | - | - | - | - | + | 16.0 | 15.0 | 11.0 | - | |
| I15AB-00457 | I15A-00457 | 线叶蜘蛛抱蛋根部 | A. tolypomycina DSM 44544T | - | 14.0 | - | - | - | - | - | - | + | - | |
| I15AA-00458 | I15A-00458 | 贵州蜘蛛抱蛋根部 | S. plicatus NBRC 13071T | - | - | 11.0 | - | - | - | - | - | - | - | |
| I15AB-00458 | I15A-00458 | 贵州蜘蛛抱蛋根部 | S. plicatus NBRC 13071T | 10.0 | - | 19.0 | - | 12.0 | 15.0 | 22.0 | 21.0 | 12.0 | 6.0 | |
| I15AA-00459 | I15A-00459 | 贵州蜘蛛抱蛋叶片 | S. vinaceusdrappus NRRL 2363T | - | - | 12.0 | - | - | - | - | - | + | - | |
| I15AB-00459 | I15A-00459 | 贵州蜘蛛抱蛋叶片 | S. vinaceusdrappus NRRL 2363T | 11.0 | - | 15.0 | 15.0 | 9.5 | 17.0 | 16.0 | 11.0 | - | ||
| I15AA-00465 | I15A-00465 | 蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | 10.0 | - | - | - | - | 12.0 | - | - | - | - | |
| I15AB-00465 | I15A-00465 | 蜘蛛抱蛋根部 | S. hydrogenans NBRC 13475T | - | - | - | - | - | - | - | - | 8.0 | + | |
| I15AB-00469 | I15A-00469 | 蜘蛛抱蛋根部 | S. badius NRRL B-2567T | - | - | - | - | - | - | 14.0 | 8.0 | - | - | |
| I15AA-00489 | I15A-00489 | 罗甸蜘蛛抱蛋叶片 | S. plicatus NBRC 13071T | + | - | 12.0 | - | - | - | - | - | - | - | |
| I15AB-00489 | I15A-00489 | 罗甸蜘蛛抱蛋叶片 | S. plicatus NBRC 13071T | - | - | 14.0 | - | 15.0 | - | 18.0 | 16.0 | 8.0 | + | |
| I15BA-00123 | I15B-00123 | 荔波蜘蛛抱蛋根部 | B. tequilensis KCTC 13622T | 12.5 | 12.0 | - | - | 21.0 | 9.5 | - | - | - | - | |
| I15BA-00133 | I15B-00133 | 荔波蜘蛛抱蛋根部 | B. tequilensis KCTC 13622T | 13.0 | - | - | - | 18.0 | 11.0 | - | - | - | - | |
| I15BA-00156 | I15B-00156 | 平塘蜘蛛抱蛋根部 | B. safensis FO-36bT | 13.6 | 20.0 | - | - | 22.0 | 18.0 | - | - | - | - | |
| I15BA-00203 | I15B-00203 | 四川蜘蛛抱蛋根部 | M. oryzae CBMB20T | - | - | - | - | + | - | - | - | - | - | |
| I15BA-00221 | I15B-00221 | 线叶蜘蛛抱蛋根部 | B. isronensis B3W22T | - | - | - | - | 12.0 | - | - | - | - | - | |
| I15BA-00222 | I15B-00222 | 线叶蜘蛛抱蛋根部 | P. borealis KK19T | - | - | - | - | 11.0 | - | - | - | - | - | |
| I15BA-00224 | I15B-00224 | 线叶蜘蛛抱蛋根部 | B. subtilis KCTC 13429T | - | - | - | - | 14.0 | - | - | - | - | + | |
| I15BA-00226 | I15B-00226 | 线叶蜘蛛抱蛋叶片 | L. macroides LMG 18474T | - | - | - | - | + | - | - | - | - | - | |
| I15BA-00227 | I15B-00227 | 线叶蜘蛛抱蛋叶片 | L. parviboronicapiens BAM-582T | - | - | - | - | 11.0 | - | - | - | - | - | |
| I15BA-00228 | I15B-00228 | 线叶蜘蛛抱蛋根部 | P. vulneris CCUG 53270T | - | - | - | - | 14.0 | - | - | - | - | - | |
| I15BA-00229 | I15B-00229 | 线叶蜘蛛抱蛋根部 | B. safensis FO-36bT | - | - | - | - | + | - | - | - | - | + | |
| I15BA-00232 | I15B-00232 | 线叶蜘蛛抱蛋根部 | B. safensis FO-36bT | - | - | - | - | + | - | - | - | - | + | |
| I15BA-00241 | I15B-00241 | 长瓣蜘蛛抱蛋叶片 | N. massiliensis NP1T | - | - | - | - | 11.0 | - | - | - | - | - | |
| I15BA-00242 | I15B-00242 | 四川蜘蛛抱蛋根部 | P. odorifer TOD45T | - | - | - | - | 12.0 | - | - | - | - | - | |
| I15BA-00244 | I15B-00244 | 四川蜘蛛抱蛋根部 | B. luciferensis LMG 18422T | - | - | - | - | 14.0 | - | - | - | - | - | |
| I15BA-00251 | I15B-00251 | 蜘蛛抱蛋根部 | M. testaceum DSM 20166T | - | - | - | - | 16.0 | - | - | - | - | - | |
| I15BA-00254 | I15B-00254 | 蜘蛛抱蛋根部 | B. safensis FO-36bT | - | - | - | - | 13.0 | - | - | - | - | - | |
| 注:发酵编号中的AA表示放线菌用A1培养基发酵得到的发酵液样品;AB表示放线菌用A2培养基发酵得到的发酵液样品;BA表示细菌用B2培养基发酵得到的发酵液样品. PXO99A:水稻白叶枯菌PXO99A;HH22:粪肠球菌HH22;ATCC 700603:肺炎雷伯ATCC 700603;ATCC 700044:耻垢分枝杆菌ATCC 700044;ATCC 10231:白色念珠菌ATCC 10231. S.:Streptomyces;B.:Bacillus;P.:Paenibacillus;L.:Lysinibacillus;St.:Stenotrophomonas;A.:Amycolatopsis;N.:Nesterenkonia;M.:Microbacterium. +:弱阳性,-:阴性. Note: AA: Fermentation with A1 medium; AB: Fermentation with A2 medium; BA: Fermentation with B1 medium. PXO99A: Xanthomonas oryzae PXO99A; HH22: Enterococcus faecalis HH22; ATCC 700603: Klebsiella pneumoniae ATCC 700603; ATCC 700044: Mycobacterium smegmatis ATCC 700044; ATCC 10231: Candida albicans ATCC 10231; S.: Streptomyces; B.: Bacillus; P.: Paenibacillus; L.: Lysinibacillus; St.: Stenotrophomonas; A.: Amycolatopsis; N.: Nesterenkonia; M.: Microbacterium. +: Weak positive; -: Negative. | ||||||||||||||
对38株初筛阳性菌株的16S rRNA基因序列对比结果(表 2)的统计分析表明,初筛阳性菌株主要集中在Streptomyces属(19株)、Bacillus属(14株)以及Paenibacillus属(3株),放线菌和芽孢菌是本研究中初筛阳性代谢产物的主要来源。
统计38株初筛阳性菌株的植物来源,发现它们广泛分布于蜘蛛抱蛋植物宿主的根和叶中(表 2)。不同发酵培养基的成分对内生细菌的抗菌活性有一定影响,12株A1培养基发酵的放线菌表现出抑菌活性,19株A2培养基发酵的放线菌表现出活性,2种培养基对放线菌发酵液的抑菌活性贡献相当,而初筛阳性细菌只有在使用B1培养基时才能发酵出活性。
3 讨论与结论本研究采用5种培养基对蜘蛛抱蛋属9个种的植株(根、叶)进行了内生细菌分离,共分离得到内生菌234株,研究表明内生菌存在于不同种植物体内,其分布具有广泛性。内生菌分离实验表明M5 (琥珀酸钠)和M6 (水琼脂)培养基的分离效果较好,进一步验证了琥珀酸钠对细菌的分离具有较好的选择作用[26]。本实验中寡营养培养基(M6)在分离过程中避免了常规菌群的快速繁殖,因而也取得了较好的分离效果。
迄今为止,蜘蛛抱蛋属药用植物内生菌的多样性研究尚未见报道。基于16S rRNA基因序列的系统发育分析表明,156株蜘蛛抱蛋内生菌分属于放线菌门、厚壁菌门和变形杆菌门。上述三大类群微生物广泛存在于不同种的蜘蛛抱蛋植株中,尤其是以链霉菌属、芽孢菌属、微杆菌属、类芽孢属和根瘤菌属等最为普遍。前期有科学家也发现人参、甘蔗、瑞香狼毒等植物的优势内生菌群为放线菌门的链霉菌、微杆菌和微球菌,厚壁菌门的芽孢杆菌、类芽孢杆菌,以及变形杆菌门的根瘤菌和葡糖醋杆菌[27-29]。除了这些普遍存在的内生菌群外,本研究还在蜘蛛抱蛋植株中发现了一些稀有的种属和7个潜在的微生物新物种,这对于开发药用植物内生细菌资源具有重要的研究意义。
目前已报道的内生菌大多可产生如抗肿瘤或抗菌类等活性成分[30]。Cho等发现一些人参内生菌(类芽孢杆菌、芽孢杆菌和假单胞菌等)具有对植物病原真菌生物防治的潜在活性[27]。本研究发现23.7%的蜘蛛抱蛋内生菌发酵液具有抑菌活性,其中链霉菌属、芽孢菌属以及类芽孢属的代谢产物在抗菌活性方面表现了较大潜力。此外,发酵培养基的选择对微生物次级代谢产物的产生具有重要的作用,本研究中得到的624份发酵液上清及菌体粗提样品中有48份样品表现出抑菌活性,它们为A1、A2及B1培养基发酵得到,而B2培养基发酵得到的发酵液没有表现出抑菌活性,提示在发酵培养基中加入适量棉籽饼粉或黄豆饼粉等有机氮源,可能有利于微生物合成活性次级代谢产物。有机氮源在其他抗生素(如青霉素)发酵过程中的应用也起到了促进效价提升作用[31]。
多样性的物种可以为寻找多样性的化合物提供机会,期望在多样性丰富的内生菌中寻找更多在化学结构和作用机理上有前景的新药以满足人类治疗的需求。目前本研究已经筛选得到38株具备不同药理活性的阳性菌。本实验分离得到的Ochrobactrum、Stenotrophomonas、Variovorax等稀有种属的菌株或者潜在新种菌株的代谢产物未表现出抗菌活性,极大可能是由于我们对这类微生物知之甚少,利用现有的培养条件不能满足它们的生长需求,或者某种代谢途径在普通培养条件下不能表达或表达较弱,而导致其代谢产物未能表现出药理活性。在前期的研究中,科学家也从植物组织中分离得到了类似的稀有微生物[32-37]。这些菌属的菌株在以往的研究中体现了丰富的生理活性潜能,如Herbaspirillum sp.、Stenotrophomonas sp.等菌株在植物的根瘤中起到了固氮作用[38-39];Leifsonia sp.、Ochrobactrum sp.等菌株能够分泌吲哚乙酸促进植物生长[40-41];在Variovorax sp.的菌株中发现了能够编码木质纤维素降解酶的基因组[42];Achromobacter sp.、Stenotrophomonas sp.等菌株对常见的根际真菌冻土毛霉(Mucor hiemalis)具有强烈的拮抗作用,并且能够抑制植物致病真菌立枯丝核菌(Rhizoctonia solani)[43-44];Leifsonia sp.能够脱去7-木糖基-10-脱乙酰基浆果赤霉素Ⅲ和7-木糖基-10-脱乙酰紫杉醇中的木糖基,产生10-脱乙酰基浆果赤霉素Ⅲ和抗癌药物紫杉醇前体10-脱乙酰紫杉醇[45]。由此推测这些较为稀有的内生菌与其宿主植物及其它微生物之间通过不同代谢途径相互作用,它们在环境压力下各自形成不同的“适应”或“御敌”策略,产生许多具有应用潜力的生物活性物质,发挥多种多样的生物学功能,从而达到自身的物质需求平衡。
在后续的研究中,积极探索这些稀有微生物的分离、培养条件,采取传统的药物模型和基因筛选相结合的策略对药用植物内生菌进行药理活性潜力评估,将为药用植物内生细菌资源的开发提供更全面的参考。
致谢: 感谢军事医学科学院放射医学研究所马百平研究员在药用植物功效判定方面的悉心指导,感谢中国医学科学院药用植物研究所郭宝林研究员在药用植物采集和鉴定方面给予的大力帮助。| [1] |
Petrini O. Fungal endophytes of tree leaves[A]//Andrews JH, Hirano SS. Microbial Ecology of Leaves[M]. New York: Springer-Verlag, 1991: 179-197
|
| [2] |
Guo LD. Advances of researches on endophytic fungi[J]. Mycosystema, 2001, 20(1): 148-152. 郭良栋. 内生真菌研究进展[J]. 菌物学报, 2001, 20(1): 148-152. |
| [3] |
Feng TX, Wang L, Chen HM, et al. Research advances on function and bioactive substances of Endophytic actinomycetes[J]. China Biotechnology, 2015, 35(4): 98-106. 冯天祥, 王玲, 陈海敏, 等. 植物内生放线菌功能及生物活性物质研究进展[J]. 中国生物工程杂志, 2015, 35(4): 98-106. |
| [4] |
Beiranvand M, Amin M, Hashemi-Shahraki A, et al. Antimicrobial activity of endophytic bacterial populations isolated from medical plants of Iran[J]. Iranian Journal of Microbiology, 2017, 9(1): 11-18. |
| [5] |
Schulz B, Boyle C. The endophytic continuum[J]. Mycological Research, 2005, 109(6): 661-686. DOI:10.1017/S095375620500273X |
| [6] |
Li JY, Strobel G, Sidhu R, et al. Endophytic taxol-producing fungi from bald cypress, Taxodium distichum[J]. Microbiology, 1996, 142(Pt 8): 2223-2226. |
| [7] |
Nalini MS, Prakash HS. Diversity and bioprospecting of actinomycete endophytes from the medicinal plants[J]. Letters in Applied Microbiology, 2017, 64(4): 261-270. DOI:10.1111/lam.2017.64.issue-4 |
| [8] |
Stone JK, Bacon CW, White JF, et al. An overview of endophytic microbes: endophytism defind[A]//Bacon CE, White JF Jr. Microbial Endophytes[M]. New York: Marcel Dekker, 2000: 29-33
|
| [9] |
Golinska P, Wypij M, Agarkar G, et al. Endophytic actinobacteria of medicinal plants: diversity and bioactivity[J]. Antonie van Leeuwenhoek, 2015, 108(2): 267-289. DOI:10.1007/s10482-015-0502-7 |
| [10] |
Dinesh R, Srinivasan V, Sheeja TE, et al. Endophytic actinobacteria: diversity, secondary metabolism and mechanisms to unsilence biosynthetic gene clusters[J]. Critical Reviews in Microbiology, 2017, 43(5): 546-566. DOI:10.1080/1040841X.2016.1270895 |
| [11] |
Matsumoto A, Takahashi Y. Endophytic actinomycetes: promising source of novel bioactive compounds[J]. Journal of Antibiotics, 2017, 70(5): 514-519. DOI:10.1038/ja.2017.20 |
| [12] |
Chen YT, Yuan Q, Shan LT, et al. Antitumor activity of bacterial exopolysaccharides from the endophyte Bacillus amyloliquefaciens sp. isolated from Ophiopogon japonicus[J]. Oncology Letters, 2013, 5(6): 1787-1792. DOI:10.3892/ol.2013.1284 |
| [13] |
Taechowisan T, Wanbanjob A, Tuntiwachwuttikul P, et al. Identification of Streptomyces sp. Tc022, an endophyte in Alpinia galanga, and the isolation of actinomycin D[J]. Annals of Microbiology, 2006, 56(2): 113-117. DOI:10.1007/BF03174991 |
| [14] |
Jasmine DJ, Agastian P. In vitro antioxidant activity and in vivo alpha glucosidase activity of endophytic actinomycetes isolated from Catharanthus roseus (l.) G. Don[J]. Journal of Pharmacy Research, 2013, 6(6): 674-678. DOI:10.1016/j.jopr.2013.06.007 |
| [15] |
Zhou H, Yang YB, Peng TF, et al. Metabolites of Streptomyces sp., an endophytic actinomycete from Alpinia oxyphylla[J]. Natural Product Research, 2014, 28(4): 265-267. DOI:10.1080/14786419.2013.830219 |
| [16] |
Indananda C, Igarashi Y, Ikeda M, et al. Linfuranone A, a new polyketide from plant-derived Microbispora sp. GMKU 363[J]. The Journal of Antibiotics, 2013, 66(11): 675-677. DOI:10.1038/ja.2013.67 |
| [17] |
Guo AJ. Study on characteristic of carbon fixation and transpiration of 10 interior foliage plants[D]. Harbin: Master's Thesis of Northeast Forestry University, 2004 (in Chinese) 郭阿君. 10种室内观叶植物固碳释氧、蒸腾、抑菌特性的研究[D]. 哈尔滨: 东北林业大学硕士学位论文, 2004 |
| [18] |
Liang XX, Kong LX, He M. A new homoisoflavone compound as a potent antibacterial agent from Aspidistra typica baill[J]. Journal of Chinese Pharmaceutical Sciences, 2016, 25(9): 700-703. |
| [19] |
Jiang XH, She CW, Zhang M, et al. A primary study on the ecology of medicinal plants A. triloba F. T. Wang et K. Y. Lang[J]. Northern Horticulture, 2010(8): 33-35. 蒋向辉, 佘朝文, 张敏, 等. 珍稀药用植物湖南蜘蛛抱蛋生态学初步研究[J]. 北方园艺, 2010(8): 33-35. |
| [20] |
Wei YZ, Zhang YQ, Zhao LL, et al. Isolation, screening and preliminary identification of endophytic actinobacteria from Mangroves at Shankou of Guangxi Province[J]. Microbiology China, 2010, 37(6): 823-828. 魏玉珍, 张玉琴, 赵莉莉, 等. 广西山口红树林内生放线菌的分离、筛选及初步鉴定[J]. 微生物学通报, 2010, 37(6): 823-828. |
| [21] |
Xu LH, Li WJ, Liu ZH, et al. Actinomycete Systematic— Principle, Methods and Practice[M]. Beijing: Science Press, 2007, 40-47, 66-80, 119-128. 徐丽华, 李文均, 刘志恒, 等. 放线菌系统学—原理、方法及实践[M]. 北京: 科学出版社, 2007, 40-47, 66-80, 119-128. |
| [22] |
Chun J, Lee JH, Jung Y, et al. EzTaxon: a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 10): 2259-2261. |
| [23] |
Thompson JD, Gibson TJ, Plewniak F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
| [24] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [25] |
Stackebrandt E, Ebers J. Taxonomic parameters revisited: tarnished gold standards[J]. Microbiology Today, 2006, 33(4): 152-155. |
| [26] |
Zhang JL, Qin YL, Xiong ZJ, et al. The selective isolation of endophytic actinomycetes[J]. Microbiology China, 2013, 40(7): 1305-1313. 张金丽, 秦玉丽, 熊子君, 等. 植物内生放线菌的选择性分离[J]. 微生物学通报, 2013, 40(7): 1305-1313. |
| [27] |
Cho KM, Hong SY, Lee SM, et al. Endophytic bacterial communities in ginseng and their antifungal activity against pathogens[J]. Microbial Ecology, 2007, 54(2): 341-351. DOI:10.1007/s00248-007-9208-3 |
| [28] |
Velázquez E, Rojas M, Lorite MJ, et al. Genetic diversity of endophytic bacteria which could be find in the apoplastic sap of the medullary parenchym of the stem of healthy sugarcane plants[J]. Journal of Basic Microbiology, 2008, 48(2): 118-124. DOI:10.1002/(ISSN)1521-4028 |
| [29] |
Du HJ, Su J, Yu LY, et al. Isolation and physiological characteristics of endophytic actinobacteria from medicinal plants[J]. Acta Microbiologica Sinica, 2013, 53(1): 15-23. 杜慧竟, 苏静, 余利岩, 等. 药用植物内生放线菌的分离和生物学特性[J]. 微生物学报, 2013, 53(1): 15-23. |
| [30] |
Tan RX, Zou WX. Endophytes: a rich source of functional metabolites[J]. Natural Product Reports, 2001, 18(4): 448-459. DOI:10.1039/b100918o |
| [31] |
Cui JQ. The function of organic nitrogen source in penicillin fermentation[J]. Journal of Science and Technology Innovation and Application, 2012(1): 41. 崔节泉. 有机氮源在青霉素发酵生产过程中的作用[J]. 科技创新与应用, 2012(1): 41. |
| [32] |
Qiu FB, Huang Y, Sun L, et al. Leifsonia ginsengi sp. nov., isolated from ginseng root[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 2): 405-408. |
| [33] |
Bertrand H, Plassard C, Pinochet X, et al. Stimulation of the ionic transport system in Brassica napus by a plant growth-promoting rhizobacterium (Achromobacter sp.)[J]. Canadian Journal of Microbiology, 2000, 46(3): 229-236. DOI:10.1139/w99-137 |
| [34] |
Rothballer M, Schmid M, Klein I, et al. Herbaspirillum hiltneri sp. nov., isolated from surface-sterilized wheat roots[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt 6): 1341-1348. |
| [35] |
Xin YH, Zhou YG, Zhou HL, et al. Ancylobacter rudongensis sp. nov., isolated from roots of Spartina anglica[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(Pt 2): 385-388. |
| [36] |
de Meyer SE, Coorevits A, Willems A. Tardiphaga robiniae, gen. nov., sp. nov., a new genus in the family Bradyrhizobiaceae isolated from Robinia pseudoacacia in Flanders (Belgium)[J]. Systematic and Applied Microbiology, 2012, 35(4): 205-214. DOI:10.1016/j.syapm.2012.02.002 |
| [37] |
Gao JL, Yuan M, Wang XM, et al. Variovorax guangxiensis sp. nov., an aerobic, 1-aminocyclopropane-1-carboxylate deaminase producing bacterium isolated from banana rhizosphere[J]. Antonie van Leeuwenhoek, 2015, 107(1): 65-72. DOI:10.1007/s10482-014-0304-3 |
| [38] |
Valverde A, Velázquez E, Gutiérrez C, et al. Herbaspirillum lusitanum sp. nov., a novel nitrogen-fixing bacterium associated with root nodules of Phaseolus vulgaris[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(Pt 6): 1979-1983. |
| [39] |
Ramos PL, van Trappen S, Thompson FL, et al. Screening for endophytic nitrogen-fixing bacteria in Brazilian sugar cane varieties used in organic farming and description of Stenotrophomonas pavanii sp. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(Pt 4): 926-931. |
| [40] |
Kang SM, Asaf S, Kim SJ, et al. Complete genome sequence of plant growth-promoting bacterium Leifsonia xyli SE134, a possible gibberellin and auxin producer[J]. Journal of Biotechnology, 2016, 239: 34-38. DOI:10.1016/j.jbiotec.2016.10.004 |
| [41] |
Yu X, Li Y, Cui Y, et al. An indoleacetic acid-producing Ochrobactrum sp. MGJ11 counteracts cadmium effect on soybean by promoting plant growth[J]. Journal of Applied Microbiology, 2017, 122(4): 987-996. DOI:10.1111/jam.2017.122.issue-4 |
| [42] |
Woo HL, Deangelis KM, Teshima H, et al. High-quality draft genome sequences of four lignocellulose-degrading bacteria isolated from Puerto Rican forest soil: Gordonia sp., Paenibacillus sp., Variovorax sp., and Vogesella sp[J]. Genome Announcements, 2017, 5(18): e00300-17. DOI:10.1128/genomeA.00300-17 |
| [43] |
Wolf A, Fritze A, Hagemann M, et al. Stenotrophomonas rhizophila sp. nov., a novel plant-associated bacterium with antifungal properties[J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(Pt 6): 1937-1944. |
| [44] |
de Boer W, Hundscheid MPJ, Gunnewiek PJAK, et al. Antifungal rhizosphere bacteria can increase as response to the presence of saprotrophic fungi[J]. PLoS One, 2015, 10(9): e0137988. DOI:10.1371/journal.pone.0137988 |
| [45] |
Hao DC, Ge GB, Yang L. Bacterial diversity of Taxus rhizosphere: culture-independent and culture-dependent approaches[J]. FEMS Microbiology Letters, 2008, 284(2): 204-212. DOI:10.1111/fml.2008.284.issue-2 |
 2018, Vol. 45
2018, Vol. 45