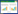扩展功能
文章信息
- 徐蕾, 王佳华, 卢烨, 徐俊
- XU Lei, WANG Jia-Hua, LU Ye, XU Jun
- 海水中主要金属盐对4株Kangiella属细菌的生长及其蛋白酶相关基因表达的影响
- Effect of major metal ions in sea water on growth and transcription of protease coding gene of 4 Kangiella strains
- 微生物学通报, 2018, 45(4): 731-743
- Microbiology China, 2018, 45(4): 731-743
- DOI: 10.13344/j.microbiol.china.170366
-
文章历史
- 收稿日期: 2017-05-16
- 接受日期: 2017-06-05
- 网络首发日期(www.cnki.net): 2017-07-11
2. 上海交通大学海洋研究院 上海 200240
2. Institute of Oceanography, Shanghai Jiao Tong University, Shanghai 200240, China
2004年,Yoon等首次从黄海的潮间带沉积物样本中分离获得菌株Kangiella koreensis DSM 16069和K. aquimarina DSM 16071,为了纪念韩国著名的微生物学家Kook Hee Kang,将此类海洋来源的革兰氏阴性杆菌的2个新种代表的新属命名为Kangiella[1]。目前,从不同的海洋环境中分离鉴定出了多个Kangiella属的新种。2010年,Romanenko等从日本海海岸附近的海水中分离出K. japonica KMM 3899[2];2011年,美国研究者从佛罗里达州附近海域的海绵体中分离鉴定了K. spongicola JCM 16211[3];2012年,Yoon等又从韩国南部的海水中分离出K. geojedonensis YCS-5[4];同年,Jean等从中国台湾北部浅海海域分离鉴定出K. taiwanensis BCRC 80330[5];2013年,Lee等从韩国南海分离鉴定出K. sediminilitoris KCTC 23892[6];2015年,Xu等在西南印度洋2 784 m的海底沉积物中分离鉴定出K. profundi FT102[7];同年,Kim等从韩国济州岛的样本中分离鉴定出K. chungangensis CAU 1040[8]。
Kangiella属按16S rRNA基因序列一直被归属于γ-变形杆菌纲海洋螺杆菌目下的食烷菌科(Alcanivoracaceae)。Wang等将新发现的与K. geojedonensis YCS-5亲缘关系较近的Aliikangiella marina GYP-15鉴定为一个新属,并据此提出在海洋螺杆菌目下建立一个新科,即康氏菌科(Kangiellaceae),其下包括Kangiella、Pleionea和Aliikangiella这3个属[9]。
目前已分离鉴定出的Kangiella菌株都来自于海洋环境,所有9株Kangiella菌株最适生长温度位于30−37 ℃范围内,最适pH为7.0左右,都具有氧化酶和过氧化氢酶;绝大多数菌株能够在无氧条件下还原硝酸盐。Kangiella菌株不能发酵大多数单糖产酸,可利用胶原、酪蛋白、酪氨酸、吐温等有机质[1-8],推测该类海洋细菌产生的胞外蛋白酶在其利用有机氮源生长的过程中起重要的作用。
盐度是影响水生系统中生物分布状态的主要因素之一。海洋环境中的盐度差异是影响该环境中微生物群落组成的决定性因素[10]。除K. chungangensis CAU 1040以外,其他已知的Kangiella菌株都必需在有NaCl (0.5%−16.0%)的复杂培养基中才能够正常生长。韩国南部半岛海域的盐度为3.0%左右[11]。西南印度洋海域的盐度相对较高,一般为3.4%左右[12]。海水中最主要的4种金属盐类分别为Na盐、Mg盐、Ca盐和K盐,在广泛使用的2216E海水培养基中这4种金属离子的摩尔浓度依次为:Na盐0.335 mol/L、Mg盐0.089 mol/L、Ca盐0.016 mol/L、K盐0.007 mol/L。本文选取分离自韩国周边海域和西南印度洋的4株Kangiella属菌株,研究了海水中主要金属离子对Kangiella属海洋细菌的生长、胞外蛋白酶活及其中3个保守的丝氨酸蛋白酶编码基因转录的影响。
1 材料与方法 1.1 菌株所用Kangiella属的4个模式菌株除K. profundi FT102为本实验室前期分离鉴定以外,其余3株皆从日本理化学研究所微生物菌种保藏中心(http://jcm.brc.riken.jp/ja/catalogue)购买获得(表 1)。
| 物种 Species |
样本来源 Sampling site |
参考文献 References |
| K. aquimarina DSM 16071 | Tidal flat sediment was collected from Daepo Beach, Yellow Sea, Korea | [1] |
| K. geojedonensis YCS-5 | Seawater off the southern coast of Korea | [4] |
| K. koreensis DSM 16069 | Tidal flat sediment was collected from Daepo Beach, Yellow Sea, Korea | [1] |
| K. profundi FT102 | A deep-sea sediment sample collected in the south-west Indian Ocean at 2 784 m | [7] |
PCR引物采用DNAMAN和Primer Express 3.0.1软件设计,由生工生物工程(上海)股份有限公司合成,引物序列见表 2。
| 基因 Genes |
序列号 Accession No. |
引物序列 Primers sequence (5′→3′) |
| A-asp1 | WP_018623495.1 | F: TCGCAACACTTGAAAGGATAGC R: ACGCAATGACCAGGTGGAA |
| A-asp2 | WP_018623492.1 | F: GGTCTTCCGCAGTTGCTTTC R: AAATCACCCAACCTGTACTGGTACT |
| A-asp3 | WP_018623491.1 | F: GAAGGCATATTGTCATTTACGTCAA R: CGCGCTAATGGCAATCG |
| A-16S rRNA | GI: 197260491 | F: CCAGCAGCCGCGGTAAT R: TTTACGCCCAGTAATTCCGATT |
| G-asp1 | WP_046560315.1 | F: CACCAGCGGCTGATGAAGA R: ACCGTGTCCTGGCTTAAATTTC |
| G-asp2 | WP_052735139.1 | F: ATGATTCGGGCGATCCAA R: TGCGACTGATATACCGCGTATG |
| G-asp3 | WP_046560317.1 | F: TTCATGACAGCGTAGCGAAAA R: GCTCGTTGCGCTAAACCATT |
| G-16S rRNA | GI: 311337312 | F: TGGCGGACGGGTGAGTAA R: GGGCTATCCCCCGCTATTC |
| K-asp1 | WP_012800119.1 | F: GCGCAGTAGCGGCATTG R: ACGGATGTCAGCTGCTGTACAA |
| K-asp2 | WP_012800121.1 | F: TGGCACAGCGTTCGAAGA R: GCTGATGCTCGCATTTGAAG |
| K-asp3 | WP_012800122.1 | F: TTCGTATTGGCAGCAGGTAATG R: TTAGCACGCGCTGGAGAAT |
| K-16S rRNA | GI: 41352514 | F: CCAGCAGCCGCGGTAA R: TTACGCCCAGTAATTCCGATTAA |
| P-asp1 | KY753907 | F: AGTGCGGTAGCTGCATTGG R: CGAATTTCTGCTGCGGTACA |
| P-asp2 | KY753908 | F: TGGTTCGCCGTTGAGCTT R: AGCCTTTTTGGCCACGAATA |
| P-asp3 | KY753909 | F: ACGGCGTGGGTCATTGATA R: TGGCATCAACGCTCAAGTCA |
| P-16S rRNA | GI: 1018196612 | F: CCAGCAGCCGCGGTAAT R: TTTACGCCCAGTAATTCCGATT |
| asp1 | KY753907 | F: ATGWMTTTTAAAKTAAAGATGGCYTGYGC R: CTAGYTWGARCTTCCDTCMAGACTAA |
| asp2 | KY753908 | F: ATGTCATTWAAAGTAAAGATGGCYTG R: TTATAVKTVGAATGAAGCTGCACAAGTTG |
| asp3 | KY753909 | F: ATGAAAAAAACRYTWATMGCYAGYGCTGT R: CTATAGATGGATRATTGGATCTGGATTBC |
| K-phoP | ACV25424.1 | F: ATGAAACTTTTATTAGTCGAA R: TCACAGATCAGCTCTTAAT |
| K-phoQ | ACV26824.1 | F: ATGAGTTTTATTCTACTTAATA R: TTAACCATTGAAACTCACTTTT |
| A-phoP | WP_018624810.1 | F: ATGAAATTATTACTGGTTGAAGAT R: TCACAGGTCCGCTCTTAGTCGA |
| A-phoQ | WP_018624811.1 | F: ATGTCAATTTCACCACGATAAGC R: GTGAGCTTAAATCACTCTTAC |
| G-phoP | AKE52099.1 | F: ATGAAAATACTCTTGATGGAAGA R: CTACTTATCTAAATCACTATTCAA |
| G-phoQ | AKE52098.1 | F: ATGCCAATACCCTGCCCCTCAAC R: GTGCAGGAACAAGCTGAACATAA |
| Note: asp1, asp2, asp3: Alkaline serine protease gene; A: K. aquimarina DSM 16071; G: K. geojedonensis YCS-5; K: K. koreensis DSM 16069; P: K. profundi FT102. | ||
福林酚试剂,北京索莱宝科技有限公司;酪蛋白、DEPC water,生工生物工程(上海)股份有限公司;PCR反应中的ExTaq酶等,TaKaRa公司;Trizol、DNase I、反转录试剂盒、PoweUpTM SYBR® Green Master Mix (AB),赛默飞世尔科技(中国)有限公司;FastDNA Spin Kit for Soil,MP Biomedicals公司。
配制培养基的原料均购自生工生物工程(上海)股份有限公司。
2216E完全培养基参考文献[13]配制。2216-Na盐:不添加NaCl的2216E培养基;2216-Mg盐:不添加MgCl2和MgSO4的2216E培养基;2216-K盐:不添加KCl的2216E培养基;2216-Ca盐:不添加CaCl2的2216E培养基。
1.4 生长曲线测定分别挑取4个菌株的单克隆接种至2216E完全培养基中,37 ℃、220 r/min培养过夜。按照0.1%的比例接入到新鲜的2216E液体培养基(pH 7.0)及4种不添加主要金属盐的培养基中,37 ℃、200 r/min培养,分别在0、6、8、12、16、20、22、26、34 h取样,在600 nm处测定光密度OD600,每个样品3个平行。
1.5 胞外蛋白酶活力检测实验按照1% (质量体积比)接入相应的培养基中,菌株生长到12 h取样,4 ℃、12 000 r/min离心5 min,上清即为蛋白酶粗酶液。采用福林酚法检测胞外蛋白酶活力,以50 mol/L Tris-HCl (pH 8.0)缓冲液配制2%的酪蛋白底物,用Tris-HCl将粗酶液稀释100倍,取1 mL稀释的粗酶液在40 ℃保温1 min,加入1 mL于42 ℃保温的2%酪蛋白。40 ℃反应30 min后,加入2 mL 0.4 mol/L三氯乙酸终止反应。在40 ℃下保温15 min后,12 000 r/min离心15 min。取1 mL上清液加入5 mL的0.4 mol/L Na2CO3,混匀加入1 mL福林酚试剂,40 ℃保温20 min,酶标仪测定OD660值。同时以灭活酶样品作为空白对照,同样按照上述步骤进行。以不同浓度的酪氨酸绘制标准曲线。酶活单位定义为:在40 ℃条件下1 min产生1 μg的酪氨酸为1 U[14]。
1.6 保守的丝氨酸蛋白酶、PhoP和PhoQ编码基因的PCR扩增参照3株Kangiella菌株基因组中3个高度保守的丝氨酸蛋白酶编码基因序列asp1、asp2和asp3设计简并引物,通过PCR扩增K. profundi FT102的基因组中对应的丝氨酸蛋白酶编码基因。PCR反应体系为:10×ExTaq Buffer 2.5 μL,dNTPs (2.5 mmol/L) 2 μL,正反向引物(10 μmol/L)各0.5 μL,ExTaq酶(5 U/μL) 0.125 μL,模板DNA 1 μL,ddH2O补至25 μL。PCR反应条件为:95 ℃ 5 min;95 ℃ 1 min,52 ℃ 45 s,72 ℃ 1 min,28个循环;72 ℃ 10 min;4 ℃保存。纯化PCR产物与pMD19-T载体连接,转化至大肠杆菌DH5α。将阳性克隆菌液送生工生物工程(上海)股份有限公司测序。
1.7 实时荧光定量PCR实验按Trizol试剂盒推荐方法制备合格的RNA样品。RNA反转录采用试剂盒,cDNA反转录条件为25 ℃ 10 min,42 ℃ 60 min,70 ℃ 10 min。反应结束后将cDNA立即置于冰上骤冷5 min,分管保存于−80 ℃。
Real-Time PCR反应体系为20 μL,各组分溶液分别为SYBR Green Master 10 μL,引物混合物(10 μmol/μL) 1.2 μL,ddH2O 7.6 μL,cDNA模板1.2 μL。扩增阶段40个循环(95 ℃ 20 s,95 ℃ 3 s,60 ℃ 30 s)。延伸阶段收集荧光信号,反应结束后对获得的信号进行处理。以16S rRNA基因为参比,2216E培养基中的菌液为对照组,每个样品3个平行。
2 结果与分析 2.1 不同金属盐对4株Kangiella菌株生长的影响测定了4株菌在2216E完全培养基和4种不添加主要金属盐培养基中的生长曲线(图 1),结果显示在培养基中不添加Na盐时,4株Kangiella菌均不能够生长;在不添加Mg盐、Ca盐、K盐的培养基中,K. aquimarina DSM 16071、K. geojedonensis YCS-5和K. profundi FT102这3株菌在稳定期的菌体浓度略低于在2216E培养基中的生长曲线,说明是否添加Mg盐、Ca盐和K盐这3类金属离子对3株菌的生长影响不大。
|
| 图 1 4株Kangiella菌在不同2216E培养基中的生长情况 Figure 1 Growth curve of 4 Kangiella strains in 2216E broth with different metal ions composition 注:A:K. aquimarina DSM 16071;B:K. geojedonensis YCS-5;C:K. koreensis DSM 16069;D:K. profundi FT102. 2216E:2216E完全培养基;2216E-Ca盐:不添加Ca盐的2216E培养基;2216E-Mg盐:不添加Mg盐的2216E培养基;2216E-K盐:不添加K盐的2216E培养基;2216E-Na盐:不添加Na盐的2216E培养基. Note: A: K. aquimarina DSM 16071; B: K. geojedonensis YCS-5; C: K. koreensis DSM 16069; D: K. profundi FT102. 2216E: Complete marine broth; 2216E-Ca salt: Complete 2216E without calcium salt; 2216E-Mg salt: Complete 2216E without magnesium salt; 2216E-K salt: Complete 2216E without potassium salt; 2216E-Na salt: Complete 2216E without sodium salt. |
|
|
对于K. koreensis DSM 16069菌株,在不添加Mg盐、Ca盐、K盐的培养基中菌株的生长明显快于2216E培养基中,在稳定期的菌体量也高于2216E培养基中。观察实验组不同金属离子对K. koreensis DSM 16069菌株生长的影响,在不添加Mg盐的培养基中(去除MgCl2和MgSO4)该菌株的生物量最高,在不添加K盐的培养基中(去除KCl)该菌株的生物量次之,在不添加Ca盐的培养基中(去除CaCl2)该菌株的生物量低于前两者,但仍然高于该菌株在2216E培养基中生长的生物量。结果表明K. koreensis DSM 16069对环境中金属离子的变化较其他3株菌株更加敏感。
2.2 4株菌胞外蛋白酶酶活力测定结果4株Kangiella菌培养12 h后菌液浓度和采用福林酚法检测的胞外蛋白酶酶活如图 2所示。

|
| 图 2 不同培养基中4株Kangiella菌生长情况及胞外蛋白酶酶活(对数期) Figure 2 Effect of different metal ions on the cell growth and extracellular protease activity of 4 Kangiella strains (Logarithmic period) 注:A:K. aquimarina DSM 16071;B:K. geojedonensis YCS-5;C:K. koreensis DSM 16069;D:K. profundi FT102. 1:2216E培养基;2:不添加Mg盐的2216E培养基;3:不添加Ca盐的2216E培养基;4:不添加K盐的2216E培养基. **:统计学差异显著(P < 0.01). Note: A: K. aquimarina DSM 16071; B: K. geojedonensis YCS-5; C: K. koreensis DSM 16069; D: K. profundi FT102. 1: 2216E complete marine broth; 2: Complete 2216E without magnesium salt; 3: Complete 2216E without calcium salt; 4: Complete 2216E without potassium salt. **: Statistically significant (P < 0.01). |
|
|
对于K. aquimarina DSM 16071菌株,在2216E培养基中该菌株的菌体浓度OD600为1.52,胞外蛋白酶活最高,为9.70 U/mL;而在2216E-Mg盐培养基中菌体浓度OD600为1.16,胞外蛋白酶活最低,为4.06 U/mL。对于K. geojedonensis YCS-5菌株,在2216E培养基中菌体浓度OD600为1.139,胞外蛋白酶活最高为6.34 U/mL;在2216E-Mg盐菌体浓度OD600仅为0.389,胞外蛋白酶活最低,为1.04 U/mL。对于K. koreensis DSM 16069菌株,在2216E-Mg盐培养基中菌体浓度OD600最高,达到1.522,此时胞外蛋白酶活为5.89 U/mL;在2216E培养基中菌体浓度反而较低,为0.689,胞外蛋白酶活仅有1.11 U/mL。对于K. profundi FT102菌株,在2216E培养基中菌体浓度最高,为1.53,胞外蛋白酶活则达到10.54 U/mL;而在2216E-Mg盐培养基中菌体浓度最低,为0.85,相对应的胞外蛋白酶活为1.56 U/mL。
从总体上看,4株Kangiella菌胞外酶酶活力的变化趋势与菌体浓度的变化趋势基本一致,说明在2216E培养基中Kangiella属海洋细菌的生物量与其胞外蛋白酶酶活之间存在正相关性。2216E培养基中不添加Mg盐、Ca盐和K盐时,K. aquimarina DSM 16071、K. geojedonensis YCS-5和K. profundi FT102这3株菌的胞外蛋白酶酶活都呈现降低的趋势。仅有K. koreensis DSM 16069菌株在不添加Mg盐、Ca盐和K盐的2216E培养基中,胞外蛋白酶活性反而增加。
2.3 K. profundi FT102菌株中phoP/phoQ基因和3个保守的丝氨酸蛋白酶编码基因的克隆丝氨酸蛋白酶在海洋细菌利用有机氮源生长的过程中起重要的作用,如与Kangiella同属于γ-变形杆菌纲的海洋细菌Pseudoaltermonas sp. SM9913菌株能分泌多种丝氨酸蛋白酶,其中包括MCP-01和MCP-03[14-17]。在已公开的K. aquimarina DSM 16071、K. geojedonensis YCS-5和K. koreensis DSM 16069这3株菌的基因组序列中也发现了多个保守的丝氨酸蛋白酶(序列号见表 2)。
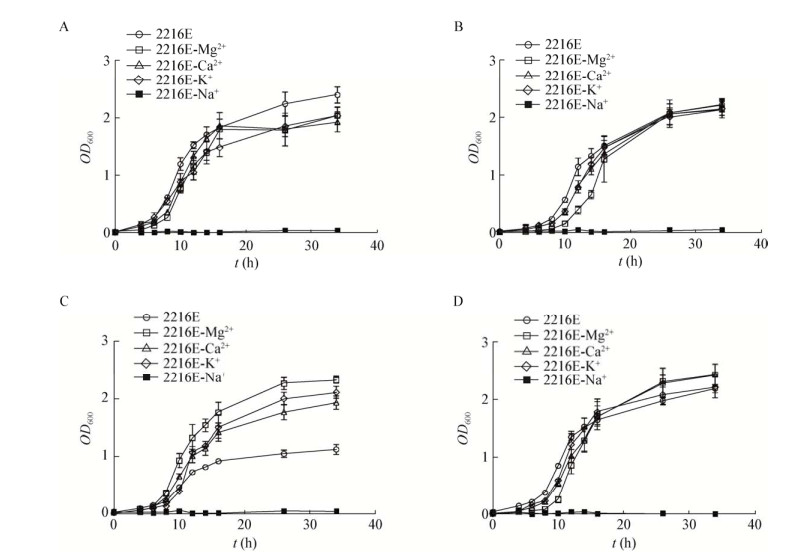
采用简并引物,以K. profundi FT102菌株的基因组DNA为模板,通过PCR扩增得到Kangiella属中保守存在的3个碱性丝氨酸蛋白酶编码基因,如图 3A所示。K. profundi FT102菌株中的P-asp1基因的长度约为2 500 bp,P-asp2基因的长度约为1 800 bp,P-asp3基因的长度约为1 100 bp,DNA测序确定的K. profundi FT102菌株的3个碱性丝氨酸蛋白酶编码基因序列已上传至GenBank (序列号为KY753907、KY753908、KY753909)。
|
| 图 3 K. profundi FT102中基因的PCR扩增 Figure 3 PCR amplification of K. profundi FT102 注:A:3个丝氨酸蛋白酶编码基因;B:phoP和phoQ基因. Note: A: 3 Serine protease genes; B: phoP and phoQ genes. |
|
|
鉴于在不添加Mg盐的2216E培养基中观察到4株菌在生长和胞外蛋白酶酶活上的差异,需进一步考察4株Kangiella菌中响应环境中Mg盐浓度变化的phoP和phoQ的表达情况。根据K. aquimarina DSM 16071菌株的phoP和phoQ基因序列设计引物,通过PCR扩增获得了K. profundi FT102菌株的phoQ基因长度约为1 350 bp,phoP基因长度约为650 bp (图 3B),基因序列已上传到至GenBank (序列号为MF179122、MF179121)。
从上述生理实验中发现菌株K. koreensis DSM 16069与K. aquimarina DSM 16071、K. geojedonensis YCS-5和K. profundi FT102在2216E培养基中不添加Mg盐条件下胞外的蛋白酶酶活上存在显著差异(P < 0.01),因此对Kangiella菌株中3个保守的碱性丝氨酸蛋白酶的氨基酸序列进行比对分析。结果如表 3所示,4株菌中的ASP1蛋白的氨基酸序列相似度高达77%;ASP2蛋白的氨基酸序列相似度大于78%;ASP3蛋白的氨基酸序列相似度为76%以上。
| 菌株 Strains |
ASP1 | ASP2 | ASP3 | ||||||||
| Amino acids | Identity (%) |
Query coverage (%) |
Amino acids | Identity (%) |
Query coverage (%) |
Amino acids | Identity (%) |
Query coverage (%) |
|||
| K | 767 | 611 | 377 | ||||||||
| P | 766 | 90 | 100 | 608 | 94 | 100 | 377 | 94 | 100 | ||
| A | 768 | 90 | 100 | 607 | 93 | 100 | 374 | 76 | 100 | ||
| G | 766 | 77 | 99 | 528 | 78 | 84 | 375 | 76 | 100 | ||
| 注:K:K. koreensis DSM 16069;P:K. profundi FT102;A:K. aquimarina DSM 16071;G:K. geojedonensis YCS-5.以K. koreensis DSM 16069菌株的3个丝氨酸蛋白酶氨基酸序列为参考,分别比对其他3株Kangiella菌株中相关蛋白与参考序列的差异性;E值=0. Note: K: K. koreensis DSM 16069; P: K. profundi FT102; A: K. aquimarina DSM 16071; G: K. geojedonensis YCS-5. Amino acid sequence of 3 conserved serine proteases from K. koreensis DSM 16069 was used as query sequence in BLASTp analysis; E-value=0. |
|||||||||||
采用MEGA系统发育分析软件包(Molecular evolutionary genetics analysis,version 6.06,http://megasoftware.net/)对来源于Kangiella属4个种中的3类保守蛋白酶的氨基酸序列构建进化树,根据NCBI在线BLASTp分析结果绘制这3类蛋白酶的保守结构域示意图。如图 4所示,ASP1、ASP2和ASP3均为丝氨酸蛋白酶家族的S8家族。ASP2和ASP3两类蛋白酶具有S8家族保守结构域;ASP1蛋白酶除了具有S8家族保守结构域外,还有PPC (Pre-peptidase C-terminal domain)结构域。此外,来自K. geojedonensis YCS-5菌株中的ASP1蛋白(WP040560315.1)还具有Peptidase inhibitor I9家族结构域。已有研究表明,海洋细菌分泌的胞外酶类除了具有催化结构域以外,在其C端还具有1−2个PPC结构域或PKD (Polycystic kidney disease I domain)结构域[18],Inhibitor结构域在蛋白前肽中具有分子伴侣的作用,有助于成熟肽的折叠。
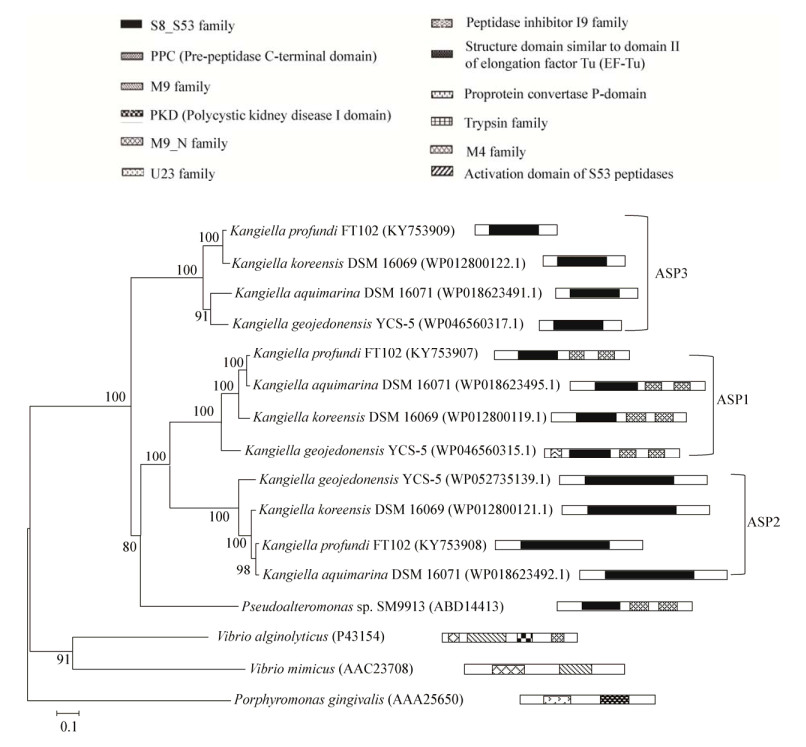
|
| 图 4 Kangiella属中3类蛋白酶中的保守结构域和系统发育树 Figure 4 Phylogenetic tree and conserved domains of the serine proteases in Kangiella strains 注:左侧为采用Neighbor-Joining法构建的进化树,右侧为ASP1、ASP2、ASP3蛋白酶保守结构域示意图;进化树中的数字表示蛋白在NCBI中对应的序列号,括号内表示该条序列所属蛋白家族;进化树采用MEGA 6.06构建,Bootstrap为1 000,分支点上的数字表示Bootstrap超过40%分支,标尺0.1表示碱基替换率为10%;蛋白保守结构域依据BLASTp结果绘制而成,保守结构域包括S8_S53家族、前肽酶C-未端结构域(PPC)、M9家族、多囊肾病Ⅰ域(PKD)、M9_N家族、U32家族、Peptidase inhibitor I9家族、类似于延伸因子Tu结构域Ⅱ的部分(EF-Tu)、Pro蛋白的P结构域、Trypsin家族、M4家族、S53肽酶的活化结构域. Note: Representative consensus Neighbor-Joining tree of the catalytic domain (left part of figure), and schematic diagrams of the domain structure (right part of figure) of ASP1, ASP2 and ASP3; The accession number of each sequence was labelled, the family of protein in brackets; The phylogenetic tree was constructed using MEGA 6.06; One thousand bootstrap replicates were used; Only bootstrap percentages > 40% are shown on the branches, 0.1 means 10% changes were observed between two sequence; The conserved domain architectures of the sequences were identified using BLASTp service available at NCBI (https://www.ncbi.nlm.nih.gov/); The conserved domains are S8_S53 family, PPC (pre-peptidase C-terminal domain), M9 family, PKD (polycystic kidney disease I domain), M9_N family, U32 family, peptidase inhibitor I9 family, structure domain similar to domain Ⅱ of elongation factor Tu (EF-Tu), proprotein convertase P-domain, trypsin family, M4 family, activation domain of S53 peptidase. |
|
|
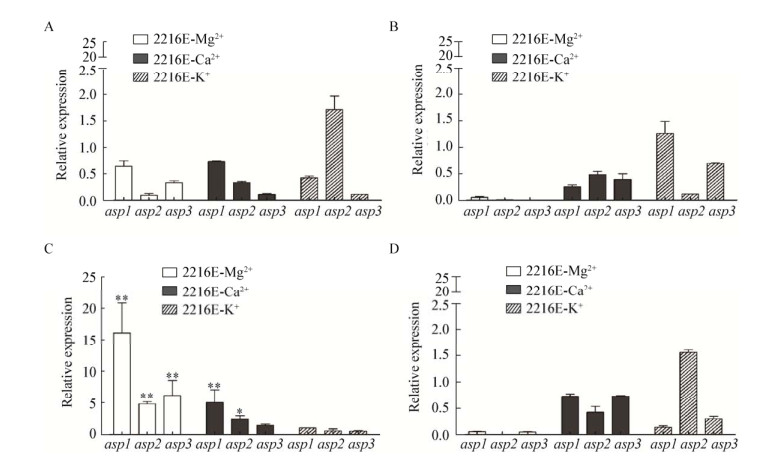
采用Real-Time PCR比较了4株Kangiella菌中3个保守的丝氨酸蛋白酶基因在2216E完全培养基及不添加3种金属盐的2216E培养基中的表达情况,结果如图 5所示。在不添加Mg盐、Ca盐和K盐的2216E培养基中,K. aquimarina DSM 16071、K. geojedonensis YCS-5和K. profundi FT102这3个菌株中的asp1、asp2和asp3基因相对表达量变化值均不超过2倍,与在2216E完全培养基中的表达水平相比无明显变化。
|
| 图 5 不同金属盐对Kangiella菌株中保守丝氨酸蛋白酶基因表达的影响 Figure 5 Differential gene expression of serine protease in Kangiella strains with different metal salts 注:A:K. aquimarina DSM 16071;B:K. geojedonensis YCS-5;C:K. koreensis DSM 16069;D:K. profundi FT102. *:具有显著差异(P < 0.05);**:具有十分显著差异(P < 0.01). Note: A: K. aquimarina DSM 16071; B: K. geojedonensis YCS-5; C: K. koreensis DSM 16069; D: K. profundi FT102. * Indicates statistically significant (P < 0.05); ** Indicates highly statistically significant (P < 0.01). |
|
|
对于K. koreensis DSM 16069菌株,在不添加Ca盐和Mg盐的2216E培养基中,这3个保守的丝氨酸蛋白酶基因的表达都表现为显著上调;而在不添加K盐的培养基中,基因相对表达量变化并不明显。在不添加Ca盐的培养基中,asp1基因相对表达量上调约6倍,asp2基因的相对表达量上调约2.5倍,asp3基因的相对表达量上调约2倍。值得注意的是,在不添加Mg盐的培养基中,asp1基因相对表达量上调约15倍,asp2基因的相对表达量上调约5倍,asp3基因的相对表达量上调约6倍。综上所述,在不添加Ca盐和Mg盐的2216E培养基中,K. koreensis DSM 16069菌株中3个丝氨酸蛋白酶基因表达的增加与其生物量和胞外酶酶活提高的趋势一致。
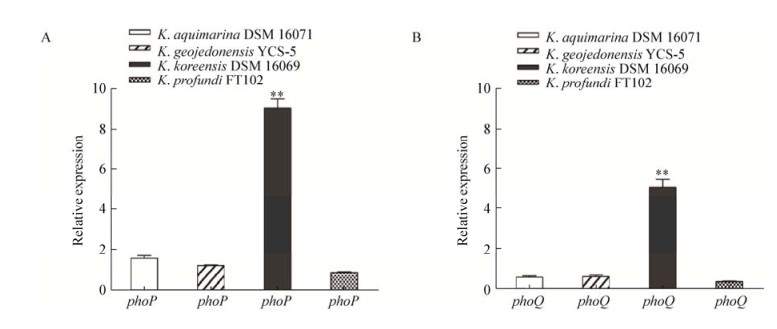
2.5 4株Kangiella菌中phoP和phoQ基因的表达水平细菌中phoP/phoQ操纵子能够对环境中的Mg盐含量变化发生响应[19]。采用Real-Time PCR比较了4株Kangiella菌的phoP和phoQ基因在2216E完全培养基及不添加Mg盐的2216E培养基中的表达情况。结果如图 6所示,在不添加Mg盐的2216E培养基中,K. koreensis DSM 16069菌株的phoP和phoQ基因表达量均显著上调。其中,phoQ基因相对表达量上调约10倍,phoP基因的相对表达量上调约5倍。而K. aquimarina DSM 16071、K. geojedonensis YCS-5、K. profundi FT102菌株中的phoQ和phoP基因在转录水平上的表达量并未发生显著变化。这说明K. koreensis DSM 16069菌株的phoP/phoQ操纵子对培养基中Mg盐浓度的变化更加敏感。
|
| 图 6 不添加Mg盐的2216E培养基对4株Kangiella菌phoP和phoQ基因表达的影响 Figure 6 Relative quantification of phoP and phoQ gene expression about 4 Kangiella strains in 2216E broth without Mg salt 注:A:phoP基因的表达;B:phoQ基因的表达. **:统计学差异显著(P < 0.01). Note: A: phoP gene expression; B: phoQ gene expression. **: Greatly statistically significant (P < 0.01). |
|
|
本实验研究了海水中主要金属离子Na盐、Mg盐、Ca盐、K盐对4株Kangiella属细菌的生长及其丝氨酸蛋白酶基因表达的影响。培养实验显示,Na盐离子为Kangiella属菌株生长所必需的金属元素。通过对该类菌株在不同培养基中的生长曲线测定结果显示,Mg盐对该类菌株生长的影响较其他金属离子更为明显。Mg盐是海水中除Na盐以外含量最多的金属离子,在维持海洋细菌细胞的渗透压平衡方面起着十分重要的作用。有研究表明,Mg盐是海洋细菌细胞内主要的无机阳离子,部分海洋细菌细胞内的Mg盐甚至是海水中Mg盐含量的10−20倍[20]。Mg盐是海洋细菌营养状态的指示物之一,Heldal等发现当环境中含有丰富有机物时,海洋细菌胞内的Mg盐浓度降低,细菌处于代谢活跃的生长状态[21]。
K. koreensis DSM 16069菌株在不添加Mg盐的2216E培养基中的胞外蛋白酶酶活高于其在2216E完全培养基中,说明Mg盐对K. koreensis DSM 16069菌株产酶具有一定的抑制作用。山东大学边斐等对分离自海洋环境的Pseudoalteromonas sp. SM9913菌株胞外酶酶活的研究结果也显示,高浓度的Mg盐对Pseudoalteromonas sp. SM9913胞外酶酶活也具有一定的抑制作用[22]。
Véscovi等报道Salmonella typhimurium中的phoP/phoQ操纵子受环境中Mg盐浓度的调控[19]。对Escherichia coli和Salmonella enterica中phoP/phoQ操纵子的调控机制研究表明,膜上的PhoQ蛋白可感知Mg盐浓度[23]。在胞外Mg盐浓度较低的情况下,磷酸化的PhoQ蛋白可以激活胞内转录调控因子PhoP,从而激活Mg盐转运蛋白以及毒力相关的多个基因表达[24]。此外,在肠杆菌科细菌中广泛存在的phoP/phoQ操纵子还可以对自身的表达进行调控,从而维持胞内Mg盐的稳态[25]。
本实验Real-Time PCR检测结果显示,在不添加Mg盐的培养基中,K. koreensis DSM 16069菌株的phoP、phoQ、asp1、asp2和asp3等基因的转录水平明显上调,而其他3株Kangiella菌的对应基因并未发生显著变化。结果说明,培养基中较低浓度的Mg盐可被K. koreensis DSM 16069菌株膜上的PhoQ感知,并通过经典的PhoQ/PhoP信号通路激活包括丝氨酸蛋白酶在内的众多基因的表达,进而使得K. koreensis DSM 16069菌株在不添加Mg盐的培养基中生长情况优于2216E完全培养基中。而对于其他3株Kangiella菌株中,phoP/phoQ操纵子对Mg盐浓度变化响应的调控机制还有待深入探究。
| [1] |
Yoon JH, Oh TK, Park YH. Kangiella koreensis gen. nov. sp. nov. and Kangiella aquimarina sp. nov. isolated from a tidal flat of the Yellow Sea in Korea[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1829-1835. DOI:10.1099/ijs.0.63156-0 |
| [2] |
Romanenko LA, Tanaka N, Frolova GM, et al. Kangiella japonica sp. nov. isolated from a marine environment[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(11): 2583-2586. DOI:10.1099/ijs.0.017087-0 |
| [3] |
Ahn J, Park JW, Mcconnell JA, et al. Kangiella spongicola sp. nov. a halophilic marine bacterium isolated from the sponge Chondrilla nucula[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(4): 961-964. DOI:10.1099/ijs.0.021733-0 |
| [4] |
Yoon JH, Kang SJ, Lee SY, et al. Kangiella geojedonensis sp. nov. isolated from seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(3): 511-514. |
| [5] |
Jean WD, Huang SP, Chen JS, et al. Kangiella taiwanensis sp. nov. and Kangiella marina sp. nov. marine bacteria isolated from shallow coastal water[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(9): 2229-2234. |
| [6] |
Lee SY, Park S, Oh TK, et al. Kangiella sediminilitoris sp. nov. isolated from a tidal flat sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(3): 1001-1006. |
| [7] |
Xu FD, Li XG, Xiao X, et al. Kangiella profundi sp. nov. isolated from deep-sea sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(7): 2315-2319. DOI:10.1099/ijs.0.000257 |
| [8] |
Kim JH, Ward AC, Kim W. Kangiella chungangensis sp. nov. isolated from a marine sand[J]. Antonie Van Leeuwenhoek, 2015, 107(5): 1291-1298. DOI:10.1007/s10482-015-0423-5 |
| [9] |
Wang G, Tang M, Wu H, et al. Aliikangiella marina gen. nov. sp. nov. a marine bacterium from the culture broth of Picochlorum sp. 122 and proposal of Kangiellaceae fam. nov. in the order Oceanospirillales[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(12): 4488-4494. DOI:10.1099/ijsem.0.000601 |
| [10] |
Hu YOO, Karlson B, Charvet S, et al. Diversity of pico-to mesoplankton along the 2000 km salinity gradient of the Baltic Sea[J]. Frontiers in Microbiology, 2016, 7: 679. |
| [11] |
Szefer P, Kim BS, Kim CK, et al. Distribution and coassociations of trace elements in soft tissue and byssus of Mytilus galloprovincialis relative to the surrounding seawater and suspended matter of the southern part of the Korean Peninsula[J]. Environmental Pollution, 2004, 129(2): 209-228. DOI:10.1016/j.envpol.2003.10.012 |
| [12] |
Donohue KA, Toole JM. A near-synoptic survey of the Southwest Indian Ocean[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2003, 50(12/13): 1893-1931. |
| [13] |
Oppenheimer CH. The growth and viability of sixty-three species of marine bacteria as influenced by hydrostatic pressure[J]. Journal of Marine Research, 1952, 11: 10-18. |
| [14] |
Xu GY. Characterization, cloning and expression of a cold-active protease from psychrophilic bacterium Pseudoalteromonas sp. QI-1[D]. Jinan: Master's Thesis of Shandong University, 2010 (in Chinese) 徐国英. 极地嗜冷菌Pseudoalteromonas sp. QI-1产适冷蛋白酶及其基因克隆与表达的研究[D]. 济南: 山东大学硕士学位论文, 2010 http://cdmd.cnki.com.cn/Article/CDMD-10422-2010103105.htm |
| [15] |
Zhao GY, Chen XL, Zhao HL, et al. Hydrolysis of insoluble collagen by deseasin MCP-01 from deep-sea Pseudoalteromonas sp. SM9913: collagenolytic characters, collagen-binding ability of C-terminal polycystic kidney disease domain, and implication for its novel role in deep-sea sedimentary particulate organic nitrogen degradation[J]. Journal of Biological Chemistry, 2008, 283(52): 36100-36107. |
| [16] |
Qiang YB, Lan CX, Hou XY, et al. Molecular analysis of the gene encoding a cold-adapted halophilic subtilase from deep-sea psychrotolerant bacterium Pseudoalteromonas sp. SM9913: cloning, expression, characterization and function analysis of the C-terminal PPC domains[J]. Extremophiles, 2009, 13(4): 725-733. DOI:10.1007/s00792-009-0263-1 |
| [17] |
Chen XL, Zhang YZ, Gao PJ, et al. Two different proteases produced by a deep-sea psychrotrophic bacterial strain, Pseudoaltermonas sp. SM9913[J]. Marine Biology, 2003, 143(5): 989-993. |
| [18] |
Guo J. Multiple mature forms of metalloprotease E495 in Thermolysin family secreted by sea ice bacteria Pseudoalteromonas sp. SM495 and function of its C-terminal PPC domain[D]. Jinan: Master's Thesis of Shandong University, 2011 (in Chinese) 郭君. 海冰细菌Pseudoalteromonas sp. SM495分泌的Thermolysin家族金属蛋白酶E495的多种成熟形式及C端PPC结构域的功能研究[D]. 济南: 山东大学硕士学位论文, 2011 http://cdmd.cnki.com.cn/Article/CDMD-10422-1011224067.htm |
| [19] |
Véscovi EG, Soncini FC, Groisman EA. Mg2+ as an extracellular signal: environmental regulation of Salmonella virulence[J]. Cell, 1996, 84(1): 165-174. DOI:10.1016/S0092-8674(00)81003-X |
| [20] |
Fagerbakke KM, Norland S, Heldal M. The inorganic ion content of native aquatic bacteria[J]. Canadian Journal of Microbiology, 1999, 45(4): 304-311. |
| [21] |
Heldal M, Norland S, Erichsen ES. Mg2+ as an indicator of nutritional status in marine bacteria[J]. ISME Journal, 2012, 6(3): 524-530. |
| [22] |
Bian F, He HL, Chen XL, et al. The effects of ions on the secretion of extracellular proteases produced by cold-adapted deep-sea bacterium Pseudoalteromonas sp. SM9913[J]. Journal of Shandong University (Natural Science), 2006, 41(5): 166-172. 边斐, 何海伦, 陈秀兰, 等. 离子对深海适冷菌Pseudoalteromonas sp. SM9913胞外蛋白酶分泌的影响[J]. 山东大学学报:理学版, 2006, 41(5): 166-172. |
| [23] |
Zwir I, Shin D, Kato A, et al. Dissecting the PhoP regulatory network of Escherichia coli and Salmonella enterica[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(8): 2862-2867. |
| [24] |
Kato A, Groisman EA. The PhoQ/PhoP regulatory network of Salmonella enterica[A]//Utsumi R. Bacterial Signal Transduction: Networks and Drug Targets[M]. New York: Springer, 2008: 7-21
|
| [25] |
Perez JC, Shin D, Zwir I, et al. Evolution of a bacterial regulon controlling virulence and Mg2+ homeostasis[J]. PLoS Genetics, 2009, 5(3): e1000428. |
 2018, Vol. 45
2018, Vol. 45