扩展功能
文章信息
- 张昆, 陈景超, 李祎, 刘磊, 王海磊
- ZHANG Kun, CHEN Jing-Chao, LI Yi, LIU Lei, WANG Hai-Lei
- CRISPR/Cas9技术在微生物研究中的应用进展
- Application progress of CRISPR/Cas9 technology in microbiological research
- 微生物学通报, 2018, 45(2): 451-464
- Microbiology China, 2018, 45(2): 451-464
- DOI: 10.13344/j.microbiol.china.170315
-
文章历史
- 收稿日期: 2017-04-21
- 接受日期: 2017-07-07
- 网络首发日期(www.cnki.net): 2017-08-08
基因组编辑(Genome editing)技术是研究基因功能的重要工具,一直是现代分子生物学的研究热点。由于同源重组的基因打靶效率较低,因此它的应用受到极大的限制。直到人工核酸内切酶的出现,这一现状才得到较大的改变。近15年来,基于人工核酸内切酶的基因组编辑技术经历了3次大的改善,分别是锌指核酸酶(Zinc-finger nucleases,ZFNs)、转录激活因子样效应物核酸酶(Transcription activator-like effector nucleases, TALENs)和CRISPR/Cas系统,这3种技术的共同点是都需要一个DNA识别结构域和一个DNA切割结构域[1]。然而,由于ZFNs和TALENs这两项技术难度较大、组装时间较长、费用较高,因此应用范围有限。2013年,一种新型的基因组编辑技术—CRISPR/Cas系统的出现,迅速受到全世界科研人员的关注。CRISPR/Cas系统广泛分布在细菌和古生菌的基因组中,是进化过程中形成的一种可以降解入侵病毒或质粒DNA的适应性免疫系统。CRISPR/Cas系统作为一种高效的基因组编辑技术,具有制备简单、靶向性强、成本低和脱靶率低等诸多优势,并在多个动植物以及微生物物种中成功应用,极大地促进了基因功能、代谢工程、转基因生物和基因治疗等方面的研究。本文将从Ⅱ型CRISPR/Cas系统的历史、基本结构、分类、作用原理、在微生物研究中的应用及其可能存在的问题等方面的研究进展进行介绍。
1 CRISPR/Cas系统的发现历史CRISPR/Cas介导的基因组编辑技术与现代分子生物学和生物信息学的发展密切相关。1987年,日本研究者Ishino等[2]发现大肠杆菌(Escherichia coli) K12菌株碱性磷酸酶基因(iap)编码区附近存在一段由长度为29 bp的重复片段(Repeats)和32 bp的非重复片段(Spacers)间隔组成的特殊序列。随后的研究发现,在已测序的大约40%细菌和90%古细菌中都能够观察到这种重复序列。Mojica等[3]和Jansen等[4-5]将这些间隔排列的串联重复序列命名为CRISPR (Clustered regularly interspaced short palindromic repeats),即成簇规律间隔的短回文重复序列。2005年,美国的3个研究小组相继发现CRISPR的间隔序列来源于外源的噬菌体或质粒的DNA序列,同时Cas9蛋白和共有序列即PAM (Protospacer adjacent motif,PAM)也被揭示出来[6-8],推测CRISPR/Cas系统可能与细菌抵抗外源遗传物质入侵的免疫保护机制有关。2007年,Barrangou等[9]发现嗜热链球菌(Streptococcus thermophiles)可利用CRISPR/Cas系统用于对噬菌体的免疫抵抗,并且免疫特异性取决于间隔序列。2008年,Marraffini等[10]发现细菌的CRISPR/Cas系统能直接作用于靶DNA,而非RNA来限制外源基因的水平转移,首次利用实验证明了CRISPR系统的功能。随后Brouns等[11]发现CRISPR的间隔区(Spacer)可被转录成CRISPR RNA (crRNA),这些crRNA和Cas蛋白一起可干扰噬菌体增殖。2010年,Garneau等[12]发现CRISPR-1/Cas系统可以在靶DNA的PAM区上游3个碱基处切割造成双链断裂。2011年,Deltcheva等[13]报道一种名为反式激活RNA分子即tracrRNA (Trans-activating CRISPR RNA),与内源性的RNase Ⅲ和Cas9蛋白共同作用,负责生成crRNA,同时推测tracrRNA和crRNA可以形成双链复合物指导Cas9降解外源DNA。Sapranauskas等[14]证明嗜热链球菌的CRISPR/Cas系统在大肠杆菌表达可避免噬菌体感染和降低质粒转化的风险。2012年,Gasiunas等[15]证明Cas9蛋白的HNN结构域和RuvC结构域分别负责切割靶DNA的互补链和非互补链,并且切割位点取决于crRNA序列。Jinek等[16]对Ⅱ型CRISPR/Cas9系统进行改造,将tracrRNA:crRNA双链复合体简化为一条嵌合的单链向导RNA。至此,Ⅱ型CRISPR/Cas系统的作用机制基本清晰,为进一步应用奠定基础。
2013年,Cong等[17]和Mali等[18]率先将CRISPR/Cas9系统应用在人类和小鼠胚胎干细胞中,不仅基因敲除效率显著提高,而且还可以实现多基因的敲除。目前该技术已成功应用到秀丽线虫(Caenorhabditis elegans)[19]、斑马鱼(Zebrafish)[20]、拟南芥(Arabidopsis thaliana)[21]、高粱(Sorghum bicolor)[21]、烟草(Nicotiana tabacum)[21]、水稻(Oryza sativa)[21]、大肠杆菌[22]和酿酒酵母(Saccharomyces cerevisiae)[23]等多种动植物和微生物中。
2 CRISPR/Cas系统的结构和作用原理CRISPR/Cas系统主要由CRISPR和CRISPR相关蛋白基因组成[24]。CRISPR由多个高度保守的重复序列和间隔序列相互串联而成,一个CRISPR位点的重复序列具有结构保守性,不同CRISPR位点重复序列的序列和结构均不相同。重复片段的序列存在二重对称性,可以形成发夹结构,这些重复序列的具体功能迄今为止尚不清楚。CRISPR的间隔序列与一些噬菌体或质粒的序列存在同源性,很可能来源于某些入侵的噬菌体或质粒。在CRISPR位点第一个重复序列的上游是前导序列(Leader sequence),前导序列可以作为启动子,负责CRISPR序列的转录,产生crRNA[24-25]。在CRISPR位点附近是CRISPR相关蛋白基因cas。cas基因是一类较大的多态性家族,编码的蛋白具有核酸酶切割活性,能将外源DNA切断,是CRISPR/Cas免疫防御途径中的重要组成成分。
CRISPR/Cas介导的免疫过程主要包括3个阶段:适应、表达和干扰[24-26]。在适应阶段,当噬菌体或者质粒DNA进入携带有CRISPR系统的细胞时,宿主的Cas1核酸结合蛋白会迅速地介导CRISPR相关蛋白质复合体与噬菌体DNA结合,通过Cas2核酸内切酶等蛋白的作用,将该DNA切割成17−84 bp不等的核酸小片段,然后在相关蛋白的作用下,Cas蛋白复合物将这些短片段DNA插入到宿主CRISPR位点的前导序列与第一段重复序列之间,形成一个新的重复序列—间隔序列单元[9]。在表达阶段,这些掺入的间隔序列被转录成crRNA前体即pre-crRNA,在Cas蛋白复合体的参与下加工成短的包括重复序列和间隔序列的成熟crRNA,这些crRNA与tracrRNA结合形成tracrRNA/crRNA复合物。在干扰阶段,当噬菌体等外源DNA再次侵染细菌时,tracrRNA/crRNA复合物通过碱基配对寻找同源的靶标核酸,然后引导Cas蛋白对与crRNA配对的外源DNA进行切割,产生一个双链DNA缺口,导致入侵DNA降解[13]。2016年2月,美国科学家Silas等[27]发现一种新的CRISPR间隔序列获取方式,在海洋细菌Marinomonas mediterranea (MMB-1)中,反转录酶RT (Reverse-transcriptase)/Cas1复合体可以将外源RNA直接插入到CRISPR位点内的DNA重复序列之间,形成新的间隔序列,这种类型的CRISPR/Cas系统与先前发现的将DNA作为间隔序列明显不同,有利于细菌防御RNA病毒。
目前应用最为成功的是Ⅱ型CRISPR/Cas9系统,最初的Cas9蛋白来源于化脓性链球菌(Streptococcus pneumoniae),其结构和功能研究已比较透彻,由1 409个氨基酸组成,含有2个核酸酶结构域:氨基端的RuvC结构域和位于中间的HNH结构域。在噬菌体或者质粒上同间隔序列对应的序列通常称为原型间隔序列(Protospacer),一般情况下Protospacer的5′或3′端有几个延伸的比较保守的碱基序列,称为PAM,Ⅱ型CRISPR/Cas系统的PAM一般为NGG。HNH结构域可以切割与crRNA互补配对的DNA链,切割位点位于PAM上游3 bp处,RuvC结构域切割与crRNA非互补的DNA链,切割位点位于PAM上游3−8 bp处。当Cas9剪切基因组时,可以在PAM序列前产生一个DNA双链断裂缺口(Double-strand breaks,DSBs),由于细胞具有修复功能,当细胞内存在靶位点的同源序列模板时,细胞就会进行同源修复(Homology-directed repair,HDR),这种方式的准确率较高;如果没有同源序列模板存在,细胞就会通过非同源末端连接(Nonhomologous end-joining,NHEJ)的方式使基因组发生随机的INDEL (Insertion and deletion)效应,由于NHEJ只是将断裂的DNA片段简单加工之后连接起来,因此这种方式的准确率较低,容易造成基因突变[28]。
由于细菌CRISPR基因位点内的间隔序列周围不存在PAM,而外源DNA则存在相应的PAM,因此CRISPR/Cas系统能够区分自身DNA和外源DNA。除此之外,对于不需要PAM的CRISPR/Cas系统而言,crRNA不仅含有间隔序列,而且在其5′端和3′端包含有部分重复序列,对于宿主菌来说,crRNA除了能与间隔区配对外,两端的重复序列也能与间隔序列两侧的基因组DNA完全配对。当外源DNA入侵宿主菌时,crRNA可以和外源DNA序列的Protospacer配对,但重复序列却不能配对,一旦发生不完全配对,核糖核蛋白复合体就会发挥切割活性,通过这样的机制,CRISPR/Cas系统能够保护宿主和避免自我免疫[29]。
3 CRISPR/Cas系统的分类随着生物信息学的发展和测序微生物的不断增多,绝大多数细菌和40%古细菌中均已发现CRISPR/Cas系统。2005年,Haft等[30]将几十个细菌的CRISPR/Cas系统划分为8个亚型。2011年,Makarova等[31]根据Cas基因核心元件序列的不同,将CRISPR/Cas系统划分为Ⅰ、Ⅱ和Ⅲ型,其核心蛋白分别是Cas3、Cas9和Cas10。这3种类型还可以进一步划分为10个亚类。Ⅰ型和Ⅲ型CRISPR/Cas系统需要多个Cas蛋白形成复合体来切割DNA双链,而Ⅱ型CRISPR/Cas系统只需要一个Cas9蛋白就可以发挥作用。值得注意的是,Ⅲ型CRISPR/Cas系统的两种亚型Ⅲ-A和Ⅲ-B的靶序列分别是DNA和RNA,它们在切割时不需要PAM。
2015年,Makarova等[32]根据CRISPR/Cas系统中CRISPR核酸酶复合体蛋白成分的不同提出2类-5型-16亚类的分类系统。其中属于第1类的核酸酶复合体由多个Cas蛋白组成,而第2类的核酸酶复合体具有单个Cas蛋白。属于第1类CRISPR/Cas系统的有Ⅰ、Ⅲ和Ⅳ型,属于第2类CRISPR/Cas系统的有Ⅱ、Ⅴ和Ⅵ型。其中Ⅳ型缺乏CRISPR系统中广泛存在的Cas1、Cas2基因和内在的CRISPR序列,因此它的作用机制尚不清楚。Ⅴ型的3个亚类Ⅴ-A、Ⅴ-B和Ⅴ-C分别具有典型的Cpf1、C2c1和C2c3蛋白,这些蛋白单独存在就可以发挥切割活性。Cpf1蛋白切割DNA时不需要tracrRNA,并且识别位点和剪切位点相距较远,产生粘性末端缺口;相比之下,Cas9蛋白需要tracrRNA才能激活,识别位点和切割位点相距较近,可产生平末端缺口。Cpf1的PAM一般位于原型间隔序列的5′端,而Cas9蛋白的PAM区则位于间隔序列的3′端。此外,Cpf1酶比标准的SpCas9要小,更容易进入组织和细胞,因此表现出良好的应用前景。C2c1蛋白需要crRNA和tracrRNA才能发挥作用,但是其PAM位于原型间隔序列的5′端。C2c3蛋白功能不详,只存在于宏基因组中。
2016年6月,Abudayyeh等[33]报告了一种新型CRISPR/Cas系统即Ⅵ-A型CRISPR/C2c2,该系统利用RNA引导酶Cas13a (先前被称为C2c2)来靶向和降解RNA,Cas13a不需要tracrRNA和PAM的向导作用,但是需要一个位于原型间隔序列3′端的侧翼位点(Protospacer flanking site,PFS)。C2c2引起的断裂位点并不固定,受靶位点单链RNA二级结构和序列组成的影响。2017年2月,Smargon等[34]利用生物信息学数据挖掘的方法,发现了两种新的Ⅵ-B型CRISPR/Cas系统,这类系统缺乏Cas1和Cas2,只利用单个的Cas酶(Cas13b)即可发挥活性。与Cas13a相同,Cas13b仅需要单个向导RNA就能找到靶序列并引起ssRNA的断裂,并且能够同时靶向多个RNA转录本,因此该型CRISPR/Cas系统可以像RNAi一样用于降低基因的表达水平。
4 CRISPR/Cas系统在微生物中的应用进展 4.1 CRISPR/Cas系统在微生物中介导的基因组编辑CRISPR/Cas系统在微生物和在真核生物中的应用几乎同时进行,2013年,Jiang等[22]利用CRISPR/Cas9系统对S. pneumoniae和E. coli进行基因组编辑。但此后,该系统并没有在微生物中广泛应用,究其原因是:(1)一些常见的细菌已经存在许多高效和便利的基因组编辑方法,缺乏发展新技术的动力;(2)多数细菌缺少NHEJ的修复途径,必需依靠DNA模板介导的HDR途径才能完成修复,否则Cas9蛋白引起的DSBs很容易引起细胞死亡,这种需要模板的要求也限制了CRISPR/Cas系统的应用。对于需要基因组编辑的微生物而言,存在两种策略:(1)利用外源的CRISPR/Cas系统,CRISPR/Cas9系统经过Jinek等改造后非常简单,用一个四碱基的连接环将crRNA和tracrRNA连接在一起构成一个具有向导作用的sgRNA (Single guide RNA)。Cas9蛋白可识别sgRNA,从而定点切割DNA[16];(2)利用自身CRISPR/Cas系统,如果细胞内在的CRISPR/Cas系统比较活跃,构建可表达一个或者多个crRNAs的pre-RNA表达系统,转化细胞后也可以用于基因组编辑。
CRISPR/Cas9系统包括Cas9蛋白和sgRNA两部分。目前使用的Cas9蛋白主要来源于S. pyogenes和S. thermophiles。由于tracrRNA和Cas9蛋白的序列相对固定,因此只需要改变crRNA序列就可以改变靶基因,再添加一个修复模板就可以用于基因组编辑。目前CRISPR/Cas9技术的基因组编辑应用策略如图 1所示,Cas9蛋白和sgRNA的表达框可以构建在1个载体上,有利于该系统的协同表达;也可以构建在2个载体上,含有Cas9基因的质粒既可以和sgRNA质粒同步转化,也可以先转化细胞,然后再转化sgRNA质粒,能更快速地检测候选sgRNA的打靶效率和脱靶情况;还可以利用体外转录的方法获得sgRNA,然后和Cas9质粒共转化,适合缺少诱导型启动子的微生物(图 1A、B、C)。对于多基因位点的编辑,目前存在两种方法:(1)在重组质粒中将多个sgRNA的DNA序列进行串联,由于多数细菌存在内源性的RNase Ⅲ,因此转录出来的crRNA前体pre-crRNA可以被加工成多个sgRNA;(2)体外转录出多个sgRNA形成一个混合池,然后和含有Cas9的质粒一起转化。需要注意的是,打靶序列必需包含PAM序列,这是由Ⅱ型CRISPR/Cas9系统依赖PAM识别外源DNA序列的特点所决定的。目前该技术已经成功运用在E. coli[22, 35-37]、枯草芽孢杆菌(Bacillus subtilis)[38-39]、丙酮丁醇梭菌(Clostridium acetobutylicum)[40]、拜氏梭菌(Clostridium beijerinckii)[40]、罗伊氏乳杆菌(Lactobacillus reuteri)[41]、蓝细菌聚球藻(Synechococcus elongatus)[42]、肺炎链球菌(Streptococcus pneumoniae)[22]、柠檬塔特姆菌(Tatumella citrea)[35]、运动发酵单胞菌(Zymomonas mobilis)[43]等(表 1)。2013年,DiCarlo等[23]首次将CRISPR/Cas9技术应用于真核微生物酿酒酵母中,敲除了细胞膜精氨酸透性酶CAN1基因和磷酸核糖酰氨基眯唑羧化酶基因ade2-101。随后,不少研究者将CRISPR/Cas9应用到丝状真菌,例如,2015年,中国科学院Liu等[44]将该技术应用到里氏木霉(Trichoderma reesei)。丹麦科学家Nødvig等[45]将该技术应用到包含构巢曲霉(Aspergillus nidulans)在内的6种丝状真菌。中国科学院Liu等[46]将该技术应用到产纤维素酶的真菌Myceliophthora thermophila和Myceliophthora heterothallica。2017年,Nayak和Metcalf首次报道在古生菌乙酸甲烷八叠球菌(Methanosarcina acetivorans)中建立CRISPR/Cas9介导的基因组编辑方法[47]。这些应用证明CRISPR/Cas9有效解决了非模式微生物难以进行基因组编辑的问题,而且为阐明微生物的基因功能、解析代谢途径提供一种有用的工具,为细菌、古生菌和真菌功能基因的挖掘、菌株改良、重要基因定点突变以及创制新的种质资源带来突破性进展。
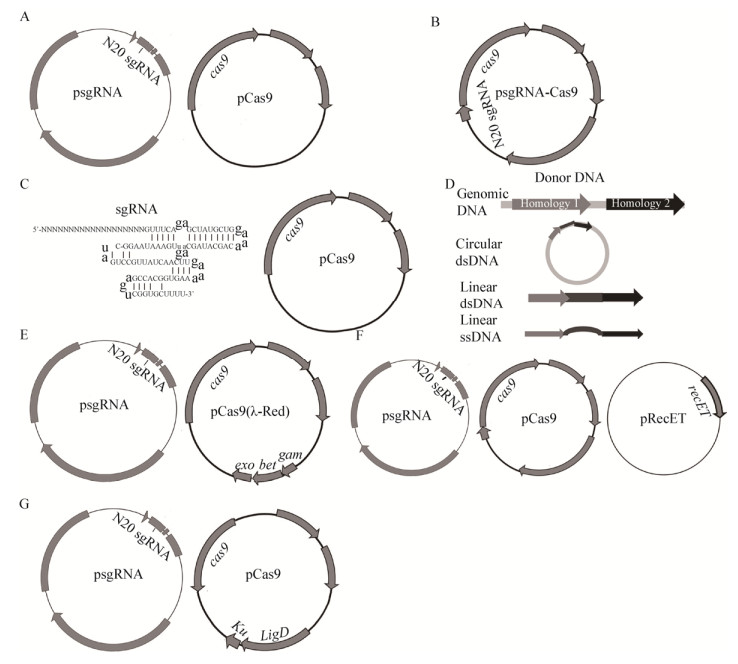
|
| 图 1 CRISPR/Cas9介导的基因组编辑策略 Figure 1 CRISPR/Cas9-mediated genome editing strategies 注:A、B:sgRNA、Cas9表达框在不同质粒和同一质粒;C:体外转录形成sgRNA和Cas9表达质粒;D:Donor的形式;E、F和G:CRISPR/Cas9系统分别和λ-Red、RecET和NHEJ系统联合使用. Note:A, B: sgRNA and Cas9 expression cassettes were constructed in two plasmids or one plasmid; C: sgRNA obtained by in vitro transcription and Cas9 expression plasmid; D: Different forms of donor; E, F and G: CRISPR/Cas9 technology was used in combination with λ-Red, RecET and NHEJ system, respectively. |
|
|
| 物种 Species |
靶基因的编辑效率 Editing efficiency of targeted genes |
修复途径 Repair pathway |
参考文献 References |
| Escherichia coli | rpsL: 65% | HDR (λ-Red) | [22] |
| cadA: 86%; maeA+maeB: 97%; cadA+maeA+maeB: 47%; LacZ: 100%; LacZ+galK: 83%; LacZ+galK+ldhA: 23% ppc/FadD: NC | [35] [36] [37] |
||
| Bacillus subtilis | srfC/spoIIAC/nprE/aprE/amyE: 33%−53%; amyE: 90%; yvmC: 97% | HDR | [38] |
| [39] | |||
| Clostridium acetobutylicum | pyrE: 30%; adc: 6.7%; agrA: 100% | HDR | [40] |
| Clostridium beijerinckii | adc/xlyR/araR/cbei3923/cbei4495/xylR: 18.8%−100% | ||
| Lactobacillus reuteri | lacL/ctrl/srtA/dp6: 90%−100% | HDR (RecT) | [41] |
| Synechococcus elongatus | nblA: 100% | HDR | [42] |
| Streptococcus pneumoniae | srtA/bgaA: 75%−100% | HDR | [22] |
| Tatumella citrea | trkA/glk: 94%−100% | HDR (λ-Red) | [35] |
| Zymomonas mobilis | rep: 5% | NHEJ | [43] |
| Saccharomyces cerevisiae | CAN1/ade2-101: 80%−100% | HDR | [23] |
| Trichoderma reesei | ura5: 100%; lae1+vib1+clr2: 4.2% | HDR | [44] |
| Aspergillus nidulans | yA: NC | NHEJ | [45] |
| Aspergillus aculeatus | albA/pyrG: NC | ||
| Myceliophthora thermophila | amdS: NC | NHEJ | [46] |
| cre-1: 95%; cre-1+res-1+gh1-1: 30%; cre-1+res-1+gh1-1+alp-1: 22% | HDR | ||
| Myceliophthora heterothallica | cre-1: 90% | HDR | |
| Methanosarcina acetivorans | ssuC/mtmCB1/mtmCB2/mcrA/hdrED: NC | HDR | [47] |
| 注:NC:没有计算编辑效率;+:多个基因同时敲除;/:单个基因敲除. Note: NC: The editing efficiency was not calculated; +: Multiple genes were simultaneously deleted; /: Single gene was deleted. |
|||
Cas9蛋白在切割靶位点后产生的DSBs具有致死效应,细胞必需修复才能存活。由于多数细菌细胞内不存在NHEJ的修复途径,因此DSBs必需依靠供体DNA (Donor)或者内源DNA才能完成HDR修复。用于修复DSBs的模板可以是环状dsDNA (质粒)、线性dsDNA或者线性ssDNA (图 1D)。由于同源重组的效率较低,并且在非分裂细胞中这种修复途径的效率会进一步降低,因此CRISPR/Cas9系统介导的编辑效率仍有改进的空间。将CRISPR/Cas9技术和噬菌体重组工程λ-Red (Exo、Beta和Gam蛋白)、RecET (RecE和RecT蛋白)或者NHEJ途径(Ku和LigD蛋白)的异源表达相结合可进一步提高编辑效率,同时还可以实现多基因和连续的基因组编辑(图 1E、F、G)。例如,天津大学的科研人员将λ-Red系统、Cas9蛋白表达系统和质粒消除系统整合在一个质粒pREDCas中,然后与sgRNA表达质粒、供体DNA一起用于大肠杆菌的基因组工程和代谢工程,单基因缺失的编辑效率接近100%,而且还可以一次打靶3个基因位点。在突变体筛选之后,经过诱导质粒消除系统还可以将sgRNA表达质粒消除掉,这样又可以进行下一轮的基因组编辑,因此十分适合需要改造多个基因的代谢工程研究[36]。中国科学院上海生命科学研究院的科研人员利用同样的策略,证明λ-Red系统和CRISPR/Cas9结合可以显著提高靶基因的编辑效率[35]。山东大学的科研人员在大肠杆菌中表达来自于结核分枝杆菌(Mycobacterium tuberculosis)的Ku蛋白和LigD蛋白,以重建一个简单的NHEJ途径,结果显示不仅编辑效率显著提高,而且不需要修复模板和筛选标记[48]。此外,CRISPR/Cas9技术在缺失基因的同时也可以通过修复模板引入新的基因和代谢途径,Ronda等[49]将含有3个sgRNA串联的载体和参与β-胡萝卜素合成的3个基因(BTS1、crtYB和crtI)的供体DNA一起转化可表达Cas9蛋白的酵母工程菌,最终使酵母能够合成β-胡萝卜素。
为了避免NHEJ途径引起的INDEL效应,哈佛大学Komor等[50]开发了一种新的CRISPR/Cas9技术,主要包括dCas9或Cas9切口酶以及胞嘧啶脱氨酶,可以对单个碱基进行编辑。胞嘧啶脱氨酶能将胞嘧啶转化为尿嘧啶,通过DNA复制或修复,尿嘧啶被转化成胸腺嘧啶,从而将最初的CAA转变为TAA,相当于引入一个终止密码子。这种技术无需切割DNA,因此不会引起意外的突变,可对靶基因精确修饰。这种酶最初在人类细胞中应用,目前已经扩展到微生物中。武汉大学Zeng等[51]将CRISPR/Cas9系统与胞嘧啶脱氨酶CodA (sm)反向筛选系统结合起来,对链霉菌(Streptomyces coelicolor)中的放线紫红素聚酮链延长因子基因actI-ORF2特异性敲除,建立了一种无标记的基因组编辑和筛选技术。
随着CRISPR/Cas9技术的发展,研究者开始关注该技术的脱靶问题,即sgRNA:Cas9蛋白复合物切割非靶基因或者非靶位点[52]。为了提高靶向效率,降低脱靶效应,不同的研究者提出不同的策略和方法,主要有以下两种:(1)提高sgRNA的特异性,可将sgRNA的3′端或者5′端缩短,或者在sgRNA的5′端添加2个鸟苷酸,这些措施均可以降低脱靶效率,但也有可能降低打靶效率[53-55]。由于染色质的状态严重影响sgRNA:Cas9蛋白复合物的活性,因此虽然基因组上可供选择的靶位点较多,但是绝大多数靶位点(> 99%)不能被sgRNA:Cas9蛋白复合物结合,因此靶位点应该尽可能在启动子、增强子和基因内部[56]。(2)突变Cas9蛋白,对野生型Cas9进行突变,使其中1个结构域失活,得到突变型D10A Cas9切口酶或者H840A Cas9切口酶。利用切口酶对DNA进行切割,发现脱靶效应可降至1/1 000[57-58]。随后,研究者将Cas9蛋白的2个结构域同时突变,产生失活的dCas9,然后将dCas9与FokI核酸酶结合形成融合蛋白(dCas9-FokI),脱靶效应可降低至1/5 000[59-60]。Slaymaker等[61]对spCas9的3个氨基酸位点进行结构指导的突变后,产生的增强型Cas9蛋白能够将脱靶突变降低到“无法检测”的水平,虽然脱靶效率是在人类胚肾细胞中检测的,但是对于细菌中Cas9蛋白改造仍具有借鉴意义。此外还可以通过调整sgRNA:Cas9蛋白的浓度来降低脱靶效应,但是由于会引起打靶效率的下降,因此需要综合平衡[62]。
4.2 CRISPR/Cas9系统介导的基因表达调控CRISPR/Cas9技术不仅可以进行基因组编辑,而且还可以在转录和翻译水平上调控基因的表达。dCas9虽无核酸酶活性,但是在sgRNA的指导下可与特定的DNA序列结合,从而对特定基因表达进行调控。如果dCas9在sgRNA引导下被用来抑制基因表达,这一方法称为CRISPR干扰技术(CRISPR interference,CRISPRi)[63]。根据Bikard等[64]和Qi等[65]的研究结果,dCas9主要通过两种方式抑制基因的转录:(1)限制RNA聚合酶与启动子结合,抑制转录起始;(2)阻止RNA聚合酶在DNA双链上的滑动,抑制转录延伸(图 2A)。目前来看,第1种方式更为有效,基因沉默的效率与目标链的位置无关,即dCas9靶向模板链与非模板链均表现相似的抑制效率。对于第2种方式,dCas9抑制基因转录的效率不仅受DNA双链位置的影响,而且还受靶位点与转录起始位点距离的影响。dCas9蛋白结合到非模板链时的抑制效率较高,而且在RBS附近的位点更为有效;dCas9蛋白结合在靶基因的启动子区域可以明显抑制基因的转录。如果结合在基因的3′末端,则抑制效率显著降低;如果结合在模板链基因的末端,抑制效率则进一步降低[66-67]。以上实验结果说明靶位点与转录起始位点的距离与dCas9抑制效率存在相反的关系。此外,有研究者发现截短的sgRNA可以结合到靶位点上,但是不会激发Cas9蛋白的核酸酶切割活性,同样可以达到抑制基因表达的效果[22, 68]。目前已有多位研究者采用该技术对E. coli[64-65]、S. pneumoniae[64]、B. subtilis]69]、Cyanobacterium synechococcus[70]、Corynebacterium glutamicum[71]、M. tuberculosis[72]和S. cerevisiae[73]等多种微生物进行了研究。

|
| 图 2 CRISPR/Cas9介导的基因表达调控策略 Figure 2 CRISPR/Cas9-mediated gene expression regulation strategies 注:A:CRISPRi,dCas9:sgRNA复合体结合在启动子区可抑制RNAP的转录起始,或者结合在基因编码区可抑制转录延伸;B:CRISPRa,dCas9与RNAP的ω亚基或者转录激活域VP64融合表达以增加基因转录水平. Note: A: CRISPRi, binding of Cas9:sgRNA complexes to promoter blocks binding of RNAP, preventing transcription initiation. Binding of dCas9 to the CDS of a gene blocks further elongation of the transcription. B: CRISPRa, binding of dCas9 fused to ω subunit of RNA polymerase (dCas9-ω) or transcription activation domain VP64 increases transcription rate. |
|
|
除转录抑制外,将dCas9蛋白与转录激活结构域融合,还可以激活特定基因的转录,这一工具称为CRISPR activation (CRISPRa)或者CRISPR-on[74] (图 2B)。靶序列位置对CRISPRa激活效率有重要影响,当靶序列与启动子的距离合适时,激活效率较高;靶序列与启动子上游相距较远时,激活效率会有一定程度的下降。当靶序列与启动子距离过近或位于开放阅读框内时则会产生抑制效应。Bikard等[64]将RNA聚合酶的ω亚基分别与dCas9的C端和N端融合,然后转入E. coli MG1655的ω亚基基因rpoZ−缺失突变体中,结果发现当dCas9-ω融合蛋白靶向LacZ启动子−35区59 nt的位置时,可以获得23倍的诱导效果。Dahlman等[75]利用截短的sgRNA和转录激活域嵌合体同Cas9蛋白共同转化,也可以增强基因表达效率。另外,通过诱导型启动子控制的载体来编码dCas9或sgRNA基因,可以精准地抑制或者激活靶基因表达,并且还具有可逆性。例如,Farzadfard等[76]将dCas9和酿酒酵母的转录激活域蛋白VP64连接起来,可以上调或者下调GFP的表达。这些研究扩大CRISPR/Cas9技术的应用范围,同时也为细菌基因表达调控的研究提供一些新的工具和思路。
4.3 细菌分型传统的细菌分型方法依赖于细菌表型特征鉴定,如生化反应、血清学特点等,这些方法存在分辨率低、重复性差、操作复杂,费时费力等缺点。与表型分型相比,分子分型技术具有快速、准确、重复性好等特点,已得到广泛应用[77]。但是,基因组测序需要操作人员较高的分析技术,脉冲场凝胶电泳技术在电泳图谱比较、识别及不同研究之间互相比对时仍存在一定的局限性,而且难以区分长期进化过程中源于同一克隆的不同菌株之间微小的DNA位点变异。因此这两种方法不适合菌株的快速分型。多位点序列分析(MLST)和多位点可变重复序列分析(MLVA)具有快速简便、分辨率高、重复性好等优点,但是需要在数据库中参比,因而其应用也受到限制。另外,如果看家基因高度保守,也不能进行精确分型。由于有的菌株种间差异小,16S rRNA基因鉴定只能定位到属的水平,不能鉴定到种的水平。CRISPR/Cas系统提供一种快速有效和高分辨率的细菌分型方法。CRISPR的间隔区来自入侵的噬菌体或者质粒DNA片段,其来源的多样性导致CRISPR位点间隔序列的多态性,通过CRISPR位点的PCR扩增和测序就可以分析间隔序列的组成和排列顺序,用于细菌分型,这种方法称为CRISPR位点基因分型[78]。这种方法已经应用在耶尔森氏菌(Yersinia)[78]、大肠杆菌属(Escherichia)[79]、沙门氏菌属(Salmonella)[80]和炭疽芽胞杆菌(Bacillus anthracis)[81]等。尽管依据CRISPR基因簇能够有效区分属、种及不同株系的菌株,但该方法也存在局限性。比如,某些细菌中的CRISPR位点出现频率低或间隔区种类较多,导致单纯依赖CRISPR位点的分辨效果降低,因此需要结合其他方法共同分析。另外,新间隔序列总是插入到前导序列和其临近的重复序列之间,而被剔除的间隔序列通常位于CRISPR位点3′端的末端,不同的间隔序列代表着不同的外源DNA侵入历史。因此,CRISPR位点间隔序列的变化可以反映细菌的进化历程,有助于研究不同细菌之间的亲缘关系。哈佛大学Shipman等[82]以CRISPR位点整合间隔序列的时序性特点为基础开发出一种简单的分子记录设备,即一种突变的大肠杆菌细胞(Cas蛋白复合体仅由Cas1和Cas2组成)能够将人工合成的特定DNA序列按照时间排序的方式整合到自身的CRISPR位点中。该技术和反转录酶联合使用,则可以记录细胞内基因的mRNA表达谱,用于基因表达调控的研究。除了细菌分型,细菌CRISPR位点的间隔序列还可以反映细菌与周围噬菌体的互作情况。由于CRISPR的间隔序列来自于噬菌体,借助于DNA二代测序技术,将环境样品中海量的CRISPR序列信息与噬菌体的序列进行比对就能推测细菌生存环境周围的病毒组(Virome),可以反映噬菌体与宿主细菌之间的进化、相互作用及其生态适应等关系。Stern等[83]对124个人肠道微生物进行宏基因组测序,通过CRISPR分析发现一个由991个噬菌体组成的病毒组。因此,CRISPR结构可以作为细菌分型溯源与进化研究的理想位点,还适用于宿主与病毒之间的互作研究。
4.4 在基因克隆和测序方面的应用长片段基因簇(尤其是合成高附加值生物分子的基因簇)的克隆是合成生物学和化学生物学研究中最为基础和关键的步骤。传统的PCR克隆方法或者通过构建基因组文库筛选等方法存在着克隆片段长度的限制、易产生突变、步骤复杂、效率低下等缺点。中国科学院微生物研究所的研究人员[84-85]利用CRISPR/Cas9技术对E. coli基因簇位点两侧进行切割,利用Gibson组装方法将切割下来的基因簇和带有30 bp同源序列的克隆载体相连,完成150 kb DNA的一步克隆。该方法(Cas9-Assisted Targeting of Chromosome segments,CATCH)大大简化细菌长片段DNA的克隆步骤,显著缩短实验时间(8 h vs数天),有利于微生物重要功能基因簇的研究。另外,Wang等[86]将CRISPR/Cas9和Gibson方法结合起来,开发出一种无缝克隆的方法。由于待插入的目的基因和载体具有相同的同源臂,首先利用Cas9蛋白对载体进行切割,经Gibson组装后转化大肠杆菌,挑选阳性克隆,这样避开酶切连接法需要载体上必需具有酶切位点或者存在多个酶切位点的限制。根据同样的原理,Lee等[87]利用CRISPR/Cas9介导的TAR (Transformation-associated recombination)克隆方法(TAR-CRISPR)可以显著增加S. cerevisiae中阳性克隆的比例,将筛选效率从0.5%−2%提高到32%。
除了应用于基因克隆,CRISPR/Cas9还被用于消除测序过程中产生的高丰度非特异序列。加州大学旧金山分校的Gu等[88]利用CRISPR/Cas9技术来优化NGS和PCR技术,这种方法称作DASH (Depletion of abundant sequences by hybridization),即通过消除来实现靶序列的富集。该方法构建一个sgRNA文库来靶向非特异序列,然后利用Cas9将其切割以消除HeLa细胞线粒体rRNA,达到富集病原菌序列的目的,有利于测序文库的构建和提高输出序列的质量。
5 总结和展望近年来,能够识别特异性序列的核酸酶已被广泛应用于动植物和微生物的基因组修饰和功能研究。虽然CRISPR/Cas9技术发展相对迅速,但是仍有不少的研究者对其进一步改进,如增加Cas9酶的来源,有研究者发现蛋白分子量更小的Cas蛋白如金黄色葡萄球菌(Staphylococcus aureus)的SaCas9,SaCas9比当前广泛使用的SpCas9小25%,解决了腺病毒等载体因负载能力有限而难以同时包装SpCas9酶和其他基因编辑必需的元件进入到单个病毒颗粒中这一问题[89]。此外,研究人员还利用分子进化系统对SaCas9进行改造,从而扩大它的靶向范围[90]。该技术的应用范围也在不断扩大,如将dCas9:sgRNA和荧光蛋白结合起来可研究细菌染色结构和动态变化。通过基因工程技术,将小片段的病毒原间隔序列整合到CRISPR的重复序列中可以增强一些工业微生物或者发酵微生物的噬菌体抵抗能力[9]。将dCas9或者sgRNA与甲基化酶或者去甲基化酶、乙酰化酶或者去乙酰化酶、激酶等参与表观调控酶相耦联用于控制基因的表达与关闭,可用于表观遗传学的研究。因此,CRISPR/Cas系统的广泛应用将对生物学的发展产生深远的影响。作为一项新技术,CRISPR/Cas系统也存在一些需要解决的问题,如脱靶效应是所有基因组编辑技术所面临的主要问题之一,但是目前Cas9的切割特异性仍然有待进一步提高。我们相信,随着越来越多研究者和实验室的加入,CRISPR/Cas9技术的功能会进一步完善,不同版本的CRISPR/Cas9技术会被开发出来用于微生物以及动植物基因功能和代谢调控合成生物学等研究领域。
| [1] |
Gaj T, Gersbach CA, Barbas CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [2] |
Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [3] |
Mojica FJM, Díez-Villaseñor C, Soria E, et al. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, bacteria and mitochondria[J]. Molecular Microbiology, 2000, 36(1): 244-246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [4] |
Jansen R, van Embden JDA, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [5] |
Jansen R, van Embden JD, Gaastra W, et al. Identification of a novel family of sequence repeats among prokaryotes[J]. OMICS: A Journal of Integrative Biology, 2002, 6(1): 23-33. DOI:10.1089/15362310252780816 |
| [6] |
Mojica FJM, Díez-Villaseñor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [7] |
Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 151(3): 653-663. DOI:10.1099/mic.0.27437-0 |
| [8] |
Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J]. Microbiology, 2005, 151(8): 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [9] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [10] |
Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA[J]. Science, 2008, 322(5909): 1843-1845. |
| [11] |
Brouns SJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [12] |
Garneau JE, Dupuis ME, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468(7320): 67-71. DOI:10.1038/nature09523 |
| [13] |
Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. |
| [14] |
Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J]. Nucleic Acids Research, 2011, 39(21): 9275-9282. DOI:10.1093/nar/gkr606 |
| [15] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): E2579-E2586. DOI:10.1073/pnas.1208507109 |
| [16] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [17] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. |
| [18] |
Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [19] |
Friedland AE, Tzur YB, Esvelt KM, et al. Heritable genome editing in C. elegans via a CRISPR-Cas9 system[J]. Nature Methods, 2013, 10(8): 741-743. DOI:10.1038/nmeth.2532 |
| [20] |
Chang NN, Sun CH, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Research, 2013, 23(4): 465-472. DOI:10.1038/cr.2013.45 |
| [21] |
Jiang WZ, Zhou HB, Bi HH, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice[J]. Nucleic Acids Research, 2013, 41(20): e188. DOI:10.1093/nar/gkt780 |
| [22] |
Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [23] |
DiCarlo JE, Norville JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135 |
| [24] |
Sorek R, Kunin V, Hugenholtz P. CRISPR—a widespread system that provides acquired resistance against phages in bacteria and archaea[J]. Nature Reviews Microbiology, 2007, 6(3): 181-186. |
| [25] |
Barrangou R, Marraffini L. CRISPR-Cas systems: prokaryotes upgrade to adaptive immunity[J]. Molecular Cell, 2014, 54(2): 234-244. DOI:10.1016/j.molcel.2014.03.011 |
| [26] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [27] |
Silas S, Mohr G, Sidote DJ, et al. Direct CRISPR spacer acquisition from RNA by a natural reverse transcriptase-Cas1 fusion protein[J]. Science, 2016, 351(6276): aad4234. |
| [28] |
Lieber MR. The mechanism of double-strand DVA break repair by the nonhomologous DNA end joining pathway[J]. Annual Review of Biochemistry, 2010, 79(1): 181-211. DOI:10.1146/annurev.biochem.052308.093131 |
| [29] |
Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity[J]. Nature, 2010, 463(7280): 568-571. DOI:10.1038/nature08703 |
| [30] |
Haft DH, Selengut J, Mongodin EF, et al. A guild of45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes[J]. PLoS Computational Biology, 2005, 1(6): e60. DOI:10.1371/journal.pcbi.0010060 |
| [31] |
Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2011, 9(6): 467-477. |
| [32] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [33] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [34] |
Smargon AA, Cox DBT, Pyzocha NK, et al. Cas13b is a Type Ⅵ-B CRISPR-associated RNA-Guided RNase differentially regulated by accessory proteins Csx27 and Csx28[J]. Molecular Cell, 2017, 65(4): 618-630. DOI:10.1016/j.molcel.2016.12.023 |
| [35] |
Jiang Y, Chen B, Duan CL, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J]. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [36] |
Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J]. Metabolic Engineering, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006 |
| [37] |
Xia J, Zheng MG, Wang L, et al. Knocking out phosphoenolpyruvate carboxylase gene by CRISPR/Cas and its influence on fatty acid metabolism in Escherichia coli[J]. Microbiology China, 2016, 48(8): 1864-1871. 夏军, 郑明刚, 王玲, 等. 运用CRISPR/Cas系统敲除大肠杆菌磷酸烯醇式丙酮酸羧化酶基因及其对脂肪酸代谢的影响[J]. 微生物学通报, 2016, 48(8): 1864-1871. |
| [38] |
Zhang K, Duan XG, Wu J. Multigene disruption in undomesticated Bacillus subtilis ATCC 6051a using the CRISPR/Cas9 system[J]. Scientific Reports, 2016, 6: 27943. |
| [39] |
Altenbuchner J. Editing of the Bacillus subtilis genome by the CRISPR-Cas9 system[J]. Applied Environmental Microbiology, 2011, 82(17): 5421-5427. |
| [40] |
Li Q, Chen J, Minton NP, et al. CRISPR-based genome editing and expression control systems in Clostridium acetobutylicum and Clostridium beijerinckii[J]. Biotechnology Journal, 2016, 11(7): 961-972. DOI:10.1002/biot.v11.7 |
| [41] |
Oh JH, van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J]. Nucleic Acids Research, 2013, 42(17): e131. |
| [42] |
Wendt KE, Ungerer J, Cobb RE, et al. CRISPR/Cas9 mediated targeted mutagenesis of the fast growing Cyanobacterium Synechococcus elongates UTEX 2973[J]. Microbial Cell Factories, 2016, 15(1): 1-8. DOI:10.1186/s12934-015-0402-6 |
| [43] |
Cao QH, Shao HH, Qiu H, et al. Using the CRISPR/Cas9 system to eliminate native plasmids of Zymomonas mobilis ZM4[J]. Bioscience, Biotechnology, and Biochemistry, 2017, 81(3): 453-459. DOI:10.1080/09168451.2016.1189312 |
| [44] |
Liu R, Chen L, Jiang YP, et al. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system[J]. Cell Discovery, 2015, 1: 15007. |
| [45] |
N dvig CS, Nielsen JB, Kogle ME, et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi[J]. PLoS One, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085 |
| [46] |
Liu Q, Gao RR, Li JC, et al. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering[J]. Biotechnology for Biofuels, 2017, 10(1): 1. |
| [47] |
Nayak DD, Metcalf WW. Cas9-mediated genome editing in the methanogenic archaeon Methanosarcina acetivorans[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(11): 2976-2981. |
| [48] |
Su TY, Liu FP, Gu PF, et al. A CRISPR-Cas9 assisted non-homologous end-joining strategy for one-step engineering of bacterial genome[J]. Scientific Reports, 2016, 6: 37895. DOI:10.1038/srep37895 |
| [49] |
Ronda C, Maury J, Jakočiunas T, et al. CrEdit: CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2015, 14(1): 97. DOI:10.1186/s12934-015-0288-3 |
| [50] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. |
| [51] |
Zeng H, Wen SS, Xu W, et al. Highly efficient editing of the actinorhodin polyketide chain length factor gene in Streptomyces coelicolor M145 using CRISPR/Cas9-CodA(sm) combined system[J]. Applied Microbiology Biotechnology, 2015, 99(24): 10575-10585. |
| [52] |
Cho SW, Kim S, Kim Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Research, 2014, 24(1): 132-141. DOI:10.1101/gr.162339.113 |
| [53] |
Fu Y, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nature Biotechnology, 2014, 32(3): 279-284. DOI:10.1038/nbt.2808 |
| [54] |
Fu Y, Reyon D, Joung JK. Targeted genome editing in human cells using CRISPR/Cas nucleases and truncated guide RNAs[J]. Methods in Enzymology, 2013, 546: 21-45. |
| [55] |
Zhang XH, Tee LY, Wang XG, et al. Off-target effects in CRISPR/Cas9-mediated genome engineering[J]. Molecular Therapy-Nucleic Acids, 2015, 4(11): e264. |
| [56] |
Wu XB, Scott DA, Kriz AJ, et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells[J]. Nature Biotechnology, 2014, 32(7): 670-676. DOI:10.1038/nbt.2889 |
| [57] |
Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389. DOI:10.1016/j.cell.2013.08.021 |
| [58] |
Mali P, Aach J, Stranges PB, et al. Cas9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering[J]. Nature Biotechnology, 2013, 31(9): 833-838. DOI:10.1038/nbt.2675 |
| [59] |
Tsai SQ, Wyvekens N, Khayter C, et al. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing[J]. Nature Biotechnology, 2014, 32(6): 569-576. DOI:10.1038/nbt.2908 |
| [60] |
Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification[J]. Nature Biotechnology, 2014, 32(6): 577-582. |
| [61] |
Slaymaker IM, Gao LY, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 |
| [62] |
Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nature Biotechnology, 2013, 31(9): 827-832. DOI:10.1038/nbt.2647 |
| [63] |
Larson MH, Gilbert LA, Wang XW, et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression[J]. Nature Protocols, 2013, 8(11): 2180-2196. DOI:10.1038/nprot.2013.132 |
| [64] |
Bikard D, Jiang WY, Samai P, et al. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system[J]. Nucleic Acids Research, 2013, 41(15): 7429-7437. DOI:10.1093/nar/gkt520 |
| [65] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [66] |
Luo ML, Leenay RT, Beisel CL. Current and future prospects for CRISPR-based tools in bacteria[J]. Biotechnology and Bioengineering, 2016, 113(5): 930-943. DOI:10.1002/bit.25851 |
| [67] |
Choi KR, Lee SY. CRISPR technologies for bacterial systems: current achievements and future directions[J]. Biotechnology Advances, 2016, 34(7): 1180-1209. |
| [68] |
Kiani S, Chavez A, Tuttle M, et al. Cas9 gRNA engineering for genome editing, activation and repression[J]. Nature Methods, 2015, 12(11): 1051-1054. DOI:10.1038/nmeth.3580 |
| [69] |
Westbrook AW, Moo-Young M, Chou CP. Development of a CRISPR-Cas9 tool kit for comprehensive engineering of Bacillus subtilis[J]. Applied and Environmental Microbiology, 2016, 82(16): 4876-4895. DOI:10.1128/AEM.01159-16 |
| [70] |
Gordon GC, Korosh TC, Cameron JC, et al. CRISPR interference as a titratable, trans-acting regulatory tool for metabolic engineering in the cyanobacterium Synechococcus sp. strain PCC 7002[J]. Metabolic Engineering, 2016, 38: 170-179. DOI:10.1016/j.ymben.2016.07.007 |
| [71] |
Cleto S, Jensen JVK, Wendisch VF, et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference (CRISPRi)[J]. ACS Synthetic Biology, 2016, 5(5): 375-385. |
| [72] |
Singh AK, Carette X, Potluri LP, et al. Investigating essential gene function in Mycobacterium tuberculosis using an efficient CRISPR interference system[J]. Nucleic Acids Research, 2016, 44(18): e143. DOI:10.1093/nar/gkw625 |
| [73] |
Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2): 442-451. |
| [74] |
Cheng AW, Wang HY, Yang H, et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system[J]. Cell Research, 2013, 23(10): 1163-1171. |
| [75] |
Dahlman JE, Abudayyeh OO, Joung J, et al. Orthogonal gene knockout and activation with a catalytically active Cas9 nuclease[J]. Nature Biotechnology, 2015, 33(11): 1159-1161. DOI:10.1038/nbt.3390 |
| [76] |
Farzadfard F, Perli SD, Lu TK. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas[J]. ACS Synthetic Biology, 2013, 2(2): 604-613. |
| [77] |
Barrangou R, Dudley EG. CRISPR-based typing and next-generation tracking technologies[J]. Annual Review of Food Science and Technology, 2016, 7(1): 395-411. DOI:10.1146/annurev-food-022814-015729 |
| [78] |
Cui YJ, Li YJ, Gorgé O, et al. Insight into microevolution of Yersinia pestis by clustered regularly interspaced short palindromic repeats[J]. PLoS One, 2008, 3(7): e2652. |
| [79] |
Toro M, Cao GJ, Ju WT, et al. Association of clustered regularly interspaced short palindromic repeat (CRISPR) elements with specific serotypes and virulence potential of shiga toxin-producing Escherichia coli[J]. Applied and Environmental Microbiology, 2014, 80(4): 1411-1420. DOI:10.1128/AEM.03018-13 |
| [80] |
Shariat N, Dimarzio MJ, Yin S, et al. The combination of CRISPR-MVLST and PFGE provides increased discriminatory power for differentiating human clinical isolates of Salmonella enterica subsp. enterica serovar enteritidis[J]. Food Microbiology, 2013, 34(1): 164-173. DOI:10.1016/j.fm.2012.11.012 |
| [81] |
Gao ZQ, Wang DS, Feng EL, et al. Clustered regularly interspaced short palindromic repeats (CRISPR) site in Bacillus anthracis[J]. Acta Microbiologica Sinica, 2014, 54(11): 1362-1368. 高志奇, 王东澍, 冯尔玲, 等. 炭疽芽胞杆菌中CRISPR位点[J]. 微生物学报, 2014, 54(11): 1362-1368. |
| [82] |
Shipman SL, Nivala J, Macklis JD, et al. Molecular recordings by directed CRISPR spacer acquisition[J]. Science, 2016, 353(6298): aaf1175. DOI:10.1126/science.aaf1175 |
| [83] |
Stern A, Mick E, Tirosh I, et al. CRISPR targeting reveals a reservoir of common phages associated with the human gut microbiome[J]. Genome Research, 2012, 22(10): 1985-1994. DOI:10.1101/gr.138297.112 |
| [84] |
Jiang WJ, Zhao XJ, Gabrieli T, et al. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters[J]. Nature Communications, 2015, 6: 8101. DOI:10.1038/ncomms9101 |
| [85] |
Jiang WJ, Zhu TF. Targeted isolation and cloning of 100-kb microbial genomic sequences by Cas9-assisted targeting of chromosome segments[J]. Nature Protocols, 2016, 11(5): 960-975. |
| [86] |
Wang JW, Wang A, Li KY, et al. CRISPR/Cas9 nuclease cleavage combined with Gibson assembly for seamless cloning[J]. BioTechniques, 2015, 58(4): 161-170. |
| [87] |
Lee NC, Larionov V, Kouprina N. Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J]. Nucleic Acids Research, 2015, 43(8): e55. DOI:10.1093/nar/gkv112 |
| [88] |
Gu W, Crawford ED, O'Donovan BD, et al. Depletion of Abundant Sequences by Hybridization (DASH): using Cas9 to remove unwanted high-abundance species in sequencing libraries and molecular counting applications[J]. Genome Biology, 2016, 17: 41. DOI:10.1186/s13059-016-0904-5 |
| [89] |
Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520(7546): 186-191. DOI:10.1038/nature14299 |
| [90] |
Kleinstiver BP, Prew MS, Tsai SQ, et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition[J]. Nature Biotechnology, 2015, 33(12): 1293-1298. DOI:10.1038/nbt.3404 |
 2018, Vol. 45
2018, Vol. 45