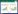扩展功能
文章信息
- 蒋程恺, 孟思童, 张菲, 胡晓婧, 谢守锋, 陶美凤, 梁晶丹, 康前进, 白林泉, 邓子新
- JIANG Cheng-Kai, MENG Si-Tong, ZHANG Fei, HU Xiao-Jing, XIE Shou-Feng, TAO Mei-Feng, LIANG Jing-Dan, KANG Qian-Jin, BAI Lin-Quan, DENG Zi-Xin
- 林可链霉菌NRRL 2936中帕马霉素生物合成基因簇的异源表达及调控基因功能研究
- Heterologous expression of the biosynthetic gene cluster of pamamycin and investigation on the functions of regulatory genes from Streptomyces lincolnensis NRRL 2936
- 微生物学通报, 2018, 45(2): 334-346
- Microbiology China, 2018, 45(2): 334-346
- DOI: 10.13344/j.microbiol.china.170268
-
文章历史
- 收稿日期: 2017-04-01
- 接受日期: 2017-05-28
- 网络首发日期(www.cnki.net): 2017-09-27
帕马霉素首次被发现于白黑链霉菌(Streptomyces alboniger)的发酵产物中,属于大环内酯类抗生素,对分枝杆菌、真菌和革兰氏阳性细菌均具有较好的抑制作用[1-2]。早期利用同位素喂养实验证实了帕马霉素的生物合成是以醋酸盐、丙酸盐和琥珀酸盐为底物组装而成[3]。此外,Rebets等克隆了来源于白黑链霉菌中帕马霉素的完整生物合成基因簇[4],在白色链霉菌(Streptomyces albus)中实现了帕马霉素的异源表达。在此基础之上,通过遗传学实验和帕马霉素生物合成中间体的质谱分析,推测了帕马霉素的生物合成途径[4]。但迄今为止,帕马霉素生物合成和调控机制并没有被详细地解析,多组分的现状与产生菌株产量较低的问题现在依然没有得到解决,但由于其具有新颖的结构(图 1)和良好的生物活性,依然使它得到了广泛的关注[5]。
|
| 图 1 帕马霉素的结构式 Figure 1 The chemical structures of pamamycin |
|
|
目前,根据链霉菌次级代谢产物生物合成的功能基因成簇排列的特征,克隆完整的次级代谢产物生物合成基因簇进行异源表达,已经成为研究目标抗生素生物合成机制和遗传改造的重要手段之一。该研究方法可以对目标活性次级代谢产物生物合成基因簇实现快捷的验证及开展其功能基因的遗传学研究[6];可以规避次级代谢产物原始产生菌的缺点,激活沉默的基因簇或提高目标化合物的产量[7-8];可以实现一些功能基因的快速替换以及利用合成生物学策略进行目标产物的结构优化等[8]。在链霉菌中,大部分抗生素生物合成基因的表达还受到了一些调控蛋白的影响。相关调控基因功能的鉴定是实现目标抗生素调控途径优化的前提,对调控途径的优化具有重要的指导作用[9]。
鉴于帕马霉素的重要研究价值以及链霉菌异源表达技术的多方面优点,本研究首先在林可链霉菌NRRL 2936的全基因组序列中发现了帕马霉素的生物合成基因簇,从该菌株的基因组文库中获得了含有完整帕马霉素生物合成基因簇的质粒pJQK450,在天蓝色链霉菌(Streptomyces coelicolor) M1154中实现了异源表达,并对帕马霉素生物合成基因簇左右两端的两个调控基因的功能进行了探讨。帕马霉素生物合成基因簇的成功异源表达,为进一步详细探讨帕马霉素生物合成机理与结构优化奠定了基础。同时,其生物合成基因簇中调控基因功能的鉴定也为帕马霉素生物合成的优化提供了研究基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒| Strains | Genotype/description | Reference/source |
| Escherichia coli | ||
| DH10B | lacZ,recA,ΔM15 | Gibco BRL |
| ET12567/pUZ8002 | recE,dcm–,dam–,hsdS,Cmr,Tetr,Strr,Kmr | [10] |
| GB08 | 基因重组菌株 | [11] |
| XL1-Blue | 基因组文库克隆宿主菌株 | [12] |
| Streptomyces | ||
| Streptomyces lincolnensis NRRL 2936 | 林可霉素产生菌,基因组文库构建菌株 | [13] |
| Streptomyces lividans SBT18 | SBT5::nusG::mdfA::ImrA::afsRs | [14] |
| Streptomyces coelicolor M1152 | Δact,Δred,Δcpk,Δcda,ΔrpoB | [15] |
| Streptomyces coelicolor M1154 | Δact,Δred,Δcpk,Δcda,ΔrpoB,ΔrpsL | [15] |
| JCK101 | pJQK450导入变铅青链霉菌SBT18中的突变株 | 本研究 |
| JCK102 | pJTU2554导入变铅青链霉菌SBT18中的突变株 | 本研究 |
| JCK105 | pJQK450导入天蓝色链霉菌M1152中的突变株 | 本研究 |
| JCK106 | pJTU2554导入天蓝色链霉菌M1152中的突变株 | 本研究 |
| JCK109 | pJQK450导入天蓝色链霉菌M1154中的突变株 | 本研究 |
| JCK110 | pJTU2554导入天蓝色链霉菌M1154中的突变株 | 本研究 |
| JCK113 | pJQK451导入天蓝色链霉菌M1154中的突变株 | 本研究 |
| JCK117 | pJQK452导入天蓝色链霉菌M1154中的突变株 | 本研究 |
| JCK121 | pJQK453导入JCK113中的突变株 | 本研究 |
| JCK125 | pJQK454导入JCK117中的突变株 | 本研究 |
| Mycobacterium smegmattis mc2155 | 生物活性测试菌株 | [16] |
| Plasmids | Relevant characteristics | Reference/source |
| pBlueScript Ⅱ SK(+) | Bla,lacZ,ori(f1) | Stratagene |
| pJTU2554 | oriT,attP(ΦC31),aac(3)IV,cos | [17] |
| pPM927 | pSAM2,tsr,oriT | [18] |
| pJQK450 | 含有帕马霉素完整生物合成基因簇的Fosmid质粒 | 本研究 |
| pJQK451 | 含有敲除pamR2的帕马霉素生物合成基因簇的Fosmid质粒 | 本研究 |
| pJQK452 | 含有敲除pamR1的帕马霉素生物合成基因簇的Fosmid质粒 | 本研究 |
| pJQK453 | pPM927的EcoRⅠ位点插入带PermE*启动子的pamR2基因 | 本研究 |
| pJQK454 | pPM927的EcoRⅠ位点插入带PermE*启动子的pamR1基因 | 本研究 |
文中所用引物见表 3。
| Primers | Sequences (5′→3′) | Size (bp) |
| Pam-Lprobe F | GGTGGCATGTTCAGGGAG | 18 |
| Pam-Lprobe R | CGTCGAGGTACAGGTGGG | 18 |
| Pam-Rprobe F | TCACCCAGACCGGACAGG | 18 |
| Pam-Rprobe R | CCCAAGAGACCGCAGCAG | 18 |
| Apr-F | GCCCCTGCCACCTCACTC | 18 |
| Apr-R | TCTTCGCATCCCGCCTCT | 18 |
| Tsr-F | CAGCGACAGCAGTCCTTT | 18 |
| Tsr-R | GCTCTGGCTCATCATGGG | 18 |
| R1-Tar-F | CAGCAGCTGCTGTACTCACTGTGTGAGGAGTTCTCCATGATTCCGGGGATCCGTCGACC | 59 |
| R1-Tar-R | CCGGTCCGGTCAGCCCCTGACCACGGTGGTCGCACCGCCTGTAGGCTGGAGCTGCTTC | 58 |
| R2-Tar-F | GGATCGATCTGGCTGCGCGGTACGAAGAAGCGAGCGGTGATTCCGGGGATCCGTCGACC | 59 |
| R2-Tar-R | TGGTCCGCGACGCGGAGGACTGACCGGCCCGGCGGCTCATGTAGGCTGGAGCTGCTTC | 58 |
| R1-T-VF | GCACCGCAACAACGTTTCCC | 20 |
| R1-T-VR | GTGCACGCGCCTTCGTCCAG | 20 |
| R2-T-VF | CGCGTTAGAATGGAAGGGTG | 20 |
| R2-T-VR | ACAGATCGCACTCCTGGTCC | 20 |
| R1-F | AACATATGATGCAAGCGTTAGGTGTTCT (Nde Ⅰ) | 28 |
| R1-R | AAGAATTCTCAGCCCCTGACCACGGTGG (EcoR Ⅰ) | 28 |
| R2-F | AACATATGGTGAAGCAACCGCTGAGCCG (Nde Ⅰ) | 28 |
| R2-R | AAGAATTCTCAGGAGGGCAGTGACCGCA (EcoR Ⅰ) | 28 |
大肠杆菌培养采用LB (Luria-Bertani)和LA培养基[4];链霉菌培养采用TSBY液体培养基(g/L):胰蛋白胨30.0,蔗糖103.0,酵母提取物10.0,pH 7.0-7.2;黄豆饼粉浸汁琼脂培养基(SFM) (g/L):黄豆饼粉20.0,甘露醇20.0,琼脂20.0,pH 7.0-7.2;链霉菌发酵培养基(SGG) (g/L):可溶性淀粉10.0,葡萄糖10.0,甘油10.0,玉米浆干粉2.5,蛋白胨5.0,酵母提取物2.0,氯化钠1.0,碳酸钙3.0,pH 7.0-7.2。
1.1.4 主要试剂和仪器阿泊拉霉素、氨苄青霉素、卡那霉素、萘啶酮酸、硫链丝菌素和壮观霉素,西格玛奥德里奇贸易有限公司;DNA连接酶,宝日医生物科技有限公司;Pfu高保真DNA聚合酶,南京诺唯赞生物科技有限公司;BamH Ⅰ、EcoR Ⅰ和Kpn Ⅰ限制性核酸内切酶,赛默飞世尔科技有限公司;甲醇、乙酸乙酯,国药集团化学试剂有限公司。恒温培养箱,韶关市泰宏医疗器械有限公司;旋转蒸发仪,布琦实验室设备贸易有限公司;LC-MS,安捷伦科技有限公司。
1.2 方法 1.2.1 基因组文库筛选[19]在帕马霉素生物合成基因簇左右边界各2 kb处设计探针引物,命名为Pam-Lprobe F/Pam-Lprobe R (左端引物)和Pam-Rprobe F/Pam-Rprobe R (右端引物),利用这两对引物对林可链霉菌NRRL 2936的Fosmid基因组文库进行筛选,获得含有完整帕马霉素生物合成基因簇的质粒。然后,再利用文库筛选的两对探针引物进行PCR的验证,将得到的PCR产物进行测序,与已知序列进行比对发现具有一致性。此外,进一步利用EcoR Ⅰ、BamH Ⅰ和Kpn Ⅰ三个限制性核酸内切酶进行限制性酶切,与通过软件Vector NTI酶切位点(EcoR Ⅰ、BamH Ⅰ和Kpn Ⅰ三个限制性核酸内切酶)预测分析所得到的条带数目及大小进行对比,酶切所得到的条带大小与数量和预期的完全符合,才能选择为后续研究的目标Fosmid质粒。
1.2.2 接合转移及突变株筛选将本实验所要用于接合转移的质粒pJQK450、pJQK451、pJQK452、pJQK453和pJQK454,通过电转化的方法转入大肠杆菌ET12567/pUZ8002中[20]。然后,通过大肠杆菌-链霉菌间的双亲接合转移导入相应链霉菌中[21],13 h后用含相应抗生素及萘啶酮酸的1 mL无菌水覆盖,待SFM平板吹干后倒置培养3-4 d可以观察到接合子。将接合子划线接种到含相应抗生素的SFM培养皿上,于30 ℃培养箱进行培养。随后用PCR扩增质粒载体上的抗生素抗性基因及基因簇中的基因确保质粒整合到菌株染色体上,得到相应突变株。
1.2.3 链霉菌培养及保藏在SFM培养基培养皿中放入20 μL冻存的链霉菌孢子,使用棉签将冻存链霉菌菌株JCK101、JCK102、JCK103、JCK106、JCK109、JCK110、JCK113、JCK117、JCK121和JCK125的孢子均匀涂布在培养基表面,待培养基表面吹干后将其放置在30 ℃恒温培养箱中静置培养7 d,然后利用棉签从培养基表面轻轻刮取孢子,将刮取的孢子浸入装有20%甘油的菌种保藏管中,振荡均匀后于-30 ℃保存。
1.2.4 微生物发酵及样品制备方法分别将获得的异源表达及基因突变菌株(JCK101、JCK102、JCK103、JCK106、JCK109、JCK110、JCK113、JCK117、JCK121和JCK125)在SFM培养基上划线培养3 d,刮取固体培养皿中的菌体接入至SGG发酵培养基中,30 ℃、220 r/min进行液体发酵4 d[4]。发酵结束后,利用50 mL离心管收集液体发酵产物,3 500 r/min离心10 min将菌体与上清液分离。上清液和乙酸乙酯1:2充分振荡混匀,菌体与甲醇1:2充分混匀。合并乙酸乙酯萃取液和甲醇萃取液,进行减压干燥,然后再用1 mL甲醇将粗提物进行溶解,经过0.22 μm有机相微孔滤膜过滤后,供生物活性和LC-MS分析使用[4]。
1.2.5 生物活性测试生物活性测试培养皿的制备:(1)将冻存的耻垢分枝杆菌于LB液体培养基中37 ℃、220 r/min条件下活化18 h,为耻垢分枝杆菌种子液;(2)将约20 mL融化后的1% LA固体培养基,在生物安全柜中加入至50 mL离心管中,冷却至50 ℃后加入25 μL活化好的耻垢分枝杆菌种子液,轻轻颠倒混匀;(3)将混合液倒入平板中,约20 min后培养皿完全凝固,则完成生物活性测试培养皿的制备。生物活性测试采用“牛津杯”法[22],在培养基表面放置牛津杯,每个牛津杯中加入100 μL处理好的菌株粗提物,在生物安全柜中待加入的液体基本干燥后,将生物测定的培养皿放置在37 ℃恒温培养箱中,静置培养18-20 h后,观察和测定抑菌圈的大小。
1.2.6 LC-MS检测色谱柱为Agilent 5 μm,Eclipse XDB-C18,4.6 mm×150 mm。洗脱条件:流动相A为20 mmol/L醋酸铵溶液,B为乙腈。0-30 min:100%-20%的A相(体积比);30-31 min:20%-100%的A相(体积比);31-35 min:100% A相(体积比)。仪器型号:Agilent 1100 Series LC/MSD Trap,电离源:ESI,干燥温度:350 ℃,流速:0.4 mL/min,阳离子模式。检测样品为菌株JCK101、JCK102、JCK103、JCK106、JCK109、JCK110、JCK113、JCK117、JCK121和JCK125的发酵产物提取液(制备方法见1.2.3)。
1.2.7 PCR-Targeting敲除基因的方法在敲除的目标基因处,设计含有同源臂的PCR-Targeting引物R1-Tar-F/R1-Tar-R和R2-Tar-F/R2-Tar-R,利用这两对引物PCR扩增得到含有卡那霉素抗性基因和同源臂DNA片段。然后在大肠杆菌GB 08培养期间加入阿拉伯糖诱导重组酶基因表达,制备大肠杆菌GB 08电转化感受态细胞[11]。最后将1 μL质粒pJQK450与5 μL获得的PCR扩增产物混合均匀,通过电转化的方法共转至大肠杆菌GB 08的电转化感受态细胞中,利用卡那霉素抗性基因作为筛选标记,筛选得到相应的质粒pJQK452和pJQK451[20]。
1.2.8 基因回补质粒的构建方法在基因起始密码子和终止密码子处设计pamR1和pamR2的PCR扩增引物R1-F/R1-R和R2-F/R2-R,以质粒pJQK450为模板,利用高保真DNA聚合酶PCR扩增出pamR1和pamR2。将扩增得到的片段与相应酶切处理后的pBlueScript Ⅱ SK(+)相连接,挑取单克隆进行片段测序。选取正确的质粒用Nde Ⅰ和EcoR Ⅰ进行酶切,使用DNA琼脂糖凝胶回收试剂盒回收两个基因片段。将回收片段与用Nde Ⅰ和EcoR Ⅰ处理后的pJTU968进行酶连,挑取单克隆验证质粒。最后将验证正确的质粒使用Mun Ⅰ和EcoR Ⅰ酶切(Mun Ⅰ和EcoR Ⅰ为同尾酶),回收得到的基因片段与用EcoR Ⅰ酶切处理后pPM927连接,验证正确后完成基因回补质粒pJQK453和pJQK454的构建。
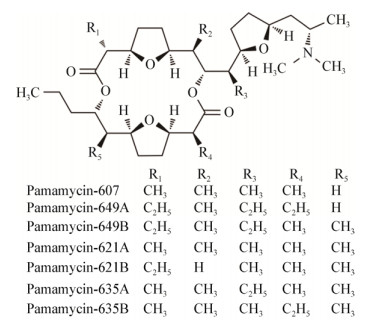
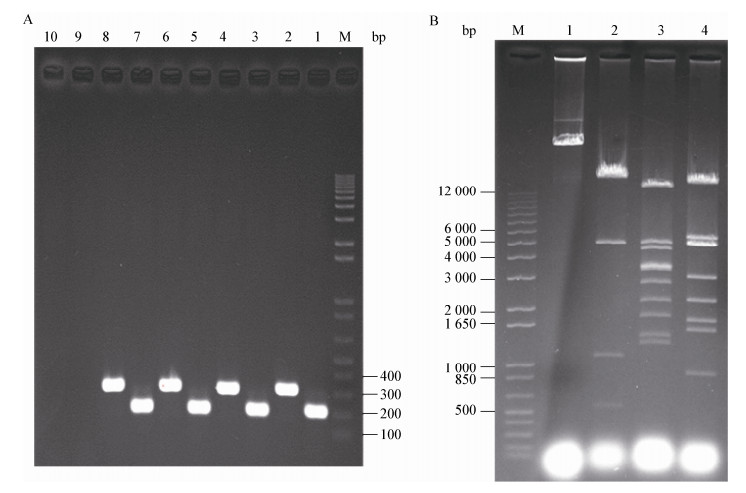
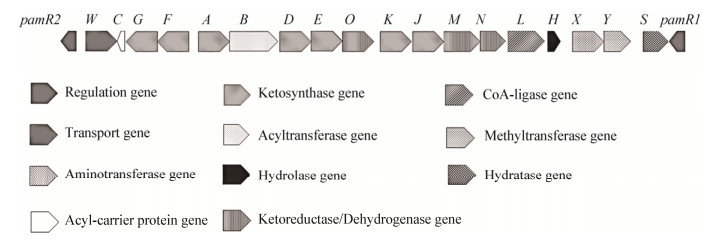
2 结果与分析 2.1 含有帕马霉素完整生物合成基因簇的文库质粒筛选通过聚合酶链式反应递缩基因组文库的方法对林可链霉菌NRRL 2936的基因组文库进行筛选[19],分别利用两对探针引物Pam-Lprobe F/Pam-Lprobe R和Pam-Rprobe F/Pam-Rprobe R进行PCR筛选,左端探针引物的PCR产物为219 bp,右端探针引物的PCR产物为318 bp (图 2A)。两对引物均为阳性时,表示该质粒可能包含完整的帕马霉素生物合成基因簇,筛选出的阳性克隆为10F11 (图 2A)。为了保证所得到质粒的可靠性,将目标质粒分别再次进行PCR和EcoRⅠ、BamHⅠ和KpnⅠ酶切验证(图 2B),PCR产物的测序和目标质粒的酶切结果均与预期一致,认为所筛选到的阳性克隆10F11包含了完整的帕马霉素生物合成基因簇(图 3),命名为pJQK450。在pJQK450质粒上,帕马霉素生物合成基因簇为32 kb,利用生物信息学分析发现其与已报道的白黑链霉菌中的帕马霉素生物合成基因簇有较高同源性[4](表 4中的Similar protein均来源于白黑链霉菌中帕马霉素生物合成的相关蛋白),其中pamA、pamB、pamD、pamE、pamF、pamG、pamH、pamJ、pamK、pamL、pamM、pamN和pamO为生物合成的功能基因,负责合成帕马霉素的分子骨架;pamR1和pamR2为调控基因,可能扮演调控生物合成基因表达的角色;pamW为编码转运蛋白的基因,可能负责帕马霉素的转运(表 4)。帕马霉素完整生物合成基因簇的克隆,为接下来开展其生物合成机制的研究和组分改造提供了很好的基础。
|
| 图 2 基因文库的PCR筛选(A)及pJQK450的酶切验证结果(B) Figure 2 The results of the PCR screening of genomic library (A) and digestion by restriction endonucleases of pJQK450 (B) 注:M:DNA ladder. A:1、3、5:左边界PCR结果;2、4、6:右边界PCR结果;7:左边界PCR阳性对照;8:右边界阳性对照;9:左边界阴性对照;10:右边界阴性对照. B:1:pJQK450;2:EcoR Ⅰ酶切图谱;3:BamH Ⅰ酶切图谱;4:Kpn Ⅰ酶切图谱. Note: M: DNA ladder. A: 1, 3, 5: PCR results of left boundary; 2, 4, 6: The PCR results of right boundary; 7: The PCR result of left boundary of positive control; 8: The PCR result of right boundary of positive control; 9: The PCR result of left boundary of negative control; 10: The PCR result of right boundary of negative control. B: 1: pJQK450; 2, 3, 4: Gel electrophoresis of pJQK450 digested by EcoR Ⅰ, BamH Ⅰ, Kpn Ⅰ. |
|
|
|
| 图 3 林可链霉菌NRRL 2936中的帕马霉素生物合成基因簇 Figure 3 The biosynthetic gene cluster of pamamycin in S. lincolnensis NRRL 2936 |
|
|
| Gene | aa | Proposed function | Similar protein (Accession No.) | Identical (%) |
| R2 | 245 | TetR family transcriptional regulator | AJS09379.1 | 76 |
| W | 558 | Drug resistance transporter | AJS09380.1 | 84 |
| C | 76 | Acyl-carrier protein | AJS09381.1 | 74 |
| G | 344 | Ketoacyl-acyl carrier protein synthase | AJS09382.1 | 85 |
| F | 439 | Ketoacyl-acyl carrier protein synthase | AJS09383.1 | 82 |
| A | 445 | Ketoacyl-acyl carrier protein synthase | AJS09384.1 | 83 |
| B | 620 | 3-Ketoacid(adipate/glutaconate) CoA transferase | AJS09385.1 | 81 |
| D | 319 | Ketoacyl-(acyl-carrier-protein) reductase | AJS09386.1 | 85 |
| E | 344 | Ketoacyl-acyl carrier protein synthase | AJS09387.1 | 87 |
| O | 264 | Ketoacyl-(acyl-carrier-protein) reductase | AJS09388.1 | 88 |
| K | 336 | Ketoacyl-acyl carrier protein synthase | AJS09389.1 | 82 |
| J | 431 | Ketoacyl-acyl carrier protein synthase | AJS09390.1 | 87 |
| M | 525 | Ketoacyl-(acyl-carrier-protein) reductase | AJS09391.1 | 75 |
| N | 275 | Ketoacyl-(acyl-carrier-protein) reductase | AJS09392.1 | 83 |
| L | 567 | Hydroxyl acids CoA ligase | AJS09393.1 | 83 |
| H | 327 | Metal-dependent hydrolase | AJS09394.1 | 86 |
| X | 448 | Aminotransferase | AJS09395.1 | 90 |
| Y | 273 | Methyltransferase | AJS09396.1 | 89 |
| S | 268 | Enoyl-CoA hydratase | AJS09397.1 | 84 |
| R1 | 215 | LuxR family transcriptional regulator | AJS09398.1 | 81 |
林可链霉菌NRRL 2936的基因组文库构建质粒为pJTU2554,该质粒含有作为筛选标记的阿泊拉霉素抗性基因,以及用于整合入宿主染色体的整合酶基因、attP位点和接合转移的oriT元件[17]。将质粒pJQK450电转化至大肠杆菌ET12567/pUZ8002中[20],得到正确转化子。然后,选取变铅青链霉菌(Streptomyces lividans) SBT18、天蓝色链霉菌M1152和天蓝色链霉菌M1154为异源表达宿主菌。通过大肠杆菌-链霉菌双亲接合转移,分别导入以上3种异源宿主中,通过Apr-F/Apr-R (图 4A)和Pam-Lprobe F/Pam-Lprobe R (图 4B)进行PCR验证,分别筛选得到突变株,挑取每个异源表达的衍生菌株,每个菌株挑取4个接合转移子,验证后分别命名为JCK101a–d、JCK105a–d和JCK109a–d,收取孢子冻存在-30 ℃备用。
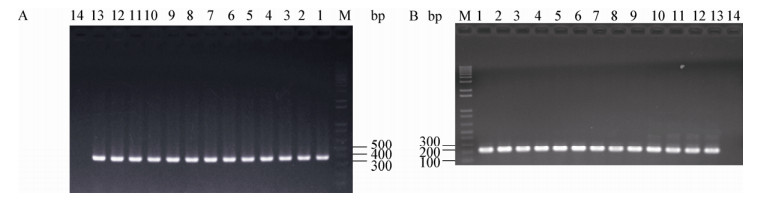
|
| 图 4 异源表达菌株的PCR验证结果 Figure 4 The PCR validation results of heterologous strains 注:M:DNA ladder. A:1、2、3、4:突变株JCK101a–d阿泊拉霉素抗性基因PCR验证结果;5、6、7、8:突变株105a–d阿泊拉霉素抗性基因PCR验证结果;9、10、11、12:突变株109a–d阿泊拉霉素抗性基因PCR验证结果;13:阳性对照;14:阴性对照. B:1、2、3、4:突变株JCK101a–d左边界PCR验证结果;5、6、7、8:突变株105a–d左边界PCR验证结果;9、10、11、12:突变株109a-d左边界PCR验证结果;13:阳性对照;14:阴性对照. Note: M: DNA ladder. A: 1, 2, 3, 4: The PCR validation results of JCK101a–d (Apr-F/Apr-R); 5, 6, 7, 8: The PCR validation result of JCK105a–d (Apr-F/Apr-R); 9, 10, 11, 12: The PCR validation result of JCK109a–d (Apr-F/Apr-R); 13: The PCR validation result of positive control (Apr-F/Apr-R); 14: The PCR validation resultsof negative control (Apr-F/Apr-R). B: 1, 2, 3, 4: The PCR validation result of left boundary for JCK101a-d; 5, 6, 7, 8: The PCR validation result of left boundary for JCK105a–d; 9, 10, 11, 12: The PCR validation result of left boundary for JCK109a–d; 13: The PCR validation result of positive control (Pam-Lprobe F/Pam-Lprobe R); 14: The PCR validation result of negative control (Pam-Lprobe F/Pam-Lprobe R). |
|
|
利用耻垢分枝杆菌作为帕马霉素生物活性测试的指示菌,每个异源表达的衍生菌株选取3个,分别为JCK101a–c、JCK105a–c和JCK109a–c进行发酵和发酵产物的制备,对照菌株(JCK102为变铅青链霉菌SBT18的空白质粒对照,JCK106为天蓝色链霉菌M1152的空白质粒对照,JCK110为天蓝色链霉菌M1154的空白质粒对照)也利用相同的方法进行发酵样品的制备。生物活性测试结果表明,异源表达菌株JCK109a–c三株菌的发酵产物均对耻垢分枝杆菌有抑制作用,表明了帕马霉素生物合成基因簇可能在天蓝色链霉菌M1154中异源表达了(图 5)。但是,在变铅青链霉菌SBT18和天蓝色链霉菌M1152两个宿主菌的异源表达菌株JCK101a–c和JCK105a–c的发酵产物中,没有发现对耻垢分枝杆菌具有抑制作用的成分,显示帕马霉素的生物合成基因簇可能在这两株菌种没有成功表达。为了进一步确定帕马霉素在JCK109a菌株中的产生情况,选用Agilent 1100 Series LC/MSD Trap液相质谱连用仪,利用阳离子模式对JCK109a (异源表达菌株)和JCK110 (空质粒对照菌株)的发酵产物进行分析,检测结果显示JCK110的发酵产物中没有帕马霉素组分的产生,而在JCK109a发酵产物中清楚发现了帕马霉素的两个组分帕马霉素-607 (m/z 608.3 [M+H]+)和帕马霉素-649 (m/z 650.2 [M+H]+)的存在(图 6)。该部分的实验结果表明,不同的链霉菌底盘菌株对于次生代谢产物的表达能力具有明显的差异。
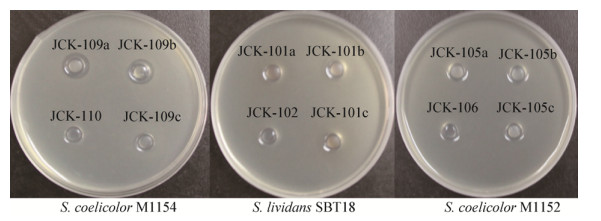
|
| 图 5 异源表达突变株JCK101、JCK105和JCK109发酵产物生物活性测试结果 Figure 5 The results of the bioactivity analysis of the cultures of JCK101, JCK105 and JCK109 |
|
|

|
| 图 6 异源表达菌株发酵产物中帕马霉素组分的LC-ESI-MS检测结果 Figure 6 The results of LC-ESI-MS analysis of pamamycin from heterologous host |
|
|
通过基因BLAST分析,帕马霉素生物合成基因簇中含有pamR1和pamR2两个调控基因,其中pamR1编码的蛋白为LuxR家族调控蛋白,pamR2编码的蛋白为TetR家族调控蛋白。为了验证这两个调控基因与帕马霉素生物合成的相关性,分别对pamR1和pamR2进行了敲除(图 7),获得质粒pJQK452和pJQK451。将这两个质粒分别电转化至ET12567/pUZ8002中[20],再通过结合转移导入至天蓝色链霉菌M1154中,每个基因突变株选取4个基因突变菌株,分别命名为JCK117a–d (pamR1基因突变株)和JCK113a–d (pamR2基因突变株)。同时,选取异源表达菌株JCK109和JCK110进行平行发酵。发酵完成后进行发酵产物的平行提取和制备,再进行生物活性测试(图 8C)。结果显示,pamR1缺失突变株(JCK117)和pamR2缺失突变株(JCK113)的发酵样品相比JCK109a的发酵样品,对于耻垢分枝杆菌的抑制能力明显增强,该结果表明这两个基因编码的蛋白均负调控了帕马霉素的生物合成过程。

|
| 图 7 基因pamR2 (A)和pamR1 (B)的基因置换示意图及PCR验证结果 Figure 7 The schematic and validation results of the gene replacement of pamR2 (A) and pamR1 (B) 注:M:DNA ladder;1:质粒pJQK450敲除pamR2前的PCR结果;2:敲除pamR2后得到的质粒pJQK451的PCR结果;3:质粒pJQK450敲除pamR1前的PCR结果;4:敲除pamR1后得到的质粒pJQK452的PCR结果. Note: M: DNA ladder; 1: The PCR result of pJQK450 before knockout of pamR2; 2: The PCR result of gene replacement of pJQK451; 3: The PCR result of pJQK450 before knockout of pamR1; 4: The PCR result of gene replacement of pJQK452. |
|
|
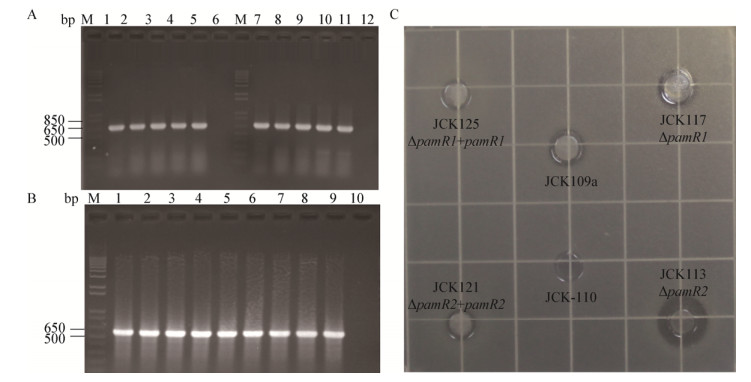
|
| 图 8 基因回补菌株的PCR验证结果(A、B)和相关菌株发酵产物抑菌能力的比较(C) Figure 8 PCR validation of the related complementation strains (A, B), comparison the bioactivity against M. smegmattis mc2155 of the cultures from related strains (C) 注:M:DNA ladder. A:1、2、3、4:菌株JCK121a–d的回补基因pamR2的PCR验证结果;5:阳性对照PCR结果;6:阴性对照PCR结果;7、8、9、10:菌株125a–d的回补基因pamR1的PCR验证结果;11:阳性对照PCR结果;12:阴性对照PCR结果. B:1、2、3、4:菌株JCK121a–d的硫链丝菌素抗性基因PCR验证结果;5、6、7、8:菌株JCK125a–d的硫链丝菌素抗性基因PCR验证结果;9:阳性对照PCR结果;10:阴性对照PCR验证结果. C:相关菌株发酵产物的生物活性测试结果. Note: M: DNA ladder. A: 1, 2, 3, 4: The PCR validation results of JCK121a–d (R2-F/R2-R); 5: The PCR result of positive control (R2-F/R2-R); 6: The PCR result of negative result (R2-F/R2-R); 7, 8, 9, 10: The PCR validation results of JCK125a–d (R1-F/R1-R); 11: The PCR result of positive control (R1-F/R1-R); 12: The PCR result of negative result (R1-F/R1-R). B: 1, 2, 3, 4: The PCR validation results of JCK121a–d (Tsr-F/Tsr-R); 5, 6, 7, 8: The PCR validation results of JCK125a–d (Tsr-F/Tsr-R); 9: The PCR result of positive control (Tsr-F/Tsr-R); 10: The PCR result of negative result (Tsr-F/Tsr-R). C: The result of bioactivity against M. smegmattis mc2155 of the cultures from related strains. |
|
|
为了进一步验证pamR1和pamR2基因的功能,通过PCR扩增得到pamR1和pamR2基因,测序正确后将其克隆到整合型载体pPM927上,并处于PermE*启动子的控制下,通过接合转移分别回补进入相应基因缺失突变株JCK117和JCK113中。每个基因回补菌株选取3个接合子,分别使用R1-F/R1-R、R2-F/R2-R和tsr-F/tsr-R引物,分别将验证正确的菌株命名为JCK125a–c (pamR1基因回补菌株)和JCK121a–c (pamR2基因回补菌株)。将JCK125、JCK121与对照菌株(JCK109a、JCK110、JCK113和JCK117)进行平行发酵,再平行提取发酵产物进行生物活性测定,结果显示pamR1和pamR2基因回补以后使得帕马霉素的合成能力恢复到了JCK109菌株的产生水平,该结果进一步说明了基因pamR1和pamR2在帕马霉素的生物合成过程中扮演着负调控的角色。
3 讨论与结论在链霉菌异源宿主中,利用异源表达技术可以快速鉴定生物合成基因和调控基因的功能。帕马霉素的生物合成基因簇全长为32 kb,完全容纳于1个Fosmid质粒中,便于在异源链霉菌宿主中进行表达研究。本研究从林可链霉菌NRRL 2936的基因组文库中快速筛选得到了含有帕马霉素生物合成完整基因簇的Fosmid质粒pJQK450,然后选择不同的链霉菌宿主进行异源表达,最终在天蓝色链霉菌M1154的发酵产物中检测到了帕马霉素的产生。该结果表明帕马霉素生物合成基因簇在不同链霉菌宿主中的表达情况差异较大,为以后对相关次生代谢产物生物合成基因簇异源表达的宿主选择提供了借鉴和参考。
天蓝色链霉菌M1152和天蓝色链霉菌M1154最主要的差别是后者含有了优化的rpsL (A262G)基因,该基因负责编码核糖体蛋白S12,引入该基因可以避免抗生素对菌株造成损伤,从而提升抗生素的产量[15, 23]。帕马霉素生物合成基因簇在天蓝色链霉菌M1154和天蓝色链霉菌M1152中异源表达结果的差异,可能暗示了rpsL (A262G)基因的引入可以提升天蓝色链霉菌M1154对帕马霉素的耐受性,促进了帕马霉素的产生。此外,rpsL基因是来自于链霉素生物合成基因簇中的抗性基因,链霉素可以与结核杆菌核糖体受体蛋白结合,从而抑制蛋白质的合成,实现杀灭或者抑制结核杆菌生长的作用[24]。帕马霉素对耻垢分枝杆菌也具有较好的抑制活性,其作用机制目前尚不清楚,但可能与链霉素的作用机制具有一定的类似性。
在链霉菌中,途径特异性调控因子一般与次生代谢产物生物合成基因簇中的功能基因成簇排列,特异性地调控了一个或多个功能基因的表达,进而影响目标次生代谢产物的生物合成的过程[9]。在帕马霉素生物合成基因簇的左右两端分别含有属于TetR家族的pamR2调控基因和属于LuxR家族的pamR1调控基因[4]。TetR家族的调控因子在许多放线菌的次生代谢产物中均有分布,大部分情况下负调控目标次生代谢产物的生物合成过程,如在阿维链霉菌中SAV576负调控阿维菌素的生物合成[25]。LuxR家族的蛋白在次生代谢产物生物合成过程中存在着正调控和负调控的现象,如在不吸水链霉菌武夷变种CK-15中WysR正调控武夷菌素的生物合成过程[26],在海洋放线菌Saccharomonospora sp. CNQ-490中Tar20负调控Taromycin A的生物合成[27]。为了详细的确定帕马霉素生物合成基因簇中pamR1和pamR2两个调控基因的功能,本研究在异源表达宿主菌株中通过基因置换与回补的策略,快速确定了这两个调控因子对帕马霉素生物合成过程起到负调控的作用。这些调控基因功能的探讨,为进一步通过功能基因的高效表达实现帕马霉素的产量优化提供了借鉴。
| [1] |
McCann PA, Pogell BM. Pamamycin: a new antibiotic and stimulator of aerial mycelia formation[J]. The Journal of Antibiotics, 1979, 32(7): 673-678. |
| [2] |
Kondo S, Yasui K, Natsume M, et al. Isolation, physico-chemical properties and biological activity of pamamycin-607, an aerial mycelium-inducing substance from Streptomyces alboniger[J]. The Journal of Antibiotics, 1988, 41(9): 1196-1204. |
| [3] |
Hashimoto M, Komatsu H, Kozone I, et al. Biosynthetic origin of the carbon skeleton and nitrogen atom of pamamycin-607, a nitrogen-containing polyketide[J]. Bioscience, Biotechnology, and Biochemistry, 2005, 69(2): 315-330. DOI:10.1271/bbb.69.315 |
| [4] |
Rebets Y, Br tz E, Manderscheid N, et al. Insights into the pamamycin biosynthesis[J]. Angewandte Chemie International Edition, 2015, 54(7): 2280-2284. DOI:10.1002/anie.201408901 |
| [5] |
Hanquet G, Salom-Roig X, Lanners S. New insights into the synthesis and biological activity of the pamamycin macrodiolides[J]. CHIMIA International Journal for Chemistry, 2016, 70(1): 20-28. DOI:10.2533/chimia.2016.20 |
| [6] |
Niu GQ, Li L, Wei JH, et al. Cloning, heterologous expression, and characterization of the gene cluster required for gougerotin biosynthesis[J]. Chemistry & Biology, 2013, 20(1): 34-44. |
| [7] |
Niu GQ, Tan HR. Biosynthesis and regulation of secondary metabolites in microorganisms[J]. Science China Life Sciences, 2013, 56(7): 581-583. DOI:10.1007/s11427-013-4501-5 |
| [8] |
Komatsu M, Uchiyama T, Ōmura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(6): 2646-2651. DOI:10.1073/pnas.0914833107 |
| [9] |
Liu G, Chater KF, Chandra G, et al. Molecular regulation of antibiotic biosynthesis in Streptomyces[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. DOI:10.1128/MMBR.00054-12 |
| [10] |
Paget MS, Chamberlin L, Atrih A, et al. Evidence that the extracytoplasmic function sigma factor sigmaE is required for normal cell wall structure in Streptomyces coelicolor A3(2)[J]. Journal of Bacteriology, 1999, 181(1): 204-211. |
| [11] |
Fu J, Bian XY, Hu SB, et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J]. Nature Biotechnology, 2012, 30(5): 440-446. DOI:10.1038/nbt.2183 |
| [12] |
Chen L, Wang YM, Guo H, et al. High-throughput screening for Streptomyces antibiotic biosynthesis activators[J]. Applied and Environmental Microbiology, 2012, 78(12): 4526-4528. DOI:10.1128/AEM.00348-12 |
| [13] |
Meng ST, Wu H, Wang L, et al. Enhancement of antibiotic productions by engineered nitrate utilization in actinomycetes[J]. Applied Microbiology and Biotechnology, 2017, 101(13): 5341-5352. DOI:10.1007/s00253-017-8292-7 |
| [14] |
Zhao ZL, Shi T, Xu M, et al. Hybrubins: bipyrrole tetramic acids obtained by crosstalk between a truncated undecylprodigiosin pathway and heterologous tetramic acid biosynthetic genes[J]. Organic Letters, 2016, 18(3): 572-575. DOI:10.1021/acs.orglett.5b03609 |
| [15] |
Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters[J]. Microbial Biotechnology, 2011, 4(2): 207-215. DOI:10.1111/j.1751-7915.2010.00219.x |
| [16] |
Poupin P, Ducrocq V, Hallier-Soulier S, et al. Cloning and characterization of the genes encoding a cytochrome P450 (PipA) involved in piperidine and pyrrolidine utilization and its regulatory protein (PipR) in Mycobacterium smegmatis mc2155[J]. Journal of Bacteriology, 1999, 181(11): 3419-3426. |
| [17] |
Li L, Xu ZN, Xu XY, et al. The mildiomycin biosynthesis: initial steps for sequential generation of 5-hydroxymethylcytidine 5'-monophosphate and 5-hydroxymethylcytosine in Streptoverticillium rimofaciens ZJU5119[J]. ChemBioChem, 2008, 9(8): 1286-1294. DOI:10.1002/cbic.v9:8 |
| [18] |
Smokvina T, Mazodier P, Boccard F, et al. Construction of a series of pSAM2-based integrative vectors for use in actinomycetes[J]. Gene, 1990, 94(1): 53-59. DOI:10.1016/0378-1119(90)90467-6 |
| [19] |
He YL, Zhu DQ, Bai LQ, et al. Cloning of antibiotic biosynthetic gene clusters from genomic library by narrow-down polymerase chain reaction[J]. Journal of Shanghai Jiao Tong University, 2009, 43(1): 5-8. 何云龙, 朱冬青, 白林泉, 等. 聚合酶链式反应递缩基因组文库以克隆抗生素生物合成基因簇[J]. 上海交通大学学报, 2009, 43(1): 5-8. |
| [20] |
Sambrook J, Russell DW. The Condensed Protocols from-Molecular Cloning: A Laboratory Manual[M]. Huang PT, Trans. Beijing: Chemical Industry Press, 2008: 50-51 (in Chinese) 萨姆布鲁克J, 拉塞尔DW. 分子克隆实验指南精编版[M]. 黄培堂, 译. 北京: 化学工业出版社, 2008: 50-51 |
| [21] |
Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces Genetics[M]. Colney: John Innes Foundation, 2000, 125-158.
|
| [22] |
Liu DM, Li L, Yang XQ, et al. Determination of the antimicrobial activity of probiotic by oxford plate assay system[J]. Food Research and Development, 2006, 27(3): 110-111. 刘冬梅, 李理, 杨晓泉, 等. 用牛津杯法测定益生菌的抑菌活力[J]. 食品研究与开发, 2006, 27(3): 110-111. |
| [23] |
Okamoto-Hosoya Y, Okamoto S, Ochi K. Development of antibiotic-overproducing strains by site-directed mutagenesis of the rpsL gene in Streptomyces lividans[J]. Applied and Environmental Microbiology, 2003, 69(7): 4256-4259. DOI:10.1128/AEM.69.7.4256-4259.2003 |
| [24] |
Kaur S, Sharma P. Protease-sensitive inhibitory activity of cell-free supernatant of Lactobacillus crispatus 156 synergizes with ciprofloxacin, moxifloxacin and streptomycin against Pseudomonas aeruginosa: an in vitro study[J]. Probiotics and Antimicrobial Proteins, 2015, 7(2): 172-180. DOI:10.1007/s12602-015-9188-4 |
| [25] |
Guo J, Zhang X, Luo S, et al. A novel TetR family transcriptional regulator, SAV576, negatively controls avermectin biosynthesis in Streptomyces avermitilis[J]. PLoS One, 2013, 8(8): e71330. DOI:10.1371/journal.pone.0071330 |
| [26] |
Liu YY, Ryu H, Ge BB, et al. Improvement of wuyiencin biosynthesis in Streptomyces wuyiensis CK-15 by identification of a key regulator, WysR[J]. Journal of Microbiology and Biotechnology, 2014, 24(12): 1644-1653. DOI:10.4014/jmb.1405.05017 |
| [27] |
Yamanaka K, Reynolds KA, Kersten RD, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(5): 1957-1962. DOI:10.1073/pnas.1319584111 |
 2018, Vol. 45
2018, Vol. 45