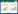扩展功能
文章信息
- 王彦杰, 侯强川, 李伟程, 余中节, 王旭, 张和平, 钟智
- WANG Yan-Jie, HOU Qiang-Chuan, LI Wei-Cheng, YU Zhong-Jie, WANG Xu, ZHANG He-Ping, ZHONG Zhi
- 不同分离源粪肠球菌的毒力基因比较
- Comparison of the virulence genes of Enterococcus faecalis isolated from different environments
- 微生物学通报, 2018, 45(12): 2695-2707
- Microbiology China, 2018, 45(12): 2695-2707
- DOI: 10.13344/j.microbiol.china.180026
-
文章历史
- 收稿日期: 2018-01-10
- 接受日期: 2018-06-12
- 网络首发日期(www.cnki.net): 2018-06-26
粪肠球菌(Enterococcus faecalis)是一种产乳酸革兰氏阳性需氧或兼性厌氧乳酸菌[1-3], 具有极强的耐受极端温度、pH值、高胆汁酸、高盐和抗菌剂的能力[1]。粪肠球菌的特点使其在自然界中广泛分布, 除人和哺乳动物肠道外, 在尿液、血液、土壤、水和食品中均有存在[2]。
作为一种重要的乳酸菌, 粪肠球菌在食品中的应用十分广泛。在一些欧美国家, 粪肠球菌常添加在鲜奶和巴氏消毒奶中制作各种干酪, 它可以通过分解脂类和蛋白质, 产生芳香类化合物改善干酪的口感和风味[4]。同时部分粪肠球菌能产生细菌素, 有效抑制致病菌如单核细胞增多性李斯特菌、梭状芽孢杆菌、金黄色葡萄球菌等的生长[5], 对食品保鲜起到重要作用。另外, 粪肠球菌是人和动物的肠道共生菌, 部分菌株具有益生作用, 在医学领域常被用于治疗腹泻[3]。
然而, 考虑到很多粪肠球菌为条件致病菌, 具有潜在的致病风险[6], 在食品医药领域应用粪肠球菌仍需要进一步评估。目前已经发现很多疾病如尿路感染[7]、败血症、血液感染[8]、心内膜炎[9]和中枢神经系统感染[10]等均与粪肠球菌密切相关。粪肠球菌成为医院致病菌与其可以获得毒力特性的能力有关[11], 同时其毒力因子基因传播性较强, 加强了粪肠球菌对人们的危害性。充分了解粪肠球菌基因组中毒力基因的携带情况对合理利用该菌种和预防其感染有重要的意义。2006年Lepage等首次大规模研究粪肠球菌基因组多样性, 给出了粪肠球菌染色体的稳定区和可变区, 该研究最重要的是在30株临床和食品粪肠球菌分离株中发现了6个乳制品分离株中不存在的基因, 它们可以成为发酵产物中粪肠球菌分离株风险评估的良好标志物[12]。Bakshi等在2016年利用比较基因组学方法评估粪肠球菌菌株的潜在致病性, 并确定了一个粪肠球菌毒力岛富集模块[13]。然而, 目前鲜有研究报道多分离源环境粪肠球菌基因组中毒力基因携带情况, 粪肠球菌乳源分离株是否更安全等都是亟需解决的问题。
为了全面评估自然发酵乳制品中粪肠球菌的安全性, 本研究利用比较基因组学方法分析19株乳源粪肠球菌与其他88株分离自血液、尿液、粪便和水源粪肠球菌携带毒力基因情况, 对比分析乳源分离株携带的毒力基因与其他分离源菌株携带毒力基因子是否存在差异, 进而评估粪肠球菌乳源分离株的安全性。
1 材料与方法 1.1 实验菌株本研究的107株粪肠球菌均具有明确的分离源, 测序质量较好, 其中包含29株血液分离株, 19株乳源分离株, 26株粪便分离株, 21株尿液分离株和12株水源分离株。所有菌株基因组均下载自NCBI的GenBank数据库(http://www.ncbi.nlm.nih.gov/genbank/), 具体菌株信息如表 1所示。
| 菌株 Isolates |
分离源 Origin |
测序技术 Sequencing technology |
基因组覆盖度 Genome coverage |
NCBI序列号 NCBI accession No. |
| V583 | Blood | - | - | AE016830.1 |
| MA1 | Blood | Illumina HiSeq | 834.0× | ANMP00000000.1 |
| R712 | Blood | Illumina | 67.4× | ADDQ00000000.1 |
| S613 | Blood | Illumina | 71.3× | ADDP00000000.1 |
| TX0012 | Blood | Illumina | 76.2× | AECD00000000.1 |
| TX0017 | Blood | Illumina | 81.3× | AEBP00000000.1 |
| TX0102 | Blood | Illumina | 76.8× | AEBD00000000.1 |
| TX0031 | Blood | Illumina | 76.1× | AEBF00000000.1 |
| DAPTO 516 | Blood | Illumina | 71.1× | AEBS00000000.1 |
| 18297 | Blood | Illumina | 216.0× | AIIS00000000.1 |
| B1290 | Blood | Illumina | 136.0× | AIJH00000000.1 |
| B1327 | Blood | Illumina | 134.0× | AIJI00000000.1 |
| B1376 | Blood | Illumina | 131.0× | AIJJ00000000.1 |
| SF21521 | Blood | Illumina | 300.0× | AJEP00000000.1 |
| RM3817 | Blood | Illumina | 150.0× | AJEV00000000.1 |
| B1005 | Blood | Illumina | 284.0× | AIPS00000000.1 |
| B1138 | Blood | Illumina | 291.0× | AIPT00000000.1 |
| B1249 | Blood | Illumina | 318.0× | AIPU00000000.1 |
| B1851 | Blood | Illumina | 126.0× | AIQG00000000.1 |
| B1921 | Blood | Illumina | 153.0× | AIQI00000000.1 |
| B2277 | Blood | Illumina | 268.0× | AIQO00000000.1 |
| B2670 | Blood | Illumina | 149.0× | AIQU00000000.1 |
| B2685 | Blood | Illumina | 149.0× | AIQV00000000.1 |
| B2687 | Blood | Illumina | 149.0× | AIQW00000000.1 |
| B2802 | Blood | Illumina | 271.0× | AIQX00000000.1 |
| B2813 | Blood | Illumina | 155.0× | AIQY00000000.1 |
| B2864 | Blood | Illumina | 301.0× | AIQZ00000000.1 |
| NGS-ED-1009 | Blood | Illumina | 222.0× | JPWN00000000.1 |
| MMH594 | Blood | Illumina | 500.0× | AOPW00000000.1 |
| DM7-2* | Dairy | Illumina HiSeq | 175.9× | MSQG00000000 |
| ELS8-4* | Dairy | Illumina HiSeq | 197.8× | MSQH00000000 |
| HS5152* | Dairy | Illumina HiSeq | 247.0× | MSQI00000000 |
| HS5302* | Dairy | Illumina HiSeq | 219.0× | MSQJ00000000 |
| MGA44-7* | Dairy | Illumina HiSeq | 225.1× | MSQK00000000 |
| NM15-4* | Dairy | Illumina HiSeq | 206.1× | MSQL00000000 |
| NM31-3* | Dairy | Illumina HiSeq | 230.2× | MSQM00000000 |
| QH29-4* | Dairy | Illumina HiSeq | 220.7× | MSQN00000000 |
| QH9-5* | Dairy | Illumina HiSeq | 219.3× | MSQO00000000 |
| WZ21-1* | Dairy | Illumina HiSeq | 216.7× | MSQP00000000 |
| WZ34-2* | Dairy | Illumina HiSeq | 189.1× | MSQQ00000000 |
| XJ76305* | Dairy | Illumina HiSeq | 222.5× | MSQR00000000 |
| XM26-4* | Dairy | Illumina HiSeq | 201.3× | MSQS00000000 |
| YM11-6* | Dairy | Illumina HiSeq | 189.5× | MSQT00000000 |
| YM39-2* | Dairy | Illumina HiSeq | 194.2× | MSQU00000000 |
| F1 | Dairy | Illumina | 292.0× | ASDL00000000.1 |
| SS-7 | Dairy | Illumina | 151.0× | ASDM00000000.1 |
| ATCC 6055 | Dairy | Illumina | 217.0× | ASDZ00000000.1 |
| LD33 | Dairy | Illumina | 328.0× | CP014949.1 |
| L9 | gut | Illumina HiSeq | 310.0× | CP018004.1 |
| L12 | Gut | Illumina HiSeq | 250.0× | CP018102.1 |
| E1Sol | Gut | 454 | 41.6× | ACAQ00000000.1 |
| PC1.1 | Gut | 454 | 30.0× | ADKN00000000.1 |
| Symbioflor 1 | Gut | - | - | HF558530.1 |
| DORA_14 | Gut | Illumina HiSeq | 255.0× | AZLY00000000.1 |
| TX1302 | Gut | 454 | 83.4× | AEBK00000000.1 |
| 7330082-2 | Gut | Illumina | 127.0× | AIIW00000000.1 |
| 7330112-3 | Gut | Illumina | 250.0× | AIIX00000000.1 |
| 7330245-2 | Gut | Illumina | 122.0× | AIIY00000000.1 |
| 7330257-1 | Gut | Illumina | 267.0× | AIIZ00000000.1 |
| 7430821-4 | Gut | Illumina | 250.0× | AIJG00000000.1 |
| HEF39 | Gut | Illumina | 143.0× | AISB00000000.1 |
| T16 | Gut | Illumina | 152.0× | AJEI00000000.1 |
| T21 | Gut | Illumina | 142.0× | AJEQ00000000.1 |
| P9-1 | Gut | Illumina | 62.0× | LKGS00000000.1 |
| Enfs51 | Gut | Illumina | 378.0× | MJEK00000000.1 |
| Enfs85 | Gut | Illumina | 297.0× | MJBV00000000.1 |
| P.En250 | Gut | Illumina | 396.0× | MJBZ00000000.1 |
| DBH18 | Gut | Illumina | 50.0× | LSFS00000000.1 |
| TX1467 | Gut | Illumina | 241.1× | AFBS00000000.1 |
| TX1346 | Gut | Illumina | 76.9× | AEBI00000000.1 |
| TX2137 | Gut | Illumina | 71.7× | AEBQ00000000.1 |
| TX4244 | Gut | Illumina | 87.3× | AEBH00000000.1 |
| X98 | Gut | 454 | 33.7× | ACAW00000000.1 |
| D32 | Gut | - | - | CP003726.1 |
| ATCC 29200 | Urine | Illumina | 134.0× | ACHK00000000.1 |
| HH22 | Urine | Illumina | 291.0× | ACIX00000000.1 |
| HS0914 | Urine | Illumina HiSeq | 102.0× | JPDQ00000000.1 |
| T14 | Urine | Illumina | 141.0× | AJBE00000000.1 |
| T13 | Urine | 454 | 283.0× | AJEJ00000000.1 |
| T3 | Urine | 454 | 39.4× | ACAF00000000.1 |
| EnGen0311 | Urine | 454 | 53.5× | AEBZ00000000.1 |
| ERV81 | Urine | Illumina | 75.0× | ALZW00000000.1 |
| ERV85 | Urine | Illumina | 79.0× | ALZX00000000.1 |
| ERV93 | Urine | Illumina | 80.0× | ALZY00000000.1 |
| SF24397 | Urine | Illumina | 123.0× | AJAW00000000.1 |
| V587 | Urine | Illumina | 147.0× | AJBB00000000.1 |
| SF28073 | Urine | Illumina | 155.0× | AJBC00000000.1 |
| WH257 | Urine | Illumina | 148.0× | AJBG00000000.1 |
| SF19 | Urine | Illumina | 119.0× | AJBH00000000.1 |
| WH571 | Urine | Illumina | 140.0× | AJBJ00000000.1 |
| TX0635 | Urine | Illumina | 144.0× | AJEF00000000.1 |
| CH136 | Urine | Illumina | 265.0× | AJEH00000000.1 |
| SF24396 | Urine | Illumina | 146.0× | AJEM00000000.1 |
| LRS29212 | Urine | Illumina | 500.0× | JPTY00000000.1 |
| F165 | Urine | Illumina | 74.0× | MBRC00000000.1 |
| AZ19 | Water | Illumina HiSeq | 100.0× | AYLU00000000.1 |
| FL2 | Water | Illumina HiSeq | 100.0× | AYKK00000000.1 |
| GA2 | Water | Illumina HiSeq | 100.0× | AYKL00000000.1 |
| GAN13 | Water | Illumina HiSeq | 100.0× | AYLV00000000.1 |
| KS19 | Water | Illumina HiSeq | 100.0× | AYND00000000.1 |
| MD6 | Water | Illumina HiSeq | 100.0× | AYLN00000000.1 |
| MN16 | Water | Illumina HiSeq | 100.0× | AYKM00000000.1 |
| MTmid8 | Water | Illumina HiSeq | 100.0× | AYKU00000000.1 |
| MTUP9 | Water | Illumina HiSeq | 100.0× | AYOJ00000000.1 |
| NJ44 | Water | Illumina HiSeq | 100.0× | AYOK00000000.1 |
| NY9 | Water | Illumina HiSeq | 100.0× | AYOL00000000.1 |
| AMB05 | Water | Illumina | 2.7× | CP015998.1 |
| 注:*:本实验室前期在自然发酵乳制品中分离得到的粪肠球菌菌株. Note: *: Isolated from naturally fermented dairy products. |
||||
将107株粪肠球菌全基因组蛋白序列分别与毒力因子数据库(Virulence factor database, VFDB; http://www.mgc.ac.cn/VFs/main.htm)[14]中的Protein sequences of full dateset进行比对, 将比对结果中相似度>50%, E < 1e-10, 匹配长度>300的蛋白序列确定为毒力基因。
1.2.2 毒力基因差异分析使用R语言软件(v3.3.2)中的Prcomp函数进行主成分分析(Principal component analysis, PCA), 比较不同环境分离株毒力基因整体差异。使用BLAST软件(v2.2.24+)将毒力基因序列与粪肠球菌全基因组序列进行对比, 获取各毒力基因在粪肠球菌基因组中的位置, 通过R语言ggplot2包将数据可视化。使用MEGA 5.0软件将共有毒力基因蛋白序列串联后构建系统发育树, 重复值为1 000。随后将人工校对后的9个共有基因以VFG000079- VFG000080-VFG005527-VFG012103-VFG016490-VFG016490-VFG030724-VFG032200-VFG032791的顺序依次导入BioNumerics v6.0软件进行等位基因型的确认, 对应的每一个等位基因有一个等位基因序列号, 最后将9个等位基因序列号组合构建107株粪肠球菌毒力基因的ST型。
1.2.3 环境特异性毒力基因分析如果一个毒力基因在某个环境出现频次显著高于其在所有环境分离株出现频次, 那么该毒力基因具有环境特异性, 称为环境特异性毒力基因。例如, 毒力基因A在107株菌中出现19次, 出现频次为17.8%, 而在19株乳源分离株中有16次, 频次为84.2%, 则该毒力基因很可能是乳源特异性的毒力基因, 为了确定结果是否具有统计学意义, 本研究采用Pearson’s chi-squared检验(SPSS 17.0软件), 将检验结果P < 0.05的毒力基因认定为环境特异性毒力基因。
2 结果与讨论 2.1 粪肠球菌毒力基因的鉴定通过与毒力因子数据库(VFDB)进行比对, 在107株粪肠球菌基因组中共找到88种毒力基因, 平均每株粪肠球菌携带42种毒力基因。其中Enterococcus faecalis B1005 (血液)、Enterococcus faecalis B2687 (血液)和Enterococcus faecalis B2802 (血液)携带毒力基因数最多, 为54种, Enterococcus faecalis NM31-3 (乳源)、Enterococcus faecalis YM11-6 (乳源)、Enterococcus faecalis AMB05 (水源)、Enterococcus faecalis FL2 (水源)和Enterococcus faecalis P9-1携带毒力基因数最少, 均携带32种。
统计结果显示, 粪肠球菌中携带的毒力因子主要包括粘附素、荚膜多糖、胞外酶、表面蛋白和溶血素激活因子等, 如表 2所示。这些毒力基因在粪肠球菌感染宿主时通过编码毒力因子使粪肠球菌对宿主细胞产生粘附、定殖及抗吞噬和入侵等作用[15]。
| 编码基因功能 Coding gene function |
毒力基因 Virulence genes name |
| 粘附素Adhesins | ace, ebpA/ebpB/ebpC, efaA, EFD32-0899, OG1RF-10869, EFD32-0908, OG1RF-10870/OG1RF-10871, ecbA, EF2347, lap, ssaB, pavA, EFD32-0900, cnm, asa1, prgB/asc10, EF0149, dltA, tig/ropA |
| 荚膜多糖Capsule | STER-1222, rfbB-1, capE/capD/cap8F/capP, galE, SGO 1723, manA, cpsG/cpsE/cpsD/cpsJ/cpsI/cpsC/ cps4I/cps2K/cps2T, ABTJ-03752, wbfV/wcvB, BCE-5394, SSP0068 |
| 溶血素Cytolysin | cylM, cylB, cylI, cylA |
| 产胞外酶Protease | gelE, EFD32-2606, EF3023, OG1RF-12303, EF0818, OG1RF-10550, EFD32-0629, lplA1, htrA/degP, eno, lisK/bsh, plr/gapA, fsrC, sigA/rpoV, tuf, katA |
| 表面蛋白Surface protein | groEL, bopD, EFD32-0765, esp, sugC, ctpV, CT396, srt2/srt1/bee1/bee3, clpC, clpE, fss1/fss2/fss3, EF0485 |
| 注:根据VFDB数据库提供的表达蛋白对菌株致病性产生的影响将毒力基因进行分类, “, ”分开的基因代表编码不同蛋白的毒力基因, “/”分开的为不同基因但编码功能相似蛋白. Note: Virulence genes were classified according to the effects of the expressed proteins provided by VFDB database on the pathogenicity of the strains, “, ”: Virulence genes encoding different proteins, “/”: Virulence genes encoding similar proteins. |
|
由表 2可知, 在粪肠球菌众多毒力基因中参与粘附的基因最多。粘附基因可以编码粘附力相关表面蛋白, 这些蛋白促使细菌粘附在宿主细胞上, 辅助细菌侵染宿主细胞, 其中胶原结合蛋白Ace和心内膜炎抗原EfaA是粪肠球菌重要的表面粘附蛋白, Ace与生物膜相关[16], EfaA和心内膜炎发病有关[17]。产细胞表面蛋白的Esp最初在粪肠球菌临床分离株中被发现, 其可以合成细胞壁相关蛋白, 促进粪肠球菌粘附和定殖[18], 带有该基因的菌株可引起多重感染。体外试验也证明Esp蛋白可在聚苯乙烯或聚氯乙稀表面形成生物膜, 形成的生物膜可以使细菌的感染更加持久[19]。在粪肠球菌中还发现了能够介导血小板、胶原和纤维蛋白原粘附, 从而引起心内膜炎的ebp菌毛相关毒力基因, 如ebpA、ebpB和ebpC, 这些基因可以编码菌毛蛋白, 调节菌毛生物作用[18], 研究证明ebp菌毛基因缺失突变株对血小板、胶原以及纤维蛋白原的粘附能力均显著降低[20]。
在粪肠球菌基因组中存在较多与荚膜多糖合成相关的毒力基因, 如capE/cap8E/capD和wbfV/wcvB。对于很多人类病原菌, 荚膜多糖在细菌免疫逃逸方面扮演着重要角色[21]。它可以通过多重机制包括抵抗吞噬和隐蔽细菌表面抗原等方法增强微生物毒力[22]。
粪肠球菌携带的细胞溶解素是唯一一种可以裂解真核细胞的羊毛硫细菌素(Lantibiotics)[23], 能裂解红细胞、血小板和巨噬细胞等, 其作用于细胞膜会造成细胞结构和功能紊乱, 并最终导致细胞死亡[24]。研究证明表达溶血素肠球菌的毒力明显高于其他同型不表达溶血素的菌株[25]。
与粪肠球菌生物膜形成相关的编码基因gelE可编码产生水解明胶、酪蛋白、血红蛋白等生物活性肽, 介导自溶素促使细胞裂解, 促进细菌扩散, 参与炎症进程。因此, gelE基因也被认为是粪肠球菌中较为重要的胞外酶基因, 其与细菌毒性密切相关。另一个重要的粪肠球菌胞外酶是丝氨酸酶, 其由基因sprE编码产生, 丝氨酸酶能增加粪肠球菌的致病性, 同时避免粪肠球菌发生自杀行为[26]。
虽然很多毒力因子编码基因单独存在于某些非致病菌中没有使细菌表现出致病性, 但是多种毒力因子之间的相互作用使得细菌的致病性明显增强。
2.2 毒力基因拷贝数分析基于上述结果发现粪肠球菌携带的毒力基因多样性较高。此外, 每种毒力基因在粪肠球菌基因组中的拷贝数对菌株安全性也非常重要, 拷贝数越多, 表达的可能性越大。鉴于此, 统计了88种毒力基因在各菌株中拷贝数分布情况, 其中有11个毒力基因出现了多拷贝, 统计结果如表 3所示。
| 毒力基因 Virulence genes |
多拷贝数分离源 Origin |
拷贝数 Copy number |
菌株数 Number of strains |
| EF0485 | Blood | 2 | 3/29, 10.3% |
| Urine | 2 | 3/21, 14.3% | |
| esp | Blood | 2 | 2/29, 6.9% |
| 3 | 2/29, 6.9% | ||
| Dairy | ≥3 | 1/19, 5.3% | |
| Urine | 2 | 2/21, 9.5% | |
| Gut | 2 | 2/26, 7.7% | |
| ≥3 | 1/26, 3.8% | ||
| galE | Blood | 2 | 21/29, 72.4% |
| Dairy | 2 | 4/19, 21.1% | |
| Water | 2 | 1/12, 8.3% | |
| Urine | 2 | 13/21, 61.9% | |
| Gut | 2 | 15/26, 57.7% | |
| fss2 | Blood | 2 | 17/29, 58.6% |
| ≥3 | 1/29, 3.4% | ||
| Dairy | 2 | 14/19, 73.7% | |
| Water | 2 | 4/12, 33.3% | |
| Urine | 2 | 6/21, 28.6% | |
| Gut | 2 | 16/26, 61.5% | |
| fss1 | Dairy | 2 | 3/19, 15.8% |
| Water | 2 | 1/12, 8.3% | |
| Gut | 2 | 1/26, 3.8% | |
| ≥3 | 1/26, 3.8% | ||
| EF3023 | Dairy | 2 | 1/19, 5.3% |
| bsh | Gut | 2 | 2/26, 7.7% |
| fss3 | Urine | 2 | 2/21, 9.5% |
| cps4I | Gut | 2 | 1/26, 3.8% |
| lplA1 | Blood | 2 | 1/29, 3.4% |
| dltA | Blood | 2 | 129, 3.4% |
根据毒力基因拷贝数分析可知, esp、fss2和galE在多个分离源的菌株中均出现了多拷贝, 并且携带多拷贝的菌株数占各分离源总菌株数的比例较高。其中esp和fss2为粘附素基因, 辅助细菌定殖, galE为荚膜多糖基因, 帮助细菌逃避免疫反应, 这两类基因在粪肠球菌毒力基因中普遍存在, 对粪肠球菌致病性起到很大的辅助作用。
2.3 毒力基因在基因组中分布情况同时, 为了了解众多毒力基因的分布规律, 将毒力基因与本研究中仅有的6株具有完成图的粪肠球菌全基因组序列进行比对, 获取各毒力基因在粪肠球菌基因组中的位置, 结果如图 1所示。
|
| 图 1 毒力基因在基因组中分布 Figure 1 Distribution of virulence factor in the genome 注:横坐标表示菌株基因组位置, 0为基因组起始, 1为基因组末尾.纵坐标表示在粪肠球菌基因组该位置存在的毒力基因个数, 即该位置毒力基因的密度. A: Enterococcus faecalis V583; B: Enterococcus faecalis L9; C: Enterococcus faecalis Symbioflor 1; D: Enterococcus faecalis LD33; E: Enterococcus faecalis L12; F: Enterococcus faecalis D32. Note: The abscissa indicates the genomic location of the strain, and the ordinate indicates the number of virulence genes present in the position of the Enterococcus faecalis genome, that is, the density of virulence genes at this position. A: Enterococcus faecalis V583; B: Enterococcus faecalis L9; C: Enterococcus faecalis Symbioflor 1; D: Enterococcus faecalis LD33; E: Enterococcus faecalis L12; F: Enterococcus faecalis D32. |
|
|
由图 1可知, 毒力基因在粪肠球菌全基因组各个位置均有分布, 同时不同菌株毒力基因分布情况不尽相同。然而, 研究发现一个共同的规律, 在以DnaA为起始基因情况下, 上述6菌株在基因组的1/3处均出现了一个毒力基因密集区。通过对这部分基因进行分析发现, 此处的毒力基因多为与细菌粘附相关的基因, 此外还存在一个ebp菌毛基因簇, 帮助粪肠球菌借助菌毛粘附到宿主组织细胞表面。
2.4 粪肠球菌共有的毒力基因分析107株粪肠球菌共有9种毒力因子编码基因, 热休克蛋白GroEL可提高细菌对宿主细胞的粘附能力[27]; ClpC和ClpE为100 kD热休克蛋白家族成员, 研究发现李斯特单核clpC突变体细胞入侵能力增强, 缺失clpC的突变体导致病毒因子InlA、InlB和ActA的转录减少, 进一步证实clpC对病原体的毒力有重要作用[28], ClpE与细菌中的4种有关毒力和致病相关的蛋白相互作用, 共同调节细菌的粘附和侵袭[29]; tuf基因编码EF-TU, EF-TU因作为病原相关分子模式而备受关注, Granato等研究发现Lactobacillus johnsonii NCC533 (La1)中携带该编码基因促进其对宿主细胞的粘附作用, 从而诱导了细胞的炎症反应[30]; 其他共有毒力因子包括Tig/RopA (起动因子), 有研究发现人畜患病病原体猪链球菌2型中的tig基因影响菌株的抗逆性和少数毒力基因的表达, 猪链球菌2型tig基因缺失株导致细菌粘附性显著降低, 溶血性缺失[31]; dltA操纵子编码D-丙氨酰, 参与整合脂磷壁酸和细胞壁磷壁酸合成, 对单增李斯特菌属的粘附和致病性有重要作用[32]。htrA/degP为丝氨酸蛋白酶编码基因, htrA/degP基因的缺失后影响肺炎链球菌表面蛋白A (PspA)、自溶素(LytA)和神经酰胺酶(NanA)这3个毒力因子的表达, 从而降低了链球菌的致病性[33]。根据这9种毒力基因编码蛋白功能发现均与致病菌粘附作用相关, 粘附是致病菌入侵宿主细胞的前提条件, 本研究的粪肠球菌中也均含有这9种毒力基因, 辅助了粪肠球菌的致病作用。
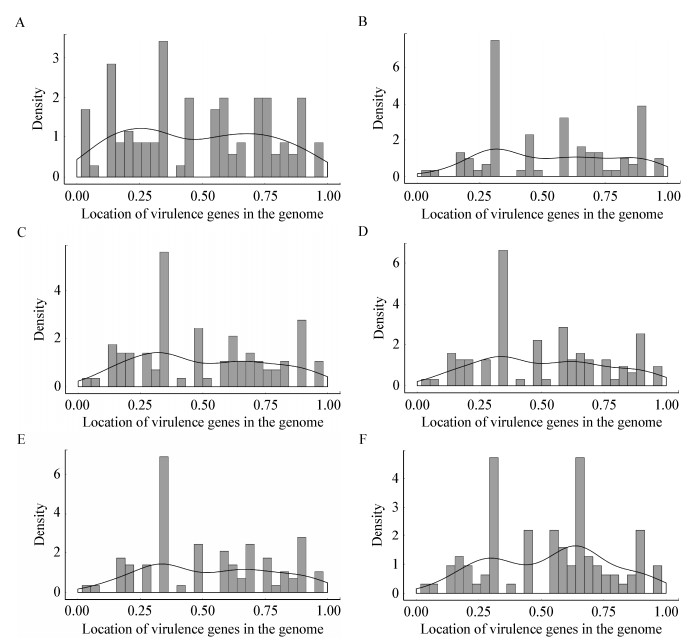
虽然所有菌株中均有这9种毒力基因, 但不同分离源菌株中携带的这些基因存在怎样的进化关系, 这些基因与分离源是否有明显的相关性?于是将每株粪肠球菌的9种毒力基因的编码序列串联后做系统发育树, 分析不同分离源菌株携带的共有毒力基因是否存在差异, 进一步评估乳源分离株中共有毒力基因是否与其他分离株之间存在分离现象, 结果如图 2所示。
|
| 图 2 共有毒力基因系统发育进化树 Figure 2 Phylogenetic tree with common virulence genes 注:红色:血液分离株; 蓝色:乳源分离株; 绿色:尿液分离株; 黄色:粪便分离株; 黑色:水源分离株; 标尺:不同菌株间遗传距离. Note: Red: Blood isolates; Blue: Dairy products isolates; Green: Urine isolates; Yellow: Gut isolates; Black: Water isolates; Scaleplate: Genetic distance between different strains. |
|
|
由图 2发现该系统发育树共分为两个分支, 分别为A分支和B分支, A分支中含有大部分血液分离株(21/29), 其中肠球菌导致的血液感染比例逐年增加, 血液分离株可能存在更大的致病风险, 由此推测处于A分支的分离株携带的毒力基因产生致病的可能性更大, 同时可以看出在该分支中存在10株粪肠球菌分离自乳源, 并没有和其他分离源出现明显分离现象, 这也预示着部分乳制品中存在的粪肠球菌也有致病的可能性。
通过多位点序列分型对107株粪肠球菌的9个关键基因进行分型, 最终将107株粪肠球菌分为70个ST型, 其中, 菌株数量最多的ST型为ST-1, 共包含17株粪肠球菌, 其中16株菌均分离自血液, 占总菌株数的15.9%, 是主要的序列型。其次含有菌株数较多的是ST-8和ST-21, 均有5株菌。其余ST型包含菌株在1-4株之间。根据菌株来源分析, 发现除了16株血液分离株存在明显相同的ST型外, 其余菌株大部分ST型不相同, 再一次显示出同一分离源菌株的基因具有多样性, 也表明不同分离源菌株毒力基因具有较大的遗传多样性。既然分离自乳源的粪肠球菌没有出现较为单一的分型, 也证实了在乳制品中的粪肠球菌毒力基因多样性较为丰富, 这增加了其致病的风险性。
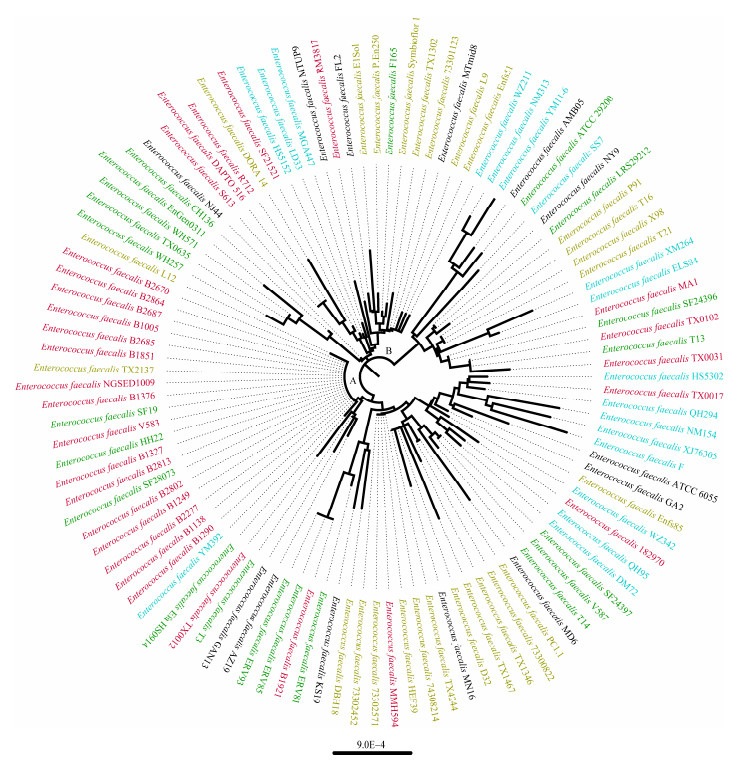
2.5 不同分离源粪肠球菌所含毒力基因粪肠球菌分布广泛, 不同分离源菌株所含毒力因子数量及种类差别是研究者所关心的问题。统计结果显示, 各分离源菌株平均携带毒力基因数量为:血液48 (37-54)种, 乳源39 (32-49)种, 粪便40 (32-47)种, 尿液44 (35-52)种, 水源36 (32-40)种, 经t检验发现水源分离株携带的毒力基因数量最少, 除乳源分离株与肠道分离株和水源分离株之间无显著差异外(P>0.05), 其他各环境分离株之间均有显著差异(P>0.05), 具体结果如图 3所示。
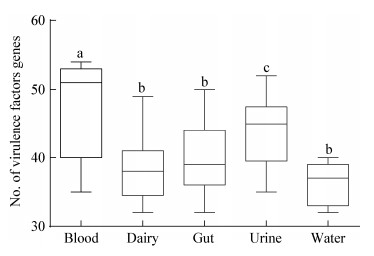
|
| 图 3 不同分离源菌株毒力基因携带情况 Figure 3 The number of virulence genes in isolates from different habitats |
|
|
然而更为关心的是不同分离源分离株所携带的毒力基因是否相同。为此, 本文进行了基于107株粪肠球菌分离株毒力基因分布结果的主成分分析, 结果如图 4所示。

|
| 图 4 菌株毒力基因聚类分析 Figure 4 PCA and clustering of virulence genes 注:基于菌株携带毒力基因存在与否的“1/0”矩阵做主坐标分析聚类图. Note: PCA and clustering of samples of 107 isolates of E. faecalis by using virulence genes presence-absence matrix ("1/0" matrix). |
|
|
由图 4发现, 除水源分离株主要集中在图片左下角, 血液、乳源、粪便和尿液分离株分布较为离散, 且不同分离源菌株相互重叠, 没有明显聚类趋势。说明上述分离源的菌株可能携带有较为相似的毒力基因, 再次证明菌株携带毒力基因与分离源无显著差异。这也预示着在粪肠球菌乳制品分离株与其他环境分离株一样, 同样存在致病风险。
值得关注的是2007年就有研究报道常作为益生菌的粪肠球菌菌株的Enterococcus faecalis Symbioflor 1与常见的致病菌株Enterococcus faecalis V583相比缺失部分编码肠球菌溶菌素, 明胶酶和透明质酸酶的毒力基因[34], 这可能是该菌株能成为益生菌的主要原因。然而在本研究分离自乳制品的粪肠球菌中发现部分菌株出现了这3种毒力基因, 这一结果再次证明粪肠球菌乳源分离株不一定安全。
2.6 环境特异性毒力基因107株菌中共发现7个环境特异性毒力基因, 其中血液特异性毒力基因3个, 乳源特异性毒力基因4个, 在尿液、粪便和水源分离株中未发现环境特异性毒力基因(表 4)。
| 毒力基因 Virulence genes |
分离源 Origin |
功能 Function |
总检出率 Detection rate (Total) (%) |
单分离源检出率 Detection rate (Single) (%) |
| esp | Blood | Enterococcal surface protein | 39/107, 35.8 | 20/29, 69 |
| EF0485 | Blood | Aggregation substance (AS) | 39/107, 35.8 | 22/29, 75.9 |
| ssaB | Blood | Manganese/Zinc ABC transporter substrate-binding lipoproteinprecursor | 51/107, 47.7 | 25/29, 86.2 |
| wbfV/wcvB | Dairy | Capsular polysaccharide | 13/107, 12.1 | 6/19, 31.6 |
| srt2 | Dairy | Biofilm enhancer in enterococci | 2/107, 1.9 | 2/19, 10.5 |
| srt1 | Dairy | Biofilm enhancer in enterococci | 2/107, 1.9 | 2/19, 10.5 |
| bee3 | Dairy | Biofilm enhancer in enterococci | 2/107, 1.9 | 2/19, 10.5 |
| bee1 | Dairy | Biofilm enhancer in enterococci | 2/107, 1.9 | 2/19, 10.5 |
血液分离株中特异性毒力基因主要包括esp基因, 在重症监护室分离的细菌中常携带该基因[35], 可增强菌株耐药能力[36]。同时在血液分离株中存在一个促进致病性的聚集物质(AS)编码基因EF0485, 该蛋白有助于粪肠球菌粘附在肠上皮细胞、肾小管细胞和心脏内皮细胞上, 常出现在粪肠球菌质粒中, 流动性较强, 容易发生转移[13]。毒力因子SsaB参与细菌金属离子转运, 尤其是介导细菌在低Mn2+环境下存活。2014年Crump等研究发现脂蛋白SsaB是一种重要的血链球菌毒力因子, 利于血链球菌感染宿主并引发心内膜炎[37]。这些血液特异性毒力因子都直接或者间接增强了粪肠球菌致病性, 促使粪肠球菌成为了医院最主要的感染菌株之一。
乳源分离株特异性毒力基因主要包括wbfV/ wcvB、srt1/srt2/bee1/bee3。wbfV/wcvB毒力基因主要参与编码荚膜多糖, 荚膜可以帮助细菌抵抗宿主免疫细胞的吞噬作用[38], 同时帮助细菌隐蔽其表面抗原[22], 荚膜多糖也因此在细菌免疫逃逸和增强细菌毒力方面扮演着重要角色[21]。毒力因子Srt1/Srt2/Bee1/Bee3为粪肠球菌生物膜增强因子[39], 其中有65%的人类细菌感染可能与细菌生物膜相关, 而这些毒力因子促进了生物膜形成, 增加了粪肠球菌的感染能力, 使细菌感染更持久。上述结果进一步说明存在于乳制品中的粪肠球菌具有一定程度致病风险性。
3 结论本研究在基因组层面揭示了不同分离源粪肠球菌菌株中所含有的毒力基因, 在107株粪肠球菌中共发现88种毒力基因, 分别参与菌株的粘附、抗吞噬和入侵。正是这些毒力基因的存在使得粪肠球菌的安全性一直受到质疑, 通过分析共有毒力基因的进化关系发现毒力基因多样性较高, 乳制品中分离的粪肠球菌也具有较多基因型, 无法证明其安全性。深入分析发现, 乳源分离株中毒力基因与其他环境分离株的毒力基因无显著差异, 因此在乳制品中使用粪肠球菌要格外注意, 即使是自然发酵乳中分离得到的粪肠球菌菌株, 在应用于食品工业中也必须经过严格的安全性评估后才能考虑应用。
| [1] |
Klare I, Werner G, Witte W. Enterococci. Habitats, infections, virulence factors, resistances to antibiotics, transfer of resistance determinants[J]. Contributions to Microbiology, 2001, 8: 108-122. DOI:10.1159/000060406 |
| [2] |
Paulsen IT, Banerjei L, Myers GSA, et al. Role of mobile DNA in the evolution of vancomycin-resistant Enterococcus faecalis[J]. Science, 2003, 299(5615): 2071-2074. DOI:10.1126/science.1080613 |
| [3] |
Franz CMAP, Huch M, Abriouel H, et al. Enterococci as probiotics and their implications in food safety[J]. International Journal of Food Microbiology, 2011, 151(2): 125-140. DOI:10.1016/j.ijfoodmicro.2011.08.014 |
| [4] |
Giraffa G. Functionality of Enterococci in dairy products[J]. International Journal of Food Microbiology, 2003, 88(2/3): 215-222. |
| [5] |
de Vuyst L, Vandamme EJ. Antimicrobial potential of lactic acid bacteria[A]//de Vuyst L, Vandamme EJ. Bacteriocins of Lactic Acid Bacteria[M]. Boston, MA: Springer, 1994: 543-550
|
| [6] |
Franz CMAP, Holzapfel WH, Stiles ME. Enterococci at the crossroads of food safety?[J]. International Journal of Food Ficrobiology, 1999, 47(1/2): 1-24. |
| [7] |
Emori TG, Gaynes RP. An overview of nosocomial infections, including the role of the microbiology laboratory[J]. Clinical Microbiology Reviews, 1993, 6(4): 428-442. DOI:10.1128/CMR.6.4.428 |
| [8] |
Dougherty SH, Flohr AB, Simmons RL. 'Breakthrough' Enterococcal septicemia in surgical patients. 19 cases and a review of the literature[J]. Archives of Surgery, 1983, 118(2): 232-238. |
| [9] |
Martínez OP, Muñoz SJ, Arriola MP, et al. Enterococcal infective endocarditis: description of 12 cases[J]. Anales de Medicina Interna, 2007, 24(11): 539-542. |
| [10] |
Bayer AS, Seidel JS, Yoshikawa TT, et al. Group D Enterococcal meningitis. Clinical and therapeutic considerations with report of three cases and review of the literature[J]. Archives of Internal Medicine, 1976, 136(8): 883-886. DOI:10.1001/archinte.1976.03630080025009 |
| [11] |
Silva N, Igrejas G, Vaz J, et al. Virulence factors in Enterococci from partridges (Alectoris rufa) representing a food safety problem[J]. Foodborne Pathogens and Disease, 2011, 8(7): 831-833. DOI:10.1089/fpd.2010.0781 |
| [12] |
Lepage E, Brinster S, Caron C, et al. Comparative genomic hybridization analysis of Enterococcus faecalis: identification of genes absent from food strains[J]. Journal of Bacteriology, 2006, 188(19): 6858-6868. DOI:10.1128/JB.00421-06 |
| [13] |
Bakshi U, Sarkar M, Paul S, et al. Assessment of virulence potential of uncharacterized Enterococcus faecalis strains using pan genomic approach-identification of pathogen-specific and habitat-specific genes[J]. Scientific Reports, 2016, 6: 38648. DOI:10.1038/srep38648 |
| [14] |
Chen LH, Yang J, Yu J, et al. VFDB: a reference database for bacterial virulence factors[J]. Nucleic Acids Research, 2005, 33(D1): D325-D328. |
| [15] |
Zhong Z. Comparative genomic analysis of the type strains of genus Enterococcus and multilocus sequence typing of Enterococcus faecalis isolated from fermented foods[D]. Hohhot: Doctor Dissertation of Inner Mongolia Agricultural University, 2015 (in Chinese) 钟智.肠球菌属模式株比较基因组分析及发酵食品中粪肠球菌多位点序列分型研究[D].呼和浩特: 内蒙古农业大学博士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10129-1015427475.htm |
| [16] |
Dong M. Virulence factors of Enterococcus faecalis expression in the biofilm of starvation phase and after two root canal conventional drugs[D]. Dalian: Master's Thesis of Dalian Medical University, 2011 (in Chinese) 董明.饥饿期粪肠球菌生物膜形成相关毒力因子的表达及两种根管常用药物对其作用研究[D].大连: 大连医科大学硕士学位论文, 2011 |
| [17] |
Zheng XH. The construction of gene efaA+ Enterococcus transformant and preliminare study of its influence on biofilm formation[D]. Fuzhou: Master's Thesis of Fujian Medical University, 2010 (in Chinese) 郑晓辉.肠球菌efaA基因阳性转化株的构建及其对生物膜形成影响的初步探讨[D].福州: 福建医科大学硕士学位论文, 2010 https://wenku.baidu.com/view/3988dcb2453610661fd9f442.html |
| [18] |
Wu EL. The influence of bacterial coculture on biological characteristics of porcine-originated Enterococcus feacalis strain[D]. Zhengzhou: Master's Thesis of Henan Agricultural University, 2016 (in Chinese) 武二丽.细菌共培养对猪源粪肠球菌生物学特性的影响[D].郑州: 河南农业大学硕士学位论文, 2016 http://cdmd.cnki.com.cn/Article/CDMD-10466-1016308455.htm |
| [19] |
Zhu B. Survey for virulence factors of enterococcus strains and prokaryotic expression of Enterococcus faecalis cytolysin cylLl, cylA[D]. Fuzhou: Master's Thesis of Fujian Medical University, 2006 (in Chinese) 朱波.肠球菌毒力因子的检测及溶血素cylLl和cylA基因的原核表达[D].福州: 福建医科大学硕士学位论文, 2006 |
| [20] |
Nallapareddy SR, Sillanpää J, Mitchell J, et al. Conservation of Ebp-type pilus genes among Enterococci and demonstration of their role in adherence of Enterococcus faecalis to human platelets[J]. Infection and Immunity, 2011, 79(7): 2911-2920. DOI:10.1128/IAI.00039-11 |
| [21] |
Luong TT, Lee CY. Overproduction of type 8 capsular polysaccharide augments Staphylococcus aureus virulence[J]. Infection and Immunity, 2002, 70(7): 3389-3395. DOI:10.1128/IAI.70.7.3389-3395.2002 |
| [22] |
Thurlow LR, Thomas VC, Fleming SD, et al. Enterococcus faecalis capsular polysaccharide serotypes C and D and their contributions to host innate immune evasion[J]. Infection and Immunity, 2009, 77(12): 5551-5557. DOI:10.1128/IAI.00576-09 |
| [23] |
Moreno MRF, Sarantinopoulos P, Tsakalidou E, et al. The role and application of enterococci in food and health[J]. International Journal of Food Microbiology, 2006, 106(1): 1-24. DOI:10.1016/j.ijfoodmicro.2005.06.026 |
| [24] |
Chen X, Suo ZW, Xu JQ, et al. Review on classification and representative species of hemolysin[J]. Chinese Agricultural Science Bulletin, 2008, 24(8): 16-22. (in Chinese) 陈希, 索占伟, 许剑琴, 等. 细菌溶血素的分类及代表性溶血素研究进展[J]. 中国农学通报, 2008, 24(8): 16-22. DOI:10.3969/j.issn.1007-7774.2008.08.007 |
| [25] |
Xu JG. Virulence genes, Multilocus sequence typing and drug resistance analysis on clinical Enterococci[D]. Hangzhou: Master's Thesis of Zhejiang University, 2010 (in Chinese) 徐建国.临床分离肠球菌耐药性、毒力基因及多位点序列分型研究[D].杭州: 浙江大学硕士学位论文, 2010 |
| [26] |
Thomas VC, Hiromasa Y, Harms N, et al. A fratricidal mechanism is responsible for eDNA release and contributes to biofilm development of Enterococcus faecalis[J]. Molecular Microbiology, 2009, 72(4): 1022-1036. DOI:10.1111/mmi.2009.72.issue-4 |
| [27] |
Tsugawa H, Ito H, Ohshima M, et al. Cell adherence-promoted activity of Plesiomonas shigelloides groEL[J]. Journal of Medical Microbiology, 2007, 56(1): 23-29. DOI:10.1099/jmm.0.46766-0 |
| [28] |
Nair S, Milohanic E, Berche P. ClpC ATPase is required for cell adhesion and invasion of Listeria monocytogenes[J]. Infection and Immunity, 2000, 68(12): 7061-7068. DOI:10.1128/IAI.68.12.7061-7068.2000 |
| [29] |
Yan WJ. Screening and inentification of ClpE interaction protein Streptococcus pneumonia[D]. Chongqing: Doctor Dissertation of Chongqing Medical University, 2013 (in Chinese) 闫文娟.肺炎链球菌ClpE相互作用蛋白的筛选鉴定[D].重庆: 重庆医科大学博士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10631-1014107226.htm |
| [30] |
Granato D, Bergonzelli GE, Pridmore RD, et al. Cell surface-associated elongation factor Tu mediates the attachment of Lactobacillus johnsonii NCC533 (La1) to human intestinal cells and mucins[J]. Infection and Immunity, 2004, 72(4): 2160-2169. DOI:10.1128/IAI.72.4.2160-2169.2004 |
| [31] |
Wu T, Zhao ZQ, Zhang L, et al. Trigger factor of Streptococcus suis is involved in stress tolerance and virulence[J]. Microbial Pathogenesis, 2011, 51(1/2): 69-76. |
| [32] |
Li XH. Distribution and pathogenicity to virulence genes of foodborne Listeria monocytogenes[D]. Baoding: Master's Thesis of Agricultural University of Hebei, 2012 (in Chinese) 李欣华.食源性单核增生性李斯特氏菌毒力基因分布及致病力的研究[D].保定: 河北农业大学硕士学位论文, 2012 |
| [33] |
Huang YS, Xu SX, Zhu D, et al. Effect of HtrA gene deletion on virulence of Streptococcus pneumoniae[J]. Journal of the Fourth Military Medical University, 2006, 27(17): 1542-1546. (in Chinese) 黄远帅, 许颂霄, 朱旦, 等. HtrA基因缺陷对肺炎链球菌毒力影响的研究[J]. 第四军医大学学报, 2006, 27(17): 1542-1546. DOI:10.3321/j.issn:1000-2790.2006.17.002 |
| [34] |
Domann E, Hain T, Ghai R, et al. Comparative genomic analysis for the presence of potential enterococcal virulence factors in the probiotic Enterococcus faecalis strain Symbioflor 1[J]. International Journal of Medical Microbiology, 2007, 297(7/8): 533-539. |
| [35] |
Wang GF, Wu LX, Xue SP. Relationship between esp gene in Enterococcus from ICU and vancomycin resistance[J]. Chinese Journal of Nosocomiology, 2012, 22(13): 2724-2726. (in Chinese) 王国富, 吴利先, 薛士鹏. ICU肠球菌属esp基因分布及对万古霉素耐药的相关性研究[J]. 中华医院感染学杂志, 2012, 22(13): 2724-2726. |
| [36] |
Shankar N, Baghdayan AS, Gilmore MS. Modulation of virulence within a pathogenicity island in vancomycin-resistant Enterococcus faecalis[J]. Nature, 2002, 417(6890): 746-750. DOI:10.1038/nature00802 |
| [37] |
Crump KE, Bainbridge B, Brusko S, et al. The relationship of the lipoprotein SsaB, manganese and superoxide dismutase in Streptococcus sanguinis virulence for endocarditis[J]. Molecular Microbiology, 2014, 92(6): 1243-1259. DOI:10.1111/mmi.12625 |
| [38] |
Kadioglu A, Taylor S, Iannelli F, et al. Upper and lower respiratory tract infection by Streptococcus pneumoniae is affected by pneumolysin deficiency and differences in capsule type[J]. Infection and Immunity, 2002, 70(6): 2886-2890. DOI:10.1128/IAI.70.6.2886-2890.2002 |
| [39] |
Tendolkar PM, Baghdayan AS, Shankar N. Putative surface proteins encoded within a novel transferable locus confer a high-biofilm phenotype to Enterococcus faecalis[J]. Journal of Bacteriology, 2006, 188(6): 2063-2072. DOI:10.1128/JB.188.6.2063-2072.2006 |
 2018, Vol. 45
2018, Vol. 45