扩展功能
文章信息
- 何世耀, 胡万金, 肖玲, 李慧琴, 朱嫚, 马燕天, 吴兰
- HE Shi-Yao, HU Wan-Jin, XIAO Ling, LI Hui-Qin, ZHU Man, MA Yan-Tian, WU Lan
- 对不同公司16S rRNA基因MiSeq测序数据的一致性分析
- Consistency analysis of MiSeq sequencing data of 16S rRNA genes from different biotech companies
- 微生物学通报, 2018, 45(12): 2648-2661
- Microbiology China, 2018, 45(12): 2648-2661
- DOI: 10.13344/j.microbiol.china.171087
-
文章历史
- 收稿日期: 2017-12-27
- 接受日期: 2018-04-26
- 网络首发日期(www.cnki.net): 2018-05-09
在分子生物学技术出现之前,微生物培养技术是整个微生物学发展的基础,然而由于绝大多数环境微生物无法在实验室条件下进行分离培养[1],使得微生物学尤其是环境微生物学的发展比较缓慢。自1965年,Zuckerkandl等首次提出了使用基因序列来区分生物间的亲缘关系后[2],Woese和Fox基于16S rRNA基因序列将原核生物分为了三大类[3],此后利用rRNA基因序列进行微生物多样性研究的技术走向成熟。其它一些基于基因序列的指纹图谱方法如DGGE[4]、T-RFLP[5]等也迅速发展起来。以上技术的出现,极大地促进了微生物学的发展,也让人们意识到微生物世界的庞大和复杂。虽然以上基于分子技术的遗传多样性分析让人们认识到了微生物的丰富性和多样性,但对于详细的微生物群落结构的研究则受益于测序技术的迅猛发展。最初的Sanger双脱氧链末端终止法可以对许多纯培养物的特定序列进行详细地研究,后来基于克隆文库的方法也可以获得一定量的环境微生物信息。但由于受限于较低的测序效率以及高昂的成本,研究者们很难对环境样品进行大规模的序列分析。自2005年454 Life Sciences公司提出焦磷酸测序法以来,先后又迅速发展了Roche 454、Illumina/Solexa以及ABI SOLiD等多个测序平台[6-8]。自此,一次测序的通量高达10 Gb以上,极大地促进了环境微生物学的高速发展。
随着高通量测序技术的大量应用,人们往往需要比较和分析大量的测序数据。但由于测序平台、测序深度以及数据处理流程的不同,往往会使相同样品的测序数据呈现出一定的差异。因此人们对高通量测序数据的可重复性提出了更高的要求。为了能够准确分析比较来自不同研究项目、不同测序公司的测序数据,我们必须事先评估测序数据的可靠性,同时了解由于测序深度造成的影响。本文将同一批次样品在基于MiSeq测序平台的A、B两家公司重复测样,采用不同的测序深度,对比两组数据间的差异性。进而探究这些测序数据间是否具有通用性,以及评估它们之间的差异对科研工作的影响。本研究对测序数据的比较分析,旨在为涉及高通量测序技术的科研工作提供一些参考和建议。
1 材料与方法 1.1 主要试剂和仪器DNA标准分子量Marker、Taq酶、PCR扩增引物,大连TaKaRa公司;PowerSoil土壤DNA提取试剂盒,MO BIO Laboratories公司;其余试剂均购自生工生物工程(上海)股份有限公司。PCR仪,Applied Biosystems公司;超微量分光光度计,Thermo公司;序列测定由上海美吉生物医药科技有限公司完成。
1.2 方法本研究收集了来自鄱阳湖4个不同区域的湖泊沉积物样品,样品采集于2014年3月,采集地点分别为鄱阳湖的松门山(S,N29°12′、E116°11′)、南矶山(N,N28°55′、E116°16′)、饶河(R,N29°01′、E116°29′)和白沙洲(B,N29°10′、E116°37′)。每个地点分别收集3份湖底1−10 cm处的沉积物,分别编号为松门山S (S1、S2、S3)、南矶山N (N1、N2、N3)、饶河R (R1、R2、R3)和白沙洲B (B1、B2、B3)。采集的沉积物样品在低温条件下运输至实验室并保存于−80 ℃。
准确称取−80 ℃保存的沉积物样品各0.3 g,使用PowerSoil土壤DNA提取试剂盒进行沉积物总微生物DNA的提取[9]。使用超微量分光光度计检验DNA的浓度以及纯度,并采用通用引物338F (5′-CTCCTACGGGAGGCWGC-3′)和1392R (5′-AC GGGCGGTGTGTACA-3′)进行16S rRNA基因PCR扩增,检验获得的DNA样品是否满足测序要求。实验中总共有11个样品符合测序要求,南矶山N2样品PCR检验失败,没有进行后续测序。
将提取到的11个DNA样品分为2份分别送至A、B两家测序公司进行16S rRNA基因的V3−V4可变区扩增并测序,二者均采用Illumina公司旗下的Solexa MiSeq 2500平台进行2×300 bp双末端测序。为便于后续比较,将所得数据分别命名为A1−A11、B1−B11。对于原始测序数据,主要使用USEARCH软件(v9.2)进行前期序列的处理,使用Mothur软件(v1.35.1)进行后续分析,同时使用Trimmomatic软件(v0.36)进行原始数据的质量控制,Flash软件(v1.2.1)进行序列拼接。统计学分析主要在SPSS 22.0与R 3.3.1软件中完成。使用Mothur软件进行主坐标分析(PCoA)以及聚类分析。Network的绘制在Gephi 0.9.1中完成,使用METAGENassist在线进行16S rRNA基因功能预测,相关图表的制作通过R、Excel 2016等软件完成。
2 结果与分析 2.1 A、B两组数据基于微生物群落结构的对比为了分析A、B两组数据在群落结构上是否存在差异,分别对A、B两组数据在OTU水平基于Bray-Curtis距离做了聚类分析(图 1)。聚类结果发现两组数据所反映的微生物群落结构在样地间的分布规律具有一致性。对两组数据中的同一样品进行相关性分析的结果也同样反映了这一点,除A-B1、B-B1样品之外,A、B两组数据的同一样品之间均存在显著相关性(表 1)。

|
| 图 1 A、B两个样品基于Bray-Curtis距离聚类 Figure 1 Cluster analysis based on Bray-Curtis distance for bacterial OTUs from A and B datasets |
|
|
| 样地Sample | 显著性Significance |
| A-B1 & B-B1 | P=0.396 2 |
| A-B2 & B-B2 | P < 0.001** |
| A-B3 & B-B3 | P < 0.001** |
| A-N1 & B-N1 | P < 0.001** |
| A-N3 & B-N3 | P < 0.001** |
| A-R1 & B-R1 | P < 0.001** |
| A-R2 & B-R2 | P < 0.001** |
| A-R3 & B-R3 | P < 0.001** |
| A-S1 & B-S1 | P < 0.001** |
| A-S2 & B-S2 | P < 0.001** |
| A-S3 & B-S3 | P < 0.001** |
| Note: **: P < 0.01. | |
然而,当我们将两组数据放在一起分别进行主坐标分析(PCoA)和聚类分析分析时发现,两种统计的结果均将测序数据按照A、B两家公司分别聚类为两个不同的集合(图 2A和B),即它们按照测序数据来源不同而被明显的区分开来。进一步在整体上比较两组数据的差异,ANOSIM相似性分析结果同样显示A、B两组数据间存在显著性差异(P < 0.001)。

|
| 图 2 A、B两组数据的PCoA分析以及聚类分析(B) (OTU水平) Figure 2 Principal co-ordinates analysis (A) and cluster (B) analysis for bacterial OTUs from A and B datasets |
|
|
以上结果表明,同一样品在不同公司的测序数据并不会对样地间微生物群落结构分布规律造成很大的影响,然而两组数据之间的确存在显著的差异。为了了解这种差异的产生原因,从A、B两组数据的测序质量以及测序深度两个方面进行进一步的分析。
2.2 A、B两组数据在测序质量上的差异由于A、B两组数据是相同样品在不同测序环境下所产生的,不可避免的会造成它们在测序质量上的差异。统计了A、B两组数据的测序质量,测序质量值是在高通量测序过程中,对每个所测碱基给予的一个质量评分,碱基的质量值为13时,错误率为5%,20的错误率为1%,30的错误率为0.1%。而为了评估下机Reads测序的准确度,一般会评估Q20或Q30 (即所有碱基质量值大于20或30所占的比例)。结果如图 3所示。图 3中分别反映了A、B两组数据的测序质量分数分布情况以及测序平均质量分布情况。从上半部分的碱基测序质量值的分布来看,A组数据的碱基测序质量值在36以上,而B组数据的整体质量值虽然也达到了30,但仍然存在一些30以下的离散值。进一步比较所有测序Reads的平均质量发现,A组的Reads质量分布比较集中,其平均质量分数在37左右,而B组数据的质量分布要更为离散,其平均质量得分在36附近。

|
| 图 3 A、B公司测序数据质量分数统计 Figure 3 Statistic of per base sequence quality and sequence quality scores 注:A:所有碱基的质量得分;B:所有序列的平均质量得分. Note: A: Quality scores across all bases; B: Quality scores distribution over all sequences. |
|
|
为了更为准确地对比A、B两组测序数据的质量情况,对两组原始数据的质控信息进行了统计,结果如表 2所示。在质控过程中,以30 bp长度为滑动窗口,筛选平均质量得分高于20的序列,经过质控后A组数据的保留率在80%以上,而B组数据仅有2个样品高于70%,说明B组数据中有大量的低质量数据被删除。同时也注意到,在经过质控步骤后,后续拼接过程中A、B两组数据的拼接效率均达到了90%以上,两者的平均拼接效率仅相差4%。此结果表明,在不同测序环境中,经过质控后,虽然不同公司的测序质量仍然存在一定差异,但质控过程可以消除或者弱化不同环境下测序质量对结果造成的影响。
| Sample | Raw data | Clean reads | Pass rate (%) | Splicing efficiency (%) |
| A-B1 | 42 791 | 34 907 | 81.58 | 96.64 |
| A-B2 | 39 309 | 32 465 | 82.59 | 96.95 |
| A-B3 | 41 570 | 33 958 | 81.69 | 96.99 |
| A-N1 | 39 842 | 32 053 | 80.45 | 96.69 |
| A-N3 | 41 443 | 34 326 | 82.83 | 96.96 |
| A-R1 | 34 020 | 28 098 | 82.59 | 96.79 |
| A-R2 | 41 511 | 34 073 | 82.08 | 96.73 |
| A-R3 | 36 671 | 29 828 | 81.34 | 96.60 |
| A-S1 | 32 026 | 27 016 | 84.36 | 97.37 |
| A-S2 | 27 286 | 23 049 | 84.47 | 97.08 |
| A-S3 | 29 217 | 24 622 | 84.27 | 97.34 |
| B-B1 | 120 885 | 80 291 | 66.42 | 95.63 |
| B-B2 | 108 866 | 73 462 | 67.48 | 94.10 |
| B-B3 | 110 814 | 75 864 | 68.46 | 95.50 |
| B-N1 | 98 096 | 64 271 | 65.52 | 96.71 |
| B-N3 | 115 811 | 81 954 | 70.77 | 97.71 |
| B-R1 | 137 864 | 99 397 | 72.10 | 95.83 |
| B-R2 | 105 380 | 66 863 | 63.45 | 95.15 |
| B-R3 | 88 675 | 56 203 | 63.38 | 94.27 |
| B-S1 | 124 984 | 91 164 | 72.94 | 96.36 |
| B-S2 | 83 324 | 49 617 | 59.55 | 94.10 |
| B-S3 | 97 481 | 64 215 | 65.87 | 94.13 |
为了进一步验证推断,对A、B两组数据的注释情况进行了统计,结果如图 4、5所示。在图 4中,稀释曲线表明A、B两组测序数据均达到了平台期,均符合测序要求。图 5中,测序数据质量较低的B组数据反而在注释率上要稍高于A,但他们的注释率并没有显著差异(P=0.771)。由于测序深度的影响,两组数据在OTU数目上相差较大,但在各个分类水平上的注释率并没有显著性差异(P=0.688)。
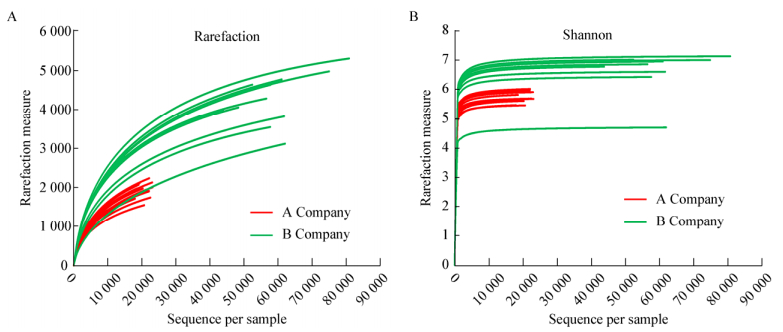
|
| 图 4 A、B数据稀疏曲线(A)与香浓曲线(B) Figure 4 Rarefaction curves and Shannon curves of A and B sequencing datasets |
|
|

|
| 图 5 A、B数据注释统计 Figure 5 Annotation results of A and B sequencing datasets |
|
|
前面的分析结果表明,不同环境下的测序质量差异并不足以影响实验结果,推测它们的差异是由测序深度不同所导致的。为了验证这一猜测,对B组数据进行了Downside处理得到数据dB,即从B组数据的每个样品中随机抽取一定数量的Reads,使同一样品在A、B两组数据中的Reads数一致,这在一定程度上削弱测序深度的影响。从序列注释情况看(表 3),在降低B组数据Reads数目的情况下,它们在各个水平注释的差异变小,但dB组数据注释出的物种数目依然要多于A。对α多样性的统计显示(表 4),A、dB两组数据在Chao1、Shannon、Sob上均无显著差异,仅在Invsimpson指数上存在显著差异。这说明两组数据在物种总的丰度上没有显著差异,但是dB物种的种类数量要显著高于A。这可能是由于dB数据与B数据相比,虽然在物种丰度上有所降低,但是对物种的种类数目并无显著改变。对B数据Downside前后数据对比也同样验证了此点(表 4),dB与B相比,虽然Chao1指数有显著降低,但是Invsimpson指数并无显著变化。
| Taxonomy | A | dB | B |
| Phylum | 45 | 52 | 52 |
| Class | 127 | 133 | 145 |
| Order | 255 | 265 | 290 |
| Family | 439 | 483 | 548 |
| Genus | 654 | 862 | 1 032 |
| Sample | Statistics | Chao1 | Shannon | Sobs | Invsimpson |
| A | NA | 3 629.78±129.69 | 6.22±0.06 | 2 656.45±81.65 | 91.47±10.85 |
| dB | NA | 3 717.44±120.77 | 6.50±0.20 | 2 924.72±113.60 | 219.04±26.79 |
| B | NA | 5 262.18±189.27 | 6.67±0.21 | 4 305.27±200.62 | 235.74±28.69 |
| A&dB | F | 0.244 7 | 1.888 1 | 3.676 9 | 19.482 2 |
| P | 0.626 2 | 0.184 6 | 0.069 6 | 0.000 2 | |
| B&dB | F | 47.335 9 | 0.345 9 | 35.855 0 | 0.181 0 |
| P | 0.000 1 | 0.563 0 | 0.000 1 | 0.675 0 |
经过前面的分析发现,不同公司的测序数据在测序质量上的差异并不能对实验结果造成显著的影响,这种差异主要是由测序深度的不同,即Reads数目导致的。为了考察这种Reads上的差异主要影响实验结果的哪一方面,以及这种影响是否重要,对A、B分别得到的微生物群落结构信息做LEfSe分析。分析结果如图 6所示,A、B两组数据的微生物群落结构存在一定差异,共发现了122个有差异的物种,隶属于8个门(13.11%) 22个纲(11.34%) 28个目(8.21%) 31个科(5.16%) 33个属(2.91%)。

|
| 图 6 A、B数据基于物种组成的LEfSe分析 Figure 6 LEfSe analysis based on bacterial species composition from A and B sequencing datasets |
|
|
为了更全面地考察A、B两组数据中微生物的群落结构分布状况,从16S rRNA基因所能达到的最低注释水平属(Genus),作A、B两组数据间的物种分布网络图(图 7)。结果显示,在属水平,拥有更大测序深度的B组数据中存在着大量的独有的低丰度属(丰度低于0.1%),而丰度高于0.1%的微生物多为A、B所共有。以上结果明确显示,测序深度的不同会造成微生物群落结构上的差异,而这种差异大多体现在稀有种方面,更深的测序深度能够捕获到一些丰度较低的菌群。

|
| 图 7 A、B两组数据的物种分布网络图 Figure 7 Network of species composition from dataset A and B 注:A、B两个点的大小代表了他们在属水平微生物的种类多少,每一条从A、B中延伸出的连线与节点代表了存在于其中的一个属,黑色连线表示微生物的丰度高于0.1%,灰色连线表示丰度低于0.1%. Note: The size of the points A and B represents the number of bacterial genera, and each line or node extending from A or B represents one genus belonging to the corresponding dataset. The blank line indicates the bacteria with high abundance (> 0.1%), and the grey lines indicate bacterial abundances below 0.1%. |
|
|
找到导致A、B两组数据差异的原因后,进一步探究了这些差异对生态系统功能造成的影响。首先对A、B两组数据的微生物群落结构在门水平(Phylum)做了关系网络关联分析(非参数Spearman)[10]。结果如图 8所示,A组数据中具有41个节点和132条边,其模块化值为0.54,网络直径为6。与之相比,B中具有51个节点以及155条边,模块化值为0.579,网络直径为7。对比发现两者的模块化值以及网络直径均相近,且其模块化值大于0.4,均存在明显的社区结构[11-12],但B的节点数以及边数均高于A。整体上看,B中微生物间的相互作用以及群落复杂程度要高于A。但从高丰度(Abundance > 1%)和中等丰度(0.1% < Abundance < 1%)的微生物门类来看,A、B两组样品相差不大。在与其它微生物相互作用较强的门(Phylum)中,高丰度的Acidobacteria (酸酐菌门)、Chloroflexi (绿弯菌门)、Ignavibacteriae等在A、B中均有出现,而其他一些中等丰度的Omnitrophica、Spirochaetae (螺旋菌门)等在A、B中也均有出现。同时也注意到,许多稀有微生物(Abundance < 0.1%)在群落中也发挥着重要的作用,例如Deferribacteres (脱铁杆菌门)、Lentisphaerae (黏胶球形菌门)、TM6_(Dependentiae)等。但是与A相比,B中出现了更多与其他微生物具有较强相互作用的低丰度微生物,例如GAL15、Deinococcus-Thermus、KSB3_(Modulibacteria)等。这一结果表明,随着测序深度的增加,更多的稀有微生物被发现,微生物的群落结构以及相互作用也更加复杂,而这些稀有的微生物在环境中可能发挥着重要的作用。

|
| 图 8 相关性网络分析 Figure 8 Network of co-occurring based on correlation analysis 注:图中每个节点代表一个门,节点颜色深浅对应其丰度高低,节点的大小代表了节点的度,即与之关联的微生物的数量,边颜色的深浅代表了正、负相关性,黑色为正相关.连线的粗细代表了相关性的强弱(Spearman’s ρ > 0.7,P < 0.01). Note: Nodes colored by abundance. The size of each node is proportional to the number of connections (equally to degree). Edges colored by correlation, black lines represent positive correlations. The edge thickness is proportional to the strength of correlation (Spearman's ρ > 0.7, P < 0.01). |
|
|
微生物的关系网络关联分析虽然能够在一定程度上反映环境中微生物之间的相互作用,但并没有直观的反映出这些作用具体表现在哪些方面,例如这些低丰度的微生物到底对整体的微生物群落结构产生了怎样的影响,如果缺失了这些微生物,我们对环境中微生物的认知会产生怎样的偏差?为了解释这一点,对A、B两组数据进行了16S rRNA基因功能预测分析,从功能的角度出发,探究低丰度菌群在环境中发挥的作用。经过功能预测,在105种代谢功能中,发现了A、B中可能存在的31种代谢功能。从整体上看,A、B两组数据在功能种类上比较一致,不存在A或者B所独有的代谢功能,未知功能的物种丰度仅相差3.39%。但是也注意到,两组数据中发挥相同功能的微生物在群落中丰度相差较大。对发现的31种代谢功能进行ANOVA分析(图 9),共发现了14种有显著性差异的代谢功能,其中4种主要的代谢功能阿特拉津代谢(Atrazine metabolism)、几丁质降解(Chitin degradation)、硫酸盐还原(Sulfate reducer)和固氮(Nitrogen fixation)在丰度上相差2.72%、2.34%、1.75%、1.36%。这一结果表明,某些低丰度的微生物在一些重要的物质代谢如氮、硫的代谢上同样发挥着重要的作用。忽视这些微生物的存在,可能会导致对整体环境的认知产生较大的偏差。

|
| 图 9 差异代谢功能丰度统计 Figure 9 The normalized abundance of differentiated metabolism |
|
|
对于测序数据而言,其测序质量的可靠性一直是所有科研工作者们所重视的,并对此开展了大量的研究[13]。以二代测序技术为例,已有研究表明不同的测序平台之间存在着一定程度的质量差异[14]。Loman比较了454 GS Junior (Roche)、Illumina MiSeq和Ion Torrent PGM (Life Technologies) 3个测序平台间的差异,发现454 GS Junior (Roche)具有最长的读长,Illumina MiSeq具有最低的错误率以及较高的通量,Ion Torrent PGM具有最高的测序通量[15]。Claesson等[16]的研究发现,随着测序长度的增加,测序的错误率会不断提升,当超过某个阈值时(一般是技术上的限制),错误率会呈指数增长,导致测序数据无法使用。而当以Q20 (错误率1%)为标准进行过滤数据时,这部分低质量数据的干扰就会被减少到可接受的范围内。这在一定程度上佐证了我们的结论,即不同环境下测序质量造成的影响可以通过质控过程减弱甚至排除,并不会对实验结论造成显著的影响。这一结论提示我们,测序的长度是决定测序质量的重要因子,为此更高的测序质量促使了测序平台能够产生更长的合格的Reads,两者相辅相成。
测序质量的提高依赖于技术革新,因此科研工作者们更加关心测序的长度以及深度。越长的测序长度蕴含了更加丰富的遗传信息,可提供更加精确的基因信息。Rajilić-Stojanović等通过测定SSU rRNA全长,鉴定了人类肠道微生物中的近1 200个种系型,并预估了可能存在3 000个种系型[17]。通过454焦磷酸测序得到的16S rRNA基因全长序列信息,能够将一个物种精确定位到种甚至亚种的水平,然而使用其它二代测序平台得到的部分16S rRNA基因片段,一般只能注释到属水平。而在测序长度受到限制的情况下,增加测序深度就成为全面挖掘环境中微生物信息的另一种手段。在测序深度较低的情况下,仅能够反映出环境中不完整的群落结构,它们大多是由高丰度微生物组成[18-19]。同时,由于PCR扩增的特性,其不能够对样品中的微生物进行等比例的扩增,高丰度的微生物会有很大的几率被扩增,而绝大多数的稀有微生物被扩增的几率则较低。这就导致了PCR过程在放大了高丰度微生物的数量的同时,进一步减少了稀有微生物被发现的概率,许多丰度小于0.1%的微生物甚至无法被扩增到[20]。虽然高丰度的微生物通常被认为是环境物质代谢过程中最活跃和最重要的部分[21],然而其仅仅占了环境中微生物多样性的一小部分。从我们的结果中可以看到,随着测序深度的增加,Genus的种类由654上升到1 032个,微生物群落结构的多样性大幅增多。如果要进一步对这部分低丰度、高多样性的“稀有生物圈”[22]展开研究,增加测序深度就显得非常必要。
Kuczynski等[23]的研究显示微生物群落多样性指数会受到测序深度的影响,Lundin等[24]研究结果表明,在淡水和沉积物样品中,5 000个降噪后的序列即可捕获超过80%的Chao1丰富度,同时他也强调,如果研究的目的是稀有微生物,则需要获得更多的Reads数。当人们的目光逐渐聚焦于稀有微生物上时,越来越多的研究也发现,稀有微生物在环境中发挥着重要的作用。Galand等[25]对海洋微生物中高丰度微生物与稀有微生物的研究中发现,稀有微生物比高丰度的微生物能够更好地区分出海洋的表层水与深层水。多方面的研究发现,稀有微生物并非是一成不变的,它们在环境中充当着重要的种质库的作用,在诸如物种形成、灭绝、扩散或物种相互作用等生态机制的作用中,部分稀有微生物与高丰度微生物之间可能会发生转变。例如Malmstrom等[26]的研究发现,在地表水中,属于稀有微生物的Gammaproteobacteria,在北极水域表面具有高比例的生物量;Walke等发现在土壤中的稀有微生物在两栖动物的皮肤上占据着较高的丰度[27]。除此之外,稀有微生物在功能上同样发挥着重要的作用,Montoya等[28]发现海洋中的氮固定是由微生物群落中的稀有微生物完成的。与之相比,我们的实验也展现出了类似的结论,随着测序深度的增加,更多稀有微生物被检出的同时,相关网络分析揭示出了微生物间更为复杂的相互作用,同时其微生物群落功能也发生了明显的改变,这些结果表明稀有微生物在群落结构中扮演着重要的角色。
已有学者指出,细菌单个基因组内16S rRNA基因多样性[29]以及测序错误[30]均会导致高通量测序所检测到的微生物多样性被高估。基于序列相似性划分OTU后进行后续分析的方法可以一定程度上降低这种误差,一般认为序列相似性高于97%时属于同一个种。Sun等的研究结果显示,随着以Unique、97%相似性、95%相似性以及90%相似性划分OTU后分析,被高估的微生物多样性逐步递减[31]。为了更进一步地降低细菌单个基因组内16S rRNA基因多样性以及测序错误造成的误差,相关科研工作者们提出了更多的解决办法,例如PyroNoise直接从焦磷酸测序仪产生的光强度流程图中检测错误序列[32],或者以低于建议的阈值(97%相似性)来划分OTU[25]。
随着科技的发展,实验技术对科研的桎梏已越来越小,不管是种类繁多的一代、二代测序,还是日趋成熟的三代测序技术,都在一刻不停地贡献着庞大的科研数据。如何归纳总结这些海量的数据,并从中提炼出有价值的信息,已变得越来越重要。Chaffron等[33]通过Greengenes数据库中的16S rRNA基因序列信息,对全球范围内的微生物进行了分析,并构建了微生物的全球网络模型,在全面分析全球范围内微生物分布规律的同时,也为进一步挖掘测序数据的潜在价值提供了新的思路。随着现今科研信息交流的越发频繁,通过对现有科研数据的整合、解析、延伸,从而获得更深刻的理解以及全新的成果已成为一种趋势。本文虽在一定程度上阐述了不同测序环境以及测序通量数据的通用性问题,但还有更多方面的问题亟待科研工作者们去解决,例如如何削弱测序通量对科研结论的影响;一代、二代、三代测序数据之间是否具有通用性等。可以预见,一旦不同测序数据间的壁垒被打通,找到一种能够将不同测序数据联用的方法,微生物学领域的研究将迎来一次更加蓬勃的发展。
| [1] |
Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169. |
| [2] |
Zuckerkandl E, Pauling L. Molecules as documents of evolutionary history[J]. Journal of Theoretical Biology, 1965, 8(2): 357-366. |
| [3] |
Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain: the primary kingdoms[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(11): 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [4] |
Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700. |
| [5] |
Liu WT, Marsh TL, Cheng H, et al. Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA[J]. Applied and Environmental Microbiology, 1997, 63(11): 4516-4522. |
| [6] |
Shendure J, Porreca GJ, Reppas NB, et al. Accurate multiplex polony sequencing of an evolved bacterial genome[J]. Science, 2005, 309(5741): 1728-1732. DOI:10.1126/science.1117389 |
| [7] |
Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376-380. DOI:10.1038/nature03959 |
| [8] |
Smith DR, Quinlan AR, Peckham HE, et al. Rapid whole-genome mutational profiling using next-generation sequencing technologies[J]. Genome Research, 2008, 18(10): 1638-1642. DOI:10.1101/gr.077776.108 |
| [9] |
Lu SM. Study on the ammonia-oxidation microorganisms in the freshwater aquaculture pond environment[D]. Wuhan: Doctoral Dissertation of Huazhong Agricultural University, 2014 (in Chinese) 陆诗敏.淡水养殖池塘环境中氨氧化微生物的研究[D].武汉: 华中农业大学博士学位论文, 2014 |
| [10] |
Barberán A, Bates ST, Casamayor EO, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [11] |
Newman MEJ. The structure and function of complex networks[J]. SIAM Review, 2003, 45(2): 167-256. DOI:10.1137/S003614450342480 |
| [12] |
Newman MEJ. Modularity and community structure in networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(23): 8577-8582. DOI:10.1073/pnas.0601602103 |
| [13] |
Kennedy K, Hall MW, Lynch MDJ, et al. Evaluating bias of Illumina-based bacterial 16S rRNA gene profiles[J]. Applied and Environmental Microbiology, 2014, 80(18): 5717-5722. DOI:10.1128/AEM.01451-14 |
| [14] |
Castelino M, Eyre S, Moat J, et al. Optimisation of methods for bacterial skin microbiome investigation: primer selection and comparison of the 454 versus MiSeq platform[J]. BMC Microbiology, 2017, 17(1): 23. DOI:10.1186/s12866-017-0927-4 |
| [15] |
Loman NJ, Misra RV, Dallman TJ, et al. Performance comparison of benchtop high-throughput sequencing platforms[J]. Nature Biotechnology, 2012, 30(5): 434-439. DOI:10.1038/nbt.2198 |
| [16] |
Claesson MJ, Wang Q, O'Sullivan O, et al. Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions[J]. Nucleic Acids Research, 2010, 38(22): e200. DOI:10.1093/nar/gkq873 |
| [17] |
Rajilić-Stojanović M, Smidt H, de Vos WM. Diversity of the human gastrointestinal tract microbiota revisited[J]. Environmental Microbiology, 2007, 9(9): 2125-2136. DOI:10.1111/emi.2007.9.issue-9 |
| [18] |
Claesson MJ, O'Sullivan O, Wang Q, et al. Comparative analysis of pyrosequencing and a phylogenetic microarray for exploring microbial community structures in the human distal intestine[J]. PLoS One, 2009, 4(8): e6669. DOI:10.1371/journal.pone.0006669 |
| [19] |
Huber JA, Welch DBM, Morrison HG, et al. Microbial population structures in the deep marine biosphere[J]. Science, 2007, 318(5847): 97-100. DOI:10.1126/science.1146689 |
| [20] |
Pedrós-Alió C. Marine microbial diversity: can it be determined?[J]. Trends in Microbiology, 2006, 14(6): 257-263. DOI:10.1016/j.tim.2006.04.007 |
| [21] |
Cottrell MT, David KL. Contribution of major bacterial groups to bacterial biomass production (thymidine and leucine incorporation) in the Delaware estuary[J]. Limnology and Oceanography, 2003, 48(1): 168-178. DOI:10.4319/lo.2003.48.1.0168 |
| [22] |
Sogin ML, Morrison HG, Huber JA, et al. Microbial diversity in the deep sea and the underexplored "rare biosphere"[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(32): 12115-12120. DOI:10.1073/pnas.0605127103 |
| [23] |
Kuczynski J, Liu ZZ, Lozupone C, et al. Microbial community resemblance methods differ in their ability to detect biologically relevant patterns[J]. Nature Methods, 2010, 7(10): 813-819. DOI:10.1038/nmeth.1499 |
| [24] |
Lundin D, Severin I, Logue JB, et al. Which sequencing depth is sufficient to describe patterns in bacterial α-and β-diversity?[J]. Environmental Microbiology Reports, 2012, 4(3): 367-372. DOI:10.1111/emi4.2012.4.issue-3 |
| [25] |
Galand PE, Casamayor EO, Kirchman DL, et al. Ecology of the rare microbial biosphere of the Arctic Ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22427-22432. DOI:10.1073/pnas.0908284106 |
| [26] |
Malmstrom RR, Straza TRA, Cottrell MT, et al. Diversity, abundance, and biomass production of bacterial groups in the western Arctic Ocean[J]. Aquatic Microbial Ecology, 2007, 47(1): 45-55. |
| [27] |
Walke JB, Becker MH, Loftus SC, et al. Amphibian skin may select for rare environmental microbes[J]. The ISME Journal, 2014, 8(11): 2207-2217. DOI:10.1038/ismej.2014.77 |
| [28] |
Montoya JP, Holl CM, Zehr JP, et al. High rates of N2 fixation by unicellular diazotrophs in the oligotrophic Pacific Ocean[J]. Nature, 2004, 430(7003): 1027-1032. DOI:10.1038/nature02824 |
| [29] |
Acinas SG, Marcelino LA, Klepac-Ceraj V, et al. Divergence and redundancy of 16S rRNA sequences in genomes with multiple rrn operons[J]. Journal of Bacteriology, 2004, 186(9): 2629-2635. DOI:10.1128/JB.186.9.2629-2635.2004 |
| [30] |
Reeder J, Knight R. The 'rare biosphere': a reality check[J]. Nature Methods, 2009, 6(9): 636-637. DOI:10.1038/nmeth0909-636 |
| [31] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [32] |
Quince C, Lanzén A, Curtis TP, et al. Accurate determination of microbial diversity from 454 pyrosequencing data[J]. Nature Methods, 2009, 6(9): 639-641. DOI:10.1038/nmeth.1361 |
| [33] |
Chaffron S, Rehrauer H, Pernthaler J, et al. A global network of coexisting microbes from environmental and whole-genome sequence data[J]. Genome Research, 2010, 20(7): 947-959. DOI:10.1101/gr.104521.109 |
 2018, Vol. 45
2018, Vol. 45