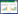扩展功能
文章信息
- 刘艳艳, 沈新迁, 王晓政, 林双君
- LIU Yan-Yan, SHEN Xin-Qian, WANG Xiao-Zheng, LIN Shuang-Jun
- 海洋链霉菌Streptomyces sp. B9173合成的2-吲哚酮生物碱
- 2-Oxindole alkaloids produced by marine-derived Streptomyces sp. B9173
- 微生物学通报, 2018, 45(11): 2470-2479
- Microbiology China, 2018, 45(11): 2470-2479
- DOI: 10.13344/j.microbiol.china.180239
-
文章历史
- 收稿日期: 2018-03-26
- 接受日期: 2018-05-23
- 网络首发日期(www.cnki.net): 2018-06-21
天然产物是药物开发的重要资源[1-2]。到目前为止,人们已从自然界中鉴定了二十多万个天然产物[3]。目前天然药物开发面临的最大问题是重复分离鉴定已知的化合物,所以在粗提物分离前期对其成分进行分析鉴别,排除已知化合物,有目标的挖掘新化合物可以极大节约资源和时间,提高发现新化合物的几率,这种方法叫做“排重(Dereplication)”[4]。目前,排重的方法主要有基因组挖掘技术及色谱数据比对技术等。基因组挖掘技术是利用生物信息学的方法分析基因组序列,鉴定基因簇,预测化合物的种类以促进新化合物的发现[5]。例如Scherlach和Hertweck等通过预测Aspergillus nidulans基因组序列中未知基因编码的产物,发现了Aspoquinolones A–D新型次级代谢产物,其具有很高的抗白血病作用[6]。色谱数据比对技术是将化合物的色谱数据建成数据库,以此来筛选未知的化合物,包括紫外吸收图谱(UV)、高分辨质谱(HRMS)、二级质谱(MS/MS)、保留时间(Retention time)、核磁共振光谱(NMR)等[7-9]。例如通过比对质谱数据筛选多肽类化合物[10]和利用LC-DAD-TOFMS可以快速地对微生物天然产物进行排重[11]。
本文研究菌株海洋链霉菌Streptomyces sp. B9173是从南美智利的太平洋沿岸沉积物中分离得到的。从该菌的发酵液里已经分离鉴定了抗白血病的生物碱类Maremycin A–F[12-13]及抗肿瘤物质色霉素A3[14],说明该菌代谢产物丰富且活性多样。我们利用HPLC-DAD和LC-MS结合的方法,排除已知化合物,筛选并成功分离鉴定了3个化合物:色胺酮(1)、甲基异靛蓝(2)、N, N-二甲基异靛蓝(3) (图 1)。其中色胺酮具有非常广泛的生物活性,是首次在细菌中被分离得到。甲基异靛蓝是临床治疗慢性粒细胞白血病的小分子药物,是首次在微生物中被分离得到。

|
| 图 1 3个化合物的化学结构 Figure 1 The chemical structure of three compounds 注:1:色胺酮;2:甲基异靛蓝;3:N, N-二甲基异靛蓝. Note: 1: Tryptanthrin; 2: Meisoindigo; 3: N, N-dimethylisoindigo. |
|
|
Streptomyces sp. B9173是德国HKI研究所馈赠的海洋链霉菌菌株。
1.1.2 培养基种子培养基(g/L):Oxide TSB 30.0。产孢培养基(g/L):甘露醇20.0,黄豆饼粉20.0,琼脂20.0。发酵培养基(g/L):甘露醇20.0,黄豆饼粉20.0。
1.1.3 主要试剂和仪器甲醇、乙酸乙酯、石油醚、氯仿、DMSO等有机试剂,上海国药集团;色谱纯乙腈(Acetonitrile,ACN),Sigma-Aldrich公司;氘代氯仿(CD3Cl-d4),Cambridge Isotope Laboratory公司;正相柱层析硅胶,青岛海洋化工有限公司;C18反相柱层析硅胶,YMC公司;凝胶色谱填料,GE医疗集团。摇床,New Brunswick Scientific公司;旋转蒸发仪,BUCHI公司;冷却循环泵,上海东玺制冷仪器设备有限公司;真空泵,VACUUBRAND公司;冷冻干燥机,LABCONCO公司;自动样品收集器,上海青浦沪西仪器厂;高效液相色谱仪(Agilent 1260 infinity HPLC),Agilent公司;超高效液相色谱-四级杆飞行时间质谱联用仪,Waters公司;600 MHz核磁共振波谱仪,Bruker公司。
1.2 方法 1.2.1 菌株的培养与发酵将菌种划线接种于产孢固体平板培养基,30 ℃恒温培养5 d至长出一层浓密的灰色孢子。滴加1 mL 20%的甘油-无菌水溶液至平板上,使用棉签轻轻地洗下孢子,用移液器将液体转移到菌种管中,−20 ℃保存;取10 μL孢子接种于含有50 mL种子培养基的250 mL三角瓶中,30 ℃、220 r/min振荡培养20 h,至菌液呈浓密粘稠状;取种子液,按8%接种量接种于装有500 mL发酵培养基的2 L三角瓶中,30 ℃、220 r/min振荡培养7 d。重复以上发酵步骤,共收集发酵液150 L。
1.2.2 次级代谢产物的提取将发酵液6 000 r/min离心10 min。取上清液进行分批萃取,每批用1.5倍发酵液体积的乙酸乙酯进行常温振荡萃取,振荡3 min,静置分层,分离水相和乙酸乙酯相。将水相再重复使用1.5倍的乙酸乙酯萃取2次。将萃取后的乙酸乙酯相30 ℃减压蒸馏浓缩,蒸除乙酸乙酯并回收再利用,共获得30.5 g黑色油状发酵粗提物。
1.2.3 次级代谢产物的检测方法取少量样品使用HPLC分析,DAD检测器全波长扫描,并选取210、254和400 nm作为检测波长。HPLC以H2O-ACN作为流动相。分析柱为XDB-C18柱(4.6 mm×250 mm,5 μm,Agilent),流速为0.4 mL/min。洗脱条件为梯度洗脱,乙腈的起始浓度为10%,0−12 min乙腈浓度由10%逐渐增加到60%;12−35 min,乙腈浓度由60%逐渐增加到95%;35−36 min,乙腈浓度由95%逐渐增加到100%;36−40 min,乙腈浓度持续为100%;40−41 min,乙腈浓度由100%逐渐降低到10%;41−46 min,乙腈浓度持续为10%,本研究中的HPLC检测方法均保持一致。
1.2.4 次级代谢产物的分离纯化首先使用正相硅胶柱进行初步分离。取约450 g 200−300目的硅胶悬浮于石油醚中,然后装于直径约10 cm的柱中,沉降完全后待用。粗提物用甲醇溶解然后用约90 g 100−200目硅胶进行拌样,采用干法上样。然后用洗脱体系(石油醚→石油醚:乙酸乙酯=5:1→石油醚:乙酸乙酯=1:1→乙酸乙酯→乙酸乙酯:甲醇=1:1→甲醇)逐级洗脱,洗脱体积都为5个柱体积,流速约10 mL/min。用250 mL三角瓶收集洗脱液,每瓶收集样品约200 mL。每瓶取样品20 μL,使用HPLC检测,根据各组分的成分,将含有相近化合物的样品合并为FR1–FR10共10个组分,通过减压蒸馏去除有机溶剂,得到棕黑色固体残渣。
使用长35 cm,直径2.5 cm的C18反相硅胶柱对各组分进行细分,采用干法上样,用甲醇和水的混合液作为流动相进行梯度洗脱,甲醇的浓度依次为35%→50%→75%→100%,每个体系洗脱5个柱体积。用试管结合自动收集器收集洗脱液,每管收集10 mL。HPLC检测后,选取含有不同代谢物的样品使用LC-MS分析。以上10个组分都按照上述步骤进行分离和检测。在组分FR5中确定3个目标化合物,将含有目标化合物的组分合并为FR5-4。
然后使用柱长200 cm,直径2.5 cm的Sephadex LH-20进行纯化,甲醇作为洗脱剂,洗脱体积为3个柱体积。收集洗脱液,每管收集5 mL。HPLC检测后,将含有目标化合物的组分合并为FR5-4-3。
对FR5-4-3使用上述相同的反相硅胶层析柱,洗脱液甲醇的浓度依次为60%、70%、80%、90%、100%,每个梯度洗脱5个柱体积。收集洗脱液,每管收集5 mL。HPLC检测后,合并含有相近化合物的组分,得到组分FR5-4-3-1、FR5-4-3-2、FR5-4-3-3、FR5-4-3-4。
对组分FR5-4-3-2使用长35 cm,直径1.5 cm的C18反相硅胶柱进一步分离,洗脱液甲醇的浓度梯度缩小为60%、65%、70%、75%、80%、100%,每个梯度洗脱3个柱体积。收集洗脱液,每管收集3 mL,HPLC检测后,将洗脱液进一步分为2个组分。第二个组分中主要为化合物1,重复组分FR5-4-3-2的分离步骤,最终得到纯的目标化合物1。
对组分FR5-4-3-3同样使用长35 cm,直径1.5 cm的C18反相硅胶层析柱进行分离,洗脱液甲醇的浓度依次为75%、80%、85%、90%、100%,每个梯度洗脱3个柱体积,收集洗脱液,每管收集3 mL。HPLC检测后合并为2个组分。对组分2重复上述分离步骤,最终得到纯的目标化合物2。
对组分FR5-4-3-4使用上述相同的层析柱进行再次分离,洗脱液甲醇的浓度依次为85%、90%、95%、100%,每个梯度洗脱3个柱体积。收集洗脱液,每管收集3 mL。HPLC检测后,排除含有杂质的组分,得到纯的目标化合物3。
取少量纯化后的样品,溶解于甲醇中,使用HRMS检测,获得样品的高分辨质谱;将剩余样品全部溶解于0.5 mL氘代氯仿中,以四甲基硅烷(TMS)作为内标,进行核磁共振光谱(NMR)分析。检测结束后,样品回收,减压蒸馏去除溶剂,将干燥的样品置于−20 ℃保存。
2 结果与分析 2.1 目标化合物的筛选当使用HPLC-DAD对FR5的组分进行分析时,在石油醚-乙酸乙酯(5:1)洗脱部分发现3个HPLC峰(图 2A),呈现出特殊的紫外吸收光谱(图 2B)。当使用反相层析柱对该组分进行细分时,纯甲醇洗脱部分(定为FR5-4)富集了这3个代谢物(图 2A),该紫外吸收光谱暗示这3个代谢物可能含有共轭结构。结合对这3个代谢物的LC-MS分析(图 2C)推测,这3个化合物虽然分子量较小,但是结构上应该具有一定的新颖性,于是将这3个化合物作为目标化合物进行后续分离。
|
| 图 2 目标化合物的检测、富集和色谱数据 Figure 2 Detection, enrichment, and the chromatographic data of three candidate compounds 注:A:目标化合物在HPLC上的保留时间;B:目标化合物的紫外吸收图谱;C:目标化合物的质谱. Note: A: The retention time of three candidate compounds on HPLC; B: The UV-vis spectra of three candidate compounds; C: The MS data of three candidate compounds. |
|
|
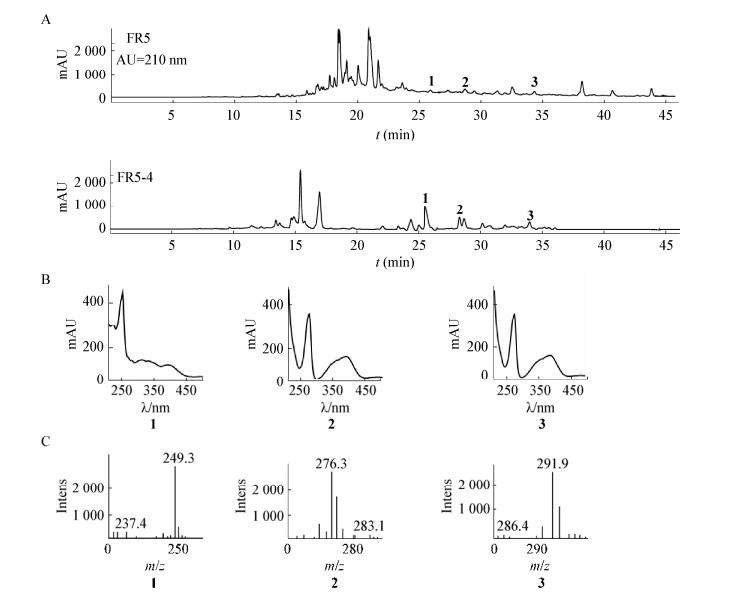
利用凝胶过滤色谱和反相层析色谱对FR5-4进行多轮反复的分离纯化,最终从组分FR5-4-3-2分离得到化合物1 (5 mg),从组分FR5-4-3-3中分离得到化合物2 (5 mg),从组分FR5-4-3-4中分离得到纯的目标化合物3 (4 mg),得到的3个纯化合物使用HPLC共进样分析的结果如图 3所示。
|
| 图 3 化合物1、2、3的HPLC谱图 Figure 3 The HPLC chromatogram of 1, 2, and 3 |
|
|
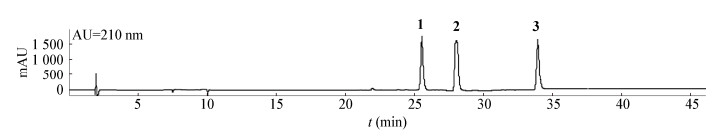
1是黄色粉末,可溶于甲醇、氯仿。紫外吸收如图 2所示,其中λmax:210 nm、252 nm。在正离子模式下,高分辨质谱分析(图 4A)给出其准分子离子峰为249.065 9 [M+H]+,而其理论分子量为248.058 6,因此确定它的分子式为C15H8N2O2。化合物1的1H NMR (600 MHz, CD3Cl)和13C NMR (150 MHz,CD3Cl)如图 5所示。1H NMR显示该化合物有8个芳香氢质子信号δH 8.64 (d,J=7.86 Hz,H-10)、δH 8.44 (d,J=7.68 Hz,H-1)、δH 8.03 (d,J=7.92 Hz,H-4)、δH 7.92 (d,J=7.32 Hz,H-7)、δH 7.86 (dd,J=7.56,7.32 Hz,H-3)、δH 7.80 (dd,J=7.62,7.56 Hz,H-9)、δH 7.68 (dd,J=7.32,7.26 Hz,H-2)、δH 7.43 (dd,J=7.26,6.89 Hz,H-8),而信号δH 1.58为水的信号,此外通过二维核磁共振谱图可以确定信号δH 1.25是体系带来的杂质,基于这些信息可以推测该结构中存在2个邻位取代苯环单元(图 1)。13C NMR和DEPT显示该化合物有2个羰基信号δc 182.6 (C-6)、δc 158.1 (C-12);5个不饱和季碳信号δc 146.6 (C-4a)、δc 146.3 (C-10a)、δc 144.3 (C-5a)、δc 123.7 (C-12a)、δc 122.2 (C-6a);8个不饱和次甲基碳信号δc 138.3 (C-9)、δc 135.2 (C-3)、δc 130.7 (C-4)、δc 130.3 (C-2)、δc 127.6 (C-1)、δc 127.2 (C-8)、δc 125.4 (C-7)、δc 118.0 (C-10) (图 5B)。结合1H-1H COSY、HSQC、HMBC等2D NMR光谱,确定化合物1为色胺酮(Tryptanthrin),化学结构如图 1所示,核磁数据与文献[15]报道一致。

|
| 图 4 3个化合物的高分辨质谱图 Figure 4 The HRMS spectra of three compounds 注:A:色胺酮的高分辨质谱图;B:甲基异靛蓝的高分辨质谱图;C:N, N-二甲基异靛蓝的高分辨质谱图. Note: A: The HRMS spectrum of Tryptanthrin; B: The HRMS spectrum of Meisoindigo; C: The HRMS spectrum of N, N-dimethylisoindigo. |
|
|
|
| 图 5 化合物1的一维核磁共振谱图 Figure 5 The 1D NMR spectra of compound 1 注:A:氢谱(600 MHz);B:碳谱(150 MHz). Note: A: 1H NMR (600 MHz); B: 13C NMR (150 MHz). |
|
|
2是紫红色粉末,可溶于甲醇、易溶于氯仿。紫外吸收如图 2所示,其中λmax:210 nm、274 nm、390 nm。在正离子模式下,质谱分析(图 4B)给出其准分子离子峰为277.098 0 [M+H]+,而其理论分子量为276.089 9,因此确定它的分子式为C17H12N2O2。1H NMR (图 6A)显示该化合物有7组芳香氢质子信号δH 9.19 (d,J=8.04 Hz, H-5')、δH 9.11 (d,J=7.98 Hz, H-5)、δH 7.38 (t,J=7.68 Hz, H-7)、δH 7.32 (t,J=7.56 Hz, H-7')、δH 7.07 (d,J=7.68 Hz, H-6')、δH 7.04 (d,J=7.68 Hz, H-6)、δH 6.81 (m,H-8')、δH 6.81 (m,H-8),推测该结构中存在2个邻位取代苯环单元,结合质谱该化合物为类二聚体;信号δH 3.28 (s)应该是氮甲基。13C NMR (图 6B)和DEPT显示该化合物有2个羰基信号δc 169.3 (C-2')、δc 167.9 (C-2);6个不饱和季碳信号δc 145.3 (C-9)、δc 142.4 (C-9')、δc 133.7 (C-3),δc 133.3 (C-3')、δc 122.5 (C-4')、δc 121.5 (C-4);8个不饱和次甲基碳信号δc 132.5 (C-7)、δc 132.5 (C-7')、δc 130.2 (C-5')、δc 129.8 (C-5)、δc 122.5 (C-6')、δc 122.4 (C-6)、δc 109.2 (C-8')、δc 107.7 (C-8);一个与氮相连的甲基碳信号δc 26.1 (C-10)。结合1H-1H COSY、HSQC、HMBC等2D NMR光谱,明确了重叠的信号峰并且排除了杂质信号(δH 1.25与δc 29.7),最终确定化合物2为甲基异靛蓝(Meisoindigo),化学结构如图 1所示,核磁数据与文献报道一致[16]。

|
| 图 6 化合物2的一维核磁共振谱图 Figure 6 The 1D NMR spectra of compound 2 注:A:氢谱(600 MHz);B:碳谱(150 MHz). Note: A: 1H NMR (600 MHz); B: 13C NMR (150 MHz). |
|
|
3是紫红色粉末,微溶于甲醇,易溶于氯仿。紫外吸收如图 2所示,与化合物2完全相同。在正离子模式下,质谱分析(图 4C)给出其准分子离子峰为291.113 5 [M+H]+,而其理论分子量为290.105 5,因此确定它的分子式为C18H14N2O2。1H NMR (图 7A)显示该化合物有4组芳香氢质子信号δH 9.21 (d,J=7.98 Hz,H-5/5′)、δH 7.38 (t,J=7.68 Hz,H-7/7′)、δH 7.07 (t,J=7.74 Hz,H-6/6′)、δH 6.79 (d,J=7.74 Hz,H-6/6′),推测该结构中存在邻位取代苯环单元;1H NMR还显示的一个氮甲基信号δH 3.26 (s),并结合该化合物的分子量,推测该化合物为二聚体;13C NMR (图 7B)和DEPT显示该化合物有一组羰基信号δc 168.0 (C-2/2′);三组不饱和季碳信号δc 145.2 (C-9/9′)、δc 133.4 (C-3/3′)、δc 121.6 (C-4/4′);四组不饱和次甲基碳信号δc 132.4 (C-7/7′)、δc 129.8 (C-5/5′)、δc 122.4 (C-6/6′)、δc 107.7 (C-8/8′);一组与氮相连的甲基碳信号δc 26.1 (C-10/10′) (图 7B)。信号δH 3.49 (s)和δc 50.9为甲醇的核磁信号。结合1H-1H COSY、HSQC、HMBC等2D NMR光谱,确定化合物3为N, N-二甲基异靛蓝(N, N-dimethylisoindigo),化学结构如图 1所示,核磁数据与文献[17]报道一致。
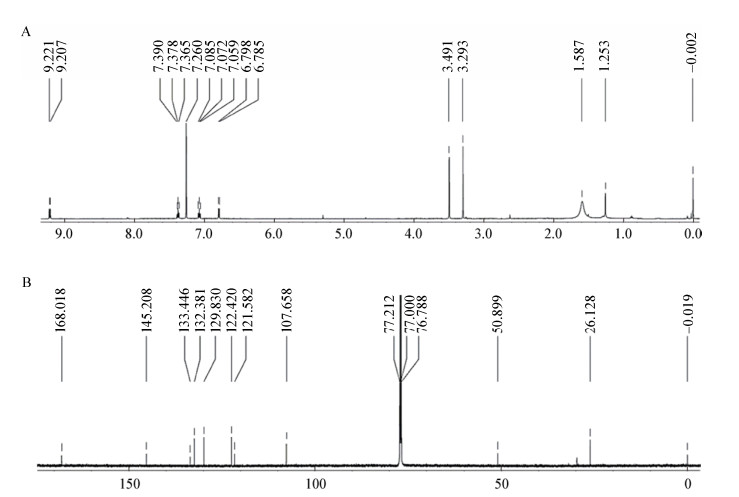
|
| 图 7 化合物3的一维核磁共振谱图 Figure 7 The 1D NMR spectra of compound 3 注:A:氢谱(600 MHz);B:碳谱(150 MHz). Note: A: 1H NMR (600 MHz); B: 13C NMR (150 MHz). |
|
|
化合物1色胺酮具有非常广泛的生物活性,主要包括抗菌、抗炎症、抗肿瘤、抗疟疾、抗原生虫、抗氧化等[18-20]。最显著的活性是对结核分枝杆菌的抑制作用,体外实验证明,它可以抑制多种类型的结核分枝杆菌,尤其是对多重抗药性的结核分枝杆菌,MIC值是0.5−1 μg/mL,活性远远高于肺结核的临床药物异烟肼(MIC 4−16 μg/mL),但体内实验尚未报道[21]。同时色胺酮在体内对多种肿瘤细胞具有抗性。目前色胺酮的来源主要有3种:一是从植物中提取;二是Hosoe等在担子菌属裂褶菌的发酵液里提取到[22];三是人工化学合成,主要方法是以吲哚醌和靛红酸酐为底物,以N-甲基哌啶为催化剂,在吡啶溶剂中回流1 h,产率达到75%−95%,但底物合成复杂,成本高,污染大[18]。本研究是首次在链霉菌发酵液里发现色胺酮,为其生物合成途径的研究奠定了基础。
化合物2甲基异靛蓝是靛玉红的衍生物,凭借活性高,副作用小,替代了靛玉红,临床上用来治疗慢性粒细胞白血病(CML)[23],且有望成为治疗急性早幼粒细胞白血病(APL)和急性骨髓性白血病(AML)的药物[24]。目前甲基异靛蓝采用人工化学方法合成,这是首次从微生物的发酵液中分离得到。化合物3是化合物2的N-CH3衍生物,水溶性较化合物2明显降低,且完全没有生物活性。
这3个化合物均含有2-吲哚酮结构单元。我们前期研究Maremycins生物合成机制过程中,阐明了血红素依赖的色氨酸双加氧酶在还原条件下特异性地催化3-取代吲哚经环氧中间体生成2-吲哚酮化合物[25]。因此,我们推测这3个化合物可能同样利用了MarE催化吲哚形成的环氧中间体,以不同的形式分别形成了化合物1和化合物2、3,如图 8所示。如果MarE催化吲哚直接形成2-吲哚酮,那么2个2-吲哚酮在氧化酶催化下发生偶联脱氢反应,形成化合物2的前体(图 8的途径A)。这个氧化酶可能类似于催化3-氧吲哚发生氧化二聚生成靛玉红的氧化酶[26],将有利于我们通过对产生菌基因组的分析挖掘到该氧化酶。该前体经氮甲基转移酶的催化下发生了2次加载甲基的过程[27],第一次加载甲基形成了化合物2,第二次加载甲基形成了化合物3。

|
| 图 8 3个化合物生物合成途径的推测 Figure 8 The proposed biosynthetic pathway for three compounds |
|
|
如果环氧中间体在邻氨基苯甲酸存在下,邻氨基苯甲酸氮上的孤对电子进攻这个中间体就形成了B途径中的化合物B1,然后B1发生脱氢和环化反应,形成内酰胺化合物B2,B2最后自发或酶催化发生脱氢反应形成芳香化合物1 (图 8的途径B)。依据推测途径,我们可以寻找相关基因和酶,继而利用代谢工程改造菌株,使之高产甲基异靛蓝和色胺酮,这将极大降低两个化合物的生产成本并保护环境,有很好的应用价值和社会效益。
| [1] |
Beutler JA. Natural products as a foundation for drug discovery[J]. Current Protocols in Pharmacology, 2009, 46: 9.11.1-9.11.21. |
| [2] |
Mishra BB, Tiwari VK. Natural products: an evolving role in future drug discovery[J]. European Journal of Medicinal Chemistry, 2011, 46(10): 4769-4807. DOI:10.1016/j.ejmech.2011.07.057 |
| [3] |
Blunt JW, Munro MHG. Is there an ideal database for natural products research?[A]//Natural Products: Discourse, Diversity, and Design[M]. Hoboken: John Wiley & Sons, Inc., 2014: 413-433
|
| [4] |
Mohamed A, Nguyen CH, Mamitsuka H. Current status and prospects of computational resources for natural product dereplication: a review[J]. Briefings in Bioinformatics, 2016, 17(2): 309-321. DOI:10.1093/bib/bbv042 |
| [5] |
Challis GL. Mining microbial genomes for new natural products and biosynthetic pathways[J]. Microbiology, 2008, 154(6): 1555-1569. DOI:10.1099/mic.0.2008/018523-0 |
| [6] |
Scherlach K, Hertweck C. Discovery of aspoquinolones A–D, prenylated quinoline-2-one alkaloids from Aspergillus nidulans, motivated by genome mining[J]. Organic & Biomolecular Chemistry, 2006, 4(18): 3517-3520. |
| [7] |
Pérez-Victoria I, Martín J, Reyes F. Combined LC/UV/MS and NMR strategies for the dereplication of marine natural products[J]. Planta Medica, 2016, 82(9/10): 857-871. DOI:10.1055/s-00000058 |
| [8] |
Zani CL, Carroll AR. Database for rapid dereplication of known natural products using data from MS and fast NMR experiments[J]. Journal of Natural Products, 2017, 80(6): 1758-1766. DOI:10.1021/acs.jnatprod.6b01093 |
| [9] |
Chervin J, Stierhof M, Tong MH, et al. Targeted dereplication of microbial natural products by high-resolution MS and predicted LC retention time[J]. Journal of Natural Products, 2017, 80(5): 1370-1377. DOI:10.1021/acs.jnatprod.6b01035 |
| [10] |
Mohimani H, Gurevich A, Mikheenko A, et al. Dereplication of peptidic natural products through database search of mass spectra[J]. Nature Chemical Biology, 2017, 13(1): 30-37. DOI:10.1038/nchembio.2219 |
| [11] |
Nielsen KF, Månsson M, Rank C, et al. Dereplication of microbial natural products by LC-DAD-TOFMS[J]. Journal of Natural Products, 2011, 74(11): 2338-2348. DOI:10.1021/np200254t |
| [12] |
Balk-Bindseil W, Helmke E, Weyland H, et al. Maremycin A and B, new diketopiperazines from a marine Streptomyces sp.[A]//Liebigs Annalen[M]. Wiley-VCH, 1995: 1291-1294
|
| [13] |
Tang YQ, Sattler I, Thiericke R, et al. Maremycins C and D, new diketopiperazines, and Maremycins E and F, novel polycyclic spiro-indole metabolites isolated from Streptomyces sp[J]. European Journal of Organic Chemistry, 2001, 2001(2): 261-267. DOI:10.1002/(ISSN)1099-0690 |
| [14] |
Xu XF, Fang Q, Liang ZT, et al. Chromomycin A3 and its analogues from the marine Streptomyces sp. B9173[J]. Genomics and Applied Biology, 2015, 34(5): 1013-1020. (in Chinese) 徐向飞, 方启, 梁祖韬, 等. 海洋链霉菌Streptomyces sp. B9173的色霉素A3及其类似物[J]. 基因组学与应用生物学, 2015, 34(5): 1013-1020. |
| [15] |
Liu Y, Ouyang F, Yu HY, et al. Chemical constituents in the leaves of Baphicacanthus cusia (Nees) Bremek[J]. Chinese Journal of Medicinal Chemistry, 2009, 19(4): 273-275, 283. (in Chinese) 刘远, 欧阳富, 于海洋, 等. 马蓝叶化学成分研究[J]. 中国药物化学杂志, 2009, 19(4): 273-275, 283. |
| [16] |
Xi KW, Yeo WK, Zhang B, et al. Synthesis and evaluation of functionalized isoindigos as antiproliferative agents[J]. Bioorganic & Medicinal Chemistry, 2009, 17(21): 7562-7571. |
| [17] |
Bergman J, Romero I. Synthesis of α-oxo-sulfines in the indole series[J]. Journal of Heterocyclic Chemistry, 2010, 47(5): 1215-1220. DOI:10.1002/jhet.v47:5 |
| [18] |
Gholamzadeh P, Ziarani GM, Badiei A, et al. Efficient green synthesis of isoindigo derivatives using sulfonic-acid- functionalized nanoporous silica (SBA-Pr-SO3H) catalyst and study of their antimicrobial properties[J]. Research on Chemical Intermediates, 2013, 39(9): 3925-3936. DOI:10.1007/s11164-012-0909-y |
| [19] |
Jung EH, Ji YJ, Ko HL, et al. Tryptanthrin prevents oxidative stress-mediated apoptosis through AMP-activated protein kinase-dependent p38 mitogen-activated protein kinase activation[J]. Archives of Pharmacal Research, 2017, 40(9): 1071-1086. DOI:10.1007/s12272-017-0947-5 |
| [20] |
Moon SY, Lee JH, Choi HY, et al. Tryptanthrin protects hepatocytes against oxidative stress via activation of the extracellular signal-regulated kinase/NF-E2-related factor 2 pathway[J]. Biological & Pharmaceutical Bulletin, 2014, 37(10): 1633-1640. |
| [21] |
Hudson A, Imamura T, Gutteridge W, et al. The current anti-TB drug research and development pipelines[R]. World Health Organization, 2003
|
| [22] |
Hosoe T, Nozawa K, Kawahara N, et al. Isolation of a new potent cytotoxic pigment along with indigotin from the pathogenic basidiomycetous fungus Schizophyllum commune[J]. Mycopathologia, 1999, 146(1): 9-12. DOI:10.1023/A:1007082619328 |
| [23] |
Xiao ZJ, Hao YS, Liu BC, et al. Indirubin and meisoindigo in the treatment of chronic myelogenous leukemia in China[J]. Leukemia & Lymphoma, 2002, 43(9): 1763-1768. |
| [24] |
Huang M, Lin HS, Lee YS, et al. Evaluation of meisoindigo, an indirubin derivative: in vitro antileukemic activity and in vivo pharmacokinetics[J]. International Journal of Oncology, 2014, 45(4): 1724-1734. DOI:10.3892/ijo.2014.2548 |
| [25] |
Zhang YY, Zou Y, Brock NL, et al. Characterization of 2-Oxindole forming heme enzyme MarE, expanding the functional diversity of the Tryptophan dioxygenase superfamily[J]. Journal of the American Chemical Society, 2017, 139(34): 11887-11894. DOI:10.1021/jacs.7b05517 |
| [26] |
Berry A, Dodge TC, Depsin M, et al. Application of metabolic engineering to improve both the production and use of biotech indigo[J]. Journal of Industrial Microbiology & Biotechnology, 2002, 28(3): 127-133. |
| [27] |
Lan YX, Zou Y, Huang TT, et al. Indole methylation protects diketopiperazine configuration in the maremycin biosynthetic pathway[J]. Science China Chemistry, 2016, 59(9): 1224-1228. DOI:10.1007/s11426-016-0026-7 |
 2018, Vol. 45
2018, Vol. 45