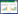扩展功能
文章信息
- 张宇, 王慧荣, 梅荣武, 李明智, 李欲如, 丁林贤, 苏晓梅, 朱瑞芬
- ZHANG Yu, WANG Hui-Rong, MEI Rong-Wu, LI Ming-Zhi, LI Yu-Ru, DING Lin-Xian, SU Xiao-Mei, ZHU Rui-Fen
- 来源于Micrococcus luteus的复苏促进因子异源表达及其对活性污泥中休眠细菌的促生长作用
- Heterologous expression of resuscitation promoting factor from Micrococcus luteus and promotes growth dormant bacteria from activated sludge
- 微生物学通报, 2018, 45(11): 2364-2375
- Microbiology China, 2018, 45(11): 2364-2375
- DOI: 10.13344/j.microbiol.china.171057
-
文章历史
- 收稿日期: 2017-12-17
- 接受日期: 2018-04-08
- 网络首发日期(www.cnki.net): 2018-04-12
2. 浙江师范大学地理与环境科学学院 浙江 金华 321004;
3. 浙江省环境科技有限公司 浙江 杭州 311121
2. College of Geography and Environmental Sciences, Zhejiang Normal University, Jinhua, Zhejiang 321004, China;
3. Zhejiang Environment Technology Co. Ltd., Hangzhou, Zhejiang 311121, China
复苏促进因子(Resuscitation promoting factor,RPF)是由某些放线菌分泌的活性蛋白质,能够复苏休眠细菌恢复生长,对正常生长的细菌也有促生长效果,复苏促进因子最初在藤黄微球菌(Micrococcus luteus)中被发现[1],随后在结核分枝杆菌属(Mycobacterium sp.)[2]、棒杆菌属(Corynebacterium sp.)[3]、链霉菌属(Streptomyces sp.)[4]等种属也发现了rpf同源基因。
来源于M. luteus RPF (MlRPF)除了对自身休眠态细胞具有复苏促进效果,还可以复苏其它几种高G+C%含量革兰氏阳性细菌的生长,表现出跨物种的活性[1-2, 5-6],而MlRPF也是迄今所发现作用效果最为敏感,在皮摩尔浓度(pmol/L)下便可复苏自身休眠态细胞[1]。目前RPF对休眠细胞复苏机制还不明确,已有的研究表明其自身具有溶菌酶活性[7],推测它利用溶菌酶活性切割细菌细胞壁组分,产生肽聚糖片段,该片段作为启动休眠态细菌生长的信号分子[8]。
目前有关RPF的研究主要集中在医学领域(致病菌的快速检测和诊断),而关于RPF对于特殊环境样品中除原始宿主以外细菌的复苏促进作用研究还处于刚起步阶段。近年来的研究均表明在污水生物处理系统、污染环境、及特殊环境样品中,RPF可以复苏除自身宿主以外的某些细菌,并且这些细菌在环保领域展现出了应用前景。Li等[9]利用M. luteus培养上清液所制备的RPF粗提液在制药废水中发现了新种细菌Gordonia jinhuaensis ZYR 51T。Su等[10]利用同样方法在多氯联苯污染的底泥中分离到了一株新型多氯联苯降解菌株Rhodococcus biphenylivorans TG9T。除此还证明了M. luteus所制备的RPF粗提液能显著提高PCBs污染土壤中菌群对联苯降解效率及菌群多样性[11-12]。Puspita等[13]利用来源于Tomitella biformata的复苏促进因子在冻土冰楔样品中分离到了一株Brevibacterium antiquum strain VKM Ac-2118相近菌株。
本文以寻找新的菌种资源、开发环境功能菌角度研究了RPF对活性污泥样品中潜在功能菌(群)的复苏促进作用,对来源于Micrococcus luteus IAM 14879的RPF (MlRPF)蛋白序列进行系统进化分析及序列比对,并同源模建了其空间结构;对其编码基因进行了克隆及大肠杆菌异源表达;利用大肠杆菌异源表达所制备的重组MlRPF,对活性污泥样品中潜在的微生物资源开展了复苏促生长研究。上述研究结果有利于更好地理解RPF促生长机理,为环境样品功能微生物资源的挖掘提供新的方法,也为最大限度地激发土著微生物对污染基质的降解效率及环保生物强化技术研发奠定理论基础。
1 材料与方法 1.1 菌株及质粒Micrococcus luteus IAM 14879为rpf基因来源菌,由浙江师范大学丁林贤教授馈赠;Escherichia coli BL21(DE3) CodonPlus用于目的基因克隆和表达,本实验室保存;表达质粒pET-28a(+)用于重组载体构建,为本实验室保存。
1.2 主要试剂、仪器及培养基限制性内切酶BamHⅠ、Hind Ⅲ、PrimeSTAR Max DNA Polymerase、T4 DNA连接酶、DNA及蛋白分子量标准,宝生物工程(大连)有限公司;细菌基因组提取试剂盒、DNA纯化试剂盒、质粒提取试剂盒及分析纯试剂,生工生物工程(上海)股份有限公司;蛋白胨、酵母提取物,OXOID公司;细菌微量培养瓶,亚速旺(上海)商贸有限公司;蛋白纯化用亲和层析介质NTA Agarose,QINGEN公司;寡核苷酸序列委托生工生物工程(上海)股份有限公司合成。
恒温摇床,上海智城分析仪器制造有限公司;生化培养箱,上海精宏实验设备有限公司;高压蒸汽灭菌锅,Panasonic公司;台式微量离心机,BECKMAN COULTER公司;NanoDrop 2000超微量紫外分光光度计、PCR仪、超低温冰箱,Thermo Fisher Scientific公司;电泳仪,上海天能科技有限公司;凝胶成像系统,Bio-Rad公司。
LMM培养基(g/L):NH4Cl 4.000,KH2PO4 1.400,生物素0.005,L-蛋氨酸0.020,维生素B1 0.040,肌苷1.000,MgSO4 0.030,L-乳酸锂盐10.000,1.0 mL矿物质盐溶液(0.375 g CuSO4·5H2O、0.785 g MnCl2·4H2O、0.183 g FeSO4·7H2O、0.029 g Na2MoO4·2H2O和0.089 g ZnSO4·7H2O),调节pH至7.5,1×105 Pa灭菌20 min。
LB固体培养基(g/L):蛋白胨10.0,酵母提取物5.0,NaCl 10.0,琼脂粉15.0,调节pH至7.0,1×105 Pa灭菌20 min,待冷却至50-60 ℃时倒制平板。
改良SOB培养基(g/L):胰化蛋白胨20.0,酵母提取物5.0,NaCl 0.5,KCl 2.5 mmol/L,将原配方中10 mmol/L MgCl2降低到8.5 mmol/L,1×105 Pa灭菌20 min。
1.3 序列比对及同源模建将M. luteus的RPF蛋白序列通过BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi)与GenBank数据库中不同细菌来源的复苏促进因子做同源性比较,选取典型的RPF序列利用ClustralX 1.83软件进行多重序列比对后应用MEGA 4.0.2软件构建系统发育树。基于结构的序列比对利用在线工具ESPript (http://espript.ibcp.fr)完成。
同源建模由Phyre2 [14]在线完成(http://www.sbg.bio.ic.ac.uk/phyre2),将MlRPF的氨基酸序列输入三级结构模建服务器Phyre2中,服务器利用穿珠法以PDB数据库中已解析的三级结构为模板分别产生多个模型,随后利用蛋白质结构评价服务器PSVS (http://psvs-1_5-dev.nesg.org/)对这些模型进行评估和排序,最后汇总评估信息确定MlRPF的三维结构模型。
1.4 rpf基因克隆提取Micrococcus luteus基因组DNA,rpf基因(GenBank编号CP001628)通过PCR从上述菌株的基因组DNA中扩增得到。上游引物:5′-GCGCGGATCCATGGACACCATGACTCTCTTCACC-3′,下游引物:5′-GATCAAGCTTTCAGGCCTG CGGCAGGACGAGCTCC-3′,划线部分为BamHⅠ和Hind Ⅲ酶切位点。PCR反应体系:M. luteus IAM 14879基因组DNA (50 ng/μL) 1 μL,上、下游引物(20 pmol/μL)各1 μL,PrimeSTAR Max Premix (2×) 25μL,灭菌去离子水补足到50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 5 min。反应结束电泳检测PCR产物。PCR产物经限制性内切酶酶切,应用同样酶切割pET-28a质粒。然后将酶切的目的基因片段与线性载体片段利用T4 DNA连接酶连接,构建重组质粒pET-28a-rpf。连接产物转化E. coli BL21(DE3) Codon Plus感受态细胞,用含50 μg/mL卡那霉素的LB固体培养基进行阳性克隆筛选,随机挑取单克隆进行测序验证。
1.5 蛋白质表达和纯化将测序验证正确的工程菌株接种到200 mL改良的SOB液体培养基[7]中(含50 μg/mL卡那霉素),37 ℃、180 r/min振荡培养,待菌体生长到OD600为0.6-0.8时,加入终浓度0.001 mol/L IPTG诱导,24 ℃、100 r/min培养12-16 h后,4 ℃、5 000 r/min离心5 min后收集菌体。加入5-10倍体积的0.05 mol/L Tris-HCl缓冲液(pH 8.0)重悬菌体,超声(500 W,超声2 s间隔2 s,共20 min)破碎后4 ℃、12 000 r/min离心10 min收集上清得到蛋白粗提液。因为所表达的目标蛋白N-端含有组氨酸标签,应用镍亲和柱对重组蛋白进行纯化,用0.1 mol/L咪唑洗脱目的蛋白,洗脱体积为10 mL。利用SDS-PAGE电泳检测目标蛋白的纯度。最后所洗脱的蛋白组分经2次透析去除咪唑,经0.22 μm滤膜抽滤灭菌后分装保存于-20 ℃待用。
1.6 M.luteus IAM 14879的休眠状态诱导、RPF生理活性验证及不同浓度RPF对复苏促进活力的影响Micrococcus luteus IAM 14879休眠状态诱导采用Puspita等[15]方法,在限制溶氧条件下诱导细胞进入不分裂状态:首先将Micrococcus luteus IAM 14879甘油保存菌液接种到含有LMM培养基(Lactate minimal medium)的微量培养瓶中,拧紧瓶盖使其密封,30 ℃、140 r/min振荡培养10 d,随后静置50 d。将100 μL缺氧状态的菌液分别接种到10 mL LMM培养基中,实验组添加终浓度为5 μg/mL纯化的重组MlRPF蛋白,对照组添加等量高温失活的重组MlRPF蛋白,30 ℃、140 r/min培养120 h,期间每间隔24 h在LMM固体平板上测定CFU,每组平行测定3次。
将缺氧诱导进入休眠状态的M. luteus IAM 14879等量接种到分别含有终浓度为5 pg/mL (0.19 pmol/L)、5 ng/mL (0.19 nmol/L)、5 μg/mL (0.19 μmol/L)纯化的MlRPF的10 mL LMM培养基中,30 ℃、140 r/min培养。每间隔24 h测定OD600,监测M. luteus生长情况,每组平行测定3次,以添加灭菌的去离子水作为对照。
1.7 RPF复苏活性污泥样品中潜在休眠细菌及其对垃圾渗滤液废水的强化处理活性污泥样品采自杭州市某垃圾渗滤液处理厂好氧池,取1 mL泥水混合样品,4 ℃、5 000 r/min离心2 min后弃上清,用1 mL灭菌去离子水涡旋混匀洗涤,4 ℃、5 000 r/min离心2 min后得上清。利用MPN (Most probable number)计数法培养:实验共分为两组(RPF处理组和RPF对照组),每组3个平行实验。取100 μL污泥上清液接种于装有LB液体培养基的微量培养瓶中,记为10-1,摇匀后再从10-1瓶取100 μL混合液到下一个培养瓶中,记为10-2,依次类推至10-6瓶,最后逐个培养瓶中添加终浓度为5 μg/mL重组MlRPF蛋白;另外一组同样操作添加等量高温失活的MlRPF蛋白作为对照。30 ℃、160 r/min振荡培养,期间观察培养液澄清度,测定菌液密度,每组3个平行实验。然后通过平板划线法对培养瓶中的菌液进行分离纯化,根据菌落形态特征初步判断菌种的种属,挑取单个菌落扩大培养后将菌液送交生工生物工程(上海)股份有限公司进行16S rRNA基因鉴定分析。
该垃圾渗滤液处理厂采用生物转盘偶联A/O工艺,现场采取该生物转盘出水,在连续流小型A/O装置中评估RPF对该废水的强化处理效果,设置2组,分别为对照组和处理组(图 1)。厌氧和好氧反应器内径均为140 mm,高度为200 mm,有效容积约为3 L,其中填充聚氨酯填料,将现场二沉池采取的活性污泥分别等量接种到2套小试装置中,装置进水取自现场生物转盘出水,调节进水流速,使2套装置停留时间均为24 h,好氧反应器溶解氧浓度控制在1-2 mg/L,温度为室温,连续运行30 d,其中前10天为污泥驯化阶段,系统内循环,处理组添加终浓度为10%(体积比)的RPF粗提液(制备方法同1.5),对照组添加等量高温灭活的RPF粗提液,随后20 d连续进水,期间定期取样,分别测定进出水的CODCr、NH3-N和TN,测定方法参照标准方法[16]。
|
| 图 1 本研究所使用的A/O装置 Figure 1 The A/O equipment used in this study 注:A:流程图;B:实物图. Note: A: Schematic illustration; B: Actual equipment. |
|
|
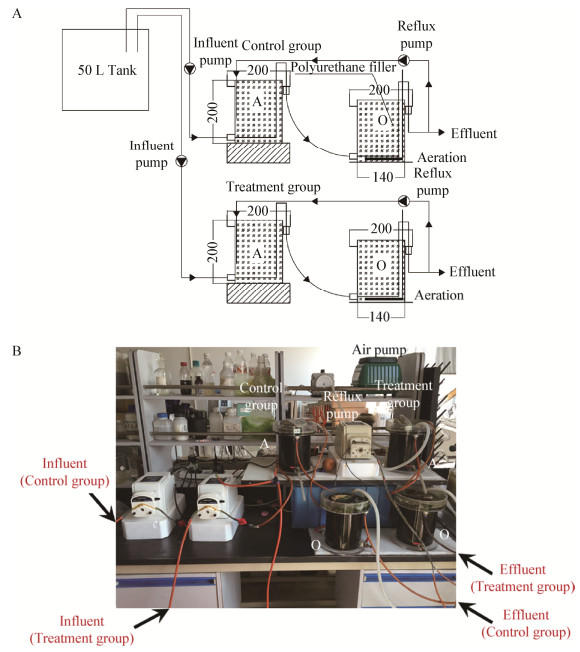
系统进化树表明MlRPF与来源于Glutamicibacter nicotianae的复苏促进因子(蛋白登录号为ABB51647)处于同一分支(图 2),二者序列相似性为93%,而一致性为34%,亲缘关系较近,而远离其它RPF家族成员;与来源于Mycobacterium tuberculosis H37Rv的5个同源蛋白Rpf A-E具有较低的序列一致性(序列一致性为10.2%-23.5%),进化树分析表明不同细菌来源的RPF在序列长度、遗传结构及宿主种类存在较大差异。
|
| 图 2 来源于M. luteus RPF (MlRPF)及其同源蛋白的系统发育树 Figure 2 Phylogenetic tree of M. luteus RPF (MlRPF) and other RPF homology proteins 注:方框内为MlRPF;括号内编号为GenBank中蛋白序列登录号;节点上的数字为Bootstrap值(1 000次重复抽样的百分比);比例尺代表遗传距离为0.02,每100个氨基酸有两个不同. Note: The MlRPF is indicated in square; Numbers in bracket are the protein sequences accession number in GenBank; Bootstrap values (expressed as percentages of 1 000 replications) are given at nodes; The scale bar represents the genetic distance is 0.02, indicates two differences per 100 amino acids. |
|
|
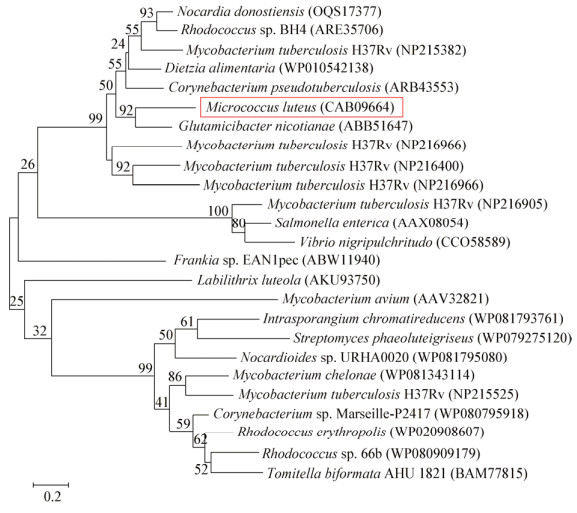
虽然在不同细菌来源的RPF蛋白序列长度存在较大差异,但这些序列N末端具有一段保守区域,称为“RPF结构域”,长度约75个氨基酸,所有的RPF结构域与C型溶菌酶家族具有同源性,活性中心均具有高度保守谷氨酸残基(图 3),该谷氨酸残基对于复苏活性至关重要[17-18]。

|
| 图 3 MlRPF及其同源蛋白“RPF结构域”区域多序列比对 Figure 3 The "RPF-domain" region of the multiple sequence alignment of MlRPF with other homology proteins 注:星号标注了RPF家族高度保守的谷氨酸残基. NP216400:RpfC (PDB ID: 2N5Z),来源于Mycobacterium tuberculosis H37Rv;CAB09664:MlRPF,来源于M. Luteus;ABB51647:来源于Glutamicibacter nicotianae;OQS17377:来源于Nocardia donostiensis;WP010542138:来源于Dietzia alimentaria;ARE35706:来源于Rhodococcus sp. BH4;ARB43553:来源于Corynebacterium pseudotuberculosis;NP215382:RpfA,来源于M. tuberculosis H37Rv;NP216966:RpfE,来源于M. tuberculosis H37Rv;NP216905:RpfD,来源于M.H37Rv. Note: Conserved glutamate residues in RPF family are labeled in asterisk. NP216400: RpfC (PDB ID: 2N5Z) from Mycobacterium tuberculosis H37Rv; CAB09664: MlRPF from M. luteus; ABB51647 from Glutamicibacter nicotianae; OQS17377 from Nocardia donostiensis; WP010542138 from Dietzia alimentaria; ARE35706 from Rhodococcus sp. BH4; ARB43553 from Corynebacterium pseudotuberculosis; NP215382: RpfA from M. tuberculosis H37Rv; NP216966: RpfE from M. tuberculosis H37Rv; NP216905: RpfD from M. H37Rv. |
|
|
对该区域(Ala42-Leu120)进行同源模建,结果表明MlRPF的“Rpf结构域”由4个α-螺旋组成,活性中心54位谷氨酸位于其中一个α-螺旋上(图 4A);结构比对发现MlRPF与Mycobacterium tuberculosis RpfC (PDB ID: 2N5Z)具有极其相似的空间结构,主链碳原子均方根偏差(Root-mean-square Deviation,RMSD)值为2.23 Å,活性中心谷氨酸残基在空间位置上几乎重叠(图 4B);而与典型的C型溶菌酶空间结构比对表明模建的MlRPF与C型溶菌酶具有相似的空间结构(图 4C),主链碳原子的RMSD为7.806 Å,这表明这2种蛋白具有相似的生物学功能,即具有溶菌酶活性,这有助于进一步解释RPF的复苏促进机制。
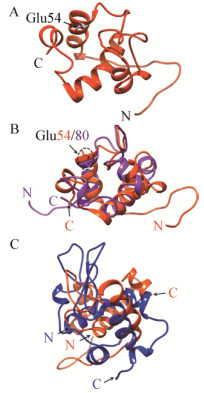
|
| 图 4 复苏促进因子结构 Figure 4 The structure of the "RPF-domain" 注:A:MlRPF模建结构,箭头指示了活性中心54位谷氨酸;B:MlRPF模建结构(桔色),与来源于Mycobacterium tuberculosis RpfC (PDB ID:2N5Z) (紫色)结构比对;C:M. luteus模建结构(桔色),与来源于Oncorhynchus mykiss的C型溶菌酶(PDB ID:1LMQ) (蓝色)结构比对. Note: A: Structure of modeled MlRPF, the arrow indicates the Glu of active center at position 54; B: Structural superimposed between modeled MlRPF (orange) and RpfC (purple) from Mycobacterium tuberculosis (PDB ID: 2N5Z); C: Structural superimposed between modeled MlRPF (orange) and C-type lysozyme (blue) from Oncorhynchus mykiss (PDB ID: 1LMQ). |
|
|
由于M. luteus IAM 14879自身分泌的复苏促进因子含量极低、提取困难,因此不利于直接利用培养藤黄微球菌来制备复苏促进因子。我们将rpf基因插入到表达质粒pET-28a(+)中,该质粒可使表达的目的蛋白在N端带有组氨酸标签,能够与特异亲和组氨酸标签的镍离子亲和柱相结合,方便纯化。测序验证正确的重组质粒转化E. coli BL21(DE3) Codon Plus,工程菌株诱导表达后,全细胞破碎液利用镍亲和柱纯化,最后经Washing buffer (含0.1 mol/L咪唑)洗脱,可以得到高纯度的目的蛋白,在20–30 kD之间获得均一蛋白条带(图 5),且与理论预测分子量相符,这表明来源于M. luteus的复苏促进因子基因在大肠杆菌中实现了可溶性表达,1 L培养液中大约能获得100 mg的目的蛋白。构建了表达MlRPF的基因工程菌,至此可以简单、快速地进行MlRPF制备。

|
| 图 5 SDS-PAGE分析MlRPF蛋白在大肠杆菌中表达 Figure 5 SDS-PAGE analysis of recombinant MlRPF expressed in E. coli 注:M:蛋白分子量标准;R:镍亲和柱纯化获得的重组MlRPFR组分(0.1 mol/L咪唑洗脱). Note: M: Protein molecular weight standard; R: Purified MlRPF fractions by Ni-NTA affinity column (eluted with 0.1 mol/L imidazole). |
|
|
为了验证大肠杆菌异源表达所制备的重组MlRPF复苏促进活性,将Micrococcus luteus IAM 14879经过60 d缺氧诱导,此时细菌总数低至3.28×103 CFU/mL。考察了MlRPF对缺氧条件诱导的M. luteus促生长效果,如图 6A所示,培养72 h后,MlRPF处理组细菌总数开始增长,并进入到对数生长期,对数期相比对照组(添加失活MlRPF)显著缩短;培养120 h后,添加MlRPF的培养液中细菌总数达到1.01×108 CFU/mL,而对照细菌总数为2.22×107 CFU/mL,提高4.5倍。这一结果表明利用大肠杆菌异源表达所制备的重组MlRPF维持了其生理活性,即能够促进休眠状态M. luteus生长。Puspita等[15]的研究也表明来源于Tomitella biformata AHU1821的复苏促进因子(经大肠杆菌表达系统制备获得)对不分裂状态的T. biformata具有明显促生长效果,且随着添加复苏促进因子浓度的提高,菌体生长加快,进入对数生长期的时间显著缩短。另外不同来源的复苏促进因子促生长活力存在显著差异,M. luteus的RPF极其灵敏,作用浓度限为皮摩尔(pmol/L)[1],而T. biformata的RPF作用浓度限为那摩尔(nmol/L)[15],不同细菌来源的RPF复苏活性差异可能与蛋白质氨基酸序列及空间结构相关。
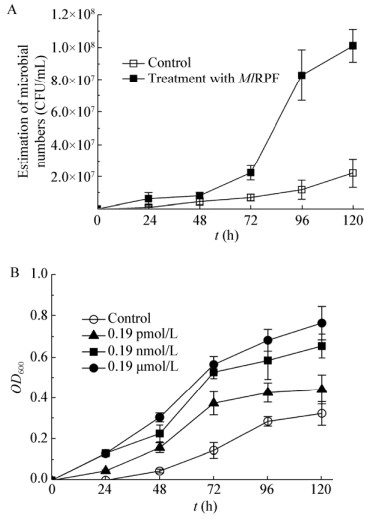
|
| 图 6 MlRPF对缺氧诱导的M. luteus IAM 14879的复苏活性 Figure 6 The growth-promoting activity of MlRPF on M. luteus IAM 14879 with induced under oxygen-limited culture conditions 注:A:MlRPF对休眠态M. luteus IAM 14879的促生长曲线;B:不同浓度MlRPF处理对休眠态的M. luteus IAM 14879的影响. Note: A: The growth-promoting curves of dormant M.luteus IAM 14879 after treatment with MlRPF; B: Effect of different MlRPF protein concentrations on dormant M. luteus IAM 14879. |
|
|
进一步考察了不同浓度量级(分别为pmol/L、nmol/L和μmol/L)的MlRPF对复苏活性的影响,如图 6B所示,添加不同浓度量级的MlRPF对于休眠状态的M. luteus均具有显著的复苏活性,且随着浓度提高复苏活性增强,在pmol/L、nmol/L和μmol/L作用浓度下,相比对照复苏活性分别提高1.4、2.0和2.4倍,该结果进一步验证了M. luteus的RPF具有极其敏感的作用浓度,这种敏感性可能取决于蛋白质活性中心氨基酸种类及独特的空间结构,该假设需要今后做更深入地研究验证。
2.4 MlRPF对活性污泥中休眠细菌的复苏促进作用利用MlRPF复苏促进作用发掘污水生物处理系统中潜在的土著微生物类群,最大限度地激发土著微生物对污染基质的降解效率,用于污水的强化处理和生物修复。采集了某垃圾渗滤液处理厂的活性污泥样品,利用纯化的MlRPF对其中菌群开展了复苏促生长研究,利用MPN计数法培养,如图 7所示,RPF处理组和对照组在低稀释浓度(10-1-10-3)情况下都存在明显混浊现象;而在10-4稀释浓度下,对照组仅有轻微浑浊现象,RPF处理组呈现明显浑浊现象;在10-5和10-6稀释浓度下对照组都表现澄清无混浊现象;而在同一稀释段的RPF处理组中的两个培养瓶均呈现明显浑浊。这些结果表明MlRPF对供试污泥样品中的菌群有较明显的促进作用,在高稀释浓度下,添加活性MlRPF处理的培养瓶中菌液密度明显比对照组高,即菌液浓度高,说明供试污泥样品中可能存在一定数量的休眠(或不占优势生长较为缓慢)的细菌种属。随后进一步对培养瓶中的细菌进行了分离和纯化,通过反复划线分离培养后,对不同形态的菌种分离纯化。经16S rRNA基因鉴定,结果见表 1,经RPF处理所获得的4个菌株分别为Dietzia、Paracoccus、Rhodococcus、Brevundimonas;而其余菌株在对照组和MlRPF处理组均分离获得。虽然我们没有对这些菌株逐一进行性质表征,但这些菌属在废水处理及生态修复领域均具有较好的应用前景,其中最近相似株D. sp. ZQ-4可以高效降解中长链烷烃[19];P. pantotrophus具有高效异养硝化好氧反硝化功能[20];R. sp. ZH8可以降解芳香族化合物[21]及长链烷烃[22];B. sp. YIM KMY42-2能够吸收重金属[23]、降解有机磷杀虫剂[24]。我们将16S rRNA基因测序所获得的序列构建了系统进化树,如图 8所示,经RPF复苏所获得的1号Dietzia和3号Rhodococcus菌株与M. luteus处于同一分支,与2号Paracoccus和5号Brevundimonas距离较远,这些结果表明MlRPF对除自身宿主(M. luteus)以外的某些微生物类群同样具有复苏促进效果,具有广谱性。
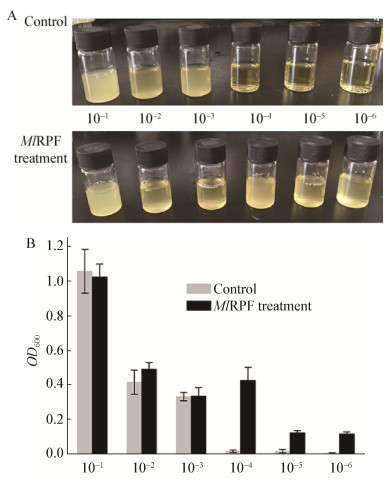
|
| 图 7 活性污泥样品经MlRPF处理培养120 h后的菌密度分析 Figure 7 Analysis of bacterial density of activated sludge sample by treatment with MlRPF after 120 h culture 注:A:不同稀释度培养瓶中菌密度观察;B:经MlRPF处理活性污泥培养120 h后细菌光密度对比. Note: A: Observation of bacterial density in different dilution bottles; B: Comparison of the bacterial optical density of activated sludge sample by treatment with MlRPF after 120 h culture. |
|
|

|
| 图 8 基于16S rRNA基因序列从活性污泥中分离获得细菌的系统进化树 Figure 8 Phylogenetic tree of bacteria strains from activated sludge sample based on 16S rRNA gene 注:括号内编号为GenBank中基因序列登录号; 节点上的数字为Bootstrap值(1 000次重复抽样的百分比);比例尺代表遗传距离为0.02,每100个核苷酸有两个不同. Note: Numbers in bracket are the nucleotide sequences accession number in GenBank; Bootstrap values (expressed as percentages of 1 000 replications) are given at nodes; The scale bar represents the genetic distance is 0.02, indicates two differences per 100 nucleotides. |
|
|
| 编号 Number |
最近相似细菌 The most similar bacteria |
GenBank登录号 GenBank No. |
一致性 Identity (%) |
| 1* | Dietzia sp. ZQ-4 | JQ809512.1 | 97 |
| 2* | Paracoccus pantotrophus | AB681113.1 | 98 |
| 3* | Rhodococcus sp. ZH8 | EU167912.1 | 98 |
| 4# | Bacillus firmus strain PAN MC15 | HQ285922.1 | 99 |
| 5* | Brevundimonas sp. YIM KMY42-2 | DQ358649.1 | 99 |
| 6# | Pseudomonas stutzeri strain LS401 | U26417.1 | 99 |
| 7# | Bacillus sp. CMJ1-20 | KC119106.1 | 99 |
| 8# | Microbacterium sp. JCM28704 | LC133735.2 | 100 |
| 9# | Bacillus amyloliquefaciens | KU922448.1 | 100 |
| 注:*:仅在MlRPF处理组中分离获得;#:在对照组和MlRPF处理组均分离获得. Note: *: Only obtained in the MlRPF treatment group; #: Both the control group and the MlRPF treatment group are isolated. | |||
随后我们利用两套平行连续流小试A/O装置,模拟取样现场的废水处理工艺,进一步表征MlRPF对废水的强化处理效果,处理组添加MlRPF粗提液,对照组添加等量高温灭活的MlRPF粗提液,两组装置活性污泥经过10 d的驯化后连续进水,两套装置出水的COD、NH4+-N和TN曲线变化相似,即前10天缓慢下降,随后稳定(图 9A-C):现场采取水样的COD平均值为505.61±36.82 mg/L,经小试装置处理后,污泥驯化期间(前10天)两套装置COD缓慢下降,随后稳定(图 9A),对照组运行后20 d出水COD平均值为170.06±20.44 mg/L,而处理组出水COD平均值为145.81±12.60 mg/L,相比对照组COD处理效率提高7.2%;现场采取水样的NH4+-N平均值为27.96±0.81 mg/L,对照组运行后20 d的出水NH4+-N平均值为11.67±1.02 mg/L,而处理组平均值为10.55±0.97 mg/L,相比对照组NH4+-N处理效率提高6.9%;现场采取水样的TN平均值为31.07±1.61 mg/L,对照组运行后20 d出水TN平均值为18.98±1.87 mg/L,而处理组出水平均值为17.05±2.64 mg/L,相比对照组TN处理效率提高16.0%,Liu等[25]研究也表明利用M. luteus培养上清所制备的RPF粗提液(SRpf)可以使小型序批式反应器对于人工模拟废水TN的去除效率提高8%。我们的研究结果进一步表明活性污泥经MlRPF驯化后可极大限度地激发污水生化处理系统中土著微生物对污染基质的降解效率,进而为基于MlRPF的废水生物强化处理及生物修复技术研发提供理论依据。

|
| 图 9 两套平行A/O装置连续运行30 d期间的水质变化 Figure 9 The variations of water qualityfrom two sets of lab-scale A/O equipment during continuous running for 30 days 注:A:化学需氧量;B:氨氮;C:总氮. Note: A: Chemical oxygen demand, COD; B: Ammonia nitrogen; C: Total nitrogen, TN. |
|
|
对来源于Micrococcus luteus的复苏促进因子(MlRPF)的蛋白序列进行了生物信息学分析及结构建模,结果表明MlRPF具有与C型溶菌酶相似的空间结构,这有助于进一步理解复苏促进因子的作用机制。
克隆了来源于M. luteus的rpf基因,在大肠杆菌中实现了可溶性表达,经大肠杆菌表达系统所制备的纯化MlRPF保持了其复苏活性,它可以使经缺氧诱导进入休眠状态的M. luteus群落总数提高4.5倍。
利用MlRPF对活性污泥样品潜在的微生物菌群开展了复苏研究,验证了污水生化处理系统中存在一定数量的休眠细菌,在经MlRPF处理的污泥样品中分离获得了Dietzia、Paracoccus、Rhodococcus、Brevundimonas菌株,表明MlRPF对除自身宿主(M. luteus)以外的某些微生物类群同样具有复苏促进效果,具有广谱复苏活性,这有助于极大限度地激发污水生化处理系统中土著微生物对污染基质的降解效率。这些结果一方面为环境样品中微生物多样性研究及特殊微生物资源的发掘提供新的途径。另一方面为MlRPF在废水生物强化处理及生物修复领域的应用奠定基础。
| [1] |
Mukamolova GV, Kaprelyants AS, Young DI, et al. A bacterial cytokine[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(15): 8916-8921. DOI:10.1073/pnas.95.15.8916 |
| [2] |
Mukamolova GV, Turapov OA, Young DI, et al. A family of autocrine growth factors in Mycobacterium tuberculosis[J]. Molecular Microbiology, 2002, 46(3): 623-635. DOI:10.1046/j.1365-2958.2002.03184.x |
| [3] |
Hartmann M, Barsch A, Niehaus K, et al. The glycosylated cell surface protein Rpf2, containing a resuscitation-promoting factor motif, is involved in intercellular communication of Corynebacterium glutamicum[J]. Archives of Microbiology, 2004, 182(4): 299-312. DOI:10.1007/s00203-004-0713-1 |
| [4] |
Ravagnani A, Finan CL, Young M. A novel firmicute protein family related to the actinobacterial resuscitation-promoting factors by non-orthologous domain displacement[J]. BMC Genomics, 2005, 6: 39. DOI:10.1186/1471-2164-6-39 |
| [5] |
Schroeckh V, Martin K. Resuscitation-promoting factors: distribution among actinobacteria, synthesis during life-cycle and biological activity[J]. Antonie Van Leeuwenhoek, 2006, 89(3/4): 359-365. |
| [6] |
Ding LX, Zhang PH, Hong HC, et al. Cloning and expression of Micrococcus luteus IAM 14879 Rpf and its role in the recovery of the VBNC state in Rhodococcus sp. DS471[J]. Acta Microbiologica Sinica, 2012, 52(1): 77-82. (in Chinese) 丁林贤, 张萍华, 洪华嫦, 等. 藤黄微球菌Rpf活性蛋白的制取及其对红球菌VBNC菌体的复苏作用[J]. 微生物学报, 2012, 52(1): 77-82. |
| [7] |
Mukamolova GV, Murzin AG, Salina EG, et al. Muralytic activity of Micrococcus luteus Rpf and its relationship to physiological activity in promoting bacterial growth and resuscitation[J]. Molecular Microbiology, 2006, 59(1): 84-98. DOI:10.1111/MMI.2006.59.issue-1 |
| [8] |
Kana BD, Mizrahi V. Resuscitation-promoting factors as lytic enzymes for bacterial growth and signaling[J]. FEMS Immunology and Medical Microbiology, 2010, 58(1): 39-50. DOI:10.1111/fim.2010.58.issue-1 |
| [9] |
Li SH, Jin Y, Cheng J, et al. Gordonia jinhuaensis sp. nov., a novel actinobacterium, isolated from a VBNC (viable but non-culturable) state in pharmaceutical wastewater[J]. Antonie Van Leeuwenhoek, 2014, 106(2): 347-356. DOI:10.1007/s10482-014-0207-3 |
| [10] |
Su XM, Liu YD, Hashmi MZ, et al. Rhodococcus biphenylivorans sp. nov., a polychlorinated biphenyl-degrading bacterium[J]. Antonie Van Leeuwenhoek, 2015, 107(1): 55-63. DOI:10.1007/s10482-014-0303-4 |
| [11] |
Su XM, Shen H, Yao XY, et al. A novel approach to stimulate the biphenyl-degrading potential of bacterial community from PCBs-contaminated soil of e-waste recycling sites[J]. Bioresource Technology, 2013, 146: 27-34. DOI:10.1016/j.biortech.2013.07.028 |
| [12] |
Su XM, Zhang Q, Hu JX, et al. Enhanced degradation of biphenyl from PCB-contaminated sediments: the impact of extracellular organic matter from Micrococcus luteus[J]. Applied Microbiology & Biotechnology, 2015, 99(4): 1989-2000. |
| [13] |
Puspita ID, Kitagawa W, Kamagata Y, et al. Increase in bacterial colony formation from a permafrost ice wedge dosed with a Tomitella biformata recombinant resuscitation-promoting factor protein[J]. Microbes and Environments, 2015, 30(2): 151-156. DOI:10.1264/jsme2.ME14119 |
| [14] |
Kelley LA, Sternberg MJ. Protein structure prediction on the Web: a case study using the Phyre server[J]. Nature Protocols, 2009, 4(3): 363-371. DOI:10.1038/nprot.2009.2 |
| [15] |
Puspita ID, Uehara M, Katayama T, et al. Resuscitation promoting factor (Rpf) from Tomitella biformata AHU 1821T promotes growth and resuscitates non-dividing cells[J]. Microbes and Environments, 2013, 28(1): 58-64. DOI:10.1264/jsme2.ME12122 |
| [16] |
Wei FS. Water and Wastewater Monitoring and Analysis Method[M]. Beijing: China Environmental Science Press, 2002: 210-462. (in Chinese) 魏复盛. 水和废水监测分析方法[M]. 北京: 中国环境科学出版社, 2002: 210-462. |
| [17] |
Cohen-Gonsaud M, Keep NH, Davies AP, et al. Resuscitation-promoting factors possess a lysozyme-like domain[J]. Trends in Biochemical Sciences, 2004, 29(1): 7-10. DOI:10.1016/j.tibs.2003.10.009 |
| [18] |
Cohen-Gonsaud M, Barthe P, Bagnéris C, et al. The structure of a resuscitation-promoting factor domain from Mycobacterium tuberculosis shows homology to lysozymes[J]. Nature Structural and Molecular Biology, 2005, 12(3): 270-273. DOI:10.1038/nsmb905 |
| [19] |
Wang XB, Chi CQ, Nie Y, et al. Degradation of petroleum hydrocarbons (C6-C40) and crude oil by a novel Dietzia strain[J]. Bioresource Technology, 2011, 102(17): 7755-7761. DOI:10.1016/j.biortech.2011.06.009 |
| [20] |
Robertson LA, van Niel EWJ, Torremans RAM, et al. Simultaneous nitrification and denitrification in aerobic chemostat cultures of Thiosphaera pantotropha[J]. Applied and Environmental Microbiology, 1988, 54(11): 2812-2818. |
| [21] |
Martínková L, Uhnáková B, Pátek M, et al. Biodegradation potential of the genus Rhodococcus[J]. Environment International, 2009, 35(1): 162-177. DOI:10.1016/j.envint.2008.07.018 |
| [22] |
Whyte LG, Hawari J, Zhou E, et al. Biodegradation of variable-chain-length alkanes at low temperatures by a psychrotrophic Rhodococcus sp.[J]. Applied and Environmental Microbiology, 1998, 64(7): 2578-2584. |
| [23] |
Resmi G, Thampi SG, Chandrakaran S. Brevundimonas vesicularis: A novel bio-sorbent for removal of lead from wastewater[J]. International Journal of Environmental Research, 2010, 4(2): 281-288. |
| [24] |
Dumas DP, Caldwell SR, Wild JR, et al. Purification and properties of the phosphotriesterase from Pseudomonas diminuta[J]. Journal of Biological Chemistry, 1989, 264(33): 19659-19665. |
| [25] |
Liu YD, Su XM, Lu L, et al. A novel approach to enhance biological nutrient removal using a culture supernatant from Micrococcus luteus containing resuscitation-promoting factor (Rpf) in SBR process[J]. Environmental Science and Pollution Research, 2016, 23(5): 4498-4508. DOI:10.1007/s11356-015-5603-3 |
 2018, Vol. 45
2018, Vol. 45