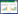扩展功能
文章信息
- 史静龙, 禹飞, 梁俊峰, 周光益, 赵厚本
- SHI Jing-Long, YU Fei, LIANG Jun-Feng, ZHOU Guang-Yi, ZHAO Hou-Ben
- 流溪河生态公益林下土壤细菌多样性
- Soil bacterial diversity in Liuxihe National Park
- 微生物学通报, 2018, 45(11): 2353-2363
- Microbiology China, 2018, 45(11): 2353-2363
- DOI: 10.13344/j.microbiol.china.171014
-
文章历史
- 收稿日期: 2017-11-30
- 接受日期: 2018-03-12
- 网络首发日期(www.cnki.net): 2018-03-19
土壤微生物在生态系统物质养分循环中发挥着积极的作用,在土壤形成、肥力演变、植物养分有效化、土壤结构形成与改良、有毒物质降解及净化等方面起着重要作用[1-2]。细菌在土壤微生物中占绝对优势[3-6]。土壤细菌是陆地生态系统中几乎所有生物地球化学循环的重要驱动者,参与了土壤中大多数养分的转化,推动着生态系统能量和物质循环,其种类组成和群落结构在很大程度上决定着土壤的生物活性[7-8]。相对于农田和草地生态系统,森林生态系统中的土壤细菌群落仍然没有被充分研究[9]。森林生态系统地下具有丰富的微生物资源,在门的分类水平上,森林土壤细菌多样性虽不如草地[8],但却高于农田[10]。有研究报道,不同的自然森林类型其土壤细菌的优势类群不同[9],土壤细菌的微生物多样性越高,则越有利于土壤的可持续利用和抗压能力[11]。通过土壤微生物结构变化特征研究,预测生态系统的结构与功能越来越受到研究者的关注[12-13]。
林分类型会影响土壤养分的积累、分布与循环,而土壤养分含量又是植被生长的重要影响因子[14]。土壤养分含量直接影响林木的生长,土壤微生物则通过分解动植物残体参与森林生态系统的物质循环和能量流动,影响树木的生长发育,是土壤肥力的重要指标之一[15]。土壤有机质的分解速率受土壤微生物种类、数量和活性的影响。有机质经过微生物的分解还可被植物再次利用,提供植物生长所需的养分,在C、N循环过程中具有重要意义[16]。土壤微生物对植物有效养分具有储备作用,对土壤C、N等养分的有效性及其在地上、地下的循环特征方面起着调控作用[17]。
流溪河位于广州市从化区东北部,地处亚热带,气候温和,雨量充沛,资源丰富,物种众多。流溪河区域现有生态公益林近7万hm2,与大多数生态公益林一样,该林区也存在森林结构不合理、林分质量较差、森林生态功能等级不高等问题[18]。
本研究选取广州流溪河公益林中不同密度林分下的土壤为研究对象,通过基于16S rRNA基因的高通量测序对表层土壤细菌群落结构进行分析,了解不同林分密度下土壤细菌群落结构的差异,揭示该区域森林生态系统中细菌多样性特征和流溪河生态公益林不同林分下土壤地力的变化,以期阐明能使林下细菌多样性得以良好维持的健康林分密度,为该区下一步进行森林抚育提供理论依据。
1 研究区概况研究区域位于广州市东北从化区北部与良口镇相连处的流溪河样地,地理位置113°17′E,23°8′N,地处亚热带,气候温和,雨量充沛,资源丰富,物种众多。年平均气温20.3 ℃,平均最高气温31.9 ℃,平均最低气温11.8 ℃,年降雨量平均2 000 mm,植被为天然次生林,坡向朝西南,土壤为红壤。
2 研究方法 2.1 样地设置和群落调查于2013年12月18-29日在流溪河林场生态公益林下划分试验样地,根据坡向、坡度、林分稀密度、林下灌木杂草情况划分为不同的样地。每样地25 m×40 m,对样地内所有胸高直径 > 3 cm的乔木进行每木检尺,记载其胸径、树高和生长状况;根据样方内胸高直径 > 3 cm的树木数量,将林分分为高密度(HD) (> 2 200株/hm2)、中密度(MD) (1 800–2 200株/hm2)和低密度(LD) (< 1 800株/hm2)。选取地理位置毗邻,环境条件(坡向、坡度等)相似但密度不同的3种林分类型,对其林下土壤微生物多样性开展研究。所选的3种不同密度林分类型分别为高密度(HD) (2 546株/hm2)、中密度(MD) (1 998株/hm2)、低密度(LD) (1 702株/hm2)。
2.2 样品采集记录土壤类型及发育状况,按照森林土壤发生层次,使用土钻在每个样地内从“S”形随机设置的5个取样点中取0-10 cm的表层土壤样品,并将土样混合均匀,每块样地重复取样3次作为重复。所取样品分为两部分,一部分土样过1 mm土壤筛,风干后用于测定土壤理化性质,这部分土壤样品保存在4 ℃;另一部分捡去石块和杂物,放入保鲜盒内,迅速带回到实验室,用自封袋分装,做好标记,用于DNA的提取,于-80 ℃保存。
2.3 主要试剂和仪器DNA抽提试剂盒、琼脂糖凝胶DNA回收试剂盒、引物,生工生物工程(上海)股份有限公司。NanoDrop 2000微量紫外分光光度计,Thermo公司;PCR仪,ABI公司;电泳仪,北京市六一仪器厂;电泳凝胶成像系统,上海嘉鹏科技有限公司。
2.4 土壤DNA提取称取0.30 g土壤样品,按照DNA抽提试剂盒中的操作步骤提取土壤微生物总DNA。将提取出来的DNA样品溶解于去离子水中,稀释10倍,使用NanoDrop 2000微量紫外分光光度计对提取的DNA浓度进行测定,使其核酸纯度A260/A280介于1.8-2.0,DNA浓度 > 10 ng/μL,提取的DNA在-20 ℃保存,以备进一步分析。对每份样品采用引物515F/806R扩增16S rRNA基因的V4高变区片段。引物序列[19]为barcode-GT-515F:5′-GCCAGCMGCCGCGGTA A-3′;barcode-806R:5′-CCGGACTACHVGGGTWTCT AAT-3′。20 μL PCR反应体系:dNTPs (2.5 mmol/L) 2 μL,10×Pyrobest buffer 2.5 μL,DNA模板(10 ng/μL) 2 μL,引物515F和806R (10 μmol/L)各1 μL,Taq DNA聚合酶(2.5 U/μL) 0.5 μL,ddH2O 16 μL。PCR反应条件:95 ℃ 3 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 30 s,共35个循环;72 ℃ 7 min;4 ℃ 10 min。PCR扩增产物经1%琼脂糖电泳检测。
土壤DNA委托北京华诺科技有限公司进行MiSeq高通量测序。
2.5 数据分析土壤pH测定按照水:土样为2.5:1使用玻璃电极测定;有机碳的测定是用1 mol/L盐酸去除碳酸盐后使用全碳分析仪分析;总氮和总硫用元素分析仪测定;碱解扩散法测定土壤碱解氮含量;土壤样品溶于H2SO4-H2O2,采用钼锑抗分光光度法测定总磷含量;样品溶于HNO3-HClO4-HF,采用火焰原子吸收分光光度计测定总钾含量。
依据PCR产物浓度将PCR扩增的所有产物等量混合,再经定量等质量控制后,构建Illumina测序文库,用Illumina MiSeq测序平台进行平行双末端测序,序列数据经BIPES (Barcoded illumina paired-end sequencing)流程进行初步处理,去除低质量数据;反向互补组装V4序列;根据Barcode将序列分拆到对应样品,去除Barcode,获得有效数据,然后开展生物信息学分析。
在97%相似度下利用Usearch (V 8.0.1623)软件对其有效数据进行操作分类单元(Operational taxonomic unit,OTU)聚类,Uparse构建OTU时会选取代表性序列,这些代表性序列使用Qiime (V 1.9.0)中的BLAST方法与NCBI (National Center for Biotechnology Information)等数据库的核苷酸序列进行比对和物种注释,在各个分类水平上统计所有样品的菌落组成,并对样品进行Alpha多样性指数计算。Chao1指数代表物种总数;ACE指数代表群落丰富度;Shannon指数代表菌落多样性;多样性指数与其所代表的值呈正比,多样性指数越大,说明所代表的多样性指标越丰富。林分内物种丰富度大小标志着林分多样性的复杂程度。物种丰富度指数值越大,则林分内物种越多,多样性越复杂。根据物种分布利用R语言中Pheatmap包绘制Heatmap图,利用Excel绘制物种组成的柱状图和饼状图。
2.6 统计学分析利用Excel作基本的数据前处理和分析;用SPSS 21.0软件作统计学分析,对数据进行正态分布和方差齐性检验,符合正态分布则采用平均值±标准误差,采用邓肯多重比较(P < 0.05),同列不同字母表示差异显著。
3 结果与分析 3.1 不同密度林分类型土壤养分含量N是植物的必需营养元素之一,也是土壤养分中最重要的元素之一,全N含量一般表示N素的供应容量,反映土壤的总体供N水平。
对采集的土壤样品做理化性质分析,由表 1可知不同密度林分类型下土壤有机碳含量表现为HD≈LD > MD,高密度样方HD和低密度样方LD有机碳含量明显高于中密度样方MD;全氮、全磷、全钾、速效磷、速效钾含量在高密度样方HD最高,而在中低密度差别不大;碱解氮含量在中密度最高,高密度次之,低密度最低。土壤样品采自相同红壤区域内3种不同林分密度类型,高密度样地林下灌木杂草较少,但中低密度样地林下灌木杂草丛生,气候条件一致;高密度林分下土壤各项理化性质均最高,说明高密度林分土壤相对比较肥沃。
| 不同密度林分 Different density stand types |
pH | 有机碳 Organic C (g/kg) |
全氮 Total N (g/kg) |
全磷 Total P (g/kg) |
全钾 Total K (g/kg) |
碱解氮 Alkalized N (mg/kg) |
速效磷 Available P (mg/kg) |
速效钾 Available K (mg/kg) |
| HD | 4.76±0.29a | 65.92±1.03a | 4.11±0.45a | 0.19±0.01a | 10.47±3.93a | 412.42±63.05a | 1.38±0.34a | 181.81±78.28a |
| MD | 4.60±0.16ab | 55.13±0.98b | 3.56±0.39a | 0.17±0.02ab | 4.87±0.17b | 460.60±26.45a | 0.80±0.41a | 107.37±6.02a |
| LD | 4.23±0.20b | 65.47±3.50a | 3.49±0.42a | 0.16±0.01b | 5.77±1.85ab | 358.52±59.32a | 1.00±0.16a | 93.85±22.72a |
| 注:数据后小写字母分别表示处理间存在P < 0.05水平显著性差异. Note: Lowercase letters are significantly at 0.05 probability level of the different treatments. | ||||||||
通过对不同样品中获得的序列进行随机抽取,以得到的序列数与基于该测序条数能代表的OTU数量构建稀释性曲线(图 1),从图 1中看出3个样方的稀释性曲线均趋于平坦,说明测序的数据量渐近合理,更多的测序只会产生较少的新OTU,测序深度已经基本覆盖到样品中所有的物种。
|
| 图 1 土壤细菌OTU稀释曲线 Figure 1 OTU rarefaction curves of soil bacteria |
|
|
通过DNA序列的相似性将大于97%的序列归为同一种OTU。多样性指数可以反映土壤细菌多样性的不同方面,对不同密度林分土壤细菌16S rRNA基因多样性指数进行分析(表 2),显示中密度林分(MD) Chao1指数和ACE指数均高于高密度和低密度林分,表明其物种总数和丰富度均高于高密度和低密度林分;而Shannon指数则是中密度(MD)和低密度林分(LD)差异较小,但略高于高密度林分(HD),表明中、低密度林分物种多样性高于高密度林分。
| Samples | Reads | OTUs | Chao1 | Shannon | ACE |
| HD | 30 151±2 847 | 963±76 | 1 067.31±68.24 | 7.66±0.26 | 1 067.23±69.04 |
| MD | 25 720±3 273 | 1 008±68 | 1 148.14±52.49 | 7.98±0.14 | 1 137.67±56.32 |
| LD | 30 776±5 877 | 943±33 | 1 043.50±33.21 | 7.89±0.11 | 1 047.48±29.99 |
通过对原始数据进行拼接、过滤得到有效数据,基于有效数据进行OTU聚类和物种分类分析,3个HD样品中共得到80 610个Tags,LD中得到80 281个Tags,MD中得到66 652个Tags。
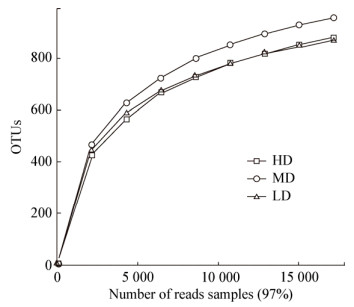
3.4.1 土壤细菌在门水平上的群落组成图 2显示了3组样品细菌群落在门水平上的组成,在HD中主要的门是酸杆菌门(Acidobacteria,占总数约35.38%)、变形菌门(Proteobacteria,30.22%)和WPS-2 (10.22%);在MD中主要门类群是变形菌门(38.48%)、酸杆菌门(28.99%)和厚壁菌门(9.68%);在LD中主要的门类群是酸杆菌门(30.19%)、变形菌门(34.77%)和放线菌门(7.27%)。可以看出,在3组样品中酸杆菌门和变形菌门是流溪河林区土壤细菌的主要类群,在高密度和低密度样方酸杆菌门含量高于变形菌门,而中密度样方变形菌门高于酸杆菌门;除上述2种主要类群外,WPS-2、放线菌门和疣微菌门(Verrucomicrobia)等类群是次要菌群,在不同密度林分下其含量略有差别;值得一提的是,厚壁菌门在高密度林分仅占0.07%,但在中、低密度林分分别占到9.68%和2.82%。上述结果表明,林分密度对土壤细菌群落结构的影响较大,特别是中密度林分,变形菌门和厚壁菌门比例明显上升而酸杆菌门比例则显著降低。
|
| 图 2 在门水平上对细菌群落进行分类 Figure 2 Taxonomic classification of bacterial community reads at phylum level |
|
|
从纲的水平对所有样品分类,总共分类到82个纲,用Excel对3种不同密度林分类型在纲水平(> 1%,16个)作物种相对丰度分布柱形图分析(图 3)。所有的样品中,相对丰度最高的纲分别为DAO52 (其含量在高、中、低3种林分密度中分别为17.75%、14.03%和13.82%)、α-变形菌纲(Alphaproteobacteria,含量依次为17.17%、17.26%和18.76%)和酸杆菌纲(Acidobacteriia,含量依次为12.29%、10.99%和11.94%),其相对含量均超过10%。除此之外,在3组样品中,浮霉状菌纲(Planctomycetia)、酸微菌纲(Acidimicrobiia)和Pedosphaerae的丰度较低,介于1.19%-2.51%之间。在3种林分密度下,γ-变形菌纲(Gammaprote- obacteria)和芽孢杆菌纲(Bacilli)变化较为明显,在高林分密度下,上述二菌含量相对较低,特别是芽孢杆菌纲仅为0.05%,但在中密度林分下,上述菌群比例明显上升,分别达到14.27%和8.20%,低密度林分介于二者之间。从以上结果可以看出,在纲水平上,不同密度林分其主要的纲较为相似,但其含量却存在明显差异,表明林分密度会影响土壤细菌的群落结构。
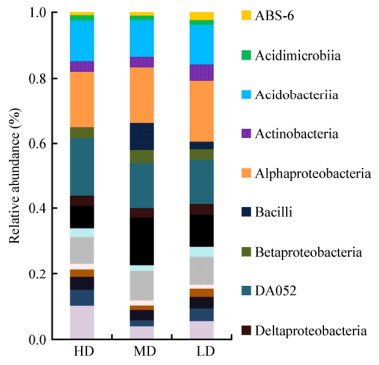
|
| 图 3 土壤细菌在纲水平上的群落结构组成分布 Figure 3 Soil bacterial community groups at class level |
|
|
在科水平上对样品和样品前50个OTU所含序列的丰度绘制Heatmap图进行聚类分析(图 4),该图能够反映出在科水平上细菌群落结构的差异性和相似性。

|
| 图 4 基于科水平上的土壤细菌热图 Figure 4 Heatmap based on soil bacterial at family level |
|
|
根据图 4菌落丰度颜色变化可见,土壤细菌菌群在科水平上的丰度受到林分密度的影响,不同密度林分细菌含量差异明显。UN--s-Ellin6513、Koribacteraceae、UN--s-WPS-2、红螺菌科(Rhodospirillaceae)、酸杆菌科(Acidobacteriaceae)、Sinobacteriaceae、Chthoniobacteraceae和生丝微菌科(Hyphomicrobiaceae)为流溪河区域土壤细菌的主要类群,但其丰富度在不同林分密度略有差异;而一些类群则受到林分密度的影响较大,比如肠杆菌科(Enterobacteriaceae)、乳杆菌科(Lactobacillaceae)、醋酸杆菌科(Acetobactereceae)和Acidithiobacteraceae在中密度林分明显高于高、低密度林分。
从总体看,不同林分类型在细菌多样性和复杂度方面的差异较明显,高密度林分土壤细菌更多集中于少数几个主要的类群,低密度林分次之,而中密度林分细菌相对更为分散,多个类群的丰度高于高、低密度林分,这说明中密度林分土壤细菌群落结构比较稳定,多样性和丰富度更高。
4 讨论 4.1 流溪河生态公益林下土壤细菌的多样性土壤细菌具有非常丰富的多样性,不同类型的土壤中存在各自的优势细菌类群,同时又有相似的细菌类群[8]。Janssen[20]研究发现森林、草地、牧场、高山草甸等不同土壤类型中,土壤细菌以变形菌门和酸杆菌门为主,两大类主要土壤微生物相对丰度依土壤类型不同而在一定范围内波动;杨菁等[21]对热带海南混交林土壤菌群结构研究发现,混交林土壤主要微生物门类包括变形菌、酸杆菌和放线菌等;Huang等[22]研究发现福建森林红壤细菌主要微生物门类包括酸杆菌门(71.5%)和变形菌门(24.1%)等;而Fierer等[6]发现在温带森林中酸杆菌是土壤中含量最丰富的细菌类群,Yuan等[23]在我国青藏草地的研究中也发现酸杆菌是土壤中含量最丰富的细菌类群,这说明虽然不同森林类型有地域差异,但其优势细菌类群基本相似,本研究中酸杆菌门含量低于变形菌门,居第二位,这也验证了Yang等[24]的观点:土壤细菌具有非常丰富的多样性,不同类型的土壤中存在各自的优势细菌类群,同时又有相似的细菌类群[8]。本研究中,由图 2可以看出酸杆菌门和变形菌门为流溪河区域土壤细菌的主要类群,但不同密度下其含量存在差异,高密度和低密度林分下酸杆菌门高于变形菌门,而中密度下正好相反;此外,WPS-2、放线菌门和疣微菌门等类群是次要菌群。
4.2 林分密度影响土壤细菌的多样性、丰富度及其群落结构林分密度通过改变光照、土壤微环境等影响土壤有机碳的输入和输出(如凋落物量及其分解速率,林木对有机质的吸收消耗等),从而影响土壤有机碳含量和碳密度,进而影响土壤微生物的生长代谢。土壤化学因子、土壤温度、植物多样性也是形成森林土壤微生物群落结构的3个主要因素[11]。Nacke等[8]也认为土壤细菌群落的结构在很大程度上受树种和土壤pH的影响;在不同的空间尺度上,pH对土壤细菌的多样性和群落组成均有很大的影响[25]。此外,几种营养成分(比如TN、TP、TK)也与细菌的群落结构存在显著的相关性。Ushio等[26]认为林分类型可以通过改变土壤性质影响细菌群落组成。
本研究的结果表明,不同林分密度下土壤细菌的多样性和丰富度存在明显差异,中密度林分比高、低密度具有更高的多样性和丰富度;而不同林分密度下细菌群落结构差异明显,也表明林分密度对土壤细菌的群落结构产生明显的影响。
4.3 流溪河区域中密度林分更适于土壤细菌的繁衍生息森林林分类型、气候类型、土壤状况等不同,适宜的林分密度会略有差异。康冰等[27]在对马尾松人工林的研究中发现,中密度(1 800株/hm2)林地养分质量分数总体较高,且土壤理化特性也优于过密或过疏的林分;陈莉莉等[28]研究发现中密度松栎混交林(中龄林)土壤有机质质量较高;吕婧娴等[29]认为密度1 717-1 867株/hm2为油松人工林较为合适的密度;王树力等[30]的研究表明在其划分的4个不同密度落叶松人工林中,中、低密度林分微生物的生长和发展状况更好;也有研究表明,凋落物分解周期和分解系数都随林分密度的变化而变化,适中的林分密度分解效果最好[31]。本研究中,中密度林分较高密度和低密度林分土壤细菌多样性和丰富度指标高(表 2),表明在3种林分密度下,中密度林分的细菌多样性最高,群落最稳定,该林分密度较高密度和低密度更适于土壤细菌的繁衍生息。
4.4 土壤的肥力状况受林分密度、灌木杂草的影响张国庆等[32]和潘辉等[33]认为土壤碳储量均随林分密度的增加而减少;McMurtrie等[34]认为土壤供氮能力较高的林分其固碳潜力较大;Scott等[35]推测高密度林分可能因降低了土壤氮有效性,进而导致土壤碳储量的下降,但目前这方面机制的研究还较少[36]。与上述研究略有差异的是,本研究中高密度林分(HD)土壤的有机碳、全氮、全磷等都最高(表 1),表明高密度林分土壤肥沃,而中、低密度林分土壤肥沃程度相对较低,原因可能在于高密度林分下杂草、灌木较少,而中、低密度林分下杂草、灌木较多。一方面浓密的灌木杂草会阻碍凋落物落到地面,延缓了凋落物的分解;另一方面灌木杂草的生长也会吸收土壤的养分,导致土壤肥沃程度降低。
4.5 16S rRNA基因的基因组异质性会导致对细菌多样性的过高估计几十年来,在原核生物中编码rRNA小亚基的16S rRNA基因已广泛应用于分类学和系统关系的确定[37-38]。由于16S rRNA基因有维护良好的数据库[39]以及易于获取,无疑是微生物生态学研究中使用最广泛的分子标记。然而,16S rRNA基因的基因组异质性已经在一些研究中被发现[40-42],并且在使用基于16S rRNA基因的方法时被认为会导致微生物多样性的高估[43-44],16S rRNA的V4和V5区因其能充分显示基因组间变异及最少的基因组异质性,被提出用做细菌16S rRNA分析的理想区域[45]。在本研究中,对16S rRNA基因的V4高变区进行序列分析,以期尽可能减少由于16S rRNA基因的基因组异质性造成的对细菌多样性的高估。尽管这样,研究结果中得到的细菌多样性仍被过高估计,目前对基因组异质性的影响研究很少[45]。
总之,林下土壤细菌的群落结构与物种多样性能反映森林土壤生态系统的结构状况,合理的林分密度可促进森林植被的良好发育,大大提高森林生态系统的稳定性,并可改善生态系统的服务功能和健康状况。林分密度调控作为森林管理的重要措施,必然对土壤质量产生一定影响。合理的经营密度能够优化林分结构,促进对森林生态系统环境要素的充分利用和林下植被、枯落物及养分循环过程的改善[46]。针对流溪河区域现有生态公益林,合理进行森林抚育管理,对过密或过稀的林分进行间伐或补栽,定期清理林下灌木杂草,能有效提高森林的生态功能。
| [1] |
Li FD. Soil Microbiology[M]. Beijing: China Agricultural Press, 1996. (in Chinese) 李阜棣. 土壤微生物学[M]. 北京: 中国农业出版社, 1996. |
| [2] |
Zhang P, Guo HJ, Dao ZL, et al. A study on quantity and diversity of soil microorganisms in Gaoligong mountains[J]. Chinese Biodiversity, 1999, 7(4): 297-302. (in Chinese) 张萍, 郭辉军, 刀志灵, 等. 高黎贡山土壤微生物的数量和多样性[J]. 生物多样性, 1999, 7(4): 297-302. DOI:10.3321/j.issn:1005-0094.1999.04.007 |
| [3] |
Gu FX, Wen QK, Pan BR, et al. A preliminary study on soil microorganisms of artificial vegetation in the center of Taklimakan Desert[J]. Chinese Biodiversity, 2000, 8(3): 287-303. (in Chinese) 顾峰雪, 文启凯, 潘伯荣, 等. 塔克拉玛干沙漠腹地人工植被下土壤微生物的初步研究[J]. 生物多样性, 2000, 8(3): 287-303. DOI:10.3321/j.issn:1005-0094.2000.03.009 |
| [4] |
Zhao XL, Zhou GS, Zhou L, et al. A preliminary study on soil microorganism in Panjin reed wetland[J]. Journal of Meteorology and Environment, 2007, 23(1): 30-33. (in Chinese) 赵先丽, 周广胜, 周莉, 等. 盘锦芦苇湿地土壤微生物初步研究[J]. 气象与环境学报, 2007, 23(1): 30-33. DOI:10.3969/j.issn.1673-503X.2007.01.007 |
| [5] |
Delmont TO, Prestat E, Keegan KP, et al. Structure, fluctuation and magnitude of a natural grassland soil metagenome[J]. The ISME Journal, 2012, 6(9): 1677-1687. DOI:10.1038/ismej.2011.197 |
| [6] |
Fierer N, Leff JW, Adams BJ, et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(52): 21390-21395. DOI:10.1073/pnas.1215210110 |
| [7] |
Zhang YY, Zhang GY, Xia J, et al. The research of wastewater treatment mechanism in wetlands[J]. Environmental Science and Technology, 2005, 28(S1): 165-167. (in Chinese) 张永勇, 张光义, 夏军, 等. 湿地污水处理机理的研究[J]. 环境科学与技术, 2005, 28(S1): 165-167. |
| [8] |
Nacke H, Thürmer A, Wollherr A, et al. Pyrosequencing-based assessment of bacterial community structure along different management types in German forest and grassland soils[J]. PLoS One, 2011, 6(2): e17000. DOI:10.1371/journal.pone.0017000 |
| [9] |
Li H, Ye DD, Wang XG, et al. Soil bacterial communities of different natural forest types in Northeast China[J]. Plant and Soil, 2014, 383(1/2): 203-216. |
| [10] |
Roesch LFW, Fulthorpe RR, Riva A, et al. Pyrosequencing enumerates and contrasts soil microbial diversity[J]. The ISME Journal, 2007, 1(4): 283-290. DOI:10.1038/ismej.2007.53 |
| [11] |
Zhou JZ, Xia BC, Treves DS, et al. Spatial and resource factors influencing high microbial diversity in soil[J]. Applied and Environmental Microbiology, 2002, 68(1): 326-334. |
| [12] |
Harris JA. Measurements of the soil microbial community for estimating the success of restoration[J]. European Journal of Soil Science, 2003, 54(4): 801-808. |
| [13] |
Lagomarsino A, Moscatelli MC, Di Tizio A, et al. Soil biochemical indicators as a tool to assess the short-term impact of agricultural management on changes in organic C in a Mediterranean environment[J]. Ecological Indicators, 2009, 9(3): 518-527. DOI:10.1016/j.ecolind.2008.07.003 |
| [14] |
Jiang HM, Li MZ, Wang Q, et al. Dynamics of soil nutrients under different vegetation types in the eastern Qilian mountains[J]. Research of Soil and Water Conservation, 2011, 18(5): 166-170. (in Chinese) 姜红梅, 李明治, 王亲, 等. 祁连山东段不同植被下土壤养分状况研究[J]. 水土保持研究, 2011, 18(5): 166-170. |
| [15] |
Jiao RZ, Yang CD, Tu XN, et al. The change of undergrowth, soil microorganism, enzyme activity and nutrient in different developing stage of the Chinese fir plantation[J]. Forest Research, 1997, 10(4): 373-379. (in Chinese) 焦如珍, 杨承栋, 屠星南, 等. 杉木人工林不同发育阶段林下植被、土壤微生物、酶活性及养分的变化[J]. 林业科学研究, 1997, 10(4): 373-379. DOI:10.3321/j.issn:1001-1498.1997.04.006 |
| [16] |
Porazinska DL, Bardgett RD, Blaauw MB, et al. Relationships at the aboveground-belowground interface: plants, soil biota, and soil processes[J]. Ecological Monographs, 2003, 73(3): 377-395. DOI:10.1890/0012-9615(2003)073[0377:RATAIP]2.0.CO;2 |
| [17] |
Spehn EM, Joshi J, Schmid B, et al. Plant diversity effects on soil heterotrophic activity in experimental grassland ecosystems[J]. Plant and Soil, 2000, 224(2): 217-230. |
| [18] |
Xu XY, Zeng F. Present situation, problem and countermeasure of classified forest management in Guangdong province[J]. Forest Resources Management, 2008(5): 37-40. (in Chinese) 许秀玉, 曾锋. 广东省森林分类经营现状问题与对策[J]. 林业资源管理, 2008(5): 37-40. DOI:10.3969/j.issn.1002-6622.2008.05.008 |
| [19] |
Caporaso JG, Lauber CL, Walters WA, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(S1): 4516-4522. |
| [20] |
Janssen PH. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes[J]. Applied and Environmental Microbiology, 2006, 72(3): 1719-1728. DOI:10.1128/AEM.72.3.1719-1728.2006 |
| [21] |
Yang J, Zhou GY, Tian YY, et al. Differential analysis of soil bacteria diversity in different mixed forests of Dalbergia odorifera[J]. Acta Ecologica Sinica, 2015, 35(24): 8117-8127. (in Chinese) 杨菁, 周国英, 田媛媛, 等. 降香黄檀不同混交林土壤细菌多样性差异分析[J]. 生态学报, 2015, 35(24): 8117-8127. |
| [22] |
Huang QG, Tian BY, Jiang XZ, et al. Community construction and function of soil bacteria in a forest decomposed-hollow stump environment[J]. Biotechnology Bulletin, 2009(3): 132-136. (in Chinese) 黄钦耿, 田宝玉, 江贤章, 等. 福州森林红壤细菌的菌群结构及其功能分析[J]. 生物技术通报, 2009(3): 132-136. |
| [23] |
Yuan YL, Si GC, Wang J, et al. Bacterial community in alpine grasslands along an altitudinal gradient on the Tibetan Plateau[J]. FEMS Microbiology Ecology, 2014, 87(1): 121-132. |
| [24] |
Yang GP, Nan L, Jia HB, et al. Bacterial genetic diversity in soils and their correlation with vegetation[J]. Acta Genetica Sinica, 2000, 27(3): 278-282. (in Chinese) 杨官品, 男兰, 贾海波, 等. 土壤细菌遗传多样性及其与植被类型相关性研究[J]. 遗传学报, 2000, 27(3): 278-282. |
| [25] |
Rousk J, Bååth E, Brookes PC, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. The ISME Journal, 2010, 4(10): 1340-1351. |
| [26] |
Ushio M, Wagai R, Balser TC, et al. Variations in the soil microbial community composition of a tropical montane forest ecosystem: does tree species matter?[J]. Soil Biology and Biochemistry, 2008, 40(10): 2699-2702. DOI:10.1016/j.soilbio.2008.06.023 |
| [27] |
Kang B, Liu SR, Cai DX, et al. Effects of Pinus massoniana plantation stand density on understory vegetation and soil properties[J]. Chinese Journal of Applied Ecology, 2009, 20(10): 2323-2331. (in Chinese) 康冰, 刘世荣, 蔡道雄, 等. 马尾松人工林林分密度对林下植被及土壤性质的影响[J]. 应用生态学报, 2009, 20(10): 2323-2331. |
| [28] |
Chen LL, Wang DX, Yu F, et al. Effects of stand density on physical and chemical properties of soil moisture[J]. Journal of Northeast Forestry University, 2013, 41(8): 61-64. (in Chinese) 陈莉莉, 王得祥, 于飞, 等. 林分密度对土壤水分理化性质的影响[J]. 东北林业大学学报, 2013, 41(8): 61-64. DOI:10.3969/j.issn.1000-5382.2013.08.014 |
| [29] |
Lü JX, Wang DX, Zhang SZ, et al. Study on the species diversity of undergrowth in the different stand densities of Pinus tabulaeformis plantation of Xiaolongshan area[J]. Journal of Northwest A & F University (Natural Science Edition), 2010, 38(11): 49-56. (in Chinese) 吕婧娴, 王得祥, 张宋智, 等. 小陇山林区不同密度油松人工林林下物种多样性研究[J]. 西北农林科技大学学报:自然科学版, 2010, 38(11): 49-56. |
| [30] |
Wang SL, Sun Y, Shen HY, et al. Changes of soil microbiological properties of Larix olgensis + Larix kaempferi plantation with the different densities[J]. Science of Soil and Water Conservation, 2009, 7(3): 59-66. (in Chinese) 王树力, 孙悦, 沈海燕, 等. 不同密度杂种落叶松人工林的土壤微生物变化特征[J]. 中国水土保持科学, 2009, 7(3): 59-66. DOI:10.3969/j.issn.1672-3007.2009.03.011 |
| [31] |
Jin HF. Effects of stand density on litter decomposition of Larix principis-rupprechtii plantation[D]. Beijing: Master's Thesis of Beijing Forestry University, 2010 (in Chinese) 金虎范.林分密度对华北落叶松人工林凋落物分解影响的研究[D].北京: 北京林业大学硕士学位论文, 2010 http://cdmd.cnki.com.cn/Article/CDMD-10022-2010128867.htm |
| [32] |
Zhang GQ, Huang CD, Guo H, et al. Spatial distribution property of carbon stocks in artificial pine ecosystems with different density[J]. Journal of Zhejiang Forestry Science and Technology, 2007, 27(6): 10-14. (in Chinese) 张国庆, 黄从德, 郭恒, 等. 不同密度马尾松人工林生态系统碳储量空间分布格局[J]. 浙江林业科技, 2007, 27(6): 10-14. DOI:10.3969/j.issn.1001-3776.2007.06.003 |
| [33] |
Pan H, Zhao K, Wang YQ, et al. Study on carbon reserves of Fujian cypress plantations with different densities[A]//A Collection of Excellent Papers on Global Climate Change and Carbon Sequestration Forestry Academic Symposium at the Fifth Annual Meeting of the Twelfth China Association for Science and Technology Association[C]. Fuzhou: The Twelfth China Association for Science and Technology, 2010: 36-39 (in Chinese) 潘辉, 赵凯, 王玉芹, 等.不同密度福建柏人工林碳储量研究[A]//2010中国科协年会第五分会场全球气候变化与碳汇林业学术研讨会优秀论文集[C].福州: 中国科学技术协会, 2010: 36-39 |
| [34] |
McMurtrie RE, Medlyn BE, Dewar RC. Increased understanding of nutrient immobilization in soil organic matter is critical for predicting the carbon sink strength of forest ecosystems over the next 100 years[J]. Tree Physiology, 2001, 21(12/13): 831-839. |
| [35] |
Scott NA, Tate KR, Ross DJ, et al. Processes influencing soil carbon storage following afforestation of pasture with Pinus radiata at different stocking densities in New Zealand[J]. Australian Journal of Soil Research, 2006, 44(2): 85-96. DOI:10.1071/SR05013 |
| [36] |
Fontaine S, Bardoux G, Abbadie L, et al. Carbon input to soil may decrease soil carbon content[J]. Ecology Letters, 2004, 7(4): 314-320. DOI:10.1111/ele.2004.7.issue-4 |
| [37] |
Woese CR. Bacterial evolution[J]. Microbiological Reviews, 1987, 51(2): 221-271. |
| [38] |
Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576 |
| [39] |
Nübel U, Engelen B, Felske A, et al. Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel electrophoresis[J]. Journal of Bacteriology, 1996, 178(19): 5636-5643. DOI:10.1128/jb.178.19.5636-5643.1996 |
| [40] |
Yap WH, Zhang ZS, Wang Y. Distinct types of rRNA operons exist in the genome of the actinomycete Thermomonospora chromogena and evidence for horizontal transfer of an entire rRNA operon[J]. Journal of Bacteriology, 1999, 181(17): 5201-5209. |
| [41] |
Klappenbach JA, Dunbar JM, Schmidt TM. rRNA operon copy number reflects ecological strategies of bacteria[J]. Applied and Environmental Microbiology, 2000, 66(4): 1328-1333. DOI:10.1128/AEM.66.4.1328-1333.2000 |
| [42] |
López-López A, Benlloch S, Bonfá M, et al. Intragenomic 16S rDNA divergence in Haloarcula marismortui is an adaptation to different temperatures[J]. Journal of Molecular Evolution, 2007, 65(6): 687-696. DOI:10.1007/s00239-007-9047-3 |
| [43] |
Lim K, Furuta Y, Kobayashi I. Large variations in bacterial ribosomal RNA genes[J]. Molecular Biology and Evolution, 2012, 29(10): 2937-2948. DOI:10.1093/molbev/mss101 |
| [44] |
Stevenson BS, Schmidt TM. Life history implications of rRNA gene copy number in Escherichia coli[J]. Applied and Environmental Microbiology, 2004, 70(11): 6670-6677. |
| [45] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. |
| [46] |
Li GL, Liu Y, Xu Y, et al. Effects of thinning intensity on the development of undergrowth in Pinus tabulaeformis plantations[J]. Journal of Beijing Forestry University, 2007, 29(2): 70-75. (in Chinese) 李国雷, 刘勇, 徐扬, 等. 间伐强度对油松人工林植被发育的影响[J]. 北京林业大学学报, 2007, 29(2): 70-75. DOI:10.3321/j.issn:1000-1522.2007.02.012 |
 2018, Vol. 45
2018, Vol. 45