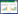扩展功能
文章信息
- 王睿, 喻晓蔚, 徐岩
- WANG Rui, YU Xiao-Wei, XU Yan
- 理性设计二硫键提高华根霉脂肪酶热稳定性
- Rationale design of disulfide bond in Rhizopus chinensis lipase to improve thermostability
- 微生物学通报, 2018, 45(11): 2311-2319
- Microbiology China, 2018, 45(11): 2311-2319
- DOI: 10.13344/j.microbiol.china.180123
-
文章历史
- 收稿日期: 2018-02-08
- 接受日期: 2018-05-04
- 网络首发日期(www.cnki.net): 2018-06-04
2. 江南大学药学院 江苏 无锡 214122;
3. 宿迁市江南大学产业技术研究院 江苏 宿迁 223814
2. School of Pharmaceutical Science, Jiangnan University, Wuxi, Jiangsu 214122, China;
3. Suqian Industrial Technology Research Institute of Jiangnan University, Suqian, Jiangsu 223814, China
脂肪酶(Triacylglycerol acylhydrolase,EC 3.1.1.3)又名三酰甘油酯水解酶,是一类能催化长链脂肪酸甘油酯水解为甘油和脂肪酸,同时也能催化酯合成和酯交换的特殊生物催化剂[1]。脂肪酶广泛应用于化学、食品、制药、洗涤剂等领域[1-2],通常需要高温反应条件,而中温脂肪酶的热稳定性欠佳,限制了它们在工业中的大规模应用。
本研究室在前期研究中从酿造浓香型大曲酒的酒曲中筛选到一株华根霉(Rhizopus chinensis CCTCC M201021)[3],从中克隆得到脂肪酶基因proRCL,并在毕赤酵母中实现了r27RCL的高效表达[4],实现了其在食品烘焙、纸张脱墨等工业生产中的应用。但是,由于华根霉脂肪酶是中温酶,热稳定性差[5],高温下的半衰期非常短[6-7]。因此,提高r27RCL的热稳定性很有意义,不仅可以拓宽该酶的工业应用范围,如饲料添加行业等,还能减少酶添加量,简化生产步骤,从而降低应用成本。
影响酶分子热稳定性的主要因素包括盐桥、氢键、疏水堆积、二硫键等,二硫键可以降低酶分子的熵值[8],提高酶分子的刚性[9],是稳定酶结构最关键的因素[10]。向酶分子中引入二硫键从而显著提高酶热稳定性的例子很多。Le等[11]用理性设计的方法,向脂肪酶CalB (Lipase B from Candida antarctica)中引入了5组二硫键,其中最佳一组将突变酶(A162C-K308C)于60 ℃下的Tm值提高了8.5 ℃,50 ℃下的半衰期延长了4.5倍;Tan等[12]用理性设计的方法,向圆弧青霉脂肪酶PcLipI (Penicillium cyclopium)中引入了一组二硫键突变(V248C-T251C),并分别于大肠杆菌(Escherichia coli BL21)和毕赤酵母(Pichia pastoris GS115)中表达,与原始酶相比,突变酶35 ℃下的半衰期分别延长了4.5倍和12.8倍。
华根霉脂肪酶r27RCL共296个氨基酸,其中7个半胱氨酸形成了3对二硫键,分别是C56-C295、C67-C70、C262-C271,3对二硫键的形成对r27RCL的正确折叠及稳定其三维构象有重要作用[13]。众所周知,蛋白质热变性最初都是从蛋白质表面的部分开始[14-15]。因此,本研究旨在向r27RCL分子表面引入半胱氨酸形成二硫键,利用Disulfide by design软件模拟,寻找位于分子表面能够形成二硫键的潜在突变位点,将其定点突变成半胱氨酸,通过增加表面二硫键的数量来提高酶的热稳定性。
1 材料与方法 1.1 主要试剂和仪器QuikChange® Lightning Site-directed Mutagenesis Kit,Stratagene公司;Sal I、Dpn I、PrimeSTAR GXL DNA Polymerase、Premix Taq、PCR产物纯化试剂盒、DL2000、DL10000 DNA marker,宝生物工程(大连)有限公司;酵母基因组DNA快速抽提试剂盒、引物、质粒提取试剂盒、YNB、DTT,生工生物工程(上海)股份有限公司。PCR仪、电转仪,Eppendorf公司。
1.2 菌株和质粒大肠杆菌JM109、巴斯德毕赤酵母Pichia pastoris GS115和华根霉Rhizopus chinensis CCTCC M201021由本实验室保存。重组菌E. coli pPIC9K-proRCL CHis和P. pastoris GS115 pPIC9K-proRCL CHis Mut+由本实验室构建。
1.3 培养基毕赤酵母培养基YPD、MD、BMGY和BMMY的配制参见Invitrogen公司的毕赤酵母表达手册。
1.4 突变位点的选择以脂肪酶r27RCL表面可能形成的潜在二硫键为研究对象,用Disulfide by design软件对r27RCL晶体结构(Protein data bank登录号为4L3W)进行预测,筛选键能(χ3 Angle)处于+97 (±20)附近的拟二硫键。以Pymol软件对脂肪酶晶体结构进行观察,弃去分子内部拟二硫键,弃去以脂肪酶催化三联体(S172-H284-D231)中心氨基酸S172为中心的9 Å范围内的突变位点,弃去催化中心上覆盖的盖子结构中的突变位点,并删除结构明显不合理的突变体。若有同一个氨基酸与不同氨基酸可形成二硫键,则按照形成二硫键的B-factor值,取值最大的一对氨基酸为研究对象。
1.5 定点突变采用快速定点突变试剂盒,以重组质粒pPIC9K-proRCL Chis Mut+为模板,设计含有突变位点的引物(表 1)进行全质粒PCR。25 μL反应体系:10×QuikChange reaction buffer 2.5 μL,模板25 ng,引物(10 μmol/L)各1 μL,QuikSolution reagent 0.75 μL,dNTP mix 0.5 μL,Quikchange lightning enzyme 0.5 μL。PCR反应条件:95 ℃ 2 min;95 ℃ 20 s,55-63 ℃ (退火温度根据引物Tm值设定) 1 min,68 ℃ 11 min,共18个循环;68 ℃ 5 min。PCR产物经Dpn I消化,转化大肠杆菌JM109感受态细胞,涂布在含氨苄青霉素(100 mg/L)的LB平板上。挑取阳性克隆子培养并提取质粒,送生工生物工程(上海)股份有限公司测序。获得的突变质粒命名为pPIC9K-proRCLm CHis。
| PCR产物名称 PCR product name |
引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
退火温度 Annealing temperature (℃) |
| m1 | G49C-Fm | GCTCACTAACTACGCTTGTGTTGCTGCTACTGCTTAC | 57.5 |
| G49C-Rm | GTAAGCAGTAGCAGCAACACAAGCGTAGTTAGTGAGC | ||
| m2 | Y73C-Fm | GCAATGTCTCAAGTATGTTCCTGATGGTAAGC | 52.1 |
| Y73C-Rm | GCTTACCATCAGGAACATACTTGAGACATTGC | ||
| m3 | I80C-Fm | GTTCCTGATGGTAAGCTTTGTAAGACCTTCACTTCTCTTC | 57.3 |
| I80C-Rm | GAAGAGAAGTGAAGGTCTTACAAAGCTTACCATCAGGAAC | ||
| m4 | I94C-Fm | GATACCAATGGCTTTTGCTTGAGAAGTGATGCTC | 55.3 |
| I94C-Rm | GAGCATCACTTCTCAAGCAAAAGCCATTGGTATC | ||
| m5 | F83C-Fm | GCTTATCAAGACCTGCACTTCTCTTCTCACTG | 55.5 |
| F83C-Rm | CAGTGAGAAGAGAAGTGCAGGTCTTGATAAGC | ||
| m6 | D149C-Fm | CCAAGTTGTCAAATGCTACTTCCCCGTCG | 55.4 |
| D149C-Rm | CGACGGGGAAGTAGCATTTGACAACTTGG | ||
| m7 | V130C-Fm | GATTATTCTCCTTGCAAGGGCGCCAAAGTTCAC | 56.3 |
| V130C-Rm | GTGAACTTTGGCGCCCTTGCAAGGAGAATAATC | ||
| m8 | A133C-Fm | CTCCTTGCAAGGGCTGTAAAGTTCACGCTGGTTTCC | 58.9 |
| A133C-Rm | GGAAACCAGCGTGAACTTTACAGCCCTTGCAAGGAG | ||
| m9 | S85C-Fm | GACCTTCACTTGCCTTCTCACTGATACCAATG | 54.8 |
| S85C-Rm | CATTGGTATCAGTGAGAAGGCAAGTGAAGGTC | ||
| m10 | Q145C-Fm | CCTTTCCTCATACAACTGCGTTGTCAAAGACTACTTCCCCG | 57.8 |
| Q145C-Rm | CGGGGAAGTAGTCTTTGACAACGCAGTTGTATGAGGAAAGG | ||
| m11 | P194C-Fm | CCAACGTGAAAAGAGATTATCTTGCAAGAACTTGAGCATC | 55.3 |
| P194C-Rm | GATGCTCAAGTTCTTGCAAGATAATCTCTTTTCACGTTGG | ||
| m12 | G220C-Fm | CGTCGACAGCACCTGTATTCCCTTCCACCGTAC | 58.9 |
| G220C-Rm | GTACGGTGGAAGGGAATACAGGTGCTGTCGACG | ||
| m13 | A213C-Fm | CGGTAACAATGCATTCTGCTACTACGTCGACAGCACC | 58.7 |
| A213C-Rm | GGTGCTGTCGACGTAGTAGCAGAATGCATTGTTACCG | ||
| m14 | L244C-Fm | GCCTTCGGTTATTGTCACCCCGGTGTC | 56.8 |
| L244C-Rm | GACACCGGGGTGACAATAACCGAAGGC | ||
| 注:下划线标出部分为半胱氨酸突变位点. Note: Underlined ‘mutant’ bases for Cysteine. | |||
将突变成功的质粒pPIC9K-proRCLm CHis用Sal I线性化,电转化毕赤酵母GS115感受态细胞。取8 μL浓度为1 g/L的线性化化质粒与80 μL感受态细胞混合均匀,转至0.2 cm冰预冷的电激杯中。冰上放置5 min, 电压1 500 V进行电击。立刻加入1 mL 1 mol/L山梨醇复苏,30 ℃培养1 h。将转化液涂布于MD平板上,30 ℃培养2 d。
将MD平板上生长出的His+转化子接种到5 mL YPD溶液中,30 ℃、250 r/min培养16 h,8 000 r/min离心5 min收集菌体,破碎细胞提取基因组DNA。以基因组DNA为模板,以α-Factor和3′-AOX为上、下游引物进行PCR扩增。25 μL反应体系:Premix Taq 12.5 μL,模板25 ng,上、下游引物(10 μmol/L)各0.5 μL,ddH2O 11 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,53 ℃ 30 s,72 ℃ 11 min,30个循环;72 ℃ 10 min。扩增成功的为阳性工程菌。
1.7 重组脂肪酶的表达、纯化和初步筛选将阳性转化子接种于含25 mL BMGY培养基的250 mL摇瓶中,30 ℃、250 r/min培养到OD600至4.0。8 000 r/min离心5 min收集菌体,用100 mL的BMMY重悬菌体,使OD600为1.0,30 ℃、250 r/min继续培养,每24 h补加0.5% (体积比)甲醇诱导表达。96 h后收集菌液,8 000 r/min离心5 min收集上清液。
取25 mL发酵上清液利用镍柱进行亲和层析,在250 mmol/L咪唑浓度时洗脱目的蛋白,收集目的蛋白洗脱峰,利用SDS-PAGE进行纯度测定后,用于脂肪酶酶活和热稳定性的初步检测。同时,以Bradford法测定纯化前后的上清液和目的蛋白洗脱液的蛋白浓度;利用pNPP法[16]测定纯化前后的上清液和目的蛋白洗脱液的酶活:在40 ℃、pH 8.5条件下,1 min产生1 μmol对硝基苯酚的酶量定义为1个脂肪酶水解酶活国际单位(U)。
将各突变酶置于55 ℃下热处理10 min,以r27RCL残留活力为100%,得到各突变酶残留活力与r27RCL的比较。将酶活及热稳定性下降严重的突变酶弃去,剩余酶测定酶学性质。
1.8 突变脂肪酶酶学性质研究利用pNPP法测定纯化后的突变脂肪酶最适pH及pH稳定性,以及最适温度和温度稳定性及60 ℃下的半衰期。60 ℃下的半衰期测定方法为:10 U纯化酶于60 ℃下处理,每间隔15 s取样,冰浴20 min,室温平衡5 min,测定酶活。取出后酶活为未热处理时50%的时间,即为酶在60 ℃下的半衰期t1/2。Tm值是在某温度下放置一定时间后,使酶活降为原来50%的温度[17]。Tm值测定方法为:10 U纯化酶于40-65 ℃的各温度下热处理30 min,冰浴20 min,于40 ℃下测定酶活。当酶活为原来的50%时,该热处理温度即为酶的Tm值。
以pNPP作为底物测定纯化后突变脂肪酶的Km值和kcat值,Km值的测定采用双倒数作图法。测定过程中,底物浓度范围是25-1 000 mmol/L。
1.9 突变脂肪酶三维结构模拟以脂肪酶r27RCL晶体结构(Protein data bank登录号为4L3W)为模板,利用PyMOL软件对突变脂肪酶的分子结构进行模拟和热稳定性分析。
2 结果与分析 2.1 突变位点的选择以Disulfide by design软件对脂肪酶r27RCL晶体结构进行预测,筛选键能(χ3 Angle)处于+97 (±20)附近的拟二硫键,共预测出33对。弃去分子内部及催化位点中心氨基酸S172的9 Å范围内突变位点,剩余13对;删除结构明显不合理的突变体,共剩下7对拟二硫键突变位点(表 2),对它们进行实验。
| 突变酶 Mutant enzyme |
二硫键突变 Cysteine pairs |
| m1/2 | G49C-Y73C |
| m3/4 | I80C-I94C |
| m5/6 | F83C-D149C |
| m7/8 | V130C-A133C |
| m9/10 | S85C-Q145C |
| m11/12 | P194C-G220C |
| m13/14 | A213C-L244C |
对2.1中筛选出来的7对突变位点设计引物(表 1)。以pPIC9K-proRCL CHis为模板,通过全质粒PCR扩增,生成含有突变位点的带缺口环状质粒pPIC9K-proRCLm Chis (图 1),Dpn I酶消化去除模板质粒,转化E. coli,构建出E. coli pPIC9K-proRCLm Chis重组菌。通过测序分析,目标突变位点突变成功。

|
| 图 1 全质粒PCR扩增突变质粒pPIC9K-proRCLm Chis的电泳图 Figure 1 Amplification of pPIC9K-proRCLm Chis by whole-plasmid PCR 注:M:DL10000 DNA分子量标准;1:扩增目的片段pPIC9K-proRCLm Chis (10 345 bp). Note: M: DL10000 DNA marker; 1: Amplified pPIC9K-proRCLm Chis (10 345 bp). |
|
|
将突变质粒pPIC9K-proRCLm以Sal I线性化后电击转化毕赤酵母GS115感受态,在MD平板上涂布培养后获得约100个转化子,通过基因组提取和PCR扩增的方法挑选阳性转化子P. pastoris GS115 pPIC9K-proRCLm CHis Mut+。利用BMGY和BMMY培养及诱导重组脂肪酶表达,甲醇诱导84 h后将发酵液8 000 r/min离心10 min,利用镍柱对25 mL发酵上清液进行亲和层析,在250 mmol/L咪唑浓度时洗脱目的蛋白,收集目的蛋白洗脱液,并利用SDS-PAGE分析纯化结果。由图 2可知,洗脱的目的蛋白分子量大小约为37 kD。同时,以Bradford法测定纯化前后的上清液和目的蛋白洗脱液的蛋白浓度,利用pNPP法测定纯化前后的上清液和目的蛋白洗脱液的酶活,纯化结果见表 3。
|
| 图 2 m9/10蛋白纯化SDS-PAGE电泳图 Figure 2 SDS-PAGE of m9/10 before/after purification 注:1:m9/10诱导96 h后的发酵上清液;2:穿透峰;3:纯化后的目的蛋白m9/10 (37 kD);M:蛋白分子量标准. Note: 1: Fermentation supernatant of m9/10 after 96 h induction; 2: Flowthrough; 3: Purified m9/10 (37 kD); M: Protein marker. |
|
|
| 纯化结果 Purification results |
r27RCL | m9/10 |
| 上清液总酶活 Total enzyme activity of supernatant (U) |
2 120.0 | 2 094.6 |
| 上清液比活 Specific activity of supernatant (U/mg) |
419.8 | 412.2 |
| 纯化目的蛋白总酶活 Total enzyme activity of purified lipase (U) |
1 569.8 | 1 506.1 |
| 纯化目的蛋白比活 Specific activity of purified lipase (U/mg) |
1 265.3 | 1 158.6 |
| 纯化倍数Purification folds | 3.0 | 2.8 |
| 纯化得率Purification yield (%) | 74.0 | 71.9 |
测定各突变酶酶活(表 4),其中m3/4和m11/12的酶活下降严重,仅保存r27RCL活力(8.90×104 U)的7%和11.8%;而m1/2、m5/6、m7/8、m9/10和m13/14的酶活略有所下降,但都还能保持70%以上。将酶液置于55 ℃下热处理10 min,以r27RCL残留活力为100%,得到各突变酶残留活力与r27RCL的比较。可以看出,m9/10热稳定性上升幅度最大,m3/4、m11/12略有上升,m5/6和m7/8有所下降,m1/2热稳定性下降最严重。即m9/10为最佳突变,在提高了热稳定性的同时没有损失催化活性,挑选m9/10进行后续实验。
| 突变酶 Mutant enzyme |
酶活 Enzyme activity (×104 U) |
残余活力比较 Increase rate of residual activity (%) |
| m1/2 | 7.45±0.25 | -56.30±1.43 |
| m3/4 | 0.62±0.12 | +6.50±1.75 |
| m5/6 | 8.56±0.34 | -6.60±0.26 |
| m7/8 | 6.77±0.29 | -13.50±1.53 |
| m9/10 | 8.78±0.09 | +49.20±2.05 |
| m11/12 | 1.05±0.08 | +2.80±0.56 |
| m13/14 | 7.49±0.14 | +1.60±1.41 |
考察原始酶r27RCL和突变株m9/10的温度稳定性和最适温度。如图 3所示,将r27RCL和突变酶m9/10于30-50 ℃测定酶活,结果表明m9/10的最适温度没有发生变化。将纯化后的酶液于25-65 ℃温度范围内处理30 min,冰上放置20 min,测定其残余酶活(图 4)。r27RCL在45 ℃下放置30 min后酶活丧失50%,50 ℃下放置30 min后几乎已无酶活残留。突变酶m9/10热稳定性明显优于r27RCL,在25-45 ℃下热处理30 min,酶活均可保存90%以上;50 ℃下热处理30 min,酶活保存约50%;但55 ℃下热处理30 min后,酶活几乎完全丧失。原始酶r27RCL和突变酶m9/10在60 ℃下的半衰期及Tm见表 5。

|
| 图 3 温度对r27RCL和m9/10活力的影响 Figure 3 Effect of temperature on the activity of r27RCL and m9/10 |
|
|

|
| 图 4 温度对r27RCL和m9/10稳定性的影响 Figure 4 Effect of temperature on the stability of r27RCL and m9/10 |
|
|
| Items | r27RCL | m9/10 |
| t1/2(min, 60 ℃) | 0.85 | 3.85 |
| Tm(℃) | 45.0 | 49.2 |
| ΔTm(℃) | 0.0 | 4.2 |
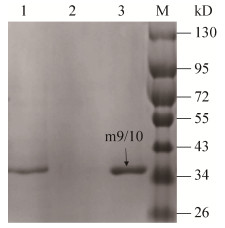
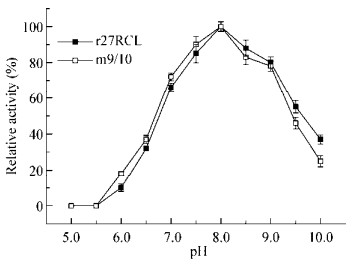
利用pNPP法对r27RCL和m9/10的最适pH和pH稳定性进行测定。结果表明,相对于原始酶r27RCL,m9/10的最适pH (图 5)和pH稳定性(图 6)几乎都没有发生变化。
|
| 图 5 pH对r27RCL和m9/10活力的影响 Figure 5 Effect ofpH on the activity of r27RCL and m9/10 |
|
|
|
| 图 6 pH对r27RCL和m9/10稳定性的影响 Figure 6 Effect ofpH on the stability of r27RCL and m9/10 |
|
|
以pNPP为底物测定r27RCL和m9/10的酶动力学参数。如表 6所示,突变酶m9/10的Km值比r27RCL下降了22.4%,说明突变酶m9/10对于底物pNPP的亲和性更高;但kcat值略有下降,kcat/Km值基本不变,说明突变后催化效率几乎未受影响。
| 酶 Enzyme |
米氏常数 Km(mmol/L) |
催化常数 kcat(s-1) |
催化效率 kcat/Km (L/(mmol·s)) |
| r27RCL | 0.304 | 18.90 | 6.22×104 |
| m9/10 | 0.236 | 14.52 | 6.15×104 |
以脂肪酶r27RCL晶体结构为模板,利用PyMOL软件对突变脂肪酶的分子结构进行模拟(图 7),研究突变位点对酶热稳定性可能产生的影响。两个半胱氨酸突变(S85C和Q145C)分别显示为红色和粉色,位于蛋白质结构表面,远离酶的催化中心(S172-H284-D231)(绿色部分)和脂肪酶活性中心上的α螺旋及两段Loop的“盖子结构”(黄色部分)。推测这两个突变不会直接引起酶催化性能的变化,动力学参数的测定也证明了这一点。

|
| 图 7 突变酶m9/10突变位点示意图 Figure 7 Mutant cysteine residues in m9/10 注:绿色部分为催化三联体(S172-H284-D231);黄色部分为覆盖在脂肪酶活性中心上的α螺旋及两段Loop的“盖子结构”;红色和粉色的2个氨基酸为突变酶m9/10中新增的二硫键突变位点(S85C和Q145C). Note: The catalytic triad residues (S172-H284-D231) are shown in green; The short α-helix linked to the 'core' of the protein structure by polypeptide hinge residues, called the 'lid' region, is shown in yellow; The two cysteine residue (S85C and Q145C), forming new added disulfide bond in mutant m9/10, is shown in red/pink sticks, respectively. |
|
|
二硫键与蛋白质的折叠过程密切相关,其键能远大于其他次级键,在维持高级结构的稳定性方面具有十分重要的作用。研究表明,二硫键通过降低蛋白质解折叠状态的熵值,用熵效应来稳定酶分子的构象。二硫键一般出现在α螺旋与另一个伸展肽链或末端不规则的α螺旋之间,在β折叠上也常有二硫键。
图 8是S85C和Q145C这一对二硫键突变前后的三维结构模拟图。85位的丝氨酸位于β2折叠上,145位的谷氨酰胺位于α4螺旋上[18],均突变成半胱氨酸后,在空间结构上有明显重叠区域形成。将m9/10突变酶结构和野生型r27RCL结构进行比较,推测突变酶85C和145C形成了二硫键,稳定了β2折叠和α4螺旋,使酶分子的结构更加紧凑,从而提高了酶的热稳定性。目前对突变体从蛋白模拟的基础上进行结构分析,今后将会进一步进行晶体结构解析,深入分析结构与功能之间的关系。
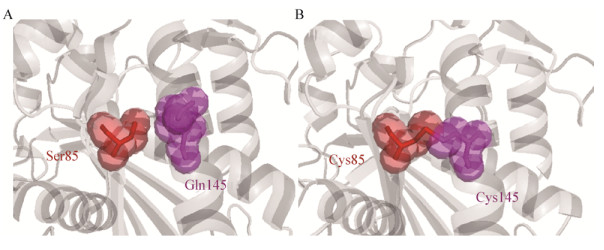
|
| 图 8 突变酶m9/10 (含突变位点S85C和Q145C)突变前(A)、后(B)三维结构模拟 Figure 8 Three-dimensional structures prior (A) and after (B) mutation of m9/10 注:位点Ser85和Gln145 (A)显示为红色和粉色,在图B中均突变成了Cys. Note: Residue Ser85 and Gln145 (A), showing as red and pink sticks, were replaced with cysteine and created a disulfide bridge (B). |
|
|
本研究以位于分子表面的氨基酸为研究对象,一方面由于蛋白质热变性最初都是从蛋白质表面的部分开始的,另一方面是由于蛋白表面位点远离酶活性中心位点和盖子结构,可以减少对酶催化活性产生的影响。但是进行实验的7组二硫键突变中,几乎所有突变酶的催化活性均有不同程度的降低,下降最严重的一组突变酶活力残存不超过10%。说明即使是酶分子表面的氨基酸,也能使得整个蛋白质分子刚性变大,使得酶在油水界面进行催化反应时,催化位点上方盖子打开幅度受影响;或者因为酶分子柔性变差,限制了底物进入活性中心进行反应,最终导致酶催化能力下降。
以突变酶分子的热稳定性为考量标准,发现7组突变酶中有3组酶的热稳定性下降了。推测原因可能是突变位点变成半胱氨酸后也未能成功形成二硫键,或者是突变位点原来可以和其他氨基酸形成稳定蛋白的如盐桥、疏水键、氢键等其他作用力,突变后丧失了原作用力导致热稳定性不升反降。
在下一步的研究中,将考量酶分子内部潜在的二硫键突变位点。一方面继续寻找能够提高热稳定性的突变位点,获得叠加效应;另一方面也可以考量是否分子内部二硫键的产生基本都以牺牲酶活为代价,为更好地开发脂肪酶提供理论基础。
| [1] |
Pandey A, Benjamin S, Soccol CR, et al. The realm of microbial lipases in biotechnology[J]. Biotechnology and Applied Biochemistry, 1999, 29(2): 119-131. |
| [2] |
Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases[J]. Enzyme and Microbial Technology, 2006, 39(2): 235-251. DOI:10.1016/j.enzmictec.2005.10.016 |
| [3] |
Xu Y, Wang D, Mu XQ, et al. Biosynthesis of ethyl esters of short-chain fatty acids using whole-cell lipase from Rhizopus chinesis CCTCC M201021 in non-aqueous phase[J]. Journal of Molecular Catalysis B: Enzymatic, 2002, 18(1/3): 29-37. |
| [4] |
Yu XW, Wang LL, Xu Y. Rhizopus chinensis lipase: gene cloning, expression in Pichia pastoris and properties[J]. Journal of Molecular Catalysis B: Enzymatic, 2009, 57(1/4): 304-311. |
| [5] |
Kamiya N, Ogawa T, Nagamune T. Enhancement of apparent thermostability of lipase from Rhizopus sp. by the treatment with a microbial transglutaminase[J]. Biotechnology Letters, 2001, 23(19): 1629-1632. DOI:10.1023/A:1011936618341 |
| [6] |
Niu WN, Li ZP, Tan TW. Secretion of pro- and mature Rhizopus arrhizus lipases by Pichia pastoris and properties of the proteins[J]. Molecular Biotechnology, 2006, 32(1): 73-82. DOI:10.1385/MB:32:1 |
| [7] |
Hiol A, Jonzo MD, Rugani N, et al. Purification and characterization of an extracellular lipase from a thermophilic Rhizopus oryzae strain isolated from palm fruit[J]. Enzyme and Microbial Technology, 2000, 26(5/6): 421-430. |
| [8] |
Matsumura M, Signor G, Matthews BW. Substantial increase of protein stability by multiple disulphide bonds[J]. Nature, 1989, 342(6247): 291-293. DOI:10.1038/342291a0 |
| [9] |
Alquati C, de Gioia L, Santarossa G, et al. The cold-active lipase of Pseudomonas fragi: Heterologous expression, biochemical characterization and molecular modeling[J]. European Journal of Biochemistry, 2002, 269(13): 3321-3328. DOI:10.1046/j.1432-1033.2002.03012.x |
| [10] |
Liebeton K, Zacharias A, Jaeger KE. Disulfide bond in Pseudomonas aeruginosa lipase stabilizes the structure but is not required for interaction with its foldase[J]. Journal of Bacteriology, 2001, 183(2): 597-603. DOI:10.1128/JB.183.2.597-603.2001 |
| [11] |
Le QAT, Joo JC, Yoo YJ, et al. Development of thermostable Candida antarctica lipase B through novel in silico design of disulfide bridge[J]. Biotechnology and Bioengineering, 2012, 109(4): 867-876. DOI:10.1002/bit.24371 |
| [12] |
Tan ZB, Li JF, Wu MC, et al. Enhancing the thermostability of a cold-active lipase from Penicillium cyclopium by in silico design of a disulfide bridge[J]. Applied Biochemistry and Biotechnology, 2014, 173(7): 1752-1764. DOI:10.1007/s12010-014-0962-7 |
| [13] |
Derewenda U, Swenson L, Wei YY, et al. Conformational lability of lipases observed in the absence of an oil-water interface: crystallographic studies of enzymes from the fungi Humicola lanuginosa and Rhizopus delemar[J]. Journal of Lipid Research, 1994, 35(3): 524-534. |
| [14] |
Eijsink VGH, Bjørk A, Gåseidnes S, et al. Rational engineering of enzyme stability[J]. Journal of Biotechnology, 2004, 113(1/3): 105-120. |
| [15] |
Johannes TW, Woodyer RD, Zhao HM. Directed evolution of a thermostable phosphite dehydrogenase for NAD(P)H regeneration[J]. Applied and Environmental Microbiology, 2005, 71(10): 5728-5734. DOI:10.1128/AEM.71.10.5728-5734.2005 |
| [16] |
Kordel M, Hofmann B, Schomburg D, et al. Extracellular lipase of Pseudomonas sp. strain ATCC 21808: purification, characterization, crystallization, and preliminary X-ray diffraction data[J]. Journal of Bacteriology, 1991, 173(15): 4836-4841. DOI:10.1128/jb.173.15.4836-4841.1991 |
| [17] |
Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability[J]. Angewandte Chemie International Edition, 2006, 45(46): 7745-7751. DOI:10.1002/(ISSN)1521-3773 |
| [18] |
Zhang M. Study of Rhizopus chinensis lipase structure, dynamic and structure-function relationship in solution using nuclear magnetic resonance assisted by X-ray diffraction[D]. Wuxi: Doctoral Dissertation of Jiangnan University, 2017 (in Chinese) 张萌. X射线衍射辅助核磁共振解析华根霉脂肪酶溶液中结构、动力学特征及构效关系[D].无锡: 江南大学博士学位论文, 2017 http://cdmd.cnki.com.cn/Article/CDMD-10295-1018063653.htm |
 2018, Vol. 45
2018, Vol. 45