扩展功能
文章信息
- 庄淼, 金锋杰
- ZHUANG Miao, JIN Feng-Jie
- 曲霉属bHLH转录因子的研究进展
- Research progress of Aspergillus bHLH transcription factors
- 微生物学通报, 2018, 45(10): 2243-2253
- Microbiology China, 2018, 45(10): 2243-2253
- DOI: 10.13344/j.microbiol.china.170913
-
文章历史
- 收稿日期: 2017-11-01
- 接受日期: 2017-01-17
- 网络首发日期(www.cnki.net): 2018-02-11
碱性螺旋-环-螺旋(Basic helix-loop-helix,bHLH)转录因子广泛存在于动植物和真菌中,其成员构成了一个蛋白家族。该蛋白家族成员在真核生物的生长发育调控过程中起着极为重要的作用,调控动物生长发育过程如神经元的发生、肌细胞的生成、肠组织的发育以及对环境毒素响应等,并且还参与调控植物花形态建成、种子萌发、气孔细胞分化、保卫细胞形成以及对胁迫作出响应等生长发育过程[1-2]。
曲霉(Aspergillus)包括构巢曲霉(Aspergillus nidulans)、烟曲霉(Aspergillus fumigatus)、米曲霉(Aspergillus oryzae)、黑曲霉(Aspergillus niger)和黄曲霉(Aspergillus flavus)等。曲霉菌广泛分布于谷物、土壤和有机体,对有机物的分解起着重要的作用。曲霉菌不仅是重要的环境微生物,同时也是发酵、食品加工业方面的重要菌株。一方面有很多菌种可被用于各种有益物质的生产,如以米曲霉为主的霉菌大都和酒、豆酱、酱油等的酿造有密切关系[3];另一方面,也有不少侵害家禽、家畜甚至人体内脏特别是呼吸器官的病菌,如引起曲霉症的烟曲霉以及能产生致癌物黄曲霉毒素(Aflatoxin)的黄曲霉。近年来,不断有研究证明bHLH转录因子对动物和植物的生长发育有着不可或缺的作用,但是真菌,尤其丝状真菌中bHLH转录因子的报道相对较少。文章对有限的曲霉属bHLH家族研究结果进行综述,以期为进一步深入研究曲霉属的生长发育及bHLH转录因子的功能提供理论参考。
1 bHLH转录因子的结构与分类碱性螺旋-环-螺旋(bHLH)转录因子因含有bHLH结构域而得名。bHLH结构域通常由近60个氨基酸组成,并且包含DNA结合域及其二聚化区域[4-5] 2个功能不同的区域,即10-15个氨基酸的碱性区域和约40个氨基酸的α螺旋-环-α螺旋(Helix-loop-helix,HLH)区[6]。碱性区域对DNA结合很重要,而HLH结构域介导其二聚化形成二聚体[7]。碱性氨基酸区位于bHLH结构域的N端,能特异性识别靶基因启动子区域中的E-box (5′-CANNTG-3′)并结合[8],其中G盒(5′-CACGTG-3′)是最常见的形式。
bHLH蛋白家族由一组在发育和分化中很重要的转录因子组成。根据与DNA的结合模式,bHLH蛋白可以分为A、B、C、D、E、F 6个组[9-10]:A组bHLH蛋白与CCGCTG结合;B组与CGCGTG结合;C组除bHLH基序外,其C端还有一个PAS结构域,能与NACGTG或NGCGTG结合;D组不与DNA结合,这一类bHLH蛋白缺乏与DNA结合的结构域[11],但可以与其他bHLH转录因子形成无活性的异源二聚体,抑制其他bHLH转录因子与DNA的结合,是bHLH蛋白的负调节因子;E组成员的碱性区域富含脯氨酸或甘氨酸[12],可优先与N-box (CACGCG或CACGAG)结合;F组含有COE结构域,该结构域在形成二聚体及与DNA结合中均发挥重要作用。根据这一分类标准及bHLH转录因子的系统发生发现,所有真菌中的bHLH蛋白均属于B组[10]。
2 曲霉属中发现的bHLH转录因子及其功能 2.1 StuAStuA是调节真菌发育和细胞周期进程的蛋白家族成员,最初在构巢曲霉中被分离并表征[13-14]。该家族的成员均具有APSES结构域,包括酿酒酵母(Saccharomyces cerevisiae)的Phd1p、Sok2p和白念珠菌(Candida albicans)的Efg1p及粗糙脉孢菌(Neurospora crassa)的Asm1p。StuA蛋白的APSES结构域与哺乳动物的bHLH蛋白MyoD、Max和E47的DNA特异性结合结构域具有结构相似性,由此将StuA归类为bHLH蛋白。构巢曲霉stuA基因缺陷株表型特征为:(1)分生孢子梗显著缩短且缺少正常的梗基和瓶梗,发育异常的分生孢子经常是从分生孢子囊泡形成的芽上直接分化而来;(2)仅有无性繁殖,不能产生有性生殖器官。由此可以得出结论:(1)虽然StuA不是营养生长所必需的,但在构巢曲霉进入繁殖生长时stuA基因被转录激活参与调控分生孢子的发育;(2) StuA是启动构巢曲霉有性生殖周期所必需的。研究表明stuA虽然在构巢曲霉成熟的分生孢子和尚未具有发育能力的体细胞菌丝中的转录水平很低,但在分生孢子形态建成期间其mRNA的表达水平增加了近50倍[15]。曲霉属分生孢子的发育主要由3个转录因子BrlA、AbaA和WetA参与调控[16]。这3个转录因子定义一个核心区域,线性调控分生孢子发育和成熟过程中相关基因时间和空间的正确表达。其中,对分生孢子生成起到关键调控作用的brlA在分生孢子形成早期阶段被激活,abaA在分生孢子发育的中期阶段被BrlA激活,而AbaA进一步诱导wetA基因表达并刺激其他相关特异性基因的表达。Miller等[13]的研究发现,stuA可以由独立的启动子启动转录产生2种mRNA——stuAα和stuAβ,BrlA蛋白诱导stuAα表达,但不诱导stuAβ的表达。这2种mRNA可以通过翻译产生相同的蛋白,并且可以与brlA、abaA的基因表达产物相互作用,进而调控分生孢子的发育,其中StuA可以通过影响AbaA的空间定位来抑制abaA的表达。
StuA还被推测与曲霉的应激反应相关。当受到氧化应激时,黄曲霉会产生次级代谢物如黄曲霉毒素,以防止细胞损伤。之前的研究发现msnA(编码酵母C2H2型锌指调节剂Msn2p的基因在曲霉中的同源物)参与调节过氧化氢酶(CAT)和超氧化物歧化酶(SOD)编码基因的表达[17]。这些抗氧化酶与黄曲霉毒素的形成是对活性氧(ROS)损伤真菌防御机制的一部分。在对黄曲霉和寄生曲霉的研究中发现,msnA缺陷株中stuA的表达水平发生了改变[18],由此推测StuA与曲霉的应激反应相关,但其确切的功能还未被发现。
2.2 AnBH1真核基因启动子区域经常存在顺式作用的CCAAT元件,与CCAAT领域结合的多聚复合物调控这些基因的转录表达[19]。在真菌构巢曲霉中,该复合物被命名为AnCF (又称构巢曲霉CCAAT结合因子)。在AnCF是否会与其他转录因子相互作用来对这些基因进行转录调控的研究中,分离出了一种新型bHLH转录因子并命名为AnBH1[20]。AnCF调控超过200个基因的转录,包括青霉素生物合成基因ipnA、aatA和aatB[21-22]。Spröte等[22]发现aatA缺陷菌株中由于aatB的存在会产生少量青霉素,而在aatA和aatB基因同时被破坏的情况下无法检测到青霉素,但aatA的过量表达也会导致青霉素合成水平降低。有趣的是aatA和aatB的转录表达被相同的AnCF和AnBH1结成的转录因子团共同调控。进一步的研究发现构巢曲霉中的蛋白激酶C (Pkc)与AnBH1在细胞中的定位相关[23]。当Pkc的活性不受抑制时,AnBH1被检测到存在于细胞核中;当Pkc的活性受到抑制时,AnBH1被定位在细胞质中。
为研究AnBH1的功能,Caruso等[21]尝试通过应用粗糙脉胞菌(Neurospora crassa)的pyr-4和构巢曲霉的argB代替anbH1从而得到anbH1敲除菌株,但均未成功获得转化菌株。由此推测anbH1的缺失对构巢曲霉来说可能是致死的。由于无法完全敲除anbH1基因,Caruso等构建了alcA(p)-anbH1融合基因取代anbH1,通过调控alcA启动子来控制anbH1基因的表达水平。研究表明在alcA(p)抑制条件下,转化株与野生型菌株相比分生孢子减少、气生菌丝增多。为进一步研究AnBH1对aatA基因表达的影响,在上述转化株的基础上进一步转化得到携带aatA(p)-lacZ基因融合单拷贝的菌株,并分别在alcA(p)诱导和抑制条件下培养。结果显示:与野生菌株相比,在抑制条件下转化株具有更高的aatA(p)-lacZ活性;在诱导条件下,则会由于alcA(p)-anbH1融合基因的过表达导致aatA(p)-lacZ表达降低。以上结果表明AnBH1阻遏aatA基因的表达,即bHLH转录因子可以抵消AnCF的作用。还有研究表明在构巢曲霉的发酵早期,AnBH1对aatA基因的表达存在一定的积极作用。另外,AnBH1对aatB基因的表达调控作用尚未明确,AnBH1的功能有待进一步的研究。
2.3 DevRTüncher等首先在构巢曲霉中分离出了一种新型bHLH转录因子并命名为DevR[24]。devR缺陷株的成功构建表明它在构巢曲霉的营养生长中并非必需。另外,在AMM (Aspergillus minimal medium)琼脂培养基中对其表型进行研究发现,与野生株相比,devR缺陷株的菌落小而褶皱,不产生分生孢子,产生黄色色素,并且所有缺陷株存在性缺陷,均不能成功自交;在AMM液体培养基中发现,缺陷株与野生株的生长状况无明显差别。这些结果证明devR是生产分生孢子所必需的,但显微镜观察发现其并不参与启动分生孢子的形成。DevR定位研究发现,在正常情况下,DevR-EGFP的融合蛋白定位于菌丝、梗基和分生孢子核中,但在tcsA缺陷株中DevR-EGFP被发现位于细胞质中。这表明TcsA参与DevR的核定位,而且2种蛋白质很可能属于相同的信号转导途径。
近来的研究表明,烟曲霉分生孢子色素产生时devR缺陷株不仅会产生轻微的生长缺陷,其分生孢子的颜色与野生株的灰绿色相比呈现浅绿色[25]。DHN-黑色素负责烟曲霉分生孢子灰绿色素的沉积,其生物合成由6个基因组成的基因簇调控,该基因簇的核心元件为pksP。对DHN-黑色素基因簇DNA序列跨物种分析发现,在pksP启动子区域中存在3个DevR的假定结合位点(E-box)。DevR与E1和E2结合可以激活pksP表达这一功能占主导作用,但与E3的结合则会抑制pksP的表达。
2.4 SrbA甾醇调节元件结合蛋白(SREBPs)是调节真核生物不同细胞反应的一类bHLH转录因子。在哺乳动物中SREBP1和SREBP2途径分别调控脂肪酸和胆固醇合成[26];在粟酒裂殖酵母和新生隐球菌中,SREBPs则作为一个重要的下游效应基因对适应低氧和麦角甾醇的生物合成至关重要[27];虽然在酿酒酵母和白念珠菌中并未发现SREBPs的直向同源物,但类似的基因Upc2和Ecm22起到与SREBPs类似的作用[28-29],这些基因的功能包括参与酵母在缺氧条件下的生长以及调节甾醇的生物合成和对抗真菌药物的抗性。固醇的生物合成、对缺氧条件的适应、对唑类药物的耐药性都与致病性酵母菌的毒力存在重要联系。烟曲霉通常可侵袭免疫受损的患者引起侵袭性肺曲霉病(Invasive pulmonary aspergillosis,IPA)[30],Willger等[31]在对烟曲霉适应缺氧所需调节因子进行研究时发现了烟曲霉中甾醇调节元件结合蛋白(SREBPs)的同源物,其氨基酸末端含有bHLH亮氨酸拉链DNA结合结构域,并将该基因命名为srbA。srbA缺陷株无法在小鼠体内引起致死性IPA。
烟曲霉中SrbA参与维持细胞极性,引导菌丝生长,适应低氧和低铁水平,它是产生毒力所必需的。在常规氧情况下野生株与srbA缺陷株均显示正常的生长速率,但显微镜观察srbA缺陷株和野生株菌丝时发现,与野生株相比,缺陷株的菌丝末端分枝明显且细胞壁-质膜界面存在着明显的缺陷。在缺氧条件下野生株以正常速率生长,但缺陷株无法生长(未观察到菌丝)。不仅如此,srbA缺陷株还显示出对氟康唑(抑制真菌细胞膜麦角固醇的生物合成)和伏立康唑(抑制形成功能性真菌膜以及维持真菌生长的甾醇生物合成)的高度敏感。野生株及缺陷株的转录谱微阵列实验显示有87个重要的基因可能受到SrbA的调节。与野生株相比,缺陷株中参与细胞壁合成(如α-半乳糖苷酶、α-葡糖苷酶B)和维持体内平衡的大量基因受到抑制,而且编码转运蛋白的几种基因也受到SrbA的调控。对缺氧的响应、甾醇的生物合成和对唑类抗真菌药物的易感性等结果与粟酒裂殖酵母和新生隐球菌中的同源物Sre1缺陷株相同[32],但维持细胞极性是SrbA在曲霉属中的独特功能。
另外,在缺氧条件下添加高水平的铁能部分恢复srbA缺陷株的生长,并恢复对三唑药物的抗药性[33]。之前的研究发现参与真菌铁代谢调节的有HapX、SreA和AcuM[34-35],Blatzer等在研究SrbA与HapX和SreA之间的关系时发现,在铁缺乏期间SrbA的转录激活与SreA和HapX无关,并且HapX的mRNA表达部分依赖于SrbA[33]。SrbA参与调节烟曲霉麦角甾醇的生物合成和铁代谢过程,因此,SrbA突变体的毒力缺陷可能不仅是因为缺氧适应的缺陷,还可能因为缺铁。
目前曲霉病诊断和治疗方面的困难主要在于这种疾病的高死亡率[36]。综合来看SrbA在烟曲霉的毒力方面起到关键作用,解开SrbA的调控网络将能更好地了解SrbA在真菌发病机制和对三唑药物敏感性中的作用,这将为治疗真菌引起的人类疾病的临床治疗提供理论依据。
2.5 SclRJin等[37]通过大片段染色体删除技术在米曲霉中发现了一种可以调控菌核形成的新型bHLH转录因子,并将其表征命名为SclR (Sclerotia regulator)[38]。Jin等[38]推测SclR含有3个糖基化位点和18个磷酸化位点。通过蛋白质基序的原位搜索(PPSearch)发现SclR可以作为依赖cAMP的蛋白激酶(PKA)、蛋白激酶C (PKC)和酪蛋白激酶2 (CK2)的磷酸化底物。这与之前的研究结果一致,许多HLH蛋白是磷酸化的底物,这是作为转录因子功能活性所必需的[39]。
麦芽琼脂培养基点培养结果表明:一方面,sclR基因缺陷株与野生株相比产生致密的分生孢子但菌核稀疏,这一表征适用于米曲霉在发酵产业上的生产利用;另一方面,sclR过表达菌株则显示分生孢子延缓产生且数量减少,其气生菌丝产生大量分枝并且相互缠结形成菌核,与野生株相比其菌核数量增加了5.5倍。菌核是致密和硬化的真菌菌丝体,核仁的一个重要作用是承受极端环境。
sclR的过表达刺激了米曲霉菌核的生产,预示其提高了菌株在极端环境中的生存耐性。在不同培养条件下的sclR转录水平调查结果表明,液体振荡培养中的米曲霉sclR表达水平远高于琼脂培养基;对其功能进一步研究发现,DPY (Dextrin-polypeptone-yeast extract)液体培养条件下sclR基因缺陷株不能形成由菌丝缠绕成球状的正常形态,菌丝极其稀疏并且生物量明显较少。SDS-PAGE结果发现,sclR基因缺陷株的菌体内部蛋白被大量分解,说明SclR功能的丧失导致了米曲霉的蛋白质迅速降解,从而进一步导致细胞快速溶解。这些结果说明sclR编码的蛋白质在维持米曲霉正常的菌株形态和细胞功能方面都有着重要的作用。另外,利用EGFP绿色荧光蛋白进行细胞定位实验的结果表明,在短期培养时间内(1 d) SclR-EGFP的融合蛋白定位于包括细胞核在内的整个细胞质中,培养3 d后大部分SclR-EGFP融合蛋白的绿色荧光仅在细胞核中被观察到。以上结果说明,SclR有可能受到上游信号刺激,仅在营养相对饥饿的条件下才被诱导聚集在细胞核中,起到转录因子的作用调控下游基因的表达。
近来的研究表明SclR参与调控和改变大部分包括糖代谢以及次级代谢产物产生相关蛋白的产生水平[40]。尤其其中参与包括糖酵解途径在内的糖代谢过程中大量蛋白的表达水平降低可能最终导致了sclR基因缺陷株的表型变化。目前的研究还表明转录因子SclR对菌核形成和菌丝融合,甚至进一步的细胞核融合也有着重要的促进作用(图 1)[41],这一发现为今后的进一步促进和实现半知菌类有性生殖开辟了新的研究视野。无论菌丝融合技术的开发还是促进未来米曲霉获得有性生殖的可能性,都意味着会促进米曲霉通过杂交方式获得新性状生产菌株的育种,有利于今后米曲霉的进一步生产利用。

|
| 图 1 由不同荧光蛋白标记细胞核的菌株形成的异核细胞的荧光显微镜观察结果[41] Figure 1 Fluorescence microscopy of heterokaryotic fusants formed by the strains with differently labeled nuclei[41] 注:细胞核中分别表达了H2B-mDsRed或H2B-EGFP融合蛋白的2个菌株在接触区形成菌核;将它们转移到CD最小培养基,并用荧光显微镜观察菌落的分生孢子和菌丝,观察到分生孢子和菌丝同时含有红色和绿色荧光,一些分生孢子显示如箭头所示的一种荧光.比例尺为5 μm. Note: The sclerotia were produced in the contact region between the strains expressing either H2B-mDsRed or H2B-EGFP fusion proteins in nuclei. They were shifted onto CD minimal medium. Conidia and hyphae from the colonies were observed by fluorescence microscopy. Note that conidia contain both red and green fluorescence and some of conidia show either of fluorescence as indicated by arrow. Scale bars=5 μm. |
|
|
Jin等[42]后续又在米曲霉中分离表征了一种表型与SclR正相反的新型bHLH蛋白编码基因并将其命名为ecdR (Early conidiophore development regulator)。对ecdR缺陷株和过表达株进行分析发现,ecdR缺陷株与参照株相比产生的菌核增多,同时分生孢子显著减少,而ecdR过表达株则表现出密集的分生孢子和稀疏的菌核形态且生长速度加快。进一步研究发现在麦芽糖琼脂培养基培养条件下,参照株低细胞密度培养会诱导形成高密度菌核,而高细胞密度培养会增加分生孢子数量;而在ecdR过表达株中,米曲霉的菌核和分生孢子密度依赖现象被完全改变。将连续稀释的分生孢子接种至麦芽琼脂培养基上(103-107)培养3 d后计数发现,参照株随着初始分生孢子密度的增加,其产生的分生孢子也明显增加,在103时产生最多菌核;ecdR过表达株比参照株更早地产生分生孢子,产生的分生孢子密度很大且随着初始密度增加无显著变化,几乎不产生菌核。
在对ecdR缺陷株与过表达株表型进行观察时,Jin等[42]发现在麦芽琼脂培养基上,ecdR缺陷株与sclR过表达株的表型相似,而ecdR过表达株的表型也与sclR缺陷株一致;通过进一步酵母双杂交实验确定了EcdR与SclR这2个bHLH蛋白通过相互作用能够形成二聚体,但这2个转录因子编码基因在不同培养条件下的表达模式完全不同(图 2)。如在琼脂培养基培养条件下,因2个转录因子的表达水平相当,SclR通过与EcdR形成异二聚体,可以竞争性阻遏SclR和EcdR与自身形成同型二聚体,从而有效抑制彼此在分生孢子或菌核形成过程中的作用(图 3)。然而在液体培养条件下,sclR的表达占完全优势,极低水平表达的EcdR不能起到任何抑制作用,使SclR转录因子的功能占据主导地位,从而严重影响菌株的表型和蛋白的产生。以上SclR-EcdR相互作用的鉴定让我们对米曲霉的生长发育乃至分化机制有了更好的理解。这也是第一次在丝状真菌曲霉属中发现bHLH家族蛋白成员之间可以通过形成异二聚体影响彼此的功能。
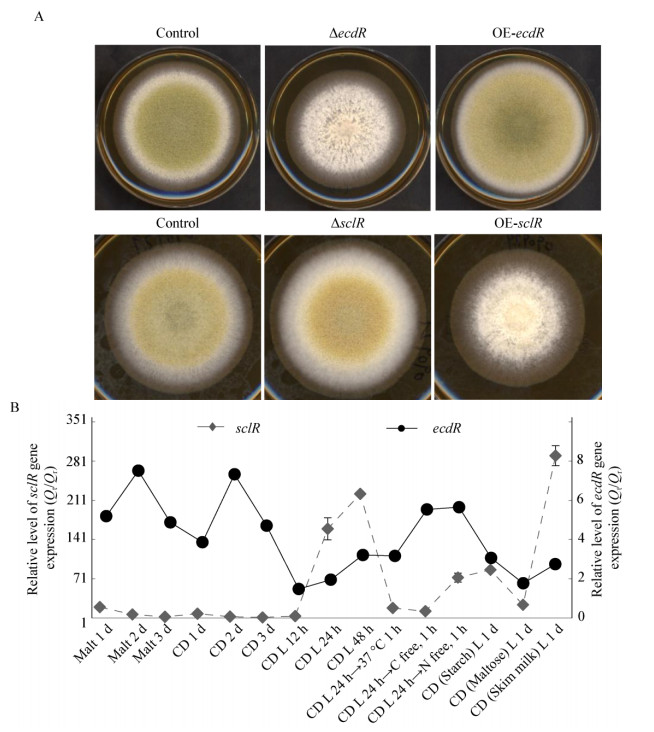
|
| 图 2 ecdR和sclR的表型和表达模式分析[42] Figure 2 Analysis of phenotypes and expression patterns of ecdR and sclR[42] 注:sclR/ecdR基因缺陷株以及过表达菌株的表型比较(A);ecdR和sclR在不同培养条件下的表达模式比较(B). Note: Comparison of phenotypes of sclR/ecdR-disruptant and overexpressing strains (A); Comparison of ecdR and sclR expression pattern (B). |
|
|
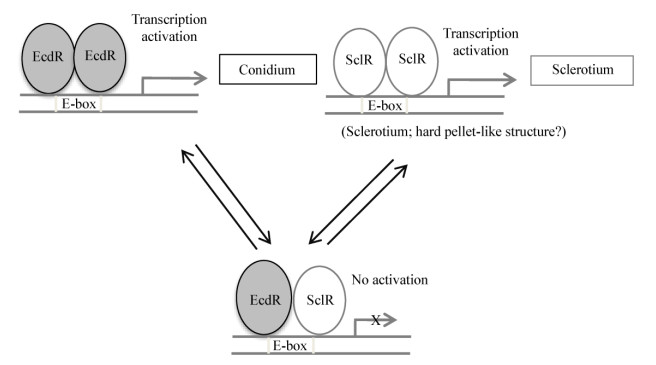
|
| 图 3 EcdR和SclR通过相互作用共同调控分生孢子和菌核的形成 Figure 3 EcdR and SclR co-regulate conidium and sclerotium formation through competitive interactions |
|
|
在酿酒酵母中,磷酸盐摄取酶由细胞周期蛋白依赖性激酶(Pho85)、细胞周期蛋白(Pho80)、细胞周期蛋白依赖性激酶抑制剂(Pho81)和bHLH转录因子Pho4 (PHO系统)参与调节。有研究发现,构巢曲霉中存在Pho4的直系同源物PalcA且参与调节PHO系统,但palcA尚未被克隆。Wu等[43]将palcA HLH结构域中的天冬酰胺密码子AAC (586)点突变为UAC酪氨酸密码子获得palcA1突变株,观察发现palcA1突变株在低磷酸盐培养基中确实无法诱导酸性磷酸酶或磷酸二酯酶的活性[44]。之前的研究发现,PalcA的确参与调节构巢曲霉磷酸盐的摄取和控制几种磷酸抑制酶的合成,也有研究猜测PalcA可能是一种磷酸通透酶[45]。迄今为止对PalcA的研究还较少,它的具体作用还有待进一步的研究。
已知的曲霉属bHLH家族蛋白成员的功能总结见表 1。
| 名称 Name |
物种 Species |
功能分析方法 Methods of function analysis |
生物学功能 Biological function |
参考文献 References |
| StuA | 构巢曲霉 Aspergillus nidulans |
删除 Deletion |
调控分生孢子发育;启动有性生殖周期 Regulate conidia development; initiate sexual reproductive cycle |
[13-14] |
| 黄曲霉 Aspergillus flavus |
实时定量PCR分析 Quantitative real-time PCR |
氧化应激反应 Response to oxidation stress |
[18] | |
| AnBH1 | 构巢曲霉 Aspergillus nidulans |
被可调节的alcA(p)-anbH1基因融合物替代 Replaced by a regulatable alcA(p)-anbH1 gene fusion |
可能是细胞必需的蛋白质;充当aatA基因表达(青霉素合成基因)的阻遏物 Likely an essential protein for the cell; act as a repressor of aatA gene expression (one of the penicillin biosynthesis) |
[21] |
| DevR | 构巢曲霉 Aspergillus nidulans |
删除 Deletion |
调控分生孢子发生和菌丝生长 Regulate conidiogenesis and hyphal growth |
[24-25] |
| 烟曲霉 Aspergillus fumigatus |
删除,实时定量PCR分析 Deletion,qPCR |
|||
| SrbA | 烟曲霉 Aspergillus fumigatus |
突变体 Mutant |
维持细胞极性,引导菌丝生长;适应低氧和低铁水平;介导对唑类抗真菌药物的抗性;产生毒力 Maintain cell polarity and direct hyphal growth; adapt to hypoxia and low iron; mediate resistance to the azole class of antifungal drugs; required for Aspergillus fumigatus virulence |
[31, 33] |
| SclR | 米曲霉 Aspergillus oryzae |
删除,过量表达 Deletion, overexpression |
维持正常的菌丝形态和细胞功能;参与无性分生孢子和菌核的形成;参与碳水化合物代谢 Retain normal hyphal morphology and cell function; involved in asexual conidiospore and sclerotial production; involved in carbohydrate metabolism |
[38, 40-41] |
| EcdR | 米曲霉 Aspergillus oryzae |
删除,过量表达,酵母双杂交 Deletion, overexpression, yeast two hybrid |
与SclR形成异源二聚体,调控分生孢子和菌核的形成 Form heterodimer with SclR, regulate asexual conidiospore and sclerotial production |
[42] |
| PalcA | 构巢曲霉 Aspergillus nidulans |
突变体 Mutant |
调节构巢曲霉磷酸盐的摄取和控制几种磷酸抑制酶的合成 Regulate phosphate acquisition, control the synthesis of several Pi-repressible enzymes |
[43-44] |
由于社会的发展和科技的进步,以及生物科技和生物信息学的快速发展,研究者们对转录因子基因家族的研究逐步加深。bHLH转录因子家族成员众多、功能多样。相对于动植物中bHLH转录因子的研究进展,曲霉中bHLH转录因子家族的研究较为缓慢。目前我们已经表征了一些曲霉属中的bHLH转录因子基因,结果表明它们在曲霉生长、分生孢子发育、次级代谢产物产生和对缺氧等胁迫方面都起着重要的作用。但该家族转录因子在曲霉属中的研究尚不深入,对这些基因的作用机理及所参与的调控网络了解还很有限,而且曲霉的相关研究虽涉及烟曲霉和米曲霉等,但主要还是侧重于构巢曲霉这一模式菌株,其他曲霉中bHLH转录因子的功能仍需要进一步挖掘。
黑曲霉和米曲霉等被广泛用于食品发酵工业、酶制剂和生物饲料等。因此,利用基因工程方法研究bHLH转录因子家族的功能,对黑曲霉和米曲霉生产酶制剂产量和效率进行改良将是未来关于bHLH转录因子家族研究的一个主要方向。对于烟曲霉等的致病菌,bHLH转录因子家族功能和作用机理的深入研究也将为治疗该疾病提供新的方向和依据。
| [1] |
Wang Y, Yao Q, Chen KP. Progress of studies on family members and functions of animal bHLH transcription factors[J]. Hereditas (Beijing), 2010, 32(4): 307-330. (in Chinese) 王勇, 姚勤, 陈克平. 动物bHLH转录因子家族成员及其功能[J]. 遗传, 2010, 32(4): 307-330. |
| [2] |
Liu WW, Li WX. Progress of plant bHLH transcription factor[J]. Current Biotechnology, 2013, 3(1): 7-11. (in Chinese) 刘文文, 李文学. 植物bHLH转录因子研究进展[J]. 生物技术进展, 2013, 3(1): 7-11. |
| [3] |
Liu LP, Liu LH. Study and application of Aspergillus oryzae in different domains[J]. China Condiment, 2008(4): 28-32. (in Chinese) 刘丽萍, 刘丽华. 米曲霉研究进展与应用[J]. 中国调味品, 2008(4): 28-32. DOI:10.3969/j.issn.1000-9973.2008.04.003 |
| [4] |
Li X, Li Y, Qu ZY, et al. The molecular mechanism and the function of bHLH regulating jasmonic acidmediated secondary metabolites synthesis[J]. Plant Physiology Journal, 2017, 53(1): 1-8. (in Chinese) 李欣, 李影, 曲子越, 等. bHLH转录因子在茉莉酸信号诱导植物次生产物合成中的作用及分子机制[J]. 植物生理学报, 2017, 53(1): 1-8. |
| [5] |
Li FM, Liu WY. Genome-wide identification, classification, and functional analysis of the basic helix-loop-helix transcription factors in the cattle, Bos Taurus[J]. Mammalian Genome, 2017, 28(5/6): 176-197. |
| [6] |
Wu DY, Yang Y, Li HY. Cloning and expression of three BpBEE genes in Betula platyphylla[J]. Journal of Zhejiang A & F University, 2017, 34(1): 137-144. (in Chinese) 武丹阳, 杨洋, 李慧玉. 3个白桦BpBEE基因的克隆与表达分析[J]. 浙江农林大学学报, 2017, 34(1): 137-144. |
| [7] |
Wiese C, Heisig J, Gessler M. Hey bHLH factors in cardiovascular development[J]. Pediatric Cardiology, 2010, 31(3): 363-370. DOI:10.1007/s00246-009-9609-9 |
| [8] |
Liu YM, Zhang L, Zhou JY, et al. Research progress of the bHLH transcription factors involved in genic male sterility in plants[J]. Hereditas (Beijing), 2015, 37(12): 1194-1203. (in Chinese) 刘永明, 张玲, 周建瑜, 等. 植物细胞核雄性不育相关bHLH转录因子研究进展[J]. 遗传, 2015, 37(12): 1194-1203. |
| [9] |
Atchley WR, Fitch WM. A natural classification of the basic helix-loop-helix class of transcription factors[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(10): 5172-5176. DOI:10.1073/pnas.94.10.5172 |
| [10] |
Wang Y, Chen KP, Yao Q. Progress of studies on bHLH transcription factor families[J]. Hereditas (Beijing), 2008, 30(7): 821-830. (in Chinese) 王勇, 陈克平, 姚勤. bHLH转录因子家族研究进展[J]. 遗传, 2008, 30(7): 821-830. DOI:10.3321/j.issn:0253-9772.2008.07.004 |
| [11] |
Wang C, Lan HY. Research progresses on functions of plant bHLH transcription factors involved in abiotic stresses[J]. Life Science Research, 2016, 20(4): 358-364. (in Chinese) 王翠, 兰海燕. 植物bHLH转录因子在非生物胁迫中的功能研究进展[J]. 生命科学研究, 2016, 20(4): 358-364. |
| [12] |
Zhang QQ, Zhu JH, Ni YM, et al. The structure and function of plant bHLH transcription factors[J]. Journal of Tropical and Subtropical Botany, 2011, 19(1): 84-90. (in Chinese) 张全琪, 朱家红, 倪燕妹, 等. 植物bHLH转录因子的结构特点及其生物学功能[J]. 热带亚热带植物学报, 2011, 19(1): 84-90. DOI:10.3969/j.issn.1005-3395.2011.01.013 |
| [13] |
Miller KY, Toennis TM, Adams TH, et al. Isolation and transcriptional characterization of a morphological modifier: the Aspergillus nidulans stunted (stuA) gene[J]. Molecular and General Genetics MGG, 1991, 227(2): 285-292. |
| [14] |
Dutton JR, Johns S, Miller BL. StuAp is a sequence-specific transcription factor that regulates developmental complexity in Aspergillus nidulans[J]. The EMBO Journal, 1997, 16(18): 5710-5721. DOI:10.1093/emboj/16.18.5710 |
| [15] |
Miller KY, Wu JG, Miller BL. StuA is required for cell pattern formation in Aspergillus[J]. Genes & Development, 1992, 6(9): 1770-1782. |
| [16] |
Mirabito PM, Adams TH, Timberlake WE. Interactions of three sequentially expressed genes control temporal and spatial specificity in Aspergillus development[J]. Cell, 1989, 57(5): 859-868. DOI:10.1016/0092-8674(89)90800-3 |
| [17] |
Chang PK, Scharfenstein LL, Luo M, et al. Loss of msnA, a putative stress regulatory gene, in Aspergillus parasiticus and Aspergillus flavus increased production of conidia, aflatoxins and kojic acid[J]. Toxins, 2011, 3(1): 82-104. DOI:10.3390/toxins3010082 |
| [18] |
El Khoury R, Caceres I, Puel O, et al. Identification of the anti-aflatoxinogenic activity of Micromeria graeca and elucidation of its molecular mechanism in Aspergillus flavus[J]. Toxins, 2017, 9(3): 87. DOI:10.3390/toxins9030087 |
| [19] |
Brakhage AA, Andrianopoulos A, Kato M, et al. HAP-like CCAAT-binding complexes in filamentous fungi: implications for biotechnology[J]. Fungal Genetics and Biology, 1999, 27(2/3): 243-252. |
| [20] |
Litzka O, Papagiannopolous P, Davis MA, et al. The penicillin regulator PENR1 of Aspergillus nidulans is a HAP-like transcriptional complex[J]. European Journal of Biochemistry, 1998, 251(3): 785-767. |
| [21] |
Caruso ML, Litzka O, Martic G, et al. Novel basic-region helix-loop-helix transcription factor (AnBH1) of Aspergillus nidulans counteracts the CCAAT-binding complex AnCF in the promoter of a penicillin biosynthesis gene[J]. Journal of Molecular Biology, 2002, 323(3): 425-439. DOI:10.1016/S0022-2836(02)00965-8 |
| [22] |
Spröte P, Hynes MJ, Hortschansky P, et al. Identification of the novel penicillin biosynthesis gene aatB of Aspergillus nidulans and its putative evolutionary relationship to this fungal secondary metabolism gene cluster[J]. Molecular Microbiology, 2008, 70(2): 445-461. DOI:10.1111/mmi.2008.70.issue-2 |
| [23] |
Herrmann M, Spröte P, Brakhage AA. Protein kinase C (PkcA) of Aspergillus nidulans is involved in penicillin production[J]. Applied and Environmental Microbiology, 2006, 72(4): 2957-2970. DOI:10.1128/AEM.72.4.2957-2970.2006 |
| [24] |
Tüncher A, Reinke H, Martic G, et al. A basic-region helix-loop-helix protein-encoding gene (devR) involved in the development of Aspergillus nidulans[J]. Molecular Microbiology, 2004, 52(1): 227-241. DOI:10.1111/j.1365-2958.2003.03961.x |
| [25] |
Valiante V, Baldin C, Hortschansky P, et al. The Aspergillus fumigatus conidial melanin production is regulated by the bifunctional bHLH DevR and MADS-box RlmA transcription factors[J]. Molecular Microbiology, 2016, 102(2): 321-335. DOI:10.1111/mmi.13462 |
| [26] |
Nomura T, Horikawa M, Shimamura S, et al. Fat accumulation in Caenorhabditis elegans is mediated by SREBP homolog SBP-1[J]. Genes & Nutrition, 2010, 5(1): 17-27. |
| [27] |
Chun CD, Liu OW, Madhani HD. A link between virulence and homeostatic responses to hypoxia during infection by the human fungal pathogen Cryptococcus neoformans[J]. PLoS Pathogens, 2007, 3(2): e22. DOI:10.1371/journal.ppat.0030022 |
| [28] |
Dunkel N, Liu TT, Barker KS, et al. A gain-of-function mutation in the transcription factor Upc2p causes upregulation of ergosterol biosynthesis genes and increased fluconazole resistance in a clinical Candida albicans isolate[J]. Eukaryotic Cell, 2008, 7(7): 1180-1190. DOI:10.1128/EC.00103-08 |
| [29] |
Vik Å, Rine J. Upc2p and Ecm22p, dual regulators of sterol biosynthesis in Saccharomyces cerevisiae[J]. Molecular and Cellular Biology, 2001, 21(19): 6395-6405. DOI:10.1128/MCB.21.19.6395-6405.2001 |
| [30] |
Nau R, Brück W. Neuronal injury in bacterial meningitis: mechanisms and implications for therapy[J]. Trends in Neurosciences, 2002, 25(1): 38-45. DOI:10.1016/S0166-2236(00)02024-5 |
| [31] |
Willger SD, Puttikamonkul S, Kim KH, et al. A sterol-regulatory element binding protein is required for cell polarity, hypoxia adaptation, azole drug resistance, and virulence in Aspergillus fumigatus[J]. PLoS Pathogens, 2008, 4(11): e1000200. DOI:10.1371/journal.ppat.1000200 |
| [32] |
Chang YC, Bien CM, Lee H, et al. Sre1p, a regulator of oxygen sensing and sterol homeostasis, is required for virulence in Cryptococcus neoformans[J]. Molecular Microbiology, 2007, 64(3): 614-629. DOI:10.1111/mmi.2007.64.issue-3 |
| [33] |
Blatzer M, Barker BM, Willger SD, et al. SREBP coordinates iron and ergosterol homeostasis to mediate triazole drug and hypoxia responses in the human fungal pathogen Aspergillus fumigatus[J]. PLoS Genetics, 2011, 7(12): e1002374. DOI:10.1371/journal.pgen.1002374 |
| [34] |
Schrettl M, Beckmann N, Varga J, et al. HapX-mediated adaption to iron starvation is crucial for virulence of Aspergillus fumigatus[J]. PLoS Pathogens, 2010, 6(9): e1001124. DOI:10.1371/journal.ppat.1001124 |
| [35] |
Liu H, Gravelat FN, Chiang LY, et al. Aspergillus fumigatus AcuM regulates both iron acquisition and gluconeogenesis[J]. Molecular Microbiology, 2010, 78(4): 1038-1054. DOI:10.1111/mmi.2010.78.issue-4 |
| [36] |
Tekaia F, Latgé JP. Aspergillus fumigatus: saprophyte or pathogen?[J]. Current Opinion in Microbiology, 2005, 8(4): 385-392. DOI:10.1016/j.mib.2005.06.017 |
| [37] |
Jin FJ, Takahashi T, Machida M, et al. Identification of a basic helix-loop-helix-type transcription regulator gene in Aspergillus oryzae by systematically deleting large chromosomal segments[J]. Applied and Environmental Microbiology, 2009, 75(18): 5943-5951. DOI:10.1128/AEM.00975-09 |
| [38] |
Jin FJ, Takahashi T, Matsushima KI, et al. SclR, a basic helix-loop-helix transcription factor, regulates hyphal morphology and promotes sclerotial formation in Aspergillus oryzae[J]. Eukaryotic Cell, 2011, 10(7): 945-955. DOI:10.1128/EC.00013-11 |
| [39] |
Gupta S, Seth A, Davis RJ. Transactivation of gene expression by Myc is inhibited by mutation at the phosphorylation sites Thr-58 and Ser-62[J]. Proceedings of the National Academy of Sciences of the United States of America, 1993, 90(8): 3216-3220. DOI:10.1073/pnas.90.8.3216 |
| [40] |
Jin FJ, Han P, Zhuang M, et al. Comparative proteomic analysis: SclR is importantly involved in carbohydrate metabolism in Aspergillus oryzae[J]. Applied Microbiology and Biotechnology, 2018, 102(1): 319-332. |
| [41] |
Wada R, Jin FJ, Koyama Y, et al. Efficient formation of heterokaryotic sclerotia in the filamentous fungus Aspergillus oryzae[J]. Applied Microbiology and Biotechnology, 2014, 98(1): 325-334. DOI:10.1007/s00253-013-5314-y |
| [42] |
Jin FJ, Nishida M, Hara S, et al. Identification and characterization of a putative basic helix-loop-helix transcription factor involved in the early stage of conidiophore development in Aspergillus oryzae[J]. Fungal Genetics and Biology, 2011, 48(12): 1108-1115. DOI:10.1016/j.fgb.2011.10.001 |
| [43] |
Wu DL, Dou XW, Hashmi SB, et al. The Pho80-like cyclin of Aspergillus nidulans regulates development independently of its role in phosphate acquisition[J]. Journal of Biological Chemistry, 2004, 279(36): 37693-37703. DOI:10.1074/jbc.M403853200 |
| [44] |
Dorn G. Genetic analysis of the phosphatases in Aspergillus nidulans[J]. Genetical Research, 1965, 6(1): 13-26. DOI:10.1017/S0016672300003943 |
| [45] |
Caddick MX, Brownlee AG, Arst HN. Phosphatase regulation in Aspergillus nidulans: responses to nutritional starvation[J]. Genetical Research, 1986, 47(2): 93-102. |
 2018, Vol. 45
2018, Vol. 45




