扩展功能
文章信息
- 郭瑞亮, 张刚, 崔钦娜, 冯婕, 陈继峰
- GUO Rui-Liang, ZHANG Gang, CUI Qin-Na, FENG Jie, CHEN Ji-Feng
- 细菌中基因组岛的转移与调控机制研究进展
- Regulation, excision and horizontal transfer of genomic islands in bacteria
- 微生物学通报, 2018, 45(10): 2234-2242
- Microbiology China, 2018, 45(10): 2234-2242
- DOI: 10.13344/j.microbiol.china.180005
-
文章历史
- 收稿日期: 2018-01-02
- 接受日期: 2018-04-13
2. 中国科学院微生物研究所 微生物资源前期开发国家重点实验室 北京 100101
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
水平基因转移(Horizontal gene transfer,HGT)能够使细菌从微环境中获得利于自身生存的基因,是细菌进化的重要驱动力。不同种属间的细菌通过移动元件(Mobile element)的转移而进行遗传信息的交流,移动元件包括基因组岛、插入序列、前噬菌体、整合子、转座子、接合性质粒和整合性接合元件等。这些移动元件内部包含不同种类的功能基因,通过水平基因转移的方式在不同种属细菌间扩散和传播。其中对人类健康产生严重威胁的是毒力因子和耐药基因的广泛传播,携带有耐药基因的临床致病菌,如鲍曼不动杆菌、金黄色葡萄球菌和肺炎克雷伯菌等极易造成病人术后的二次感染,危及生命。我们实验室通过对基因组和宏基因组数据的分析发现,土壤、动物粪肥、海水养殖等环境中存在多种耐药基因,移动元件能够介导环境中的耐药基因形成基因簇并在不同宿主中扩散和传播[1-4]。基因组岛(Genomic islands,GI)则是移动元件中最重要的一大类,其内部不仅是毒力因子、耐药基因、代谢调控相关等功能基因的聚类[5-6],而且携带有转座子、插入序列等移动元件。因此,研究基因组岛复杂的转移调控机制对控制病原菌耐药的发展是非常重要的。目前,由基因组岛介导的细菌获得性耐药已成为研究热点。
基因组岛是相对独立的DNA片段,分为可移动和不可移动两大类。前者能够整合进宿主基因组,在特定的环境压力下切离成环并通过转化、转导等方式整合到新的宿主基因组中。基因组岛的转移有助于微生物对外界环境的适应,因此对宿主基因组的可塑性和进化演变方面发挥了关键作用[7]。
1 基因组岛的结构及功能1990年,Hacker等在对大肠杆菌536和J96的溶血素基因的研究中发现,该毒力基因存在于染色体可删除片段中,并将这段两端带有重复序列的片段定义为毒力岛(Pathogenicity islands,PAIs)[8]。近一步研究发现,整合性接合元件是一类可自主转移的基因组岛,ICEberg数据库收录了400多个该类元件,其中188个携带耐药基因,136个编码致病因子[9]。迄今为止大多数基因组岛都存在以下特征(图 1):

|
| 图 1 基因组岛的基本特征 Figure 1 General features of genomic island |
|
|
(1) 基因组岛是存在于原核生物染色体上相对独立的DNA片段,片段长度通常在10-200 kb之间[10]。例如,Hacker等在大肠杆菌536中发现的致病岛PAI-Ⅰ、PAI-Ⅱ、PAI-Ⅲ和PAI-Ⅳ的长度在25-190 kb之间[8];Song等在铜绿假单胞菌中确定的可移动基因组岛PAO1GI、PA14GI、PA7GI和LESB58GI的大小在10-110 kb之间[11]。
(2) 基因组岛的G+C%含量、寡聚核苷酸分布和密码子使用等序列特征上与其所在染色体骨架区存在显著差异。例如,在假单胞菌PAO1和PA14中,染色体骨架区的G+C%含量在66%以上,其内部基因组岛的G+C%含量都低于该值,在47%-61%之间[12]。其中基因组岛PA14GI-1的G+C%含量比其染色体骨架区的G+C%含量低5%以上。
(3) 基因组岛通常整合在tRNA、tmRNA以及GMP合酶基因的3′末端。例如,Mantri等[13]通过对141个基因组岛分析发现,tRNA和tmRNA的下游是基因组岛的整合热点区域;Zhang等[14]通过对28株肺炎克雷伯菌的环境和临床菌的基因组分析发现,tRNA和tmRNA是外来基因组岛的插入热点区域;Song等[11]通过对34个细菌基因组分析,确定了34个整合在GMP合酶基因下游的基因组岛;Szabó等[15]和Harmer等[16]研究发现,dcm-1基因是基因组岛GIsul2的插入整合区域。
(4) 基因组岛内部存在位点特异性重组酶,根据氨基酸序列同源性和催化机制的不同,分为酪氨酸重组酶和丝氨酸重组酶[17]。同时在基因组岛两侧会形成16-20 bp的正向重复序列(Direct repeats,DRs)。正向重复序列由位点特异性重组产生,并作为整合、切离过程的识别位点序列[18]。
(5) 基因组岛常携带转座子、插入序列等移动元件以及功能基因簇。根据其内部所附加的功能基因不同,可将其分为代谢岛(Metabolic islands)、共生岛(Symbiosis islands)、抗性岛(Resistance islands)、毒力岛(Pathogenicity islands)和适应性岛(Fitness islands)等[6, 19] (图 1)。基因组岛赋予了宿主菌丰富多样的特性,例如:携带有溶血毒素的基因组岛使大肠杆菌536和J96具有致病性[8];接合性转座子SXT使霍乱弧菌具有抗新诺明、甲氧苄氨嘧啶和链霉素的耐药表型[20];在金黄色葡萄球菌(Staphylococcus aureus N315)中发现了对甲氧苯青霉素耐药的基因组岛[21]等。
2 基因组岛的转移机制基因组岛中起核心作用的是整合酶,这些酶多是酪氨酸重组酶,与噬菌体来源的整合酶相似。我们实验室就曾据此对携带有多重耐药基因的移动元件ISCR2进行过相关研究[2, 22]。根据噬菌体整合酶位点特异性重组的机制,基因组岛的转移(位点特异性重组)如图 2所示;基因组岛通过其内部的整合酶与边界的正向重复序列相互作用,从宿主染色体上切离环出成为环状中间体;成环后的基因组岛也可在整合酶与其他辅助因子的作用下与染色体发生位点特异性重组,进而整合到染色体中。Schubert等在2004年提出了假结核耶尔森HPI (High-pathogenicity island)基因组岛转移机制模型,即整合酶介导的位点特异性重组系统[23]。有研究者使用双质粒系统对大肠杆菌毒力岛(PAI)的整合进行了研究,发现PAI-Ⅱ和PAI-Ⅲ重组位点attB的碱基数目可以分别降至16 bp和20 bp而不影响重组效率[24]。
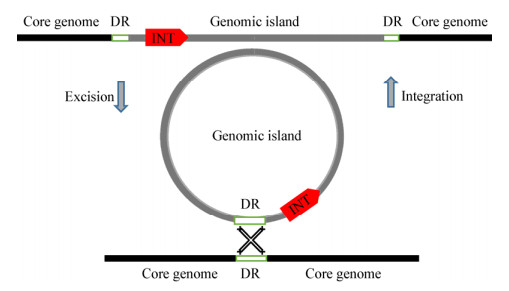
|
| 图 2 可移动基因组岛的环出与整合 Figure 2 Excision and integration of mobile genomic island |
|
|
基因组岛的转移实际上是由一套位点特异性重组系统来实现的。最简单的位点特异性重组系统包含整合酶以及该酶所作用的重复序列(重组位点)。在基因组岛转移的过程中,整合酶催化结构域中的一个保守氨基酸残基攻击靶序列,产生断裂切口并形成DNA-蛋白的联会复合体,通过复合体构象的改变完成DNA链交换,从而使基因组岛与宿主染色体间靶序列特异性重组,并最终完成基因组岛的转移[25-26]。重组所需的靶序列通常是20 bp左右的双链DNA序列,靶序列的中心是DNA断裂-重组区域,两侧是整合酶的识别位点。大肠杆菌噬菌体P1编码的Cre-loxP位点特异性重组系统就具有如此最简单的位点识别结构[27-28](图 3A)。

|
| 图 3 整合酶识别位点结构 Figure 3 Structure of recombination site |
|
|
除了Cre-loxP这种简单的识别位点,许多整合酶的重组位点更加复杂,不仅包含20 bp左右的链交换区域,而且还包含100 bp左右的附加序列,如λ整合酶、XerCD整合酶[17, 24]。附加序列不仅为整合酶提供结合位点,还提供了辅助因子的作用位点。近年来研究发现,辅助因子大多数是位点特异性重组系统所必需的。宿主整合因子可使DNA序列形成复杂的二级结构,从而使整合酶更精准地识别与结合靶位点。辅助因子可能参与整合酶构象的改变,或在重组过程发挥调控功能,或者在基因组岛切离和整合阶段与整合酶起协同作用。多数基因组岛正是通过复杂的位点特异性重组系统来实现高效、精准的整合与转移,如大肠杆菌λ-整合酶重组系统[29](图 3B)。在λ-整合酶介导attP (Attachment site of phage)位点与attB (Attachment site of bacteria)位点特异性重组的过程中,宿主整合因子(Integration host factor,IHF)结合到H1、H2和H′区域中,使DNA序列发生不同程度的弯曲,形成复杂的二级结构,在λ-整合酶与靶序列结合后,有利于DNA-蛋白联会复合体形成四聚体的空间结构。此外,attP位点还包含了切离酶(Excisionase,Xis)结合位点以及辅助因子Fis (Factor for inversion stimulation)的结合区域[24, 30]。在Fis蛋白及其他辅助蛋白因子的协同作用下,四聚体联会复合物通过依次在不同的臂型结合区(Arm type sites) P1、P2、P′1、P′2和P′3结合,从而完成DNA链的交换,并最终完成基因组岛与宿主整合位点的特异性重组。
基因组岛的内部还存在与整合酶具有协同作用的因子。例如,soj基因调控环化后基因组岛整合到宿主染色体的时机[31]。Cox蛋白与切离酶Xis参与基因组岛环化的过程[32]。alpA基因与整合酶相互作用,从而调控基因组岛转移[33]。基因组岛常携带细菌Ⅳ型分泌系统(T4SS)基因簇。T4SS是一种在革兰氏阴性菌和革兰氏阳性菌中均广泛存在的多功能、多组分的跨膜通道结构,可介导单链DNA的接合转移。例如,携带T4SS的嗜血杆菌基因岛ICEHin1056和假单胞菌基因岛ICEclc可介导耐药基因和毒力因子的传播[34]。SecReT4数据库收录了811个T4SS基因簇,分布于286条细菌染色体和352个质粒上[35]。此外,基因组岛和质粒等可移动遗传元件常编码Ⅱ型毒素-抗毒素系统(Toxin-antitoxin,TA)。Ⅱ型TA基因通常组成一个独立的操纵子,抗毒素蛋白能与相应的毒素蛋白结合以中和其毒性;该系统与岛和质粒在受体菌中的遗传稳定性密切相关。TADB数据库收录了800多个复制子中的6 000多对Ⅱ型TA,并提供了预测工具TAfinder[36]。因此,基因组岛的转移是由多种复杂机制调控的结果。
3 ICE转移调控分析整合性结合元件(Integrative and conjugative element,ICE)属于基因组岛中的一类,ICE转移的过程中涉及到多种蛋白的相互作用以及调控因子对ICE的调节作用。ICE不仅能够以特异性位点重组的方式整合到染色体中,还可以从染色体上环出,并以类似于结合性质粒的转移方式在不同细胞间进行传播。因此,对ICE转移所涉及调控因子的研究是非常重要的。
Beaber等在对霍乱弧菌中SXT元件的转移研究中发现,喹诺酮类抗生素的使用会导致霍乱弧菌DNA损伤并产生SOS应激修复[37]。SOS反应会导致RecA蛋白被激活,激活的RecA蛋白有助于setR的自我分解,从而减少setR对setC和setD因子的抑制作用,转录因子setC和setD激活了INT整合酶的表达,提高了SXT元件的转移频率(图 4)。

|
| 图 4 SXT元件的调控 Figure 4 The regulatory pathway of SXT transfer |
|
|
Sentchilo等对假单胞菌B13中的clc基因组岛研究发现,在基因组岛clc转移的过程中,参与位点特异性重组的整合酶intB13在不同阶段的表达量不同[38]。即在clc元件处于染色体整合态和环状中间态时整合酶intB13的表达受到两个不同启动子的调控。当clc元件处于环状中间态时,整合酶intB13由启动子Pcirc进行转录,整合酶的高表达使clc元件更频繁地切除和环化;当clc元件处于染色体整合态时,整合酶intB13的转录由启动子Pint调控,弱转录活性的启动子Pint使得clc元件更趋向于染色体整合状态(图 5)。

|
| 图 5 clc基因组岛的调控 Figure 5 The regulatory pathway of clc genomic island |
|
|
Auchtung等对枯草芽孢杆菌中的基因组岛ICEBs1研究发现,基因组岛ICEBs1的转移至少由2种不同的通路进行调控[39-42]。一种是由SOS反应所引起的基因组岛的转移,另一种则是由细菌群落群感效应所产生的多肽物质对基因组岛的转移进行调控。ICEBs1的环出需要切离酶xis的表达,阻遏蛋白immR通过抑制启动子Pxis的转录进而调控ICEBs1的环出,抗阻遏蛋白immA则会抑制immR的活性,从而激活ICEBs1的表达与转移。SOS反应通过RecA蛋白激活抗阻遏蛋白immA,进而增强ICEBs1的转移。在群感效应的调控通路中,RapI蛋白会激活抗阻遏蛋白immA的表达,与此同时Pre-Phr蛋白被转运到胞外,形成成熟的Phr多肽。通过寡肽通透酶(Opp)进入细胞的Phr多肽则会抑制RapI蛋白的活性进而间接地抑制转录因子的活性,并调节ICEBs1的转移(图 6)。
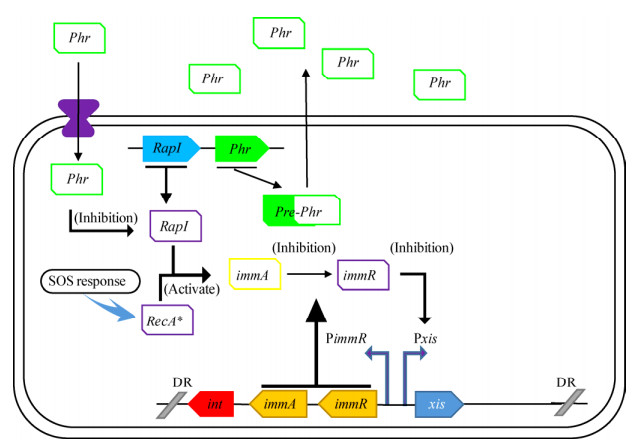
|
| 图 6 ICEBs1基因组岛的调控 Figure 6 The regulatory pathway of ICEBs1 genomic island |
|
|
目前,基因组岛的预测主要分为两类:基于单个基因组的序列特征差异方法和基于多个亲缘菌的比较基因组方法[43-45]。单个基因组的序列特征差异法是根据不同种属细菌间基因组序列构成的差异预测基因组岛,主要有:SIGI-HMM[46]和Island Path-DIMOB[47]等方法。通常来说,基因组岛的G+C%含量、二核苷酸偏好性以及密码子偏好性与其所在的基因组存在显著性差异。若基因组中存在序列特征差异的片段,那么则预测该片段是水平基因转移的候选片段[48]。但基因突变以及高表达基因的存在所引起的序列差异会导致该方法预测的基因组岛出现假阳性。此外,随着时间的推移,基因组岛的序列构成会趋同于所在基因组的序列构成,从而导致部分基因组岛未能被准确识别。对此,VRprofile软件采用“功能模块为主,序列特征为辅”的方法来预测基因组岛。内置Profile-HMM方法搜索岛的整合和接合模块特征蛋白同源基因,经基因簇共定位方法识别整个岛,再结合序列特征获得更高的预测精度[49]。比较基因组法则是利用全基因组序列比对,寻找独立存在于某基因组而不存在于亲缘菌基因组中的大片段DNA。比较基因组法有:Island Pick方法[50]和Mobilome FINDER方法[51]。但该方法存在基因组分化前所插入的基因组岛不能被准确识别的问题。
目前常用的基因组岛预测方法是Island Viewer[52],内置3种精确的预测方法:Island Pick、Island Path-DIMOB和SIGI-HMM。Island Viewer则通过内置方法的差异互补预测基因组岛。Island Viewer提供了可视化分析工具,能够对用户提供的序列信息提供交互式基因组可视化体验。在预测基因组岛的同时,还能够对毒力因子和耐药基因进行注释,是当前预测基因组岛较为全面的互联网服务器。
5 展望目前,细菌获得性耐药问题是威胁人类健康的难题之一。细菌可以通过基因突变和基因水平转移的方式获得耐药性,近几年来病原菌基因组大规模分析结果显示,基因水平转移使多数病原菌获得耐药特性,特别是多重耐药菌的形成[1, 3, 53-54]。例如,基因组岛ICEPmiJpnl和ICEPmiSpnl携带blaCMY-2基因,从而使宿主菌具有广谱β-内酰胺抗性[55]。因此,抑制基因组岛的转移是控制细菌耐药性传播的重要手段。
近来我们通过敲除实验等技术手段初步发现,多数基因组岛的转移过程存在辅助因子的参与和调控。但相关辅助因子的作用仍未被完全阐明。例如,基因组岛成环后转移并整合到新宿主过程中辅助蛋白的调控机制和作用靶点问题。此外,整合酶介导重组的体外验证实验以及重要整合酶的晶体结构解析都尚未有新进展。因此,研究并解决以上这些问题,进而系统揭示基因组岛转移的精细调控机制将对最终抑制基因组岛传播的频率具有重要意义。未来对重要基因组岛重组转移及其调控分子机制的深入研究将是一个非常重要的方向。
| [1] |
Yang J, Wang C, Wu JY, et al. Characterization of a multiresistant mosaic plasmid from a fish farm sediment Exiguobacterium sp. isolate reveals aggregation of functional clinic-associated antibiotic resistance genes[J]. Applied and Environmental Microbiology, 2014, 80(4): 1482-1488. DOI:10.1128/AEM.03257-13 |
| [2] |
Zhou WQ, Yao KH, Zhang G, et al. Mechanism for transfer of transposon Tn2010 carrying macrolide resistance genes in Streptococcus pneumoniae and its effects on genome evolution[J]. Journal of Antimicrobial Chemotherapy, 2014, 69(6): 1470-1473. DOI:10.1093/jac/dku019 |
| [3] |
Leclercq SO, Wang C, Zhu YX, et al. Diversity of the tetracycline mobilome within a chinese pig manure sample[J]. Applied and Environmental Microbiology, 2016, 82(21): 6454-6462. DOI:10.1128/AEM.01754-16 |
| [4] |
Leclercq SO, Wang C, Sui ZH, et al. A multiplayer game: species of Clostridium, Acinetobacter, and Pseudomonas are responsible for the persistence of antibiotic resistance genes in manure-treated soils[J]. Environmental Microbiology, 2016, 18(10): 3494-3508. DOI:10.1111/1462-2920.13337 |
| [5] |
Bardaji L, Echeverría M, Rodríguez-Palenzuela P, et al. Four genes essential for recombination define GInts, a new type of mobile genomic island widespread in bacteria[J]. Scientific Reports, 2017, 7: 46254. DOI:10.1038/srep46254 |
| [6] |
Coetzee JN, Datta N, Hedges RW. R factors from Proteus rettgeri[J]. Journal of General Microbiology, 1972, 72(3): 543-552. DOI:10.1099/00221287-72-3-543 |
| [7] |
Juhas M, van der Meer JR, Gaillard M, et al. Genomic islands: tools of bacterial horizontal gene transfer and evolution[J]. FEMS Microbiology Review, 2009, 33(2): 376-393. DOI:10.1111/j.1574-6976.2008.00136.x |
| [8] |
Hacker J, Bender L, Ott M, et al. Deletions of chromosomal regions coding for fimbriae and hemolysins occur in vitro and in vivo in various extra intestinal Escherichia coli isolates[J]. Microbial Pathogenesis, 1990, 8(3): 213-225. DOI:10.1016/0882-4010(90)90048-U |
| [9] |
Bi DX, Xu Z, Harrison EM, et al. ICEberg: a web-based resource for integrative and conjugative elements found in Bacteria[J]. Nucleic Acids Research, 2012, 40(D1): D621-D626. DOI:10.1093/nar/gkr846 |
| [10] |
Hacker J, Kaper JB. Pathogenicity islands and the evolution of microbes[J]. Annual Review of Microbiology, 2000, 54(1): 641-679. DOI:10.1146/annurev.micro.54.1.641 |
| [11] |
Song L, Pan YT, Chen SH, et al. Structural characteristics of genomic islands associated with GMP synthases as integration hotspot among sequenced microbial genomes[J]. Computational Biology and Chemistry, 2012, 36: 62-70. DOI:10.1016/j.compbiolchem.2012.01.001 |
| [12] |
Song L, Zhang XH. Innovation for ascertaining genomic islands in PAO1 and PA14 of Pseudomonas aeruginosa[J]. Chinese Science Bulletin, 2009, 54(21): 3991-3999. DOI:10.1007/s11434-009-0598-0 |
| [13] |
Mantri Y, Williams KP. Islander: a database of integrative islands in prokaryotic genomes, the associated integrases and their DNA site specificities[J]. Nucleic Acids Research, 2004, 32(S1): D55-D58. |
| [14] |
Zhang J, van Aartsen JJ, Jiang XF, et al. Expansion of the known Klebsiella pneumoniae species gene pool by characterization of novel alien DNA islands integrated into tmRNA gene sites[J]. Journal of Microbiological Methods, 2011, 84(2): 283-289. DOI:10.1016/j.mimet.2010.12.016 |
| [15] |
Szabó M, Nagy T, Wilk T, et al. Characterization of two multidrug-resistant IncA/C plasmids from the 1960s by Using the MinION sequencer device[J]. Antimicrobial Agents and Chemotherapy, 2016, 60(11): 6780-6786. DOI:10.1128/AAC.01121-16 |
| [16] |
Harmer CJ, Hamidian M, Hall RM. pIP40a, a type 1 IncC plasmid from 1969 carries the integrative element GIsul2 and a novel class Ⅱ mercury resistance transposon[J]. Plasmid, 2017, 92: 17-25. DOI:10.1016/j.plasmid.2017.05.004 |
| [17] |
Zhang L, Zhao GP, Ding XM. Site-specific recombination systems: mechanisms and applications[J]. Scientia Sinica Vitae, 2010, 40(12): 1090-1111. (in Chinese) 张霖, 赵国屏, 丁晓明. 位点特异性重组系统的机理和应用[J]. 中国科学:生命科学, 2010, 40(12): 1090-1111. |
| [18] |
Schmidt H, Hensel M. Pathogenicity islands in bacterial pathogenesis[J]. Clinical Microbiology Review, 2004, 17(1): 14-56. DOI:10.1128/CMR.17.1.14-56.2004 |
| [19] |
Dobrindt U, Hochhut B, Hentschel U, et al. Genomic islands in pathogenic and environmental microorganisms[J]. Nature Reviews Microbiology, 2004, 2(5): 414-424. DOI:10.1038/nrmicro884 |
| [20] |
Waldor MK, Tschäpe H, Mekalanos JJ. A new type of conjugative transposon encodes resistance to sulfamethoxazole, trimethoprim, and streptomycin in Vibrio cholerae O139[J]. Journal of Bacteriology, 1996, 178(14): 4157-4165. DOI:10.1128/jb.178.14.4157-4165.1996 |
| [21] |
Ito T, Katayama Y, Hiramatsu K. Cloning and nucleotide sequence determination of the entire mec DNA of Pre-methicillin-resistant Staphylococcus aureus N315[J]. Antimicrobial Agents and Chemotherapy, 1999, 43(6): 1449-1458. DOI:10.1128/AAC.43.6.1449 |
| [22] |
Xu YX, Wang C, Zhang G, et al. ISCR2 is associated with the dissemination of multiple resistance genes among Vibrio spp. isolated from farmed fish[J]. Archives of Microbiology, 2017, 199(6): 891-896. DOI:10.1007/s00203-017-1365-2 |
| [23] |
Schubert S, Rakin A, Heesemann J. The Yersinia high-pathogenicity island (HPI): evolutionary and functional aspects[J]. International Journal of Medical Microbiology, 2004, 294(2-3): 83-94. DOI:10.1016/j.ijmm.2004.06.026 |
| [24] |
Rajeev L, Malanowska K, Gardner JF. Challenging a paradigm: the role of DNA homology in tyrosine recombinase reactions[J]. Microbiology and Molecular Biology Reviews, 2009, 73(2): 300-309. DOI:10.1128/MMBR.00038-08 |
| [25] |
Grindley NDF, Whiteson KL, Rice PA. Mechanisms of site-specific recombination[J]. Annual Review of Biochemistry, 2006, 75: 567-605. DOI:10.1146/annurev.biochem.73.011303.073908 |
| [26] |
Landy A. The λ integrase site-specific recombination pathway[J]. Microbiology Spectrum, 2015, 3(2): 91-118. |
| [27] |
Abremski K, Hoess R. Bacteriophage P1 site-specific recombination: purification and properties of the Cre recombinase protein[J]. Journal of Biology Chemistry, 1984, 259(3): 1509-1514. |
| [28] |
van Duyne GD. A structural view of cre-loxP site-specific recombination[J]. Annual Review of Biophysics and Biomolecular Structure, 2001, 30: 87-104. DOI:10.1146/annurev.biophys.30.1.87 |
| [29] |
Landy A, Ross W. Viral integration and excision: structure of the Lambda att sites[J]. Science, 1977, 197(4309): 1147-1160. DOI:10.1126/science.331474 |
| [30] |
Warren D, Laxmikanthan G, Landy A. A chimeric Cre recombinase with regulated directionality[J]. Proceedings of the National Academy of Sciences of United States of America, 2008, 105(47): 18278-18283. DOI:10.1073/pnas.0809949105 |
| [31] |
Qiu XY, Gurkar AU, Lory S. Interstrain transfer of the large Pathogenicity island (PAPI-1) of Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Sciences of United States of America, 2006, 103(52): 19830-19835. DOI:10.1073/pnas.0606810104 |
| [32] |
Lewis JA, Hatfill GF. Control of directionality in integrase-mediated recombination: examination of recombination directionality factors (RDFs) including Xis and Cox Proteins[J]. Nucleic Acids Research, 2001, 29(11): 2205-2216. DOI:10.1093/nar/29.11.2205 |
| [33] |
Kirby JE, Trempy JE, Gottesman S. Excision of a P4-like cryptic prophage leads to Alp protease expression in Escherichia coli[J]. Journal of Bacteriology, 1994, 176(7): 2068-2081. DOI:10.1128/jb.176.7.2068-2081.1994 |
| [34] |
Juhas M. Type IV secretion systems and genomic islands-mediated horizontal gene transfer in Pseudomonas and Haemophilus[J]. Microbiological Research, 2015, 170: 10-17. DOI:10.1016/j.micres.2014.06.007 |
| [35] |
Bi DX, Liu LM, Tai C, et al. SecReT4: a web-based bacterial type IV secretion system resource[J]. Nucleic Acids Research, 2013, 41(D1): D660-D665. DOI:10.1093/nar/gks1248 |
| [36] |
Xie YZ, Wei YQ, Shen Y, et al. TADB 2.0: an updated database of bacterial type Ⅱ toxin-antitoxin loci[J]. Nucleic Acids Research, 2018, 46(D1): D749-D753. DOI:10.1093/nar/gkx1033 |
| [37] |
Beaber JW, Hochhut B, Waldor MK. SOS response promotes horizontal dissemination of antibiotic resistance genes[J]. Nature, 2004, 427(6969): 72-74. DOI:10.1038/nature02241 |
| [38] |
Sentchilo V, Zehnder AJB, van der Meer JR. Characterization of two alternative promoters for integrase expression in the clc genomic island of Pseudomonas sp. strain B13[J]. Molecular Microbiology, 2003, 49(1): 93-104. DOI:10.1046/j.1365-2958.2003.03548.x |
| [39] |
Auchtung JM, Lee CA, Monson RE, et al. Regulation of a Bacillus subtilis mobile genetic element by intercellular signaling and the global DNA damage response[J]. Proceedings of the National Academy of Sciences of United States of America, 2005, 102(35): 12554-12559. DOI:10.1073/pnas.0505835102 |
| [40] |
Auchtung JM, Lee CA, Garrison KL, et al. Identification and characterization of the immunity repressor (ImmR) that controls the mobile genetic element ICEBs1 of Bacillus subtilis[J]. Molecular Microbiology, 2007, 64(6): 1515-1528. DOI:10.1111/j.1365-2958.2007.05748.x |
| [41] |
Bose B, Auchtung JM, Lee CA, et al. A conserved anti-repressor controls horizontal gene transfer by proteolysis[J]. Molecular Microbiology, 2008, 70(3): 570-582. DOI:10.1111/mmi.2008.70.issue-3 |
| [42] |
Bose B, Grossman AD. Regulation of horizontal gene transfer in Bacillus subtilis by activation of a conserved site-specific protease[J]. Journal of Bacteriology, 2011, 193(1): 22-29. DOI:10.1128/JB.01143-10 |
| [43] |
Lu BX, Leong HW. Computational methods for predicting genomic islands in microbial genomes[J]. Computational and Structural Biotechnology Journal, 2016, 14: 200-206. DOI:10.1016/j.csbj.2016.05.001 |
| [44] |
Zhang R, Ou HY, Gao F, et al. Identification of horizontally-transferred genomic islands and genome segmentation points by using the GC profile method[J]. Current Genomics, 2014, 15(2): 113-121. DOI:10.2174/1389202915999140328163125 |
| [45] |
Ou HY, Chen LL, Lonnen J, et al. A novel strategy for the identification of genomic islands by comparative analysis of the contents and contexts of tRNA sites in closely related bacteria[J]. Nucleic Acids Research, 2006, 34(1): e3. DOI:10.1093/nar/gnj005 |
| [46] |
Waack S, Keller O, Asper R, et al. Score-based prediction of genomic islands in prokaryotic genomes using hidden Markov models[J]. BMC Bioinformatics, 2006, 7: 142. DOI:10.1186/1471-2105-7-142 |
| [47] |
Hsiao WWL, Ung K, Aeschliman D, et al. Evidence of a large novel gene pool associated with prokaryotic genomic islands[J]. PLoS Genetics, 2005, 1(5): e62. DOI:10.1371/journal.pgen.0010062 |
| [48] |
Bosi E, Fani R, Fondi M. The mosaicism of plasmids revealed by atypical genes detection and analysis[J]. BMC Genomics, 2011, 12: 403. DOI:10.1186/1471-2164-12-403 |
| [49] |
Li J, Tai C, Deng ZX, et al. VRprofile: gene-cluster-detection- based profiling of virulence and antibiotic resistance traits encoded within genome sequences of pathogenic bacteria[J]. Briefings in Bioinformatics, 2017. DOI:10.1093/bib/bbw141 |
| [50] |
Langille MGI, Hsiao WWL, Brinkman FSL. Evaluation of genomic island predictors using a comparative genomics approach[J]. BMC Bioinformatics, 2008, 9: 329. DOI:10.1186/1471-2105-9-329 |
| [51] |
Ou HY, He XY, Harrison EM, et al. MobilomeFINDER: web-based tools for in silico and experimental discovery of bacterial genomic islands[J]. Nucleic Acids Research, 2007, 35(W1): W97-W104. |
| [52] |
Langille MGI, Brinkman FSL. IslandViewer: an integrated interface for computational identification and visualization of genomic islands[J]. Bioinformatics, 2009, 25(5): 664-665. DOI:10.1093/bioinformatics/btp030 |
| [53] |
Martínez JL, Coque TM, Lanza VF, et al. Genomic and metagenomic technologies to explore the antibiotic resistance mobilome[J]. Annals of the New York Academy of Sciences, 2017, 1388(1): 26-41. DOI:10.1111/nyas.2017.1388.issue-1 |
| [54] |
Yang J, Wang C, Shu C, et al. Marine sediment bacteria harbor antibiotic resistance genes highly similar to those found in human pathogens[J]. Microbial Ecology, 2013, 65(4): 975-981. DOI:10.1007/s00248-013-0187-2 |
| [55] |
Mata C, Navarro F, Miró E, et al. Prevalence of SXT/R391-like integrative and conjugative elements carrying blaCMY-2 in Proteus mirabilis[J]. Journal of Antimicrobial Chemotherapy, 2011, 66(10): 2266-2270. DOI:10.1093/jac/dkr286 |
 2018, Vol. 45
2018, Vol. 45




