扩展功能
文章信息
- 尹有勤, 马德慧, 李岩松, 蒙小刚, 霍晓伟, 刘锴
- YIN You-Qin, MA De-Hui, LI Yan-Song, MENG Xiao-Gang, HUO Xiao-Wei, LIU Kai
- 鹅源奇异变形杆菌的分离鉴定及毒力基因检测
- Isolation, identification of goose Proteus mirabilis and it's virulence genes detection
- 微生物学通报, 2018, 45(10): 2152-2163
- Microbiology China, 2018, 45(10): 2152-2163
- DOI: 10.13344/j.microbiol.china.170998
-
文章历史
- 收稿日期: 2017-11-26
- 接受日期: 2018-04-20
- 网络首发日期(www.cnki.net): 2018-05-09
2. 内蒙古自治区肉牛疾病防控工程技术研究中心 内蒙古 通辽 028000
2. Inner Mongolia Beef & Cattle Disease Control Center, Tongliao, Inner Mongolia 028000, China
奇异变形杆菌(Proteus mirabilis)是一种人兽共患的机会致病菌,在自然界分布广泛,人和动物携带致病菌的机率高,存在于污水、泥土、粪便当中[1-3]。它可致人和动物的尿道感染,引起尿道炎和肾炎[4]。同时,大量的研究表明,奇异变形杆菌还是引发婴幼儿急、慢性腹泻和成人慢性腹泻的致病菌[5]。截至目前,有大量关于奇异变形杆菌潜在毒力基因的报道,主要包括菌毛、“雾蔓”迁徙能力、尿素酶、金属蛋白酶及溶血素等[6]。关于奇异变形杆菌的研究报告呈逐渐上升趋势,对人的健康安全和畜禽养殖的危害越来越大[7]。
2017年,通辽市某鹅场送检的18只发病和病死雏鹅,发病雏鹅表现为体温升高、精神欠佳、食欲减退、站立不稳、张口呼吸、呼吸急促、咳嗽、打喷嚏、鼻腔流出粘液、排灰白色和浅绿色稀粪。本实验室从送检的病死雏鹅内脏中分离到一株细菌,对其进行了生化鉴定、耐药性分析、人工感染试验、16S rRNA基因序列分析,综合鉴定为有耐药性和致病性的鹅源奇异变形杆菌并将其编号为AYQ-1。目前关于鹅源奇异变形杆菌的研究报道较少,本研究对其进行了部分毒力基因检测分析,了解AYQ-1的毒力基因型,以期对该病原的研究与临床诊断和临床用药提供参考。
1 材料与方法 1.1 材料 1.1.1 样品与试验动物病死雏鹅由通辽市某鹅场送检,55只1周龄的健康雏鹅(购自于某大型雏鹅孵化场)用于人工感染试验,在实验室暂养7 d。
1.1.2 主要试剂和仪器麦康凯琼脂、SS琼脂、酵母浸粉、葡萄糖、氯化钠、蛋白胨、普通营养琼脂,青岛海博生物技术有限责任公司;16S rRNA基因引物和8种毒力基因引物均由吉林省库美生物有限公司合成;无菌脱纤维兔血,上海信裕生物技术有限公司;Rxn Buffer、dNTPs、MgCl2、Taq酶、6×DNA Loading Buffer,Invitrogen公司;JM109、18-T Vector、凝胶回收试剂盒、质粒提取试剂盒、EB终结者、内切酶、异丙基-β-D-硫代吡喃半乳糖苷(IPTG),北京天根生化科技有限公司。电泳仪,Bio-Rad公司;凝胶成像系统,北京原平皓生物技术有限公司;PCR仪,Thermo Fisher Scientific公司;BD PhoenixTM 100全自动微生物鉴定仪、革兰氏阴性菌鉴定板、鉴定肉汤管,BD医疗器械有限公司。
1.1.3 参考菌株从GenBank下载了12株已发表的奇异变形杆菌的atfA、atfC、pmfA、ucaA、ureC、rsbA、zapA、mrpA基因序列,作为参考的对比序列(表 1)。
| Strains | Accession No. | Regions | Year | Hosts | Isolation sources |
| BB2000 | CP004022.1 | America | 2013 | - | - |
| T18 | CP017085.1 | Zhejiang, China | 2017 | Homo sapiens | Urine |
| T12 | CP017082.1 | Zhejiang, China | 2017 | Homo sapiens | Sputum |
| AR_0155 | CP021694.1 | America | 2017 | - | - |
| AR_0159 | CP021550.1 | America | 2017 | - | - |
| AR_0059 | CP020052.1 | America | 2017 | - | - |
| AR_0156 | CP021852.1 | America | 2017 | - | - |
| HI4320 | AM942759.1 | UK | 2008 | - | - |
| CYPV1 | CP012675.1 | Taiwan, China | 2015 | Homo sapiens | Urine |
| CYPM1 | CP012674.1 | Taiwan, China | 2015 | Homo sapiens | Urine |
| AOUC-001 | CP015347.1 | Italy | 2016 | Homo sapiens | Blood |
| Pm7 | HM217133.1 | Shandong, China | 2011 | - | Marine environments |
| 注:-:未找到相关信息. Note: -: No information. | |||||
琼脂浓度为1%的普通营养琼脂平板(g/L):普通营养琼脂33.0,琼脂粉10.0;普通营养琼脂平板(g/L):普通营养琼脂33.0,琼脂粉20.0;麦康凯平板(g/L):麦康凯琼脂52.0,琼脂粉20.0;SS平板(g/L):SS琼脂62.5,琼脂粉20.0;LB液体培养基(g/L):蛋白胨10.0,酵母浸粉5.0,葡萄糖5.0,氯化钠5.0;X-Gal培养基(g/L):普通营养琼脂33.0,琼脂粉10.0,20 μg/L二甲基甲酰胺溶液,238.31 g/L IPTG;Amp LB液体培养基(g/L):蛋白胨10.0,酵母浸粉5.0,葡萄糖5.0,氯化钠5.0;50 μg/L Amp溶液;5%的脱纤维兔血平板(g/L):蛋白胨10.0,酵母浸粉5.0,葡萄糖5.0,氯化钠5.0,琼脂粉20.0,50 mL/L无菌脱纤维兔血。
1.2 方法 1.2.1 病原菌的鉴定(1) 病原菌的分离与培养。无菌采集症状典型的雏鹅肝脏和脾脏,并接种在脱纤维兔血平板中,37 ℃厌氧培养24 h后挑取脱纤维兔血平板上生长的细菌菌落,涂片、革兰氏染色和显微镜检查;再将其接种于普通营养平板进行分离、纯化。
(2) 生长特性观察。挑取1.2.1(1)中在普通营养平板上纯化后的单个菌落接种于麦康凯平板、琼脂浓度为1%的普通营养平板、SS琼脂平板,37 ℃厌氧培养24 h后再观察分离株在各培养基上的生长特性。
(3) 生化鉴定。使用BD PhoenixTM 100全自动微生物鉴定仪对菌株进行生化特性鉴定和药物敏感性测试。用无菌棉签从纯培养过夜的普通营养琼脂平板上挑取单个菌落,在鉴定肉汤管中制成0.6麦氏单位的菌悬液,将制备好的鉴定肉汤管菌悬液倒入革兰氏阴性菌鉴定板中对应的鉴定孔中,封闭。检测板准备完毕后,扫描板条,输入资料,将检测板放入仪器中反应。试验重复2次,反应结束后仪器自动读卡并打印最终的结果。
1.2.2 人工感染试验挑取1.2.1 (1)中纯化后的单菌落于LB液体培养基中,37 ℃、150 r/min恒温培养24 h,用无菌生理盐水10倍稀释法稀释菌悬液并进行细菌计数。取健康的雏鹅55只(7日龄),随机分成6组(5组实验组,1组对照组),实验组每组10只,对照组5只。将菌悬液制成4.75×104-4.75×108 CFU/mL 5个稀释度,每个稀释度肌肉注射1 mL;另外选取5只雏鹅肌肉注射无菌LB培养基作为对照组。每组雏鹅进行常规的隔离饲养,并观察雏鹅的临床症状并记录死亡数量,连续观察5 d,取死亡雏鹅的内脏再次进行细菌的分离、纯化和鉴定,参照熊浩明等方法[8]计算半数致死量(LD50)。
1.2.3 药物敏感性测试采用K-B纸片扩散法,用移液枪无菌吸取培养过夜的新鲜菌悬液于普通营养琼脂平板中,再使用一次性无菌涂布棒将其均匀涂布于整个平板。用无菌镊子取药敏片轻轻贴附于培养基上,试验设立2个平行,37 ℃培养18-24 h后观察并测量抑菌圈直径。试验选用红霉素、氧氟沙星、环丙沙星等多种药物。
1.2.4 16S rRNA基因PCR鉴定(1) 煮沸法提取细菌DNA模板。挑取1.2.1(2)中麦康凯平板上的单个菌落,接种于LB液体培养基中,37 ℃、120 r/min振荡培养过夜。取菌液1 mL于1.5 mL的离心管中,室温12 000 r/min离心5 min后弃去上清。加入1 mL蒸馏水,将菌沉淀重新悬浮,12 000 r/min离心10 min,弃去上清。再次用相同体积的蒸馏水重悬菌沉淀2次,然后加入150 μL TE缓冲液(10 mmol/L Tris-HCl、1 mmol/L EDTA,pH 8.0),100 ℃水浴3 min,10 000 r/min离心2 min后取上清作为DNA模板,-20 ℃或-80 ℃保存DNA提取物[9]。
(2) 16S rRNA基因PCR扩增。PCR扩增反应的总体系为25 μL:ddH2O 15 μL,50 ng/μL DNA模板1 μL,10 mmol/L上、下游引物各1 μL,10×Rxn buffer 2 μL,10 mmol/L dNTPs 1 μL,50 mmol/L MgCl2 2 μL,5 U/μL Taq酶2 μL。PCR扩增条件参照Shukla等[10](94 ℃ 5 min;94 ℃ 30 s,退火温度见表 2,30 s,72 ℃延伸时间见表 2,35个循环;72 ℃ 10 min)。取5 μL PCR产物于含EB终结者的1.5%的琼脂糖凝胶中85 V电泳35 min,后在凝胶成像系统下观察结果。
| 目的基因 Target gene |
引物 Primers |
序列 Primers sequence (5′→3′) |
参考文献 References |
PCR扩增条件 Amplification condition |
| 16S rRNA gene | 27F 1492R | AGAGTTTGATCCTGGCTCAG GGTTACCTTGTTACGACTT | [10] | 94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min |
| ureC | ureC-F ureC-R | GTTATTCGTGATGGGATGGG ATAAAGGTGGTTACGCCAG | [11] | 94 ℃ 5 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| mrpA | mrpA-F mrpA-R | ATTTCAGGAAACAAAAGATG TTCTTACTGATAAGACATTG | [11] | 94 ℃ 5 min;94 ℃ 30 s,39 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| zapA | aapA-F zapA-R | ACCGCAGGAAAACATATAGCCC GCGACTATCTTCCGCATAATCA | [11] | 94 ℃ 5 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| atfC | atfC-F atfC-R | AGAAAGGGATCCTACAAATTAA TATAGCATGCATTTAAATTGCC | [11] | 94 ℃ 5 min;94 ℃ 30 s,49 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| atfA | atfA-F atfA-R | CATAATTTCTAGACCTGCCCTAGCA CTGCTTGGATCCGTAATTTTTAACG | [11] | 94 ℃ 5 min;94 ℃ 30 s,49 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| ucaA | ucaA-F ucaA-R | GTAAAGTTGTTGCGCAAAC TTGAGCCACTGTGGATACA | [11] | 94 ℃ 5 min;94 ℃ 30 s,49 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| pmfA | pmfA-F pmfA-R | GGATCATCTATAATGAAACTG CTGATAATCAACTTGGAAGTT | [11] | 94 ℃ 5 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min |
| rsbA | rsbA-F rsbA-R | TCGATTTCAGTGTTTGGCCAT CCGAGCTTCATCATGGCTG | [11] | 94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 105 s,35个循环;72 ℃ 10 min |
(3) PCR产物胶回收纯化、连接转化。用琼脂糖凝胶回收试剂盒回收纯化PCR产物,具体操作参照说明书进行。回收PCR产物后用18-T载体连接后转化至大肠肝菌JM109感受态中,接菌于X-Gal培养基37 ℃培养18-24 h后,挑取白色阳性菌落接菌于Amp (50 μg/mL)抗性的LB培养基中,培养12-16 h后取菌液用质粒提取试剂盒提取质粒。将所提取的质粒酶切鉴定正确后送至生工生物工程(上海)股份有限公司进行测序。
1.2.5 毒力基因PCR检测(1) 细菌基因组DNA的提取参照1.2.4 (1)中的方法提取。
(2) 毒力基因引物设计与合成。根据已报道的文献[11],设计奇异变形杆菌编码尿素酶的功能亚单位(ureC)、金属蛋白酶的主要功能亚单位(zapA)、MPR菌毛主要结构亚单位(mrpA)、尿道上皮粘附素结构亚单位(ucaA)、“雾蔓”迁徙能力调节因子(rsbA)、Pmf菌毛主要结构亚单位(pmfA)、适温菌毛结构亚单位(atfA)、适温菌毛合成分子引物编码的(atfC)等8种潜在的毒力基因引物,由吉林省库美生物有限公司合成。反应条件及引物信息见表 2,PCR反应体系参照1.2.4(2),PCR产物用含EB终结者的1.5%的琼脂糖凝胶中85 V电泳35 min。
1.2.6 16S rRNA基因与毒力基因序列分析(1) 16S rRNA基因序列分析。测序成功后将测得的序列提交至GenBank进行BLAST比对分析,然后选取得分最高的几条序列利用DNAStar 8.0软件中的MegAlign与所测序列进行相似性比对,并建立系统进化树。
(2) 毒力基因序列分析。将测序所得的8种毒力基因序列利用DNAStar 8.0软件中的MegAlign与表 1中12株参考菌株序列进行相似性比对,并建立系统进化树,再利用MEGA 7.0进行氨基酸序列分析。
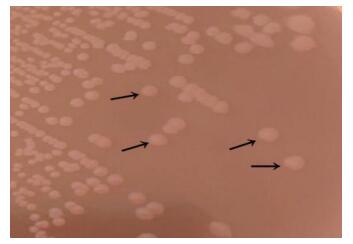
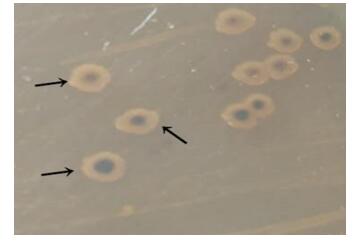
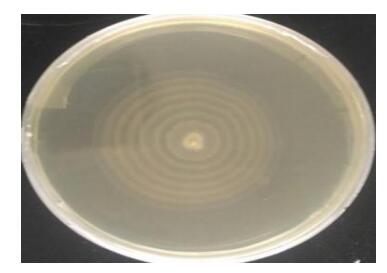
2 结果与分析 2.1 分离株鉴定结果 2.1.1 分离株菌落形态在麦康凯琼脂平板上形成扁平、边缘不整齐、圆形、无色(不发酵乳糖)半透明菌落(图 1);在SS琼脂培养基上37 ℃培养过夜,形成扁平、圆形、中央为黑色,周围不平整且半透明的菌落(产硫化氢) (图 2)。在普通营养琼脂平板上呈辐射性生长(图 3)。
|
| 图 1 AYQ-1在麦康凯培养基上菌落形态 Figure 1 Colony morphology of AYQ-1 on MacConkey culture medium 注:箭头所指为AYQ-1菌落. Note: The arrows refer to colony morphology of AYQ-1. |
|
|
|
| 图 2 AYQ-1在SS培养基上的菌落形态 Figure 2 Colony morphology of AYQ-1 on SS culture medium 注:箭头所指为AYQ-1菌落. Note: The arrows refer to colony morphology of AYQ-1. |
|
|
|
| 图 3 AYQ-1在1%琼脂的普通营养平板上生长情况 Figure 3 Growth situation of AYQ-1 on 1% agar of general nutrition medium |
|
|
显微镜观察可见形状不一的革兰氏阴性菌,其形状多为单个或成对的球状、杆状、球杆状、长杆状的无芽孢菌(图 4)。

|
| 图 4 AYQ-1菌体形态(1 000×) Figure 4 Morphology thallus of AYQ-1 (1 000×) 注:箭头所指为AYQ-1菌体. Note: The arrows refer to thallus of AYQ-1. |
|
|
健康的雏鹅腹腔注射菌株AYQ-1后,实验组雏鹅出现精神不振、站立不稳等症状。每天观察雏鹅出现的症状和人工感染死亡数(表 3),参照熊浩明等[8]方法计算出AYQ-1的LD50为1.51×107 CFU/mL。从死亡雏鹅的内脏内可分离到菌落形态、生化特性、16S rRNA基因序列都与菌株AYQ-1相同的菌株。
| 注射菌含量 Bacterial dose (CFU/mL) |
每日死亡量 Number of goslings dead |
死亡数 Mortality |
||||
| 1 d | 2 d | 3 d | 4 d | 5 d | ||
| 4.75×104 | 0 | 0 | 0 | 0 | 0 | 0 |
| 4.75×105 | 0 | 0 | 0 | 0 | 0 | 0 |
| 4.75×106 | 0 | 0 | 0 | 0 | 3 | 3 |
| 4.75×107 | 0 | 0 | 2 | 3 | 0 | 5 |
| 4.75×108 | 0 | 3 | 5 | 0 | 0 | 8 |
| Control group | 0 | 0 | 0 | 0 | 0 | 0 |
利用BD PhoenixTM 100全自动微生物鉴定仪对AYQ-1进行生化鉴定,AYQ-1被鉴定为奇异变形杆菌(表 4),置信度为99%。
| 生化实验项目 Biochemical tests |
结果 Results |
| BETA-阿洛糖BALL | - |
| BETA-龙胆二糖BGEN | - |
| DEDXTROSE DEX | + |
| D-半乳糖DGAL | + |
| D-果糖DFRU | - |
| D-葡糖酸DGUA | + |
| D-蜜二糖DMLB | - |
| L-阿拉伯糖LARA | - |
| 甲基-B-葡糖苷MBGU | - |
| N-乙酰-氨基半乳糖NGA | - |
| N-乙酰-葡糖胺NGU | + |
| 山梨醇DSBT | - |
| 蔗糖DSUC | - |
| 半乳糖醛酸GRA | - |
| 麦芽酮糖MTU | - |
| L-鼠李糖多粘菌素E LRHA | - |
| D-甘露醇DMNT | - |
| 柠檬酸盐CIT | + |
| 多粘菌素CLST | + |
| L-色氨酸-AMC LTRY | - |
| L-焦谷氨酸-AMC LPYR | - |
| L-脯氨酸-AMC LPORB | - |
| L-精氨酸-AMC LARGH | - |
| 多粘菌素B PXB | + |
| L-苯丙氨酸-AMC LPHET | - |
| 4MU-N-乙酰-BD-氨基葡糖苷NAG | - |
| L-谷氨酸-AMC LGTA | + |
| 鸟氨酸ORN | + |
| L-脯氨酸-NA LPROT | + |
| GAMMA-L-谷氨酰-NA LGGH | + |
| 七叶酸乙酸ESC | - |
| 福寿草醇ADO | - |
| 丙二酸MLO | - |
| ALPHA-酮戊二酸KGA | + |
| 顺芷酸TIG | - |
| 赖氨酸-丙氨酸-AMC LYALD | - |
| 戊二酰-苷氨酸-精氨酸-AMC GUGAH | - |
| 苷氨酸-脯氨酸-AMC GLPRB | - |
| 乙酸ACT | + |
| 苷氨酸-AMC GLYB | - |
| 精氨酸-精氨酸-AMC ARARR | + |
| L-亮氨酸-AMC LLEUH | + |
| 尿素S_URE | + |
| BIS (PNP)磷酸盐BPHO | + |
| PNP-BD=葡糖苷BDGLU | - |
| H2S | + |
| 注:+:阳性;-:阴性.
Note: +: Positive; -: Negative. | |
试验采用了K-B纸片扩散对菌株AYQ-1进行药物敏感性测试,结果显示:菌株AYQ-1对链霉素、氧氟沙星和氨曲南等12种药物高度敏感,对庆大霉素、新霉素和诺氟沙星等7种药物表现为中度敏感,对万古霉素、青霉素和四环素等15种药物耐药(表 5)。
| 抗菌药物 Drugs |
药物浓度 Dosage (μg/disc) |
抑菌圈直径 Inhibition zone diameter (mm) |
敏感性 Sensitivity |
| 红霉素Erythromycin | 15.00 | 0 | R |
| 庆大霉素Gentamicin | 10.00 | 14.00 | I |
| 新霉素Norfloxacin | 30.00 | 14.00 | I |
| 头孢曲松Ceftriaxone | 30.00 | 18.00 | I |
| 诺氟沙星Norfloxacin | 10.00 | 13.50 | I |
| 氯霉素Chloramphenicol | 30.00 | 12.00 | R |
| 哌拉西林Piperacillin | 100.00 | 14.20 | R |
| 丁胺卡那Amikacin | 30.00 | 12.00 | R |
| 青霉素Penicillin | 10U | 0 | R |
| 四环素Achromycin | 30.00 | 0 | R |
| 链霉素Streptomycin | 10.00 | 19.00 | S |
| 氧氟沙星Ofloxacin | 5.00 | 28.00 | S |
| 呋喃妥因Furadantin | 300.00 | 12.30 | R |
| 多西环素Doxycycline | 30.00 | 6.00 | R |
| 万古霉素Vancomycin | 30.00 | 0 | R |
| 氨苄西林Ampicillin | 10.00 | 16.00 | I |
| 卡那霉素Kanamycin | 30.00 | 7.50 | R |
| 头孢拉定Cefradine | 30.00 | 7.00 | R |
| 头孢噻肟Cefotaxime | 30.00 | 25.00 | I |
| 甲氧苄氨嘧啶Trimethoprim | 5.00 | 20.00 | S |
| 氨曲南Aztreonam | 10.00 | 21.00 | S |
| 克拉维酸Clavulanate | 3.00 | 15.00 | S |
| 氨苄青霉素Ampicillin | 10.00 | 18.00 | S |
| 头孢唑啉Cefazolin | 30.00 | 18.00 | S |
| 头孢他啶Ceftazidime | 30.00 | 0 | R |
| 粘菌素Colistin | 300.00 | 15.50 | S |
| 环丙沙星Ciprofloxacin | 5.00 | 16.30 | S |
| 亚胺培南Lmipenem | 10.00 | 16.00 | S |
| 左氧氟沙星Levofloxacin | 5.00 | 18.00 | S |
| 莫西沙星Moxifloxacin | 5.00 | 12.30 | I |
| 美罗培南Meropenem | 10.00 | 17.50 | S |
| 头孢吡肟Cefepime | 30.00 | 8.00 | R |
| 复方新诺Cotrimexazole | 23.00 | 0 | R |
| 阿莫西林Amoxicillin | 20.00 | 6.00 | R |
| 注:R:耐药;I:中等敏感;S:敏感.
Note: R: Resistance; I: Moderate; S: Sensitivity. | |||
16S rRNA基因的PCR产物经1.5%的琼脂糖凝胶电泳后,在凝胶成像系统下观察PCR结果,可在1 000-2 000 bp之间观察到一条带,与试验预期相符,测定的产物大小为1 443 bp (图 5)。

|
| 图 5 16S rRNA基因PCR扩增结果 Figure 5 Results of 16S rRNA gene by PCR Note: M: DNA marker DL2000; 1: Product 16S rRNA gene by PCR. |
|
|
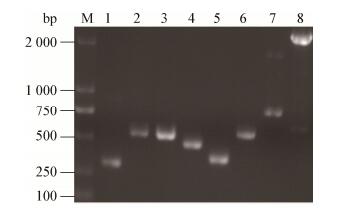
毒力基因用设计的8种毒力基因特异性引物进行PCR扩增,经过琼脂凝胶电泳后,8种毒力基因均被检测到:ureC (292 bp)、mrpA (520 bp)、zapA (510 bp)、atfC (439 bp)、atfA (331 bp)、pmfA (537 bp)、ucaA (764 bp)、rsbA (2 297 bp) (图 6)。
|
| 图 6 8个毒力基因PCR扩增结果 Figure 6 The PCR results of eight kinds of virulence genes 注:M:Marker;1、2、3、4、5、6、7和8分别用ureC、mrpA、zapA、atfC、atfA、pmfA、ucaA、rsbA基因的引物进行扩增所获的PCR产物. Note: M: DNA marker DL2000; 1, 2, 3, 4, 5, 6, 7 and 8:PCR amplification used the primers of ureC, mrpA, zapA, atfC, atfA, pmfA, ucaA and rsbA gene respectively. |
|
|
测序结果在GenBank中进行BLAST比较分析,结果与奇异变形杆菌的相似性达98%以上。选取得分较高的奇异变形杆菌序列进行相似性分析,构建系统发育树(图 7)。结果表明:AYQ-1和10个代表菌株相似性均在98%以上,与CP017085.1 (中国浙江株)和CP017082.1 (中国浙江株)相似性最高,分别为99.7%和99.8%。系统进化树中分离菌株与CP017085.1和CP017082.1处于同一分支,亲缘关系较近,进一步证明分离株AYQ-1为奇异变形杆菌。
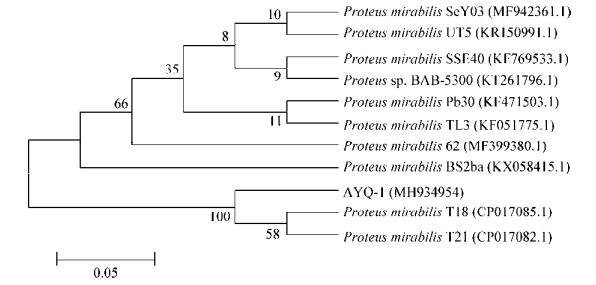
|
| 图 7 菌株AYQ-1的16S rRNA基因系统进化树 Figure 7 Phylogenetic tree based on 16S rRNA gene sequences of strain AYQ-1 注:标尺为序列差异的分支长度;括号内的数字为GenBank序列号;节点处的数字为Bootstrap值;括号内为GenBank数据库的登录号. Note: Bar: Nucleotide divergence; Numbers in parenthesis represented GenBank accession number; Numbers at the branch points indicated the Bootstrap values; Those in parentheses are GenBank accession number. |
|
|
毒力基因的测序结果在NCBI进行对比分析,结果如下:ureC基因与P. mirabilis (CP017085.1和CP017082.1)的相似性达99%;mrpA基因与P. mirabilis (CP021852.1和CP021694.1)的相似性达99%;zapA基因与P. mirabilis (CP021852.1和CP015347.1)的相似性99%;atfC基因与P. mirabilis (CP004022.1和CP026571.1)的相似性达99%;atfA基因与P. mirabilis (CP026051.1和CP026581.1)的相似性达99%;pmfA基因与P. mirabilis (CP026059.1和CP021694.1)的相似性达100%;ucaA基因与P. mirabilis (CP004022.1和CP021852.1)的相似性达99%;rsbA基因与P. mirabilis (CP017085.1和CP017082.1)的相似性达99%。
同源性分析结果表明,菌株AYQ-1的atfA、atfC、mrpA、pmfA、rsbA和ureC基因序列与表 1中的参考菌株中国台湾株CYPV1 (CP012675.1)、中国浙江株T18 (CP017085.1)、中国台湾株CYPM1 (CP012674.1)、英国株HI4320 (AM942759.1)、美国株BB2000 (CP004022.1)对应基因序列的相似性为96.9%-99.7%,遗传进化树处于同一分支,与表 1中其它参考菌株相似性均低于50%;而AYQ-1的ucaA和zapA基因序列与参考菌株美国株AR_0059 (CP020052.1)、美国株AR_0155 (CP021694.1)、意大利株AOUC-001 (CP015347.1)、中国浙江株T12 (CP017082.1)、美国株AR_0159 (CP021550.1)、美国株AR_0156 (CP021852.1)对应基因序列的相似性为97.7%-98.4%,遗传进化树显示也处于同一分支,与表 1中其它参考菌株相似性均低于50%;氨基酸序列分析显示,atfA、atfC、ureC和pmfA基因未检出变异,rsbA基因序列在8位存在碱基C的缺失,其所在位置的遗传密码子CCT编码脯氨酸,导致脯氨酸的缺失;ucaA基因序列在7位存在碱基T的缺失,其所在位置的遗传密码子ACT编码苏氨酸,导致苏氨酸的缺失;zapA基因序列在13位碱基A的缺失,其所在位置的遗传密码子ATA编码异亮氨酸,导致异亮氨酸氨酸的缺失,在4位存在A→G点突变,其所在位置的遗传密码子GAA编码谷氨酸,GGA编码甘氨酸;mrpA基因序列在514、515、516位存在A→C、A→G、T→A点突变,突变所在位置的遗传密码子CAA/TAT分别编码谷氨酰胺和酪氨酸,CAC编码组氨酸,TGA为终止密码子,且在521位存在碱基T的缺失,导致异亮氨酸的缺失。
3 讨论与结论随着畜禽养殖的规模化,高密度的养殖模式导致各种传染疾病频发,奇异变形杆菌病日益严重。奇异变形杆菌可感染猪、鸡、鸭、水貂、羔羊和人[12],是一种人兽共患机会致病菌。但近几年关于鹅感染奇异变形杆菌的报道是空白的,本研究首次报道鹅养殖过程中感染奇异变形杆菌致死的病例,这表明P. mirabilis的宿主范围在扩大,需要引起足够的重视[13]。
本实验从病死的雏鹅体内分离出致病菌,通过细菌形态学观察、动物致病性试验、BD PhoenixTM 100全自动微生物鉴定仪检测,初步鉴定为奇异变形杆菌。再用分子生物学方法测定16S rRNA基因序列,16S rRNA基因具有的高度保守性和特异性是种属鉴定的分子生物学基础[14]。16S rRNA基因序列在NCBI GenBank上进行BLAST在线比对进一步证明分离菌株AYQ-1为P. mirabilis,同源性比对和系统进化树结果显示菌株AYQ-1与人尿源CP017085.1 (中国浙江株)的相似性为99.8%,遗传进化树处于同一分支,说明其亲缘关系较近。选择10株来源不同的奇异变形杆菌菌株,来自人、动物和环境中,其中KR150991.1株与CP017082.1的来源相同,但地理位置不同,与分离株AYQ-1的相似性差别较大;KF051775.1和MF942361.1与分离株AYQ-1的地理位置相差不大,但其来源不同,与分离株AYQ-1的相似性也不高。这表明奇异变形杆菌的亲属亲缘关系与地理位置分布和种属之间有一定的关系。
本次试验选取测试的药物并非均适用于养殖,其中氯霉素、万古霉素、呋喃妥因和头孢类等药物已被农业部列为禁药,结合养殖场所常用的药物,推荐使用甲氧苄氨嘧啶、链霉素、氨苄青霉素和环丙沙星对鹅奇异变形杆菌病进行治疗。在本次试验中AYQ-1表现耐受的药物占总药物的44%,这说明AYQ-1对多数药物产生了抗药性。奇异变形杆菌产生β-内酰胺酶[15-17],分布于世界各地且类型众多,包括超广谱β-内酰胺酶(ESBLs)、头孢菌素酶(AmpC)、金属β-内酰胺酶和碳青霉烯酶[18-19]。近年来,奇异变形杆菌对第三代头孢菌素的耐药性[20]不断攀升,产ESBLs和AmpC是奇异变形杆菌对第三代头孢菌素耐药的最重要机制。尤其是一些养殖场长期不合理地使用抗生素,使得细菌的耐药性变得更加严重[21],同时也增加了治疗的难度。因此,对于该病的预防和治疗显得尤为重要,当发生感染时,应当依据药敏试验筛选出来的敏感药物进行治疗[22]。
杨巧玲等[11]对32株犬腹泻源性的奇异变形杆菌进行了8种潜在毒力基因检测,结果显示32株奇异变形杆菌均含有atfA、atfC、mrpA、pmfA、ureC和zapA基因,27株菌含有rsbA基因,25株菌含有ucaA基因。从GenBank上下载了11株参考菌株的atfA、mrpA、rsbA、pmfA、ucaA、atfC和ureC基因序列,下载了12株参考菌株的zapA基因序列,参考菌株的对应毒力基因间的相似性在39.8%-100%,遗传进化树都分为2个大分支,这表明参考菌株间存在一定的差异性,这可能与不同的血清型和亚型群有关。8种毒力基因分别与参考菌株对应毒力基因之间存在较大的差异,说明这8种毒力基因保守度不高,不可作为奇异变形杆菌PCR检测的特异性引物。氨基酸序列分析显示,AYQ-1的atfA、atfC、ureC和pmfA基因未检出变异,rsbA、ucaA、zapA和mrpA基因存在少部分碱基点突变和碱基缺失,与部分参考菌株相似性较高,表明从通辽地区雏鹅脏器分离到的AYQ-1的这几个毒力基因没有发生大的变异。对病原菌毒力特征的掌握可为其致病性、传播性提供基础[23],可为进一步研究核酸疫苗、亚单位疫苗和快速检测试剂盒的开发奠定基础,为该菌的快速检测和防治奠定基础[24]。本次试验从病原菌中检测出P. mirabilis的8种潜在毒力基因,对该菌毒力基因的携带情况有了进一步的了解。
雏鹅感染奇异变形杆菌发病后,虽然经过及时治疗控制了疫情,后续没有再发生死亡,但是会影响雏鹅后期的生长发育,如果诊断治疗不及时就会造成雏鹅的死亡,将会造成巨大的经济损失。奇异变形杆菌为条件致病菌,当动物机体抵抗力下降,尤其发生其他疾病时更易继发感染或混合感染,且该菌血清型多达340-360个[25],免疫预防难度较大。加强对该病原菌引起动物的临床症状、防治技术和免疫原性的研究十分重要,而本研究为鹅奇异变形杆菌感染的临床诊断、防治提供了一定的基础数据。
| [1] |
Lee CC, Wang JL, Lee CH, et al. Clinical benefits of antimicrobial de-escalation in adults with community-onset monomicrobial Escherichia coli, Klebsiella species and Proteus mirabilis bacteremia[J]. International Journal of Antimicrobial Agents, 2017, 50(3): 371-376. DOI:10.1016/j.ijantimicag.2017.03.024 |
| [2] |
Zhang WW, Niu ZL, Yin K, et al. Quick identification and quantification of Proteus mirabilis by polymerase chain reaction (PCR) assays[J]. Annals of Microbiology, 2013, 63(2): 683-689. DOI:10.1007/s13213-012-0520-x |
| [3] |
Wang HJ, Ni L, Yang HJ, et al. Isolation and identification of Proteus mirabilis from goat and the analysis of 16S rDNA and ZapA gene by PCR-RFLP[J]. Chinese Journal of Veterinary Science, 2011, 31(11): 1594-1598. (in Chinese) 王豪举, 倪莉, 杨红军, 等. 山羊奇异变形杆菌的分离鉴定及16S rDNA和ZapA基因的PCR-RFLP分析[J]. 中国兽医学报, 2011, 31(11): 1594-1598. |
| [4] |
Li XN, Xia X, Li YC, et al. The research progress of Proteus mirabilis animal origins[J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2011(12): 73-75. (in Chinese) 李欣南, 夏欣, 李永才, 等. 奇异变形杆菌研究进展[J]. 现代畜牧兽医, 2011(12): 73-75. DOI:10.3969/j.issn.1672-9692.2011.12.051 |
| [5] |
Christopoulos G, Christopoulou V, Routsias JG, et al. Greek rheumatoid arthritis patients have elevated levels of antibodies against antigens from Proteus mirabilis[J]. Clinical Rheumatology, 2017, 36(3): 527-535. DOI:10.1007/s10067-016-3441-4 |
| [6] |
Senior BW, Anderson GA, Morley KD, et al. Evidence that patients with rheumatoid arthritis have asymptomatic 'non-significant' Proteus mirabilis bacteriuria more frequently than healthy controls[J]. The Journal of Infection, 1999, 38(2): 99-106. DOI:10.1016/S0163-4453(99)90076-2 |
| [7] |
Barbour EK, Hajj ZG, Hamadeh S, et al. Comparison of phenotypic and virulence genes characteristics in human and chicken isolates of Proteus mirabilis[J]. Pathogens and Global Health, 2012, 106(6): 352-357. DOI:10.1179/2047773212Y.0000000042 |
| [8] |
Xiong HM, Wei BQ, Wei RJ, et al. Calculation of median lethal dose (LD50) for Yersinia pestis by SPSS package[J]. Chinese Journal of Zoonoses, 2013, 29(11): 1127-1130. (in Chinese) 熊浩明, 魏柏青, 魏荣杰, 等. 用SPSS软件计算鼠疫菌半数致死量(LD50)[J]. 中国人兽共患病学报, 2013, 29(11): 1127-1130. DOI:10.3969/cjz.j.issn.1002-2694.2013.11.020 |
| [9] |
Xia LX, Sun WJ, Shen Z, et al. Comparison of genomic DNA extraction from bioleaching acidophiles by two methods: Boiling and kit methods[J]. Progress in Modern Biomedicine, 2014, 14(1): 31-35, 17. (in Chinese) 夏乐先, 孙文娟, 沈振, 等. 煮沸裂解法和试剂盒法提取浸矿菌基因组DNA的比较[J]. 现代生物医学进展, 2014, 14(1): 31-35, 17. |
| [10] |
Shukla J. 16S rRNA PCR for differentiation of pathogenic and non-pathogenic leptospira isolates[J]. Indian Journal of Medical Microbiology, 2003, 21(1): 25-30. |
| [11] |
Yang QL, Gao J, Luo LP, et al. Virulence tests of canine Proteus mirabilis[J]. Chinese Journal of Veterinary Medicine, 2013, 49(8): 60-63. (in Chinese) 杨巧玲, 高健, 罗立平, 等. 犬源性奇异变形杆菌的毒力试验[J]. 中国兽医杂志, 2013, 49(8): 60-63. DOI:10.3969/j.issn.0529-6005.2013.08.023 |
| [12] |
Kaca W, Arabski M, Fudala R, et al. Human complement activation by smooth and rough Proteus mirabilis lipopolysaccharides[J]. Archivum Immunologiae et Therapiae Experimentalis, 2009, 57(5): 383-391. DOI:10.1007/s00005-009-0043-8 |
| [13] |
Zhang XZ, Zhang ZX, Chang CY, et al. Isolation, identification and phylogenetic analysis of Proteus mirablilis from raccoon dog[J]. Heilongjiang Animal Science and Veterinary Medicine, 2016(12): 193-195. (in Chinese) 张香斋, 张召兴, 常超越, 等. 貉源奇异变形杆菌分离鉴定及系统发育分析[J]. 黑龙江畜牧兽医, 2016(12): 193-195. |
| [14] |
Zhu MH, Zhu RL, Ma RD, et al. Isolation, identification of chicken Proteus mirabilis and phylogenetic analysis of 16S rRNA gene sequence[J]. Chinese Journal of Veterinary Science, 2011, 31(6): 804-808. (in Chinese) 朱明华, 朱瑞良, 马荣德, 等. 鸡奇异变形杆菌的分离鉴定和16S rRNA基因序列测定与系统进化分析[J]. 中国兽医学报, 2011, 31(6): 804-808. |
| [15] |
Liu YQ. Analysis of resistance to antibiotics and detection of inducible β-lactamase produced in Proteus mirabilis[J]. Chinese Journal of Microecology, 2015, 27(10): 1195-1198. (in Chinese) 刘英其. 奇异变形杆菌β-内酰胺酶检测及耐药性分析[J]. 中国微生态学杂志, 2015, 27(10): 1195-1198. |
| [16] |
Lee CC, Lee CH, Hong MY, et al. Propensity-matched analysis of the impact of extended-spectrum β-lactamase production on adults with community-onset Escherichia coli, Klebsiella species, and Proteus mirabilis bacteremia[J]. Journal of Microbiology, Immunology and Infection, 2017. DOI:10.1016/j.jmii.2017.05.006 |
| [17] |
Li WJ, Liu YH, Zhou CL, et al. Distribution and drug resistance of Proteus mirabilis[J]. Chinese Journal of Nosocomiology, 2017, 27(12): 2660-2663. (in Chinese) 李文静, 刘晔华, 周春雷, 等. 奇异变形菌分布与耐药性分析[J]. 中华医院感染学杂志, 2017, 27(12): 2660-2663. |
| [18] |
Su GJ, Wang GQ. Detection of ESBLs and AmpC β-lactamase producing Enterobacter cloacae and Proteus mirabilis and analysis of drug resistance[J]. Chinese Journal of Laboratory Diagnosis, 2015, 19(5): 719-722. (in Chinese) 苏国娟, 王国庆. 阴沟肠杆菌、奇异变形杆菌AmpC酶和ESBLs的检测及其耐药性研究[J]. 中国实验诊断学, 2015, 19(5): 719-722. |
| [19] |
Sun L. Study on the biological characters, molecular typing and carbapenem resistant mechanisms of Proteus mirabilis deficient in swarming motility[D]. Hangzhou: Master's Thesis of Zhejiang University, 2016 (in Chinese) 孙龙.无迁徙生长碳青霉烯耐药奇异变形杆菌生物学特性、耐药机制及分子分型研究[D].杭州: 浙江大学硕士学位论文, 2016 |
| [20] |
Ying CM, He C, Wang YP, et al. The genotypes of extended-spectrum β-lactamases in 119 clinical isolates of Proteus mirabilis[J]. Chinese Journal of Infection and Chemotherapy, 2009, 9(5): 343-346. (in Chinese) 应春妹, 何澄, 汪雅萍, 等. 奇异变形杆菌产超广谱β内酰胺酶基因检测[J]. 中国感染与化疗杂志, 2009, 9(5): 343-346. DOI:10.3321/j.issn:1009-7708.2009.05.005 |
| [21] |
Tao SL. Study on the resistance and molecular epidemiology in P. mirablis isolates[D]. Hefei: Master's Thesis of Anhui Medical University, 2012 (in Chinese) 陶胜来.临床分离奇异变形杆菌的耐药性及分子流行病学研究[D].合肥: 安徽医科大学硕士学位论文, 2012 http://cdmd.cnki.com.cn/Article/CDMD-10366-1012468146.htm |
| [22] |
Wu XR, Zhou H, Yang Q, et al. Resistance of Proteus mirabilis and clinical features of infection by imipenen non-susceptible strains[J]. Chinese Journal of Microecology, 2014, 26(9): 1035-1039. (in Chinese) 吴晓茹, 周华, 杨青, 等. 奇异变形杆菌耐药性及亚胺培南不敏感株感染的临床特点研究[J]. 中国微生态学杂志, 2014, 26(9): 1035-1039. |
| [23] |
Cheng JM, Wan MY, Zhou JY, et al. Identification and virulence genes detection of Edwardsiella tarda isolated from Carassius auratus gibelio[J]. Microbiology China, 2017, 44(10): 2380-2390. (in Chinese) 程俊茗, 万明月, 周晋扬, 等. 鲫源迟缓爱德华氏菌的分离鉴定及其毒力基因的检测[J]. 微生物学通报, 2017, 44(10): 2380-2390. |
| [24] |
Cui GL. Phyletic evolution analysis, distribution regularity in chick and the subunit vaccine development of Proteus mirabilis[D]. Tai'an: Master's Thesis of Shandong Agricultural University, 2013 (in Chinese) 崔国林.奇异变形杆菌的系统发育分析、在雏鸡体内分布规律及亚单位疫苗研制[D].泰安: 山东农业大学硕士学位论文, 2013 |
| [25] |
Kaca W, Glenska J, Lechowicz L, et al. Serotyping of Proteus mirabilis clinical strains based on lipopolysaccharide O-polysaccharide and core oligosaccharide structures[J]. Biochemistry (Moscow), 2011, 76(7): 851-861. DOI:10.1134/S0006297911070169 |
 2018, Vol. 45
2018, Vol. 45




