扩展功能
文章信息
- 郑福成, 黄轶群, 潘裕添, 欧一新
- ZHENG Fu-Cheng, HUANG Yi-Qun, PAN Yu-Tian, OU Yi-Xin
- 放线菌来源的糖肽类抗生素的药学机制研究进展
- Advances in pharmacological mechanism of glycopeptide antibiotics from actinomycetes
- 微生物学通报, 2018, 45(1): 197-206
- Microbiology China, 2018, 45(1): 197-206
- DOI: 10.13344/j.microbiol.china.160757
-
文章历史
- 收稿日期: 2017-01-20
- 接受日期: 2017-09-19
- 优先数字出版日期(www.cnki.net): 2017-09-26
2. 闽南师范大学生物科学与技术学院 菌物产业工程技术中心 福建 漳州 363000
2. Engineering Technological Center of Mushroom Industry, School of Biological Science and Biotechnology, Minnan Normal University, Zhangzhou, Fujian 363000, China
以万古霉素(Vancomycin)和替考拉宁(Teicoplanin)为代表的天然第一代糖肽类抗生素均是从放线菌发酵产物中被发现的(图 1),由于它们可以特异性地抑制细菌细胞壁的合成,几乎对所有的革兰氏阳性细菌感染都表现出较好的治疗作用[1]。这两个糖肽类抗生素在临床上的应用,大大缓解了人们治疗一些顽固性革兰氏阳性细菌感染疾病的压力,被称为“人类对抗顽固性耐药菌的最后一道防线”。但继1988年在欧洲发现第一株耐受万古霉素的粪肠球菌以来[2],临床上多耐药性、高耐药性病原菌及新型感染性疾病不断出现,迫使世界各国研究者不断利用新技术、新方法,快速改造和发现新的药源分子来对抗顽固性病原菌感染。随着对糖肽类抗生素研究的不断深入,许多研究者利用化学方法对天然来源的糖肽类抗生素的结构进行修饰,产生了第二代糖肽类抗生素[3],为糖肽类抗生素的临床应用拓展了新的机遇。此外,随着糖肽类抗生素生物合成机制的深入解析,为进一步利用组合生物合成和合成生物学技术实现糖肽类抗生素的定向创制奠定了基础。

|
| 图 1 糖肽类抗生素的结构特征 Figure 1 The chemical structures of glycopeptides |
|
|
天然糖肽类抗生素的多肽骨架是由7个氨基酸通过肽键相连,多以同系物的形式出现(如万古霉素)。其中,有5个保守的芳香氨基酸残基几乎存在于每一个糖肽类抗生素的骨架中,而且糖肽类抗生素生物合成后修饰的模式较为统一,在七肽骨架的第4个芳香氨基酸与第2个和第6个芳香氨基酸,分别通过两个二苯基醚键氧桥相连。同时,第5个芳香氨基酸残基与第7个芳香氨基酸残基间通过特异性的C-C键形成分子内的环形结构(图 1)[4]。糖肽类抗生素的结构差异主要由肽链骨架上氨基酸残基的修饰类型、数量和不同的取代位置所造成。另外,在糖肽类抗生素芳香氨基酸残基上还存在着多样化的糖基修饰,一些糖基的位置还被疏水的脂肪链取代(如:替考拉宁[5])。糖肽类抗生素的氯代修饰对该类化合物的生物活性的贡献也较大。
1.2 天然糖肽类抗生素的结构与活性的关系糖肽类抗生素氨基酸残基的不同修饰对终产物的生理活性具有重要的影响[6]。一般来说,万古霉素类的第1位亮氨基酸的去除会导致生物活性的丧失,氨基酸氮上的甲基化以及第3位天冬酰胺的甲基化均会导致活性的下降。第2位的芳香氨基酸氯代以后可以增强抗生素的生理活性。第4位芳香氨基酸的对位羟基通常连接着二糖的取代,第1个糖为葡萄糖,第2个糖为L-万古糖胺或4-L-表-万古糖胺。第2个氨基糖的取代与否会导致药物在体内的活性下降5倍左右,该氨基糖上的烷基化修饰可以增加药物的疏水性,提高药物的活性以及较长的保留时间。第7位氨基酸羧基端酰胺化修饰后会增加糖肽类抗生素对抗葡萄球菌的活力。第6位的芳香氨基酸上氯代的存在对化合物生理活性的贡献不是太大,但是在其羟基位置上的糖基取代会提高化合物的生理活性[7]。
1.3 第二代半合成糖肽类抗生素的结构特征与药效活性的关系糖肽类抗生素的化学修饰大大改善了天然糖肽类抗生素的结构缺点,通过化学方法增强糖肽类抗生素的生理活性以及延长药效的修饰方法主要遵循以下两种原则:一是在不直接接触结合口袋的位点,以及在一些容易接近的功能区域进行改造,主要包括了糖基的替代、改造和移除,或者在第7位芳香氨基酸游离羧基上进行修饰;二是直接改造化合物的修饰区域,该方面的改造容易导致结合口袋的破坏而丧失抗菌活性[6, 8]。例如以达巴万星、奥利万星和特拉万星为代表的第二代糖肽类抗生素,在临床的应用中表现出了较天然结构更加优越的药学特征。
在天然糖肽类化合物A40926结构中的C-端羧基位置,通过化学方法引入3-(二甲氨基)-1-丙胺获得了达巴万星。该抗生素主要由Pfizer公司主导开发,与天然糖肽类抗生素的活性机制类似,但生物活性明显增强,且对万古霉素耐受的VanA型肠球菌的生理活性不是太强。达巴万星的药物代谢动力学研究结果表明,该药物的代谢及排泄可能比较缓慢,该药物最常见的不良反应主要为胃肠道症状[9-10]。
奥利万星是在天然糖肽类抗生素A82846B的4-氨基酸二糖的氨基上加载了4′-氯联苯基甲基的结构衍生物,主要作用机制与万古霉素和替考拉宁相似,而且还可以抑制细菌RNA的合成。糖基上修饰侧链基团的引入还促进了该药物与靶向菌株细胞膜的锚定,增强了抗菌活性,但不足之处是该药物的抗菌能力与给药浓度具有较强的相关性。药物代谢动力学研究发现,该药物在体内的半衰期较长,对万古霉素耐受的VanA型肠球菌也具有较好的抑制活性,在治疗皮肤和软组织感染上已经取得了良好的效果,具有较好的安全性和耐受性[11-12]。
特拉万星最初是由Theravance公司以万古霉素为结构基础,通过化学修饰在糖基的氨基上引入脂肪链,并在第7个芳香氨基酸引入磷酸甲胺甲基获得,该药物属于脂糖肽类,为快速杀菌的注射用抗生素。该药物除了与万古霉素有类似的作用机制外,还可以直接作用于细菌的细胞膜,引起细菌细胞膜电位的快速去极化并增加细胞膜的通透性,快速发挥其抗菌的功能。药物代谢动力学研究结果显示,该药物在人体内无蓄积,给药剂量2/3的药物以原始结构从肾脏排泄。特拉万星不良反应较轻,主要表现在味觉改变、呕吐和瘙痒等,一般在停药后的短期内可恢复正常[13]。
2 糖肽类抗生素对抗微生物感染的机理革兰氏阳性细菌细胞壁的组成主要由肽聚糖骨架、四肽侧链和五肽交联桥组成。糖肽类家族的抗生素通过抑制病原菌细胞壁的生物合成来显示其杀菌和抑菌的能力,其作用机制主要表现在以下两个方面。
一方面,万古霉素家族的糖肽类抗生素特异性的与细菌细胞壁合成中五肽末端的D-丙氨酰-D-丙氨酸残基结合,进而阻碍细菌细胞壁合成过程中的转糖基或(和)转肽作用,阻止细菌细胞壁生物合成的顺利进行,最终导致细菌无法生长或死亡(图 2) [14]。糖肽类抗生素与D-丙氨酰-D-丙氨酸残基结合需要肽聚糖末端的D-丙氨酸的羧基端为游离的状态,而且五肽中的第3个氨基酸为乙酰-L-氨基酸时结合能力较强[15]。

|
| 图 2 糖肽类抗生素抑制细菌细胞壁合成的机理及肽聚糖的生物合成过程 Figure 2 The bioactive mechanism of glycopeptide and the biosynthesis of peptidoglycan 注:DdI:羧肽酶;D-Ala:D-丙氨酸连接酶;MurF:合成酶;UDP:二磷酸尿嘧啶. Note: DdI: D, D-carboxypeptidase; D-Ala: D-Ala ligase; MurF: a synthetase protein; UDP: uracil diphosphate. |
|
|
另一方面,在革兰氏阳性细菌细胞壁生物合成的最后阶段,双糖肽插在细胞外的细胞壁生长点中,交联后形成肽聚糖。肽聚糖类抗生素可以与细菌细胞膜上的转糖-转肽酶结合,从而抑制细胞壁合成过程中糖基转移酶、肽基转移酶和羧肽酶所催化的肽聚糖之间的交联,破坏了细胞壁的有效合成,从而实现杀菌和抑菌的功能(图 2)[15]。
3 细菌对糖肽类抗生素的耐药机制根据耐药菌株的抗性基因编码的蛋白对细胞壁合成过程中五肽末端修饰结构的不同,其抗性机制主要分为D-Ala-D-Lac[16-17](包括VanA、VanB、VanD和VanM)和D-Ala-D-Ser[18-19](包括VanC、VanE、VanG、VanL和VanN)两种主要类型[20]。
3.1 细菌细胞壁五肽末端修饰为D-Ala-D-Lac类型的糖肽类抗生素耐受机制在糖肽类抗生素的产生菌及许多临床的耐受菌株中,VanA类型的耐受基因簇中具有一套保守的vanHAX基因开放阅读框和vanRS双组分调控系统[21](图 3)。其中,vanH编码了D-乳酸脱氢酶(Lactate dehydrogenase,LDH),催化了丙酮酸(Pyruvate)和D-乳酸(D-lactate)之间的相互转化[22];vanX所编码的基因为D, D-二肽酶(Dipeptidase),负责水解D-Ala-D-Ala底物,在vanY基因编码的羧肽酶VanY的协助下,将细胞壁合成过程中的五肽最末端的D-Ala残基进行水解[23];vanA编码了D-Ala-D-Lac连接酶(Ligase),催化D-Ala和D-Lac之间酯键的形成。最终,在4个蛋白的共同作用下,将细菌细胞壁合成过程中的五肽末端修饰为D-Ala-D-Lac[24](图 3)。VanS的N末端具有细胞膜跨膜结构域,C末端为组氨酸(His)激酶结构域,N末端主要负责感受细胞膜外糖肽类抗生素的存在情况,当感受到细胞外有糖肽类抗生素存在时,开始启动其C-端His的磷酸化。VanR是一个转录激活因子,N端负责接受VanS感知的糖肽类抗生素的信号,C端是效应子,负责激活相关抗性基因的快速表达,开启细胞壁生物合成过程中五肽末端的修饰[25]。
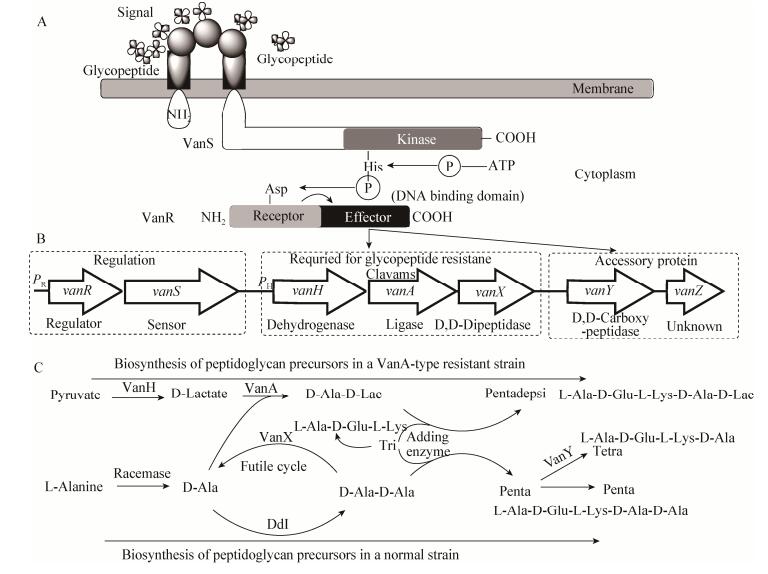
|
| 图 3 细菌VanA类型耐受糖肽类抗生素的机制 Figure 3 VanA-type glycopeptide resistance model 注:A:VanA类型耐受糖肽类抗生素系统的信号传导通路;B:vanA类型的基因组装特征;C:VanA抗性相关蛋白对于细胞壁的修饰过程. Note: A: signal transduction pathway of VanA-type glycopeptide resistance; B: Organization of vanA-type operon; C: the biosynthetic modification of cell-wall by VanA-type proteins. |
|
|
该类型的抗性机制以VanC类型最具有代表性[26],其抗性基因主要由vanC、vanXYc和vanT组成,vanT编码了丝氨酸消旋酶(Serine racemase),催化L-丝氨酸转化为D-丝氨酸;vanXYc编码的二肽酶功能与羧肽酶VanY的功能类似,将细胞壁合成过程中的五肽最末端的D-Ala-D-Ala残基末端的D-Ala进行水解;vanC编码的蛋白为D-Ala-D-Ser连接酶(图 4)。最后,在这3个蛋白的协调作用下,将细胞壁合成过程中正常的D-Ala-D-Ala残基末端修饰为D-Ala-D-Ser[27],从而避免了糖肽类抗生素对细胞壁合成过程的干扰。
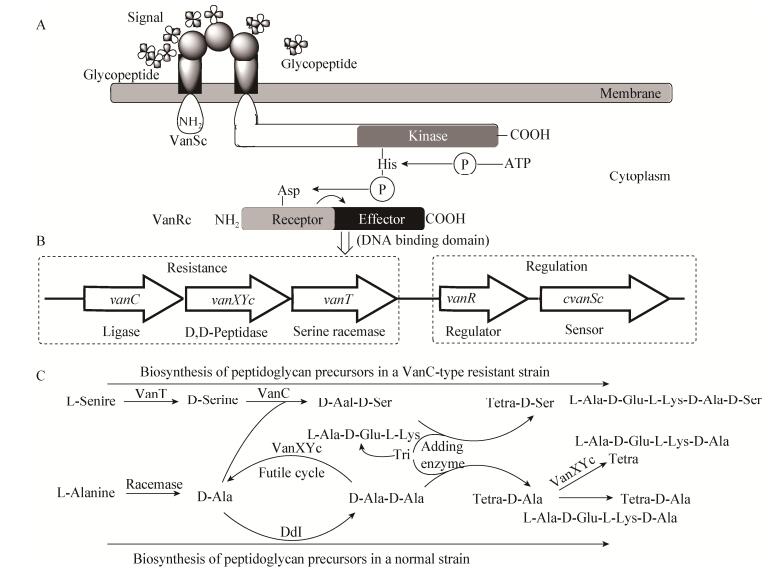
|
| 图 4 细菌VanC类型耐受糖肽类抗生素的机制 Figure 4 VanC-type glycopeptide resistance model 注:A:VanC类型耐受糖肽类抗生素系统的信号传导通路;B:vanC类型的基因组装特征;C:VanC抗性相关蛋白对于细胞壁的修饰过程. Note: A: signal transduction pathway of VanC-type glycopeptide resistance, B: Organization of vanC-type operon, C: the biosynthetic modification of cell-wall by VanC-type proteins. |
|
|
与VanA类型的糖肽类抗生素的抗性基因簇不同的是,VanC的双组分调控系统vanRcSc位于VanC类型抗性基因簇的下游。同样,vanSc负责细胞外糖肽类抗生素的感知,当有相关的糖肽类抗生素存在时,VanSc将C末端的His位点进行磷酸化。然后,VanRc的N末端负责接收VanSc将C末端传输过来的信号,从而开启C端的效应子功能,激活相关抗性基因的表达,开始对细胞壁生物合成过程中五肽末端的修饰,从而使得菌株获得抗性[28](图 4)。
4 糖肽类抗生素生物合成机制与新结构定向衍生化创制糖肽类抗生素聚肽骨架的较高一致性表明了其生物合成具有较高的相似性,糖肽类抗生素聚肽骨架的形成是通过非核糖体肽类合成酶(Non-ribosomal peptide synthase,NRPS)催化形成,共含有7个聚肽模块,每一个模块负责催化一个氨基酸的加载形成一个肽键[29]。
每个模块主要有腺苷化结构域(Adenylation domain,A)、缩合结构域(Condensation domain,C)和肽酰载体蛋白结构域(Peptidyl carrier protein domain,PCP)组成。此外,在一些聚肽延伸模块(如:在替考拉宁的第2、4和5的3个聚肽延伸模块)中还含有氨基酸的异构化结构域(Epimerization domain,E),负责将连接在肽酰载体蛋白结构域(PCP)上的氨基酸残基由L构型转变为D构型。聚肽末端中还含有一个X结构域,与PCP共同作用来招募负责聚肽骨架氨基酸侧链的修饰[30](图 5)。最后,聚肽末端的硫酯酶结构域(Thioesterase,TE)对最终的产物进行环化成肽键和释放,形成糖肽类抗生素的苷元。聚肽延伸过程中的特殊氨基酸残基Dpg是以三型聚酮途径催化形成,Hpg是来自酪氨酸的降解途径[31]。
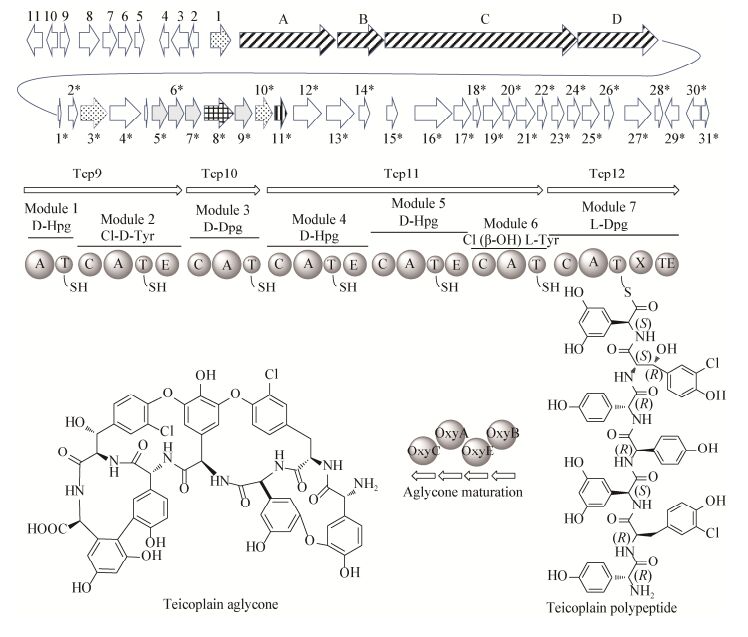
|
| 图 5 替考拉宁的生物合成基因簇和生物合成机制 Figure 5 The biosynthetic gene cluster and biosynthetic pathway of teicoplain |
|
|
研究发现糖肽类抗生素肽链骨架中的氨基酸侧链交联的聚肽母核成熟过程需要四步P450催化的反应。在整个催化过程中,经过聚肽模块催化延伸完成的肽链连接在PCP上,X-结构域和PCP共同负责招募OxyB、OxyE、OxyA和OxyC四个氧化酶,分别形成C-O-D环、D-O-E环、F-O-G环和A-B环的交联反应,形成刚性分子结构(图 6)。最后,在糖基化、甲基化和其他的后修饰酶的作用下,分别形成最终的结构[30, 32-33]。

|
| 图 6 替考拉宁生物合成过程中氧化环化的顺序及过程 Figure 6 Order of cyclization reactions and enzymes involved during the biosynthesis of teicoplain |
|
|
随着微生物研究方法的快速更新和抗生素生物合成研究的不断深入,利用合成生物学研究思路和方法,设计和建造新的基因部件和系统来合成新结构或新活性衍生物成为了新的研究热点。糖肽类抗生素具有相似性较高的聚肽骨架,多样化的氯代、甲基化、糖基化和酰基化等修饰(图 7),为糖肽类抗生素定向化的结构修饰提供了丰富的结构修饰位点和基因元件[34-36]。
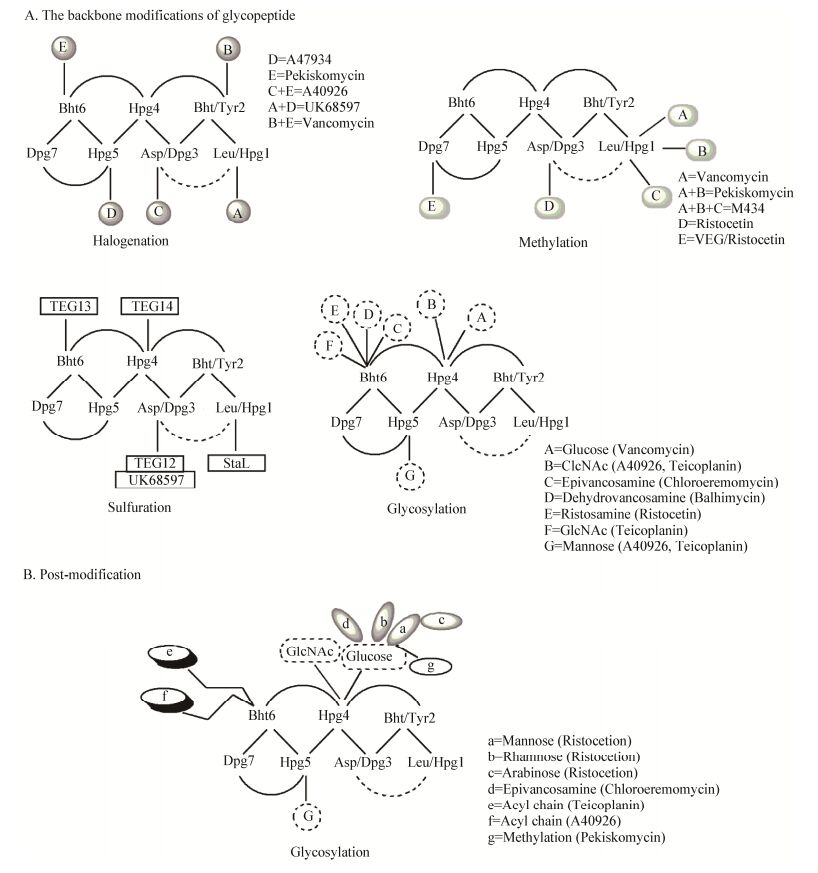
|
| 图 7 糖肽类抗生素多样化的结构修饰位点 Figure 7 The diversity of GPA modifications offers tremendous opportunity for chemical diversification |
|
|
糖肽类抗生素在治疗微生物感染疾病中具有不可替代的地位,其生物活性和病原菌的耐受机制的深入研究,为糖肽类抗生素的合理用药和分子结构优化提供了坚实的保障。但是,糖肽类抗生素抗性基因的系统发掘以及在致病菌间水平转移的机制、评测以及如何控制,依然是糖肽类抗生素耐药机制研究需要解决的问题。同时,在临床使用糖肽类抗生素对抗病原菌感染的同时,势必对人体的有益菌群造成巨大干扰,实时监控和快速恢复给药患者体内的益生菌,保证患者的健康依然是需要探索的研究内容。
在糖肽类抗生素新药源发现方面,伴随着微生物药物生物合成及合成生物学技术的飞速发展,利用微生物遗传改造和合成生物学策略对糖肽类抗生素的改造越来越受到关注。接下来,以糖肽类抗生素的活性和病原菌耐受机理为基础,利用计算机辅助的药物设计技术为指导,集成微生物遗传学改造、合成生物技术和有机化学技术,来快速实现糖肽类抗生素结构多样化衍生创制和筛选生理活性优异的抗生素分子,是糖肽类抗生素新药研发的重要研究策略。
| [1] |
Butler MS, Hansford KA, Blaskovich MA, et al. Glycopeptide antibiotics: back to the future[J]. The Journal of Antibiotics, 2014, 67(9): 631-644. DOI:10.1038/ja.2014.111 |
| [2] |
Li G, Hou S, Li Y, et al. Surveillance of gram-positive cocci infections and drug resistance[J]. Cellular and Molecular Biology (Noisy-le-Grand, France), 2015, 61(4): 90-93. |
| [3] |
Tie Y, Liu L. Progress in research of the new lipoglycopeptide antimicrobial agents dalbavancin, oritavancin and telavancin[J]. Chinese Journal of New Drugs, 2016, 22: 2542-2549. |
| [4] |
Xie J, Okano A, Pierce JG, et al. Total synthesis of [Ψ[C(═S)NH]Tpg4]vancomycin aglycon, [Ψ[C(═NH)NH]Tpg4] vancomycin aglycon, and related key compounds: reengineering vancomycin for dual D-Ala-D-Ala and D-Ala-D-Lac binding[J]. Journal of the American Chemical Society, 2012, 134(2): 1284-1297. DOI:10.1021/ja209937s |
| [5] |
López-Sánchez C, Rodríguez-Pardo D, Pigrau C, et al. Teicoplanin-an old new treatment for enterococcal prosthetic joint infections[J]. The Journal of Infection, 2016, 72(6): 764-766. DOI:10.1016/j.jinf.2016.03.002 |
| [6] |
Jeya M, Moon HJ, Lee KM, et al. Glycopeptide antibiotics and their novel semi-synthetic derivatives[J]. Current Pharmaceutical Biotechnology, 2011, 12(8): 1194-1204. DOI:10.2174/138920111796117382 |
| [7] |
Ashford PA, Bew SP. Recent advances in the synthesis of new glycopeptide antibiotics[J]. Chemical Society Reviews, 2012, 41(3): 957-978. DOI:10.1039/C1CS15125H |
| [8] |
Binda E, Marinelli F, Marcone GL. Old and new glycopeptide antibiotics: action and resistance[J]. Antibiotics, 2014, 3(4): 572-594. DOI:10.3390/antibiotics3040572 |
| [9] |
Smith JR, Roberts KD, Rybak MJ. Dalbavancin: a novel lipoglycopeptide antibiotic with extended activity against gram-positive infections[J]. Infectious Diseases and Therapy, 2015, 4(3): 245-258. DOI:10.1007/s40121-015-0077-7 |
| [10] |
Cheng M, Ziora ZM, Hansford KA, et al. Anti-cooperative ligand binding and dimerisation in the glycopeptide antibiotic dalbavancin[J]. Organic & Biomolecular Chemistry, 2014, 12(16): 2568-2575. |
| [11] |
Mattox J, Belliveau P, Durand C. Oritavancin: a novel lipoglycopeptide[J]. The Consultant Pharmacist, 2016, 31(2): 86-95. DOI:10.4140/TCP.n.2016.86 |
| [12] |
Sweeney D, Stoneburner A, Shinabarger DL, et al. Comparative in vitro activity of oritavancin and other agents against vancomycin-susceptible and -resistant enterococci[J]. The Journal of Antimicrobial Chemotherapy, 2017, 72(2): 622-624. DOI:10.1093/jac/dkw451 |
| [13] |
Chen CH, Li GH. Telavancin: a novel lipoglycopeptide antibiotic[J]. Chinese Journal of Infection and Chemotherapy, 2012, 12(2): 153-157. 陈春辉, 李光辉. 新型脂糖肽类抗生素特拉万星[J]. 中国感染与化疗杂志, 2012, 12(2): 153-157. |
| [14] |
Tan HL, Guo H, Wang SL, et al. Chemistry and biology of glycopeptides with antibiotic activity[J]. Protein and Peptide Letters, 2014, 21(10): 1031-1047. DOI:10.2174/0929866521666140626110327 |
| [15] |
Hesketh A, Deery MJ, Hong HJ. High-resolution mass spectrometry based proteomic analysis of the response to vancomycin-induced cell wall stress in Streptomyces coelicolor A3(2)[J]. Journal of Proteome Research, 2015, 14(7): 2915-2928. DOI:10.1021/acs.jproteome.5b00242 |
| [16] |
Bender JK, Kalmbach A, Fleige C, et al. Population structure and acquisition of the vanB resistance determinant in German clinical isolates of Enterococcus faecium ST192[J]. Scientific Reports, 2016, 6: 21847. DOI:10.1038/srep21847 |
| [17] |
Song JY, Cheong HJ, Seo YB, et al. Clinical and microbiological characteristics of vancomycin-resistant enterococci with the VanD phenotype and vanA genotype[J]. Japanese Journal of Infectious Diseases, 2013, 66(1): 1-5. DOI:10.7883/yoken.66.1 |
| [18] |
Panagiota G, Vangelis E, Petros B, et al. Vancomycin-resistance phenotypes, vancomycin-resistance genes, and resistance to antibiotics of enterococci isolated from food of animal origin[J]. Foodborne Pathogens and Disease, 2015, 12(3): 214-220. DOI:10.1089/fpd.2014.1832 |
| [19] |
Peltier J, Courtin P, El Meouche I, et al. Genomic and expression analysis of the vanG-like gene cluster of Clostridium difficile[J]. Microbiology, 2013, 159(Pt 7): 1510-1520. |
| [20] |
Chen CC, Xu XG. Genetic characteristics of vancomycin resistance gene cluster in Enterococcus spp[J]. Hereditas (Beijing), 2015, 37(5): 452-457. |
| [21] |
Jung YH, Lee YS, Lee SY, et al. Structure and transfer of the vanA cluster in vanA-positive, vancomycin-susceptible Enterococcus faecium, and its revertant mutant[J]. Diagnostic Microbiology and Infectious Disease, 2014, 80(2): 148-150. DOI:10.1016/j.diagmicrobio.2014.06.012 |
| [22] |
Lozano C, Gonzalez-Barrio D, Camacho MC, et al. Characterization of fecal vancomycin-resistant enterococci with acquired and intrinsic resistance mechanisms in wild animals, Spain[J]. Microbial Ecology, 2016, 72(4): 813-820. DOI:10.1007/s00248-015-0648-x |
| [23] |
Muthyala R, Rastogi N, Shin WS, et al. Cell permeable vanX inhibitors as vancomycin re-sensitizing agents[J]. Bioorganic & Medicinal Chemistry Letters, 2014, 24(11): 2535-2538. |
| [24] |
Eshaghi A, Shahinas D, Li AM, et al. Characterization of an Enterococcus gallinarum isolate carrying a dual vanA and vanB cassette[J]. Journal of Clinical Microbiology, 2015, 53(7): 2225-2229. DOI:10.1128/JCM.03267-14 |
| [25] |
Gudeta DD, Moodley A, Bortolaia V, et al. vanO, a new glycopeptide resistance operon in environmental Rhodococcus equi isolates[J]. Antimicrobial Agents and Chemotherapy, 2014, 58(3): 1768-1770. DOI:10.1128/AAC.01880-13 |
| [26] |
Périchon BCP. Glycopeptide resistance[A]//Dougherty TJ, Pucci MJ. Antibiotic Discovery and Development[M]. US: Springer, 2012: 515-542
|
| [27] |
Werner G, Klare I, Fleige C, et al. Vancomycin-resistant vanB-type Enterococcus faecium isolates expressing varying levels of vancomycin resistance and being highly prevalent among neonatal patients in a single ICU[J]. Antimicrobial Resistance and Infection Control, 2012, 1: 21. DOI:10.1186/2047-2994-1-21 |
| [28] |
Sting R, Richter A, Popp C, et al. Occurrence of vancomycin-resistant enterococci in turkey flocks[J]. Poultry Science, 2013, 92(2): 346-351. DOI:10.3382/ps.2012-02652 |
| [29] |
Yim G, Thaker MN, Koteva K, et al. Glycopeptide antibiotic biosynthesis[J]. The Journal of Antibiotics, 2014, 67(1): 31-41. |
| [30] |
Haslinger K, Peschke M, Brieke C, et al. X-domain of peptide synthetases recruits oxygenases crucial for glycopeptide biosynthesis[J]. Nature, 2015, 521(7550): 105-109. DOI:10.1038/nature14141 |
| [31] |
Kastner S, Müller S, Natesan L, et al. 4-Hydroxyphenylglycine biosynthesis in Herpetosiphon aurantiacus: a case of gene duplication and catalytic divergence[J]. Archives of Microbiology, 2012, 194(6): 557-566. DOI:10.1007/s00203-012-0789-y |
| [32] |
Brieke C, Peschke M, Haslinger K, et al. Sequential in vitro cyclization by cytochrome P450 enzymes of glycopeptide antibiotic precursors bearing the X-domain from nonribosomal peptide biosynthesis[J]. Angewandte Chemie International Edition, 2015, 54(52): 15715-15719. DOI:10.1002/anie.201507533 |
| [33] |
Peschke M, Haslinger K, Brieke C, et al. Regulation of the P450 oxygenation cascade involved in glycopeptide antibiotic biosynthesis[J]. Journal of the American Chemical Society, 2016, 138(21): 6746-6753. DOI:10.1021/jacs.6b00307 |
| [34] |
Yushchuk O, Ostash B, Pham TH, et al. Characterization of the post-assembly line tailoring processes in teicoplanin biosynthesis[J]. ACS Chemical Biology, 2016, 11(8): 2254-2264. DOI:10.1021/acschembio.6b00018 |
| [35] |
Park JW, Nam SJ, Yoon YJ. Enabling techniques in the search for new antibiotics: Combinatorial biosynthesis of sugar-containing antibiotics[J]. Biochemical Pharmacology, 2017, 134: 56-73. DOI:10.1016/j.bcp.2016.10.009 |
| [36] |
Thaker MN, Wright GD. Opportunities for synthetic biology in antibiotics: expanding glycopeptide chemical diversity[J]. ACS Synthetic Biology, 2015, 4(3): 195-206. DOI:10.1021/sb300092n |
 2018, Vol. 45
2018, Vol. 45




