扩展功能
文章信息
- 刘文容, 陈双, 徐岩
- LIU Wen-Rong, CHEN Shuang, XU Yan
- 黄酒贮存酸败关键微生物的分离鉴定
- Identification and characterization of spoilage microorganisms in aging Chinese rice wine by culture-independent and culture-dependent technologies
- 微生物学通报, 2018, 45(1): 120-128
- Microbiology China, 2018, 45(1): 120-128
- DOI: 10.13344/j.microbiol.china.170204
-
文章历史
- 收稿日期: 2017-03-12
- 接受日期: 2017-05-11
- 优先数字出版日期(www.cnki.net): 2017-05-25
黄酒(Chinese rice wine)是以稻米、黍米等为主要原料,经加曲、酵母等糖化发酵剂酿制而成的发酵酒,是我国独具民族特色的酒精饮料[1]。黄酒又称“老酒”,其典型特征是“越陈越香”。新酿成的黄酒需要经过一定时间的贮存来提高其风味品质,此过程称为黄酒的老熟或陈酿[2]。但由于黄酒中糖分、氨基酸等营养物质含量丰富,黄酒贮存陈酿过程中时常出现酸败变质的现象,尤其是大罐陈酿过程中的酸败对黄酒行业造成较大的经济损失[3-4]。由于目前对于引起黄酒陈酿酸败的关键微生物及其酸败机理认识不清,缺乏有效的控制措施,黄酒酸败问题已经成为限制黄酒全程规模化、机械化的“瓶颈”问题,成为黄酒大罐贮存陈酿新技术推广的关键限制因素[5-6]。
黄酒的陈酿酸败作为黄酒行业的一大危害,早在20世纪80年代已有学者对黄酒陈酿酸败的原因进行了研究探索。根据黄酒酸败的现象不同可以将黄酒酸败分为生菌膜酸败(醋酸酸败)和非菌膜酸败(乳酸酸败)[7]。醋酸酸败主要在低酒精度、好氧情况下容易发生,一般是由于酒坛破损或密封不严造成酒液酒精挥发到一定程度后酵母菌和醋酸菌大量繁殖造成。这类微生物往往不耐受较高浓度乙醇,对正常贮存的黄酒危害较小[8-9]。而乳酸酸败则是在较高的酒精度(18%)条件下、密闭环境中容易发生,由于经常在包装完好的酒坛中出现,对黄酒贮存危害更大。但由于这类微生物对营养环境要求苛刻,常规培养方法一般难以培养,仅根据酸败黄酒中微生物形态推测为乳酸菌类[10]。毛青钟[11]采用一种保密培养基从酸败陈酿黄酒中分离得到两种乳酸杆菌,通过形态分析初步鉴定为乳酸杆菌新种。但由于研究手段的限制,对黄酒陈酿危害更大的乳酸酸败关键微生物种类和生物学特征目前还未完全解析,这也严重限制了对黄酒贮存过程中酸败微生物控制策略的研究。
酒精饮料作为一种特殊的生长基质,具有高酒精度、低pH的特点,一般不适合微生物的生长,但是仍有部分微生物适应了酒精饮料中的生长环境而大量繁殖导致酸败问题的发生。在啤酒的研究中,直到近几年,耐酸乳杆菌(Lactobacillus acetotolerans)才作为一种典型的啤酒酸败微生物被检测出来。究其原因,耐酸乳杆菌是一种难培养的啤酒酸败微生物,在常规的MRS培养基中难以生长。Deng等[12]通过添加过氧化氢酶,在酸败啤酒中检测出耐酸乳杆菌。在清酒的研究中,酒液浑浊、有异味等酸败现象被称为“火落”[13],一直是清酒行业面临的一大难题,而造成这种现象的微生物却一直没有被检测出来。直到20世纪70年代,Sugama等专门设计了针对性培养基——S.I.培养基[14],培养得到了酸败清酒中的难培养微生物同型腐酒乳杆菌(Lactobacillus homohiochi)和食果糖乳杆菌(Lactobacillus fructivorans)[15]。1982年,Colwell实验室提出活的但难培养微生物的概念[16],指出自然界中存在着大量具有细胞活性但在实验室条件下难以分离培养的微生物[17],通过改进微生物的培养环境和营养条件可实现部分难培养微生物的可培养。
在本研究中,针对黄酒陈酿过程中的难培养酸败微生物,通过高通量测序技术解析酸败黄酒中的主要微生物,设计针对性培养基实现难培养微生物的可培养,并对培养得到的微生物酸败性能进行验证。
1 材料与方法 1.1 样品正常贮存(未出现贮存容器破损)条件下发生酸败的黄酒样品分别由浙江省和江苏省的5个黄酒生产企业提供,共13个样品。其中1-5#样品由1号黄酒厂提供,为大罐贮存酒样;6-7#样品由2号黄酒厂提供,为陶坛贮存酒样;14#样品由3号黄酒厂提供,为陶坛贮存酒样;15-16#和18-19#样品由4号黄酒厂提供,为大罐贮存酒样;20#样品由5号黄酒厂提供,为大罐贮存酒样。未发生酸败的陈酿黄酒由5号黄酒厂提供。
1.2 主要试剂和仪器磷酸氢二钠、磷酸二氢钠、醋酸钠、10 mmol/L Tris-HCl (pH 8.0)、氯化钠、饱和酚溶液、10% SDS (pH 8.0)、氯仿、异戊醇、无水乙醇,国药集团化学试剂有限公司;过氧化氢酶、甲瓦龙酸,百灵威科技有限公司;2×Taq PCR Master Mix,天根生化科技(北京)有限公司;MRS培养基,Oxoid公司;细菌基因组DNA快速抽提试剂盒,生工生物工程(上海)股份有限公司。珠磨器,Biospec公司;高速冷冻离心机,Beckman Coulter公司;电泳仪,Bio-Rad公司;冷冻干燥器,Labconco公司;pH计,Mettler-Toledo公司。
1.3 微生物DNA提取取酸败陈酿黄酒70 mL,10 000 r/min离心15 min收集细胞沉淀,用5 mL PBS缓冲液(57.7 mmol/L Na2HPO4,2.3 mmol/L NaH2PO4)洗涤3次,至洗涤液干净无色,10 000 r/min离心10 min得细胞沉淀提取DNA。具体步骤如下[18]:将细胞沉淀重悬于1 mL buffer Z (10 mmol/L pH 8.0 Tris-HCl,150 mmol/L NaCl)中混匀,转移至加有0.3 g玻璃珠(直径0.1 mm)的螺帽管中,加入150 μL苯酚,珠磨器最大速度击打4 min (每击打80 s将螺帽管冰浴1 min,共3次);加入110 μL 10% SDS,轻轻混匀后冰浴10 min (每5 min轻轻颠倒混匀);加入150 μL氯仿-异戊醇溶液(24:1,体积比),轻轻混匀后12 000 r/min离心12 min收集上清,加入1/10体积的3 mol/L醋酸钠溶液,用等体积苯酚、苯酚-氯仿(1:1,体积比)、氯仿各抽提一次,12 000 r/min离心12 min收集上清;加入2倍体积冰乙醇,置于-20 ℃过夜沉淀后,12 000 r/min离心20 min;弃上清,所得沉淀经真空干燥后溶于30 μL无菌水中,备用。
1.4 高通量测序及分析针对13个酸败黄酒样品的DNA,利用通用引物扩增其16S rRNA基因的V3-V4高变异区,通用引物分别为338F (5′-ACTCCTACGGGAGGC AGCAG-3′)和806R (5′-GACTACHVGGGTWTCTA AT-3′)[19],这些引物含有8个碱基的特异性标签,用以区分不同的黄酒样品。25 μL PCR反应体系:10×Pyrobest Buffer 2.5 μL,dNTP Mixture (2.5 mmol/L) 2 μL,10 μmol/L上、下游引物各1 μL,Pyrobest DNA聚合酶0.4 U,DNA模板15 ng。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,25个循环;72 ℃ 10 min。PCR产物经纯化后等摩尔数混合,利用北京奥维森公司的Illumina Miseq PE300测序仪测序分析。测序后获得的数据首先要进行提取和优化,去除一些不符合要求的序列,然后再进行物种的分类,包括分类操作单元(OTU)聚类和分类学分析,具体步骤参考文献[20]。
1.5 培养基用到的培养基有MRS培养基,添加浓缩黄酒的MRS培养基,添加过氧化氢酶的MRS培养基,以及添加甲瓦龙酸的MRS培养基,培养基成分如表 1所示,其中浓缩黄酒是以旋蒸法将正常黄酒浓缩10倍的黄酒浓缩液。固体培养基是在液体培养基中加入2%的琼脂。灭菌条件:1×105 Pa灭菌15 min。
| 培养基 Medium |
成分 Composition (L–1) |
| MRS broth | Peptone 10.0 g, Yeast extract 4.0 g, Glucose 20.0 g, “Lab-lemco” powder 8.0 g, Sorbitan mono-oleate 1 mL, K2HPO4 2.0 g, CH3COONa·3H2O 5.0 g, (NH4)3C6H5O7 2.0 g, MgSO4·7H2O 0.2 g, MnSO4 0.05 g, Final pH 5.7 |
| MRS-Rice wine inspissation | MRS broth (powder) 52.0 g, Rice wine inspissation 100 mL, Ethanol 150-200 mL, ddH2O 750-700 mL, Final pH 5.0 |
| MRS-Catalase | MRS broth (powder) 52.0 g, Catalase* 0.1 mL, Ethanol 150-200 mL, ddH2O 850-800 mL, Final pH 5.0 |
| MRS-Mevalonic acid | MRS broth (powder) 52.0 g, Mevalonic acid 5.0 mg, Ethanol 150-200 mL, ddH2O 850-800 mL, Final pH 5.0 |
| 注:*:过氧化氢酶溶液浓度为10 000 U/mL. Note: *: The concentration of catalase solution was 10 000 U/mL. |
|
采用细菌基因组DNA快速抽提试剂盒提取培养得到的微生物基因组DNA,并进行PCR扩增,所用引物序列如表 2所示。3组PCR反应体系均为25 μL:ddH2O 10.5 μL,2×Taq PCR Master Mix 12.5 μL,上、下游引物(20 μmol/L)各0.5 μL,DNA模板1 μL。16S rRNA基因扩增程序:94 ℃ 4 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 2 min,共30个循环;72 ℃ 10 min。基因recA、tuf扩增程序:94 ℃ 4 min;94 ℃ 1 min,55 ℃ 30 s,72 ℃ 45 s,共30个循环;72 ℃ 10 min。PCR扩增产物在1%的琼脂糖凝胶中电泳检测,送生工生物工程(上海)股份有限公司测序部测序,测序结果提交到NCBI (http://www.ncbi.nlm.nih.gov/) GenBank数据库做同源性序列比对及相关信息检索。
| 引物名称 Primers |
引物序列 Sequences (5′→3′) |
引物长度 Primers size (bp) |
目标基因 Target genes |
| 27F | AGAGTTTGATCCTGGCTCA | 19 | 16S rRNA |
| 1492R | GGTTACCTTGTTACGACTT | 19 | |
| LA1 | AAAATTTCAACTGTGCCATCCGG | 23 | recA |
| LA2 | TCAAAGCACGACCACCGG | 18 | |
| LF1 | GACTTAGTTGAGATGGAAGTCCG | 23 | tuf |
| LF2 | GACCAGTAAACTTCTTGTGGGTATG | 25 |
将筛选出的微生物接入到针对性培养基中,30 ℃厌氧条件下静置培养。待微生物培养至生长对数中后期(OD600为1.80左右),将种子液以2%的比例接入到未发生酸败的黄酒中(10 mL/试管),然后置于30 ℃恒温培养箱中厌氧静置培养2周后,以未发生酸败的黄酒为对照组,测定加菌黄酒总酸含量的变化。黄酒中总酸含量的测定参照国标GB/T 1366-2008规定的滴定法执行。
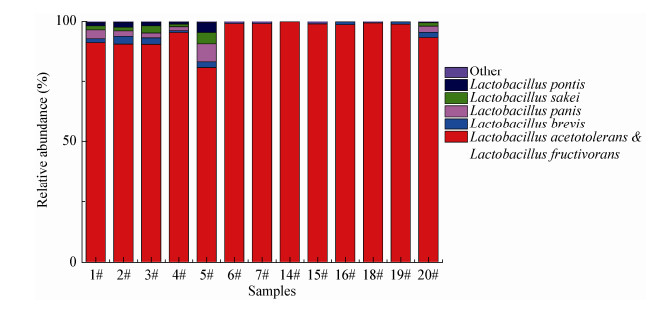
2 结果与分析 2.1 采用高通量测序解析酸败黄酒中的主要微生物为了解析酸败黄酒中的主要微生物,利用高通量测序技术对酸败黄酒中污染微生物V3-V4高变异区进行测序,测序结果已上传至DDBJ数据库,序列号为DRA005614。在本次测序中共得到416 221条有效序列,平均每个样品的有效序列数为32 017,序列平均长度为397 bp。在97%水平下对样品序列进行分类,共得到2 053个OTU。对这些OTU序列进行微生物比对分类,结果显示酸败黄酒中的微生物有3门4纲5目7科11属。其中对酸败微生物属水平分类分析可知(图 1),乳酸杆菌属为优势菌属,其相对丰度达到95%以上,其他菌属的相对丰度较低(< 1%),说明酸败黄酒中的主要微生物为乳酸杆菌属微生物。进一步对这类乳酸杆菌属微生物进行种水平比对分析,如图 2所示,酸败黄酒中乳酸杆菌属微生物包含9种乳酸杆菌,其中主要的一类乳酸杆菌的相对丰度达到82%以上,为绝对优势微生物。在对这类微生物进行种水平比对分析时发现,比对结果中同时出现了耐酸乳杆菌和食果糖乳杆菌这2个可能的种,而且相似性均为99%。分析原因发现,耐酸乳杆菌和食果糖乳杆菌在16S rRNA基因V3-V4高变异区的序列相似性很高,仅存在1个碱基的差异,基于高通量测序技术无法准确将两种微生物区分鉴定。但通过高通量测序分析将黄酒酸败的主要微生物锁定为乳酸杆菌属微生物,且微生物种类较为单一,可能为耐酸乳杆菌或食果糖乳杆菌。

|
| 图 1 黄酒酸败微生物在属水平上的相对丰度 Figure 1 Microbial community structure of spoilage microorganisms at genus level 注:相对丰度小于0.1%的菌属归为other. Note: Genus relative abundance of less than 0.1% classified as other. |
|
|
|
| 图 2 黄酒酸败微生物在种水平上的相对丰度 Figure 2 Microbial community structure of spoilage microorganisms at species level 注:相对丰度小于0.1%的菌种归为other. Note: Species relative abundance of less than 0.1% classified as other. |
|
|
已有相关研究表明,耐酸乳杆菌是啤酒中主要的酸败微生物,由于其难培养的特征,近几年才被发现培养出来[12]。食果糖乳杆菌作为清酒中主要的酸败微生物,具有很高的酒精耐受性(18%-25%)和低pH耐受性(4.0-5.0),以及较高的耐热性,巴氏灭菌对微生物的影响较小[15],其对清酒的危害较大。这两类微生物都属于难培养微生物,需要特殊的培养基才能实现分离培养。
2.2 酸败黄酒中主要微生物的分离根据高通量测序的分析结果,酸败黄酒中的主要微生物可能是耐酸乳杆菌或食果糖乳杆菌,目前已有相关文献证明[12, 15],这两种微生物对生长环境的营养需求十分苛刻,在常规培养基中难以生长,是典型的难培养微生物。为了分离培养出黄酒中主要的酸败微生物,根据微生物的特征设计不同的分离培养基,包括模拟酸败微生物的生存环境,在MRS培养基中添加浓缩黄酒,或者添加文献[12, 15]报道的酸败微生物生长所需要的生长因子——过氧化氢酶或甲瓦龙酸,对酸败黄酒中的污染微生物进行培养。采用平板分离法,分别取酸败黄酒样品涂布于4种不同的固体培养基上,培养基成分见表 1。接种后置于厌氧盒内,由厌氧产气袋确保厌氧盒内的厌氧环境,并于30 ℃培养箱内静置培养,观察并记录培养结果(表 3)。结果显示,在酒精浓度为17%的MRS培养基中没有微生物生长,与已有报道结果吻合[9];而在设计的针对性培养基中均有微生物生长,微生物菌落呈白色,湿润,边缘整齐。
| 培养基 Medium |
培养结果 Cultivate results |
菌落数量 Number of colonies |
培养周期 Period |
样品来源 Samples source |
| MRS* | Failure | - | N | 7#, 19# |
| MRS-Rice wine inspissation | Success, small and milky white colonies | + | 5-6 d | 7#, 19# |
| MRS-Catalase | Success, small and milky white colonies | ++ | 4-5 d | 7#, 19# |
| MRS-Mevalonic acid | Success, small and milky white colonies | +++ | 4-5 d | 7#, 19# |
| 注:*:MRS培养基酒精浓度为17%. -:无菌落;+:有菌落,+数量越多表示菌落数量越多. N:无培养周期.
Note: *: The alcohol concentration of the medium was 17%. -: There was no colony; +: Colonies grew on the media and the more the number of +, the more the number of colonies. N: No culture period. |
||||
经过前期培养基的筛选与验证,选择培养结果最好的MRS-甲瓦龙酸培养基进行微生物的培养,经过多次划线纯化得到微生物共36株,为了进一步明确酸败黄酒中的主要微生物种类,采用传统方法对微生物进行鉴定。以提取到的微生物基因组为模板,选择引物27F和1492R对16S rRNA基因序列进行PCR扩增,扩增产物测序后与NCBI (http://www.ncbi.nlm.nih.gov/) GenBank数据库进行同源性比对。结果发现,同一条扩增序列的鉴定结果中会出现2种微生物(食果糖乳杆菌和耐酸乳杆菌),相似性均为99%,与高通量测序比对结果相似,无法准确判断该序列所代表的微生物种类。通过对耐酸乳杆菌和食果糖乳杆菌16S rRNA基因序列比对发现,两者仅存在3个碱基的差异,所以传统的16S rRNA鉴定方法不能区分鉴定耐酸乳杆菌和食果糖乳杆菌。为了准确区分两种微生物,对两者的所有基因进行比对和普查,选择耐酸乳杆菌和食果糖乳杆菌各自的特异性基因recA和tuf为模板,针对两种微生物分别设计特异性引物LA1、LA2和LF1、LF2 (表 2),经PCR扩增及测序后,再与NCBI GenBank数据库进行同源性比对。比对结果显示,每一条扩增序列仅对应一种微生物,且序列相似性达到99%,从前期培养得到的36株微生物中鉴定出28株耐酸乳杆菌和8株食果糖乳杆菌。从两株菌的电镜扫描结果(图 3)中可以看出,两者虽然都为杆菌、单个排列,但是从细胞形态分析,食果糖乳杆菌的平均长度、宽度分别为8.26 μm和0.65 μm,耐酸乳杆菌的平均长度、宽度分别为3.04 μm和0.43 μm,呈现较为明显的差异。

|
| 图 3 食果糖乳杆菌(A)和耐酸乳杆菌(B)扫描电镜照片 Figure 3 Scanning electron microscope of L. fructivorans (A) and L. acetotolerans (B) |
|
|
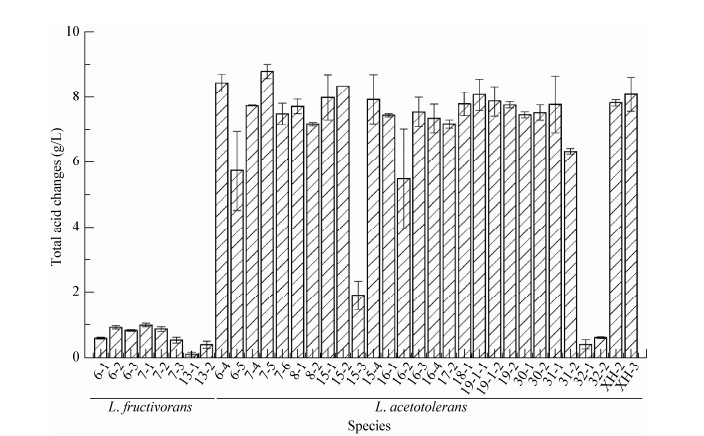
为验证分离得到的微生物是否具有导致黄酒酸败的能力,对其酸败性能做了相关研究(图 4)。前8种微生物为食果糖乳杆菌,后面的微生物均为耐酸乳杆菌。结果显示,食果糖乳杆菌虽然可以导致黄酒的总酸含量升高(0.2-1.1 g/L),但其变化量较小,而耐酸乳杆菌能显著提高黄酒的总酸含量(5.6-8.8 g/L),导致黄酒酸败,但菌株15-3、32-1和32-2造成的黄酒总酸含量变化较小(0.5-2.0 g/L),说明菌种之间导致黄酒酸败的能力存在差异。从筛选出的酸败微生物数量和性能差异分析,耐酸乳杆菌是导致黄酒酸败的主要微生物,食果糖乳杆菌次之。
|
| 图 4 酸败微生物导致的黄酒总酸含量变化 Figure 4 Changes of total acid content caused by spoilage microorganism |
|
|
黄酒贮存陈酿过程中发生的微生物乳酸酸败现象一直是黄酒行业难以解决的问题[21]。由于这类微生物生长条件苛刻,采用传统的研究手段难以对其进行培养鉴定,目前无法阐明其微生物本质。近年来发展起来的基于高通量测序的微生物未培养技术为解决这一难题提供了新的技术手段。本研究将未培养技术应用于黄酒中难培养污染微生物的解析,为食品中污染微生物的快速分析提供了新的研究思路。基于未培养技术的应用,本研究首次发现耐酸乳杆菌和食果糖乳杆菌是造成黄酒酸败的主要微生物,这两类微生物也在黄酒中首次被检出。耐酸乳杆菌和食果糖乳杆菌一般存在于酒、食醋等特殊环境中,是典型的难培养微生物,也是啤酒、日本清酒酸败的主要微生物[12, 15]。以未培养技术分析结果为指导,根据两种微生物的营养需求特征,通过针对性培养基的开发,首次在酸败黄酒中分离培养出耐酸乳杆菌和食果糖乳杆菌,这也为后期对这两种黄酒酸败微生物生理生化特征的解析及控制措施的研究奠定了基础。
| [1] |
Chen S, Xu Y. The influence of yeast strains on the volatile flavour compounds of Chinese rice wine[J]. Journal of the Institute of Brewing, 2010, 116(2): 190-196. DOI:10.1002/(ISSN)2050-0416 |
| [2] |
Yang GJ. Aging of yellow rice wine[J]. Liquor-making Science and Technology, 2006(6): 74-76. 杨国军. 黄酒的陈化[J]. 酿酒科技, 2006(6): 74-76. |
| [3] |
Qi CL. Causes of deterioration & rancidity of yellow rice wine storaged in big pots and the relative control measures[J]. Liquor-making Science & Technology, 2005(2): 111-112. 祁传林. 大罐贮存黄酒出现变质酸败的因素及控制[J]. 酿酒科技, 2005(2): 111-112. |
| [4] |
Fan WG, Zhang YM, Qiao XJ, et al. Research on the storage of yellow rice wine by high-capacity stainless steel tanks[J]. Liquor-making Science & Technology, 2014(5): 67-70. 范伟国, 张艳梅, 乔新建, 等. 大容量不锈钢罐贮存黄酒的研究及应用[J]. 酿酒科技, 2014(5): 67-70. |
| [5] |
Mao QZ, Zhang SJ, Lu RG, et al. Yellow rice wine storage warehouse altar wine rancidity of the reasons for research and control, measures[J]. Liquor Making, 2009(4): 42-46. 毛青钟, 张水娟, 鲁瑞刚, 等. 黄酒仓库贮存坛酒的酸败原因及控制措施研究[J]. 酿酒, 2009(4): 42-46. |
| [6] |
Qian YL, Fei XZ. Study on the storage of yellow rice wine by big pots[J]. Jiangsu Food and Fermentation, 1993(3): 1-3. 钱裕良, 费雪忠. 黄酒大罐贮存的技术研究[J]. 江苏食品与发酵, 1993(3): 1-3. |
| [7] |
Lin MC. A preliminary study on the spoiled yellow rice wine[J]. China Brewing, 1993(5): 44-46. 林茂丛. 黄酒酸败问題初探[J]. 中国酿造, 1993(5): 44-46. |
| [8] |
Zhang YZ, Yao WR. Separation and identification of microorganisms from spoiled traditional aging rice wine[J]. China Brewing, 2008, 27(21): 62-66. 章银珠, 姚卫蓉. 变质传统发酵陈化黄酒微生物的分离和鉴定[J]. 中国酿造, 2008, 27(21): 62-66. |
| [9] |
Zhang YZ. Studies on the isolation, identification and characteristics of spoilage microbes from aging yellow rice wine[D]. Wuxi: Master's Thesis of Jiangnan University, 2008 (in Chinese) 章银珠. 陈化传统黄酒腐败微生物的分离鉴定和特性研究[D]. 无锡: 江南大学硕士学位论文, 2008 http://cdmd.cnki.com.cn/Article/CDMD-10295-2009013045.htm |
| [10] |
Mao QZ. Chinese rice wine mash fermentation of lactic acid bacteria detection and unique media research and preliminary identification[J]. Liquor Making, 2014, 41(4): 75-80. 毛青钟. 黄酒发酵醪中特有乳酸杆菌检测和培养基的研究及初步鉴定[J]. 酿酒, 2014, 41(4): 75-80. |
| [11] |
Mao QZ. Cause aging Chinese rice wine rancidity test method of Lactobacillus and media research and preliminary identafication[J]. Liquor Making, 2014, 41(3): 45-50. 毛青钟. 引起陈酿黄酒酸败乳酸杆菌的检测方法和培养基研究及初步鉴定[J]. 酿酒, 2014, 41(3): 45-50. |
| [12] |
Deng Y, Liu JY, Li HP, et al. An improved plate culture procedure for the rapid detection of beer-spoilage lactic acid bacteria[J]. Journal of the Institute of Brewing, 2014, 120(2): 127-132. DOI:10.1002/jib.v120.2 |
| [13] |
Wada Y, Mizoguchi H. New insertion sequence in Lactobacillus fructivorans strains isolated from spoiled sake[J]. Journal of Bioscience and Bioengineering, 2007, 103(5): 399-405. DOI:10.1263/jbb.103.399 |
| [14] |
Sugama S, Iguchi T. A study on the prevention of sake spoilage—development of S. I. medium and its applications to prediction of hiochi phenomena[J]. Journal of the Brewing Society of Japan, 1970, 65: 720-725. DOI:10.6013/jbrewsocjapan1915.65.720 |
| [15] |
Suzuki K, Asano S, Iijima K, et al. Sake and beer spoilage lactic acid bacteria—a review[J]. Journal of the Institute of Brewing, 2008, 114(3): 209-223. DOI:10.1002/(ISSN)2050-0416 |
| [16] |
Xu HS, Roberts N, Singleton FL, et al. Survival and viability of nonculturable Escherichia coli and Vibrio cholerae in the esturarine and marine environment[J]. Microbial Ecology, 1982, 8(4): 313-323. DOI:10.1007/BF02010671 |
| [17] |
Zeng JM, Zeng ZS, Yuan HJ, et al. Advances in culture methods of oligotrophic microbes[J]. Current Biotechnology, 2012, 2(3): 165-170. 曾建民, 曾振顺, 原红娟, 等. 难培养微生物培养方法的研究进展[J]. 生物技术进展, 2012, 2(3): 165-170. |
| [18] |
Wang HY, Zhang XJ, Zhao LP, et al. Analysis and comparison of the bacterial community in fermented grains during the fermentation for two different styles of Chinese liquor[J]. Journal of Industrial Microbiology and Biotechnology, 2008, 35(6): 603-609. DOI:10.1007/s10295-008-0323-z |
| [19] |
Zhang XL, Tian XQ, Ma LY, et al. Biodiversity of the symbiotic bacteria associated with toxic marine dinoflagellate Alexandrium tamarense[J]. Journal of Biosciences and Medicine, 2015, 3(6): 23-28. DOI:10.4236/jbm.2015.36004 |
| [20] |
Li Z, Dong L, Huang Q, et al. Bacterial communities and volatile compounds in Doubanjiang, a Chinese traditional red pepper paste[J]. Journal of Applied Microbiology, 2016, 120(6): 1585-1594. DOI:10.1111/jam.2016.120.issue-6 |
| [21] |
Zhai JH. Study on the control measures of spoiled yellow rice wine[J]. China Food Safety Magazine, 2015(9): 50-52. 翟嘉华. 黄酒酸败控制措施的研究[J]. 食品安全导刊, 2015(9): 50-52. |
 2018, Vol. 45
2018, Vol. 45




