扩展功能
文章信息
- 范中菡, 张波, 廖敏, 李小林, 张小平, 陈熊春蕊
- FAN Zhong-Han, ZHANG Bo, LIAO Min, LI Xiao-Lin, ZHANG Xiao-Ping, CHEN-Xiong-Chun-Rui
- 四川阿坝不同生境绿绒蒿内生放线菌多样性
- Diversity of endophytic actinomycetes from Entire meconopsis sampled in Aba, Sichuan
- 微生物学通报, 2018, 45(1): 81-90
- Microbiology China, 2018, 45(1): 81-90
- DOI: 10.13344/j.microbiol.china.170050
-
文章历史
- 收稿日期: 2017-01-17
- 接受日期: 2017-10-12
- 优先数字出版日期(www.cnki.net): 2017-10-23
2. 四川省农业科学院植物保护研究所 四川 成都 610066;
3. 绵阳师范学院 四川 绵阳 621000;
4. 四川省农业科学院土壤肥料研究所 四川 成都 610066
2. Institute of Plant Protection, Academy of Agricultural Sciences, Chengdu, Sichuan 610066, China;
3. Mianyang Normal University, Mianyang, Sichuan 621000, China;
4. Soil and Fertilizer Research Institute, Academy of Agricultural Sciences, Chengdu, Sichuan 610066, China
绿绒蒿(Entire meconopsis)为一年生或多年生草本植物,有止咳、镇痛、抗菌、治疗热性水肿、肝硬化、肠炎等药用功效[1-2]。绿绒蒿生长于海拔2 800−4 300 m的山坡草地,自然分布于西藏东北部、四川西北部、甘肃南部和青海东南部,古代草本学家典籍很少有记载(仅在藏药典籍有过相关记载)。绿绒蒿作为珍贵野生资源,具有较高的观赏价值和药用价值,如今却面临日益枯竭的危险。
放线菌可产生抗生素及其他生物活性物质[3],目前临床及农业上应用的抗生素3/4是由放线菌产生的[4-5]。现在人们已将目光投向植物内生放线菌的研究,并发现了其丰富的生物多样性和功能多样性[6],及其与宿主植物密切复杂的关系[7]。然而纯培养手段只能分离出环境样品中极少的微生物,据国内外研究结果表明,尚有90%以上的微生物不能被纯培养[8],放线菌也是如此。近年来,多种免培养技术逐渐应用于极端环境微生物资源的开发利用[9],攻克了纯培养的缺陷。刘菲菲等[10]对香蕉植株内生细菌群落进行PCR-DGGE分析,结果表明香蕉健康菌株与病株各组织中所含内生细菌的种群丰富度为根部 > 假茎 > 叶片;陈诚等[11]对镍矿区紫茎泽兰进行PCR-DGGE分析,结果表明紫茎泽兰根际与不同器官内生细菌多样性有着明显差异。由于免培养技术不能获得所需菌株,因此结合纯培养与免培养技术有利于研究者更全面了解植物内生环境微生物群落结构。目前国内外对绿绒蒿的研究主要集中于化学成分,对其内生放线菌的研究鲜有报道。本研究结合纯培养和免培养方式(PCR-DGGE)对绿绒蒿不同器官内生放线菌多样性和群落结构进行研究,一方面能系统了解绿绒蒿内生放线菌多样性,另一方面为发掘放线菌天然活性物质提供资源基础。
1 材料与方法 1.1 样品采集2013年6月从四川省阿坝地区6个不同地理位置采集野生绿绒蒿样品,每个采样点采集3株健康植株,植株生长海拔及地理位置如表 1所示。样品采集时,将切口部位乙醇消毒,进行蜡封,装入无菌塑料袋,带回四川农业大学微生物实验室4 ℃保存备用。
| 编号 No. |
采样地点 Location |
海拨 Altitude (m) |
地理位置 Geographical position |
| A | 松潘尕力台At Galitai in Songpan | 3 553 | N 32°55′44.1″,E 103°25′35.9″ |
| B | 红原安曲乡At Anqu Village in Hongyuan | 3 523 | N 32°43′14.1″,E 102°23′29.6″ |
| C | 马尔康梦笔山On Mount Mengbi in Barkam | 3 894 | N 31°41′23.3″,E 102°19′29.0″ |
| D | 小金县In Xiaojin County | 3 789 | N 31°41′12.3″,E 102°19′17.4″ |
| E | 小金县巴郎山On Mount Balang in Xiaojin County | 4 120 | N 30°55′52.8″,E 102°52′56.0″ |
| F | 小金县In Xiaojin County | 4 114 | N 31°42′23.9″,E 102°18′49.7″ |
细菌基因组DNA试剂盒、master mix,生工生物工程(上海)股份有限公司;Plant Genomic DNA Kit试剂盒,美国Sigma-aldrich公司。超净工作台,苏州安泰空气技术有限公司;生化培养箱,宁波莱福科技有限公司;涡旋混匀器,美国Scilogex公司;PCR扩增仪、电泳仪、琼脂糖凝胶成像系统,美国Bio-Rad公司。
1.3 分离培养样品采用10种培养基进行分离,琼脂含量均为2%。为抑制细菌、真菌生长,培养基添加终浓度为50 µg/mL萘啶酮酸和K2Cr2O7[12]。10种培养基为:HV培养基[13];棉籽糖组氨酸培养基[14];低营养矿物盐培养基[14];琥珀酸钠-天冬酰胺培养基(CaCl2 0.20 g,K2HPO4 0.60 g,KH2PO4 0.90 g,KCl 0.30 g,琥珀酸钠1.00 g,MgSO4 0.10 g,FeSO4 0.001 g,L-天冬酰胺0.20 g,蒸馏水定容至1 000 mL);高氏一号培养基[13];甲壳素培养基[14];海藻糖酪蛋白培养基(CaCO3 0.02 g,KNO3 0.10 g,K2HPO4 0.05 g,NaCl 0.50 g,MgSO4 0.50 g,FeSO4 0.000 1 g,海藻糖1.00 g,酪蛋白3.00 g,蒸馏水定容至1 000 mL);改良脯氨酸培养基[13];SCN培养基(KNO3 2.00 g,NaCl 0.02 g,FeSO4 0.01 g,可溶性淀粉10.0 g,酪蛋白0.30 g,蒸馏水定容至1 000 mL);葡萄糖-天冬酰胺培养基(K2HPO4 0.50 g,葡萄糖10.00 g,天冬酰胺0.50 g,蒸馏水定容至1 000 mL)。
采集的样品按照廖敏等[13]的方法进行表面消毒,处理好的植物样品一部分用于内生放线菌分离,另一部分用于总DNA提取。选取部分样品置于无菌研钵捣碎,取匀浆均匀涂布于分离培养基,28 ℃培养30 d。ISP4培养基[13]用于菌株纯化,菌株纯化后接种于ISP4斜面,于4 ℃保存备用。实验分离所得内生放线菌统一编码为SCAUE&培养基编号&植物编号&植株部位&菌株自然序号,免培养所得内生放线菌统一编码为SCAUE&植物编号&植株部位&菌株自然序号。其中植株部位根、茎、叶、花分别用R、S、L及F表示。
1.4 16S rDNA-RFLP分析放线菌DNA采用细菌基因组DNA试剂盒进行提取,16S rDNA的PCR引物使用8-27f (5′-CCGTCG ACGAGCTCAGAGTTTGATCCTGGCTCAG-3′)[15]和1523-1504r (5′-CCCGGGTACCAAGCTTAAGGAGG TGATCCAGCCGCA-3′)[15]。PCR反应体系(30 μL):master Mix 15 μL,正、反向引物(10 μmol/L)各0.5 μL,模板DNA 1 μL,ddH2O补足至30 μL。PCR反应条件[14]:94 ℃ 5 min;94 ℃ 1 min,56 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃ 10 min,4 ℃保存。PCR结束后用1%琼脂糖凝胶电泳检测,电压120 V,20 min。
选用限制性内切酶Hae Ⅲ及Hha Ⅰ分别对放线菌扩增产物进行酶切,反应体系(10 μL):Hae Ⅲ和Hha Ⅰ (10 U/µL)各0.5 µL,10×Buffer 1 μL,PCR产物5 μL,ddH2O补足至10 μL。充分混匀体系后于37 ℃反应8−16 h,反应结束后用2.5%琼脂糖凝胶进行电泳(100 V,4 h)。成像仪照相保存。酶切图谱分析处理采用Excel、NTSYS等软件,并运用非加权平均法(UPGMA)构建树状图谱[16]。
1.5 PCR-DGGE分析绿绒蒿各组织总DNA使用Plant Genomic DNA Kit试剂盒提取,做3次重复。植物组织总DNA采用Touch-down程序[17-18]进行2轮PCR扩增。第1轮PCR扩增体系为(30 μL):master mix 15 µL,引物F243 (5′-GGATGAGCCCGCGGCCTA-3′) (10 μmol/L)和R1378 (5′-GTAGTCATATGCTTGTC TC-3′) (10 μmol/L)[17]各0.5 µL,模板DNA 1 µL,ddH2O补足至30 µL。第2轮PCR扩增体系为(50 μL):master Mix 25 µL,引物F984GC (5′-GCAA CGCGAAGAACCTTAC-3′)(10 μmol/L)和R1378 (10 μmol/L)[17]各0.5 µL,第1轮PCR产物为模板1 µL,ddH2O补足至50 µL。完成PCR后进行琼脂糖凝胶电泳检测,条件同1.3。
采用Bio-Rad Dcode进行DGGE凝胶电泳,所用凝胶变性剂梯度为30%−60%。取第2次PCR产物20 µL,电泳缓冲液为1×TAE,50 V预跑30 min,之后150 V电泳6 h,温度60 ℃。电泳结束后采用银染法染色[19],并拍照保存。
绿绒蒿DGGE图谱多样性分析使用Quantity One软件,描述并比较其各组织内生放线菌多样性则引入多样性指数(H)、丰度(S)和均匀度(EH)等指标,计算公式如下[17]:
H=−∑ Pi·lnPi;
EH=H/Hmax=H/lnS。
Pi为绿绒蒿某样品中单一条带强度占该样品所有条带总强度的比率,S为绿绒蒿某样品总条带数。数据处理采用Excel 2003、SPSS 13.0统计软件。
1.6 菌株测序及构建系统发育树内生放线菌RFLP分析后选择代表菌株测序,同时切割DGGE较特异条带清洗、扩增、纯化、克隆[18]后进行测序,测序由成都擎科梓熙生物技术有限公司完成。采用Blast程序在GenBank中找出与所测序列相似度较高的相关序列,ClustalX多序列比对后,MEGA 6.0软件构建系统发育树(Neighbor-Joining)[20]。
2 结果与分析 2.1 影响绿绒蒿内生放线菌分离两因素及RFLP分析影响内生放线菌分离的因素较多,实验探究了培养基、植株部位两因素对内生放线菌分离的影响,结果见图 1。采用10种培养基共分离到32株内生放线菌,海藻糖酪蛋白培养基与改良脯氨酸培养基分离效果最佳,共分离到10株,占到31%,棉籽糖组氨酸培养基、高氏一号培养基次之,甲克素培养基分离效果最差。绿绒蒿不同部位内生放线菌分离情况差异也较大,根部分离数量最多,占到34%,花组织最少,总体为:根 > 叶 > 茎 > 花。

|
| 图 1 不同培养基及植株不同部位分离内生放线菌数量比较 Figure 1 Comparison of isolated endophytic actionbacteria with different culture media in different plant tissues |
|
|
将分离到的32株内生放线菌酶切聚类,NTSYS软件处理后见图 2。Hha Ⅰ/Hae Ⅲ酶切综合聚类结果显示,32株放线菌在70%相似度水平聚在一起,而在85%相似度水平分散为7个遗传类型,其中Ⅲ遗传类型占主导地位,含16株内生放线菌,占到总数的50%。Ⅲ遗传类型主要分离于海藻糖酪蛋白培养基,且大部分分离自植株根、叶,采样地是小金县巴郎山。

|
| 图 2 内生放线菌16S rDNA Hae Ⅲ/Hha Ⅰ酶切综合聚类图 Figure 2 Integrated clustering diagram for Hae Ⅲ/Hha Ⅰ digestion of endophytic actionbacteria 16S rDNA |
|
|
7株内生放线菌分别选自RFLP分析中7个不同遗传类群,代表菌株测序并构建系统发育树见图 3。代表菌株包括链霉菌属(Streptomyces)、北里孢菌属(Kitasatospora)和厄氏菌属(Oerskovia),与参比菌株一起大致聚为3簇。分离的内生放线菌中链霉菌属为优势属,占71%。酶切聚类及系统发育分析揭示了绿绒蒿内生放线菌较为丰富的多样性。菌株SCAUEjDS1比对序列只鉴定到纲;菌株SCAUEbEL3与SCAUEhFS1聚为一簇,与其他菌属亲缘关系较远。
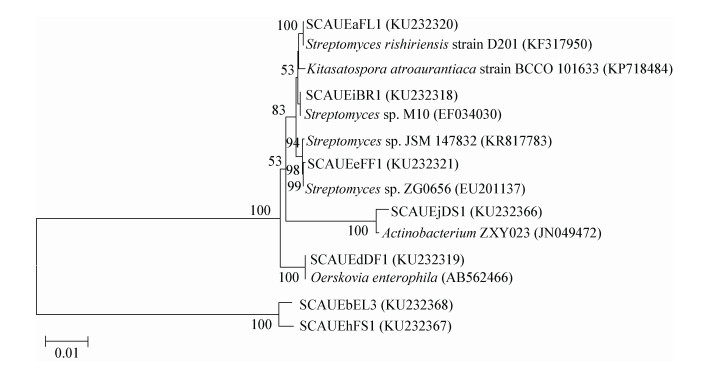
|
| 图 3 绿绒蒿内生放线菌代表菌株系统发育分析 Figure 3 Phylogenetic analysis of representive endophytic actinomycetes in Entire meconopsis 注:括号内序号为菌株登录号;分支点上的数字为系统发育树靴值;标尺长度代表遗传距离. Note: the serial numbers in the brackets were accession numbers of the strains. The numbers on the branch points of the phylogenetic tree represented bootstrap value. Scale length represented genetic distance |
|
|
从采自6个不同地理位置的绿绒蒿各组织样品分离出若干电泳条带(图 4)。由图 4可知,各泳道存在一定差异,体现在条带数量、迁移率、亮度、强度等方面,这也直观反映出绿绒蒿内生放线菌的遗传多样性。不同地理位置样品条带差异明显,而同一地理位置绿绒蒿根、茎、叶、花DGGE条带也各不相同,采集点B (红原安曲乡)绿绒蒿根部DGGE条带迁移率不同于茎、叶、花,茎和叶条带丰富、亮度大,而花则反之。

|
| 图 4 绿绒蒿内生放线菌DGGE图谱 Figure 4 Agarose gel electrophoresis of endophytic actinobacterial PCR-DGGE for different samples |
|
|
通过DGGE图谱比较分析绿绒蒿内生放线菌多样性指数(H)、丰度(S)和均匀度(EH),其中多样性指数是研究群落物种数及其个体数分布均匀度的综合指标[20]。比较发现(表 2),绿绒蒿内生放线菌多样性指数、丰度、均匀度在不同地理位置、不同植物组织都有所差异。采样点A (松潘尕力台)植株根部多样性指数、丰度、均匀度皆为最高:多样性指数2.72,与采样点B (红原安曲乡)、C (马尔康梦笔山)、D (小金县)植株根部多样性指数呈显著差异;其丰度为15,与其他5个采样点植株根部丰度呈显著差异。总体而言,同一采样点绿绒蒿各组织内生放线菌多样性指数为:A:茎 > 叶 > 根 > 花;B:花 > 叶 > 茎 > 根;C:叶 > 花 > 茎 > 根;D:花 > 茎 > 叶 > 根;E:茎 > 花 > 叶 > 根;F:花 > 茎 > 根 > 叶。而不同采样点绿绒蒿各组织内生放线菌多样性指数为:根:A > F > E > D > C > B;茎:A > D > E > B > C;叶:A > B > D > C > E > F;花:B > D > E > C > F > A。
| 样品 Samples |
多样性指数 Shannon-weaver index |
丰度 Richness |
均匀度 Evenness |
| Root A | 2.72±0.04c | 15±1c | 0.998 3±0.000 1c |
| Root B | 1.79±0.01a | 6±0a | 0.996 6±0.000 4bc |
| Root C | 1.91±0.27a | 7±2a | 0.994 9±0.002 6ab |
| Root D | 2.38±0.01b | 11±0b | 0.994 0±0.000 1a |
| Root E | 2.50±0.10bc | 12±1b | 0.997 5±0.000 1c |
| Root F | 2.53±0.09bc | 13±1b | 0.997 1±0.001 8c |
| Stem A | 2.87±0.06c | 18±1d | 0.998 2±0.000 3d |
| Stem B | 2.60±0.11b | 14±2b | 0.996 4±0.000 2bc |
| Stem C | 2.38±0.17a | 11±2a | 0.996 9±0.000 6c |
| Stem D | 2.78±0.03bc | 16±1cd | 0.994 6±0.000 6a |
| Stem E | 2.67±0.08bc | 15±1bc | 0.996 9±0.000 8c |
| Stem F | 2.22±0.16a | 9±2a | 0.995 8±0.000 1b |
| Leaf A | 2.83±0.06d | 17±1d | 0.998 2±0.000 4d |
| Leaf B | 2.72±0.10cd | 15±2cd | 0.996 9±0.000 2bc |
| Leaf C | 2.58±0.04bc | 13±1bc | 0.996 6±0.000 8b |
| Leaf D | 2.69±0.01cd | 15±0cd | 0.994 6±0.000 9a |
| Leaf E | 2.51±0.05ab | 12±1ab | 0.997 6±0.000 2cd |
| Leaf F | 2.38±0.19a | 11±2a | 0.996 7±0.000 3a |
| Flower A | 2.12±0.06a | 8±1a | 0.998 1±0.000 1c |
| Flower B | 3.10±0.05d | 22±1d | 0.997 6±0.000 2c |
| Flower C | 2.46±0.24b | 12±3b | 0.996 6±0.000 5b |
| Flower D | 2.80±0.03cd | 17±1c | 0.994 6±0.000 3a |
| Flower E | 2.58±0.09bc | 13±1b | 0.997 9±0.000 5c |
| Flower F | 2.30±0.32ab | 10±3ab | 0.996 7±0.000 6b |
构建系统发育树(图 5)显示,所有序列大致分为8个簇,大部分为红球菌属(Rhodococcus),占到70%,而非稀有放线菌链霉菌属(Streptomyces)仅2株(FF2-1和BR1-1)。与参比菌株独立成簇的菌株序列包括AS1-1、EL1-1、FS3-1和CL1-1,分别为棒杆菌属(Corynebacterium)、栖霉菌属(Mycetocola)、微杆菌属(Microbacterium)和假诺卡氏菌属(Pseudonocardia)。此外,序列BR2-1、BS1-1、BR1-2和BS3-1聚在一起,为诺卡氏菌属(Nocardia);ES1-1、ES3-1为微球菌属(Micrococcus)。尽管红球菌属占绝大多数,但比较发现,同是红球菌属DGGE条带的迁移率却差异较大,可见菌株亲缘关系与DGGE条带迁移率无必然联系。系统发育树较为丰富的分支表明了绿绒蒿内生放线菌丰富的多样性,同时绿绒蒿茎部体现出最丰富的放线菌资源。
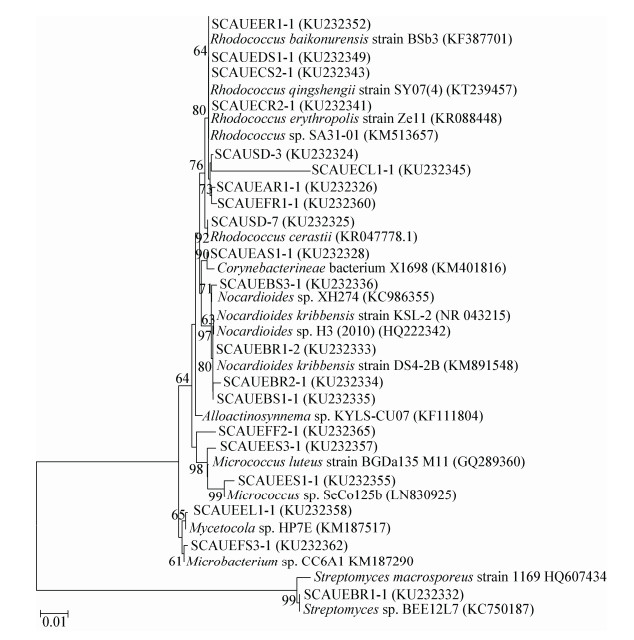
|
| 图 5 绿绒蒿内生放线菌DGGE条带系统发育分析 Figure 5 Phylogenetic analysis of gene sequences of endophytic actinobacteria in Entire meconopsis from DGGE bands |
|
|
四川省阿坝州地处青藏高原东南缘,海拔高、气温低,适合绿绒蒿的生长,而且该系统分布着种类繁多、数量极大的微生物群落。本研究采用10种不同培养基分离绿绒蒿内生放线菌,分离放线菌的数量与种属不如预期,可见绿绒蒿内生放线菌的分离存在一定难度,这可能与绿绒蒿的生长环境及培养条件等多方面因素有关,培养基的成分也至关重要。在本实验中高氏一号培养基分离放线菌效果较好,但是关统伟等[21]用不同盐浓度条件下的培养基对新疆罗布泊盐湖的土壤样品进行分离,结果证明高盐浓度的培养基比高氏一号培养基分离效果好,获得18个属的放线菌且包括8个放线菌新种;贾文文[22]采用不同培养基对南大西洋沉积物进行放线菌分离培养,棉籽糖组氨酸培养基和甘油-精氨酸培养基培养获得的放线菌株数量较多。可见不同生长环境的放线菌对培养基营养成分的需求有所不同。
从PCR-DGGE结果可看出,不同采样点的绿绒蒿各组织间内生放线菌种类、数量存在差异,整体看来花和茎的多样性最好。但可培养分离结果中,从根部分离得到的内生放线菌最多,一方面可能是因为可培养仅能分离极少数内生放线菌,另一方面可能是花、茎、叶器官比较脆弱,消毒过程导致较多内生放线菌死亡,所以根部最易分离到内生放线菌菌株。研究结果中,不同采样点的内生放线菌存在差异,其中采样点A (松潘尕力台)绿绒蒿的内生放线菌最丰富,可见从不同生境的角度进行探索,能挖掘更丰富的内生放线菌资源。
本研究通过可培养方法共分离出内生放线菌32株,包括3个遗传类型,其中链霉菌属是最主要的类群;DGGE结果中绿绒蒿内生放线菌包括8个菌属,其中红球菌属是最主要的类群,免培养的内生放线菌更丰富,这一方面是因为可培养结果仅代表极少绿绒蒿的内生放线菌群落。此外,由于培养条件及处理方法的局限性也导致分离结果不佳。从结果可看出,北里孢菌属和厄氏菌属在DGGE结果中未检测到,但在可培养中分离到了相关菌株,这可能因为实验材料的差异性、PCR扩增过程引物的偏好性、DGGE技术的局限性等因素影响[23]。在未来的研究中,需要采用更先进的技术和方法,更加充分地挖掘内生放线菌资源,从而为活性物质和药用功效的研究奠定基础。
| [1] |
Liu YS, Gao LY, Wang H, et al. The research status of Meconopsis punicea[J]. Xiandai Horticulture, 2012(6): 14-15. 刘玉珊, 高兰阳, 王辉, 等. 红花绿绒蒿的研究现状[J]. 现代园艺, 2012(6): 14-15. |
| [2] |
Zhou XF. Application of aquatic plants in garden water features[J]. Beijing Gardens, 2009, 25(1): 27-30. 周小芳. 水生植物在园林水景中的应用[J]. 北京园林, 2009, 25(1): 27-30. |
| [3] |
Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces Genetics[M]. Norwich: John Innes Foundation, 2000.
|
| [4] |
Bérdy J. Thoughts and facts about antibiotics: where we are now and where we are heading[J]. The Journal of Antibiotics, 2012, 65(8): 385-395. DOI:10.1038/ja.2012.27 |
| [5] |
Bérdy J. Bioactive microbial metabolites, a personal review[J]. Journal of Antibiotics, 2005, 58(1): 1-26. DOI:10.1038/ja.2005.1 |
| [6] |
Zou WX, Tan RX. Recent advances on endophyte research[J]. Acta Botanica Sinica, 2001, 43(9): 881-892. 邹文欣, 谭仁祥. 植物内生菌研究新进展[J]. 植物学报, 2001, 43(9): 881-892. |
| [7] |
Sturz AV, Christie BR, Matheson BG, et al. Biodiversity of endophytic bacteria which colonize red clover nodules, roots, stems and foliage and their influence on host growth[J]. Biology and Fertility of Soils, 1997, 25(1): 13-19. DOI:10.1007/s003740050273 |
| [8] |
Alain K, Querellou J. Cultivating the uncultured: limits, advances and future challenges[J]. Extremophiles, 2009, 13(4): 583-594. DOI:10.1007/s00792-009-0261-3 |
| [9] |
Jiang HC, Dong HL, Zhang GX, et al. Microbial diversity in water and sediment of Lake Chaka, an athalassohaline lake in Northwestern China[J]. Applied and Environmental Microbiology, 2006, 72(6): 3832-3845. DOI:10.1128/AEM.02869-05 |
| [10] |
Liu FF, Li C, Liu YQ, et al. Analysis of entophytic bacteria community structure in banana by PCR-DGGE technique[J]. Journal of Fruit Science, 2012, 29(2): 235-240. 刘菲菲, 李赤, 刘勇勤, 等. 香蕉植株内生细菌群落的PCR-DGGE分析[J]. 果树学报, 2012, 29(2): 235-240. |
| [11] |
Chen C, Zhao K, Li XL, et al. Diversity of rhizospheric and entophytic bacteria of Eupatorium adenophorum in nickel mine[J]. Journal of Sichuan University (Natural Science Edition), 2015, 52(6): 1387-1392. 陈诚, 赵柯, 李小林, 等. 镍矿区紫茎泽兰根际及内生细菌多样性研究[J]. 四川大学学报:自然科学版, 2015, 52(6): 1387-1392. |
| [12] |
Si MR, Xue QH, Lai HX. Studies on selection of the isolation medium for actinomycetes and inhabition methods to miscellaneous microorganism[J]. Microbiology China, 2004, 31(2): 61-65. 司美茹, 薛泉宏, 来航线. 放线菌分离培养基筛选及杂菌抑制方法研究[J]. 微生物学通报, 2004, 31(2): 61-65. |
| [13] |
Liao M, Zhang B, Fan ZH, et al. Diversity and anti-microbial activity of endophytic actinomycetes isolated from Stellera chamaejasme sampled in Aba, Sichuan[J]. Acta Prataculturae Sinica, 2016, 25(3): 43-51. 廖敏, 张波, 范中菡, 等. 阿坝地区狼毒内生放线菌多样性及抗菌活性[J]. 草业学报, 2016, 25(3): 43-51. DOI:10.11686/cyxb2015262 |
| [14] |
Zhao K. Study on diversity and antibiotic activity of endophytic and rhizospheric actinomycetes from medicinal plants in Panxi region[D]. Chengdu: Doctoral Dissertation of Sichuan Agricultural University, 2010 (in Chinese) 赵珂. 攀西地区药用植物内生及根际放线菌的多样性与抗菌活性研究[D]. 成都: 四川农业大学博士学位论文, 2010 http://cdmd.cnki.com.cn/Article/CDMD-10626-1011222371.htm |
| [15] |
Heuer H, Krsek M, Baker P, et al. Analysis of actinomycete communities by specific amplification of genes encoding 16S rRNA and gel-electrophoretic separation in denaturing gradients[J]. Applied and Environmental Microbiology, 1997, 63(8): 3233-3241. |
| [16] |
Li XL, Yuan HM, Qi SS, et al. Isolation and genetic diversity of the endophytic actinomycetes from Salvia miltiorrhiza Bge. and Polygonatum sibiricum Red[J]. Microbiology China, 2010, 37(9): 1341-1346. 李小林, 袁红梅, 戚珊珊, 等. 丹参、黄精内生放线菌的分离及遗传多样性分析[J]. 微生物学通报, 2010, 37(9): 1341-1346. |
| [17] |
Li XL, Gu YF, Zhang XP, et al. Nitrobacteria community and diversity in the rhizosphere soil of mature tobacco[J]. Scientia Agricultura Sinica, 2011, 44(12): 2462-2468. 李小林, 辜运富, 张小平, 等. 烟草成熟期根际硝化细菌种群的结构及其多样性[J]. 中国农业科学, 2011, 44(12): 2462-2468. DOI:10.3864/j.issn.0578-1752.2011.12.007 |
| [18] |
Zhang B, Li XL, Jiang HM, et al. Diversity and community structures of the endophytic actinomycetes isolated from Medicago ruthenica[J]. Acta Prataculturae Sinica, 2013, 22(5): 113-119. 张波, 李小林, 江华明, 等. 花苜蓿内生放线菌多样性及群落结构[J]. 草业学报, 2013, 22(5): 113-119. DOI:10.11686/cyxb20130513 |
| [19] |
Bassam BJ, Caetano-Anollés G, Gresshoff PM. Fast and sensitive silver staining of DNA in polyacrylamide gels[J]. Analytical Biochemistry, 1991, 196(1): 80-83. DOI:10.1016/0003-2697(91)90120-I |
| [20] |
Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
| [21] |
Guan TW, Teng Y, Che ZM, et al. Comparison of isolation media for actinobacteria from LopNur Salt Lake[J]. Biotechnology, 2013, 23(3): 56-60. 关统伟, 腾芸, 车振明, 等. 罗布泊盐湖放线菌分离培养基比较研究[J]. 生物技术, 2013, 23(3): 56-60. |
| [22] |
Jia WW. Isolated and diversity analysis of deep sea actinobacteria in the South Atlantic[D]. Harbin: Master's Thesis of Harbin Institute of Technology, 2013 (in Chinese) 贾文文. 南大西洋深海放线菌的分离与多样性分析[D]. 哈尔滨: 哈尔滨工业大学硕士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10213-1014003053.htm |
| [23] |
Sheng P, Yu YZ, Wang DS, et al. Analysis of bacterial diversities in different estuarine sediments of Poyang Lake by PCR-DGGE[J]. Jiangxi Science, 2015, 33(3): 312-317, 329. 圣平, 于一尊, 王东升, 等. 通过PCR-DGGE技术分析鄱阳湖不同河口底泥中微生物多样性[J]. 江西科学, 2015, 33(3): 312-317, 329. |
 2018, Vol. 45
2018, Vol. 45




