扩展功能
文章信息
- 范凯, 王栋, 徐岩
- FAN Kai, WANG Dong, XU Yan
- 华根霉液态培养生产脂肪酶不同形态菌体的转录组差异分析
- Transcriptome comparison of Rhizopus chinensis mycelia with different morphology in submerged fermentation for lipase production
- 微生物学通报, 2018, 45(1): 29-41
- Microbiology China, 2018, 45(1): 29-41
- DOI: 10.13344/j.microbiol.china.170198
-
文章历史
- 收稿日期: 2017-03-09
- 接受日期: 2017-05-12
- 优先数字出版日期(www.cnki.net): 2017-05-17
丝状真菌在食品、医药、化工和能源等众多领域有着广泛的应用[1-4]。在工业生产中,真菌的生长和代谢除了考虑遗传因素和物理化学等培养环境因素外,真菌形态也被认为是工业生产中重要的影响因素之一[5]。丝状真菌在液态培养中可形成分散的菌丝、聚集的菌球或菌块等不同形态,如黑曲霉(Aspergillus niger)和产黄青霉(Penicillium chrysogenum)等菌体形态的不同对特定产物的形成影响显著,相关研究已有较多报道[5-7]。一般认为,丝状真菌菌体形态差异与其病原性、代谢活性和酶的生产有关[8-10],有研究者提出聚集的菌体可以看作是一种分化的组织[11],特定的菌体形态可能更有利于目的产物的代谢和工业生产。但是相关研究目前仍主要是基于发酵液的流变特性、传质传氧等物理化学过程参数,关于真菌形态差异及其影响代谢的内在关系仍不清楚,缺乏从分子水平对其本质较为细致的阐述[12]。
随着后基因组时代的来临,各种系统生物学技术和方法为解决类似的复杂问题提供了可能。转录组分析技术作为一种可以定性和定量评价细胞特定时期所有转录样本的有效手段,已应用于丝状真菌的研究中[13-15],为明确基因功能及相关分子机制提供了非常有价值的信息。目前,具有较高动态范围和分辨率的RNA测序(RNA-Seq)技术已成为转录组分析揭示细胞基因表达模式的有力工具。RNA-Seq除了可以分析基因转录的相对丰度,还可以检测转录样本的绝对丰度,从而不仅可以确定差异最大的、最有影响的转录基因,还可以分析转录最多最快的基因[16-18]。转录组学分析常用方法包括基因功能富集分析、表达差异基因分析等。但Kr ber等[19]利用RNA-Seq技术,得到不同基因表达量RPKM (Reads Per Kilo bases per Million reads)数据,进而进行表达差异基因分析,得到了一些新的发现。
华根霉(Rhizopus chinensis) CCTCC M201021是从我国传统固态酿造白酒大曲中筛选得到的一株丝状真菌,不仅在传统酿造过程中具有重要功能,其发酵生产的脂肪酶在食品、生物香料、表面活性剂、生物柴油等领域也具有良好的工业应用潜力[20-22]。该菌株的全基因组序列已测序完成[23],并在美国Joint Genome Institute (JGI)的MycoCosm真菌基因组数据库发布(http://genome.jgi.doe.gov/Rhich1/Rhich1.home.html)。研究发现,华根霉在液态培养中也会出现菌体形态的明显差异,显著影响脂肪酶的生产水平,聚集态的菌体表现出较高的脂肪酶活性[24]。本研究基于RNA-Seq技术对华根霉两种典型形态,即聚集态和分散态菌体进行转录组分析,探究两种形态菌体的内在差异,分析菌体形态影响脂肪酶表达的可能机制,为研究丝状真菌华根霉的形态控制培养提供一定的线索。
1 材料与方法 1.1 材料 1.1.1 菌株华根霉(R. chinensis) CCTCC M201021[25]由江南大学酿造微生物学及应用酶学研究室筛选保藏。
1.1.2 培养基马铃薯-葡萄糖琼脂斜面培养基(g/L):马铃薯200.0,葡萄糖20.0,琼脂2.0,pH自然。液态培养基(20.0 mL/瓶) (g/L):鱼粉蛋白胨40.0,麦芽糖10.0,K2HPO4·3H2O 2.0,ΜgSO4·7H2O 0.5,橄榄油22.5,pH 5.5。
1.1.3 主要试剂和仪器Trizol total RNA extraction kit提取试剂盒,美国Invitrogen公司;1%琼脂糖凝胶、RNA酶抑制剂,美国Sigma公司;DNase I,美国Thermo公司;Illumina TruSeq™ RNA Sample Prep Kit,美国Illumina公司;AMPure XP beads试剂,美国BeckMan Coulter公司(上海);液氮及消耗器材,生工生物工程(上海)股份有限公司;其它试剂均为分析纯,国药集团化学试剂有限公司。
贝克曼Avanti J-E离心机,美国Beckman Coulter公司;立式冷冻离心机,美国Sigma公司;冷冻干燥机,美国Labconco公司;Nanodrop分光光度计,美国Nanodrop Technologies公司;Agilent 6820气相色谱仪,美国Agilent公司;测序仪为Illumina NextSeq™ 500 platform,测序公司为上海派森诺公司。
1.2 方法 1.2.1 培养方法及条件菌体培养参照Teng等[24]报道的方法进行操作。孢子培养及孢子悬浮液制备:将保藏的华根霉菌株划线到马铃薯-葡萄糖琼脂斜面培养基,30 ℃培养72 h。向斜面培养基中加入适量无菌生理盐水,用接种环收集菌体,将其转移到含有玻璃珠的锥形瓶中,并不断振荡使孢子分散。将锥形瓶中含有华根霉孢子的生理盐水通过两层擦镜纸过滤除去菌丝制成孢子悬浮液备用,用血球计数法计算孢子浓度。液态培养条件:20 mL液态培养基于150 mL三角瓶中,30 ℃、200 r/min培养70 h,菌体生长基本达到稳定期。以接种量不同来控制液体发酵菌体形态,分别以107个/mL和109个/mL孢子浓度接种培养,形成典型的聚集态华根霉和分散态华根霉菌体[24]。
1.2.2 脂肪酶酯化活性及生物量的测定菌体生物量的测定,将发酵所得菌体过滤收集,用无菌水洗涤,无菌纱布过滤,−70 ℃预冻6 h,再经冷冻干燥机冻干(约24 h)后得到冻干菌体,称重并计算生物量(g/L发酵液)。参照Wang等[26]报道的方法进行脂肪酶酯化活性(U/g生物量)测定。在正庚烷反应体系中脂肪酶催化辛酸与乙醇的酯化反应,利用气相色谱仪测定反应产物辛酸乙酯的含量。在测定条件下每分钟生成1 μmol辛酸乙酯所需的酶量定义为1个脂肪酶酯化活性单位。
1.2.3 RNA样品的制备参照熊毅等[27]报道的Trizol法提取样本RNA,操作方法略有改变,主要步骤包括:液态培养结束后,两种形态华根霉发酵液分别经12 000 r/min离心30 min,去除上清液收集菌体。将0.1 g菌体在研钵中用液氮研磨成粉状,迅速分装至盛有1.0 mL Trizol试剂的1.5 mL离心管中,分别加入10 μL DNase I与200 μL氯仿,提取菌体总RNA。使用1%琼脂糖凝胶电泳进行检测,用分光光度计检测提取的RNA,分别测定230、260、280 nm下的吸光度。
1.2.4 RNA-Seq测序及数据处理依据Dang等[28]报道的方法结合操作指南操作,Illumina TruSeq™ RNA Sample Prep Kit试剂盒构建cDNA库,PCR扩增构建序列库,使用Illumina NextSeqTM 500 Platform测序。使用Fastq格式存储原始数据,数据经过质量过滤处理后,使用FastQC来分析测序结果(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)。
1.2.5 基因比对及功能注释使用Bowtie2/tophat2 (http://tophat.cbcb.umd.edu/)软件将测序结果比对到华根霉基因组(GenBank登录号ANKS00000000)数据库(http://genome.jgi.doe.gov/Rhich1/Rhich1.home.html)[23]。将比对到的基因进行KOG (Eukaryotic orthologous groups)功能注释[29] (https://www.ncbi.nlm.nih.gov/COG/)。
1.2.6 转录组数据分析在转录组中,使用RPKM (Reads Per Kilobase per Million mapped reads)值来衡量每个基因在样本中的表达量[30]。通过HTSeq (http://www-huber.embl.de/users/anders/HTSeq)软件统计比对到基因上的数值,然后通过DESeq (http://www-huber.embl.de/users/anders/DESeq)软件分析表达差异基因,按照表达量倍数差异Fold change大于2.0或小于0.5及表达差异显著性P < 0.05进行筛选,确定差异表达基因。将差异表达基因RPKM值进行标准化处理转换成Base mean值[31-32](BM值)后,进行基因差异比较。
2 结果与讨论 2.1 不同形态华根霉菌体的发酵表现及转录组差异通过控制接种量,培养得到了华根霉聚集和分散两种典型的菌体形态。对这两种形态菌体分别进行生物量和脂肪酶酯化活性分析,结果如图 1所示。两种形态华根霉菌体的生物量分别为27 g/L和29 g/L,较为接近。但是聚集态菌体脂肪酶活性达到247.5 U/g,而分散态菌体的脂肪酶活性只有94.9 U/g,两者的酶活比约为2.6倍。这一结果与之前的研究报道较为一致[33],表明华根霉的菌体形态对脂肪酶的合成有显著影响。
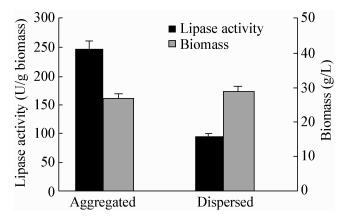
|
| 图 1 不同形态华根霉的发酵表现 Figure 1 Fermentation performance of the aggregated and dispersed mycelia of R. chinensis |
|
|
为了探究两种形态华根霉的内在差异,对这两种菌体分别提取RNA进行转录组测序,并将转录组测序结果与华根霉基因组进行比对,对所有基因表达进行初步分析。根据测序结果,在聚集态和分散态华根霉菌体中,分别有15 401和14 992个基因得到了表达。利用RPKM值对两种形态菌体的转录样本表达量进行比较[34],结果如表 1所示。聚集态和分散态华根霉菌体RPKM值范围分别为0.013−38 428和0.023−67 780。两者的平均RPKM值较为接近,分别为68.36和64.13。两者的RPKM中值均较低,分别1.52和2.28。通过RPKM的中值与平均值的比较,说明无论是聚集态还是分散态菌体,一半以上的转录基因此时都处在较低的转录水平,即大部分基因处于低水平表达。
| 形态 Morphology |
最大值 Maximum |
平均值 Average |
中值 Median |
| Aggregated mycelia of R. chinensis | 38 427.98 | 68.36 | 1.52 |
| Dispersed mycelia of R. chinensis | 67 780.26 | 64.13 | 2.28 |
进一步比较两种形态菌体转录组数据,按照表达量差异倍数(Fold change)大于2.0或小于0.5以及表达差异显著性P < 0.05确定差异转录基因,绘制火山图,结果如图 2所示。数据显示,两种形态菌体基因转录模式存在明显的差异,聚集态菌体共有865个显著上调表达的基因,而分散菌体只有134个基因显著上调表达。火山图分析表明,聚集态菌体中可能存在一些更为活跃的生物过程。
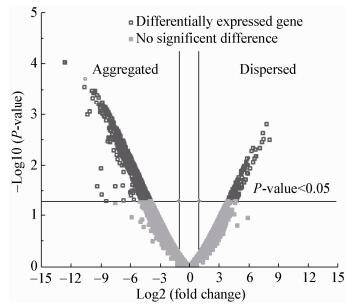
|
| 图 2 不同形态华根霉表达差异基因火山图 Figure 2 Volcano plot of differential gene transcription for R. chinensis with different morphology |
|
|
为了进一步探究两种形态华根霉基因表达差异,根据RPKM值分别统计了两种形态华根霉表达量最高的前20个基因,这些高表达基因可能与细胞中最活跃的生物学过程相关。对这些基因按KOG功能注释进行分类和功能分析,并按表达量从高到低分别列于表 2、3中。在聚集和分散的华根霉菌体表达量最高的20个基因中,分别有15个和12个基因具有KOG注释。
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
RPKM值 RPKM |
| Information Storage and Processing | ||
| Translation, ribosomal structure and biogenesis (J) | ||
| A12821 | 60S ribosomal protein L44 | 38 427.98 |
| A05628 | 60S ribosomal protein L31 | 18 985.09 |
| A04276 | 60S ribosomal protein L31 | 17 709.55 |
| A06801 | 60S ribosomal protein L14/L17/L23 | 15 436.65 |
| A11751 | 60S ribosomal protein L34 | 13 212.06 |
| A14552 | 40S ribosomal protein S25 | 12 901.19 |
| A09437 | 60S ribosomal protein L34 | 12 278.05 |
| A06355 | 40S ribosomal protein S20 | 10 628.63 |
| A03750 | 60S ribosomal protein L23 | 10 192.61 |
| A10301 | 60S ribosomal protein L27 | 9 982.22 |
| A16564 | 60S acidic ribosomal protein P2 | 9 616.07 |
| A07218 | 60S ribosomal protein L14 | 8 949.74 |
| Metabolism | ||
| Carbohydrate transport and metabolism (G) | ||
| A16838 | Glyceraldehyde 3-phosphate dehydrogenase | 12 964.82 |
| A11039 | Glyceraldehyde 3-phosphate dehydrogenase | 11 135.12 |
| Secondary metabolites biosynthesis, transport and catabolism (Q) | ||
| A04505 | Alcohol dehydrogenase, class V | 16 326.61 |
| Function unknown | ||
| A08829 | Unknown | 37 660.96 |
| A01509 | Unknown | 32 101.34 |
| A07922 | Unknown | 23 668.11 |
| A05703 | Unknown | 20 480.22 |
| A01519 | Unknown | 16 297.08 |
| 注:a:类别指KOG功能类别. Note: a: KOG function category. |
||
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
RPKM值 RPKM |
| Information Storage and Processing | ||
| Translation, ribosomal structure and biogenesis (J) | ||
| A12821 | 60S ribosomal protein L44 | 20 480.14 |
| A05628 | 60S ribosomal protein L31 | 11 555.25 |
| A04276 | 60S ribosomal protein L31 | 11 033.60 |
| A10301 | 60S ribosomal protein L27 | 10 134.83 |
| A09437 | 60S ribosomal protein L34 | 9 676.16 |
| A11751 | 60S ribosomal protein L34 | 9 234.64 |
| A06582 | 60S ribosomal protein L13 | 8 761.11 |
| Metabolism | ||
| Carbohydrate transport and metabolism (G) | ||
| A12858 | Chitinase | 11 074.77 |
| A08357 | Glyceraldehyde 3-phosphate dehydrogenase | 8 522.19 |
| Cellular Process and Signaling | ||
| Signal transduction mechanisms (T) | ||
| A05762 | Circadian clock protein period | 15 620.14 |
| A04753 | Circadian clock protein period | 10 274.76 |
| Poorly Characterized | ||
| General function prediction only (R) | ||
| A01860 | Protein involved in thiamine biosynthesis and DNA damage tolerance | 8 545.15 |
| Function unknown | ||
| A06840 | Unknown | 67 780.25 |
| A16951 | Unknown | 53 498.96 |
| A10486 | Unknown | 29 658.33 |
| A00017 | Unknown | 28 337.12 |
| A16305 | Unknown | 13 402.58 |
| A14531 | Unknown | 9 627.28 |
| A09855 | Unknown | 9 299.04 |
| A07019 | Unknown | 8 329.13 |
| 注:a:类别指KOG功能类别. Note: a: KOG function category. |
||
对比两种形态菌体高表达基因,发现这些基因中只有4个是相同的,此外均为不同的基因。由于高表达基因所参与的可能是较活跃的细胞生理活动,说明这两种形态菌体中活跃的细胞生理活动大部分是不相同的。4个相同的基因分别是A12821、A05628、A04276、A11751,都属于“翻译,核糖体结构与形成(Translation,ribosomal structure and biogenesis)”类别中,是编码不同核糖体蛋白(Ribosomal protein)的基因,可能与华根霉细胞基本的生物过程有关。但是在聚集菌体中发现有更多的高表达核糖体蛋白基因(12个)。一般研究认为,核糖体蛋白的主要功能是构成核糖体,参与蛋白质的翻译合成。也有研究报道,核糖体蛋白参与了除合成蛋白质外的其他细胞活动[35-36],包括DNA修复、RNA伴侣活动、调节细胞分化、抵抗病原机制等。聚集菌体中多种核糖体蛋白的高水平表达可能增强了菌体某些蛋白质的合成,也可能执行了其他一些与菌体形态分化相关的功能,值得进一步研究。
聚集和分散菌体中各有2个高表达基因属于“碳水化合物转运与代谢(Carbohydrate transport and metabolism)”类别。聚集菌体中的这两个基因(A16838与A11039)同为3-磷酸甘油醛脱氢酶(表 2);分散菌体中的基因A08357也为3-磷酸甘油醛脱氢酶,另一个A12858为几丁质酶(表 3)。3-磷酸甘油醛脱氢酶在碳水化合物代谢糖酵解中起着重要作用,此外该酶也参与转录活化、细胞凋亡、代谢调节和囊泡运输等细胞活动[37]。3-磷酸甘油醛脱氢酶不同基因在不同形态菌体中的高表达,可能与其不同的功能有关。此外,由于几丁质是真菌细胞壁的重要组分,几丁质酶在真菌细胞形态重塑中起着重要作用[38]。分散菌体中几丁质酶的高表达,可能与华根霉菌体形态的分化有关。分散菌体中还有2个高水平表达的基因A04753和A05762与“信号传导机制(Signal transduction mechanisms)”有关,均为生物钟蛋白(Circadian clock protein period),真菌中的生物钟蛋白机制较为复杂,目前其作用尚不清楚,但这些基因的高表达可能暗示分散菌体存在较为复杂的信号调控机制。
以上功能分析结果表明,不仅不同菌体形态的高表达量基因有明显不同,且功能相同的蛋白在不同形态下也多是由不同基因表达,可能承担着不同的作用。此外还有一些表达量最高的基因功能并不明确,如聚集菌体表达量最高的前5个基因中的4个(表 2)和分散菌体表达量最高的前4个基因(表 3),功能均为未知。这说明与菌体形态分化相关的一些重要基因及其功能目前尚不清楚,相关研究亟需开展。
2.3 不同形态华根霉显著差异表达的基因分析在分析高表达基因的基础上,接着对显著差异表达基因,即表达差异倍数最大的基因进行分析。对表达差异显著性P < 0.05的差异基因,将其RPKM值进行标准化转换成Base mean (BM)值进行比较,按照差异倍数高低,分别选取聚集态和分散态菌体上调表达倍数最大的20个基因进行分析,结果如表 4、5所示。
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
聚集态BM值 Base mean of the aggregated mycelia |
分散态BM值 Base mean of the dispersed mycelia |
差异倍数b Fold changeb |
| Metabolism | ||||
| Energy production and conversion (C) | ||||
| A11119 | NADH-dehydrogenase (ubiquinone) | 5 034.82 | 5.590 | 899.87 |
| A03509 | Monooxygenase involved in coenzyme Q (ubiquinone) biosynthesis | 5 291.12 | 10.250 | 515.82 |
| A17422 | Monooxygenase involved in coenzyme Q (ubiquinone) biosynthesis | 3 337.24 | 6.520 | 511.25 |
| Carbohydrate transport and metabolism (G) | ||||
| A15746 | Beta-1, 6-N-acetylglucosaminyltransferase, contains WSC domain | 879.35 | 0.930 | 943.00 |
| A03893 | Beta-1, 6-N-acetylglucosaminyltransferase, contains WSC domain | 2 078.27 | 2.790 | 742.90 |
| A13906 | Chitinase | 4 473.97 | 6.520 | 685.40 |
| A11039 | Glyceraldehyde 3-phosphate dehydrogenase | 175 347.08 | 266.690 | 657.47 |
| Lipid transport and metabolism (I) | ||||
| A12841 | Squalene monooxygenase | 18 913.57 | 14.920 | 1 267.65 |
| Inorganic ion transport and metabolism (P) | ||||
| A12511 | Na+/dicarboxylate, Na+/tricarboxylate and phosphate transporters | 1 089.53 | 1.860 | 584.20 |
| A15169 | Na+/dicarboxylate, Na+/tricarboxylate and phosphate transporters | 542.62 | 0.930 | 581.90 |
| Poorly Characterized | ||||
| General function prediction only (R) | ||||
| A02512 | FOG: Zn-finger | 5 715.78 | 0.930 | 6 129.50 |
| A14573 | FOG: Zn-finger | 5 889.51 | 3.730 | 1 578.95 |
| Function unknown | ||||
| A03347 | FOG: Low-complexity | 14 447.11 | 13.050 | 1 106.62 |
| A03801 | Unknown | 5 840.18 | 0.932 | 6 262.90 |
| A04837 | Unknown | 1 382.29 | 0.932 | 1 482.35 |
| A15644 | Unknown | 793.56 | 0.932 | 851.00 |
| A04157 | Unknown | 1 412.32 | 1.860 | 757.27 |
| A01824 | Unknown | 1 765.13 | 2.790 | 630.96 |
| A15472 | Unknown | 489.00 | 0.930 | 524.40 |
| A12438 | Unknown | 483.64 | 0.930 | 518.65 |
| 注:a:类别指KOG功能类别;b:差异倍数指聚集态与分散态菌体BM值之比. Note: a: KOG function category; b: Fold change is the ratio of BM value between the aggregated and dispersed of R. chinensis. |
||||
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
聚集态BM值 Base mean of the aggregated mycelia |
分散态BM值 Base mean of the dispersed mycelia |
差异倍数b Fold changeb |
| Cellular Process and Signaling | ||||
| Posttranslational modification, protein turnover, chaperones (O) | ||||
| A13911 | Predicted E3 ubiquitin ligase | 1.070 | 96.98 | 0.011 0 |
| Signal transduction mechanisms (T) | ||||
| A07819 | Tyrosine kinase specific for activated (GTP-bound) p21cdc42Hs | 2.140 | 134.28 | 0.015 0 |
| A14224 | Protein tyrosine phosphatase | 2.140 | 126.82 | 0.016 0 |
| Cytoskeleton (Z) | ||||
| A16109 | Profilin | 16.085 | 973.53 | 0.016 0 |
| Information Storage and Processing | ||||
| Chromatin structure and dynamics (B) | ||||
| A16862 | Chromatin-associated protein Dek and related proteins, contains SAP DNA binding domain | 19.300 | 1 588.98 | 0.012 0 |
| Metabolism | ||||
| Energy production and conversion (C) | ||||
| A01943 | NADP/FAD dependent oxidoreductase | 1.070 | 80.19 | 0.013 0 |
| Amino acid transport and metabolism (E) | ||||
| A14421 | Alpha-isopropylmalate synthase/homocitrate synthase | 1.070 | 187.43 | 0.005 7 |
| A02789 | Glutamate decarboxylase/sphingosine phosphate lyase | 91.150 | 5 305.95 | 0.017 0 |
| Lipid transport and metabolism (I) | ||||
| A12883 | Acyl-CoA synthetase | 2.140 | 377.66 | 0.005 6 |
| A09120 | Acyl-CoA synthetase | 2.140 | 222.86 | 0.009 6 |
| A11247 | Acyl-CoA synthetase | 4.280 | 398.17 | 0.010 0 |
| Secondary metabolites biosynthesis, transport and catabolism (Q) | ||||
| A10500 | Sorbitol dehydrogenase | 37.530 | 10 624.95 | 0.003 5 |
| A12160 | Sorbitol dehydrogenase | 2.140 | 497.02 | 0.004 3 |
| Function unknown | ||||
| A05981 | Unknown | 1.070 | 207.94 | 0.005 1 |
| A01258 | Unknown | 1.070 | 154.79 | 0.006 9 |
| A10101 | Unknown | 1.070 | 118.42 | 0.009 0 |
| A00590 | Unknown | 1.070 | 117.49 | 0.009 1 |
| A10425 | Unknown | 1.070 | 110.03 | 0.009 7 |
| A03006 | Unknown | 27.880 | 1 866.87 | 0.014 0 |
| A04737 | Unknown | 1.070 | 67.14 | 0.015 0 |
| 注:a:类别指KOG功能类别;b:差异倍数指聚集态与分散态菌体BM值之比. Note: a: KOG function category; b: Fold change is the ratio of BM value between the aggregated and dispersed of R. chinensis. |
||||
在聚集态菌体上调表达差异倍数最大的20个基因中,有10个具有KOG功能注释的基因都属于“代谢”类别(表 4),主要涉及能量、碳水化合物和无机离子等的代谢与转运。其中,几丁质酶基因A13906也得到了较高的差异表达,但与表 3中分散态菌体高表达的几丁质酶A12858并不相同,说明在细胞壁分解与重塑中两种形态菌体可能受到了不同的调控。在分散态菌体上调表达差异倍数最大的20个基因中,也有8个具有KOG功能注释的基因属于“代谢”类别(表 5),但主要涉及氨基酸、脂质和次级代谢等过程,与聚集态菌体代谢差异比较明显。此外,除了与代谢相关的差异基因外,分散态菌体另有4个具有KOG功能注释的基因属于“细胞过程与信号”类别,包括参与翻译后修饰的泛素连接酶基因A13911,两个参与信号转导机制的酪氨酸激酶基因A07819与酪氨酸磷酸酶基因A14224,以及作为肌动蛋白结合蛋白的细胞骨架前纤维蛋白Profilin基因A16109[39]。这些结果再次表明,分散菌体可能受到了更为复杂的信号传导和调控。而前纤维蛋白Profilin在细胞移动和形态变化过程中也有重要作用[39],可能影响了分散菌体在培养基中的状态。
此外,在这些高差异表达基因中,聚集态和分散态菌体分别有8个和7个基因功能未知,且有一些表达差异非常大,这些基因功能还需要进一步研究。
除了表达差异倍数最大的基因,进一步对这两种形态菌体独有表达基因进行了分析。根据转录组测序结果,聚集态和分散态菌体独有表达的基因分别为218个和26个,其中有KOG功能注释的基因分别为114个和12个。聚集态菌体大量的独有表达基因说明这种形态的菌体中可能存在更为复杂的生理活动。分别对这两种形态华根霉的独有表达基因的表达量前20进行分类分析,结果如表 6和表 7所示。
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
RPKM值 RPKM |
| Metabolism | ||
| Amino acid transport and metabolism (E) | ||
| A07760 | H+/oligopeptide symporter | 68.41 |
| Coenzyme transport and metabolism (H) | ||
| A12057 | Coproporphyrinogen Ⅲ oxidase CPO/HEM13 | 32.63 |
| Energy production and conversion (C) | ||
| A14739 | NADP+-dependent malic enzyme | 23.56 |
| Cell cycle control, cell division, chromosome partitioning (D) | ||
| A02897 | G-protein alpha subunit (small G protein superfamily) | 20.44 |
| Information Storage and Processing | ||
| Transcription (K) | ||
| A05800 | Regulator of arginine metabolism and related MADS box-containing transcription factors | 31.04 |
| Cellular Process and Signaling | ||
| Cell wall/membrane/envelope biogenesis (M) | ||
| A00080 | Glucosamine-phosphate N-acetyltransferase | 19.10 |
| Function unknown | ||
| A13955 | Unknown | 137.36 |
| A04678 | Unknown | 106.68 |
| A04897 | Unknown | 105.42 |
| A00520 | Unknown | 85.89 |
| A01816 | Unknown | 80.54 |
| A01033 | Unknown | 69.94 |
| A10735 | Unknown | 43.08 |
| A13787 | Unknown | 30.45 |
| A00868 | Unknown | 27.82 |
| A12428 | Unknown | 26.78 |
| A00934 | Unknown | 26.22 |
| A00301 | Unknown | 19.40 |
| A00685 | Unknown | 19.33 |
| A05480 | Unknown | 16.90 |
| 注:a:类别指KOG功能类别. Note: a: KOG function category. |
||
| 基因ID及类别a Gene ID and categorya |
功能描述 KOG description |
RPKM值 RPKM |
| Information Storage and Processing | ||
| Transcription (K) | ||
| A04197 | Transcriptional activator FOSB/c-Fos and related bZIP transcription factors | 8.56 |
| Replication, recombination and repair (L) | ||
| A12580 | DNA repair and recombination protein RAD52/RAD22 | 4.70 |
| A17356 | DNA repair and recombination protein RAD52/RAD22 | 2.75 |
| Cellular Process and Signaling | ||
| Signal transduction mechanisms (T) | ||
| A04979 | FOG: FHA domain | 2.31 |
| Metabolism | ||
| Lipid transport and metabolism (Q) | ||
| A13259 | Cytochrome P450 CYP4/CYP19/CYP26 subfamilies | 2.03 |
| Poorly Characterized | ||
| Function unknown | ||
| A09548 | Uncharacterized conserved protein | 4.29 |
| A05597 | Unknown | 112.01 |
| A13661 | Unknown | 15.38 |
| A02239 | Unknown | 13.20 |
| A11719 | Unknown | 11.15 |
| A01906 | Unknown | 10.42 |
| A14838 | Unknown | 7.95 |
| A06754 | Unknown | 7.25 |
| A14241 | Unknown | 6.22 |
| A06706 | Unknown | 5.00 |
| A17133 | Unknown | 4.33 |
| A13355 | Unknown | 4.21 |
| A15494 | Unknown | 3.89 |
| A10487 | Unknown | 2.41 |
| A17621 | Unknown | 2.03 |
| 注:a:类别指KOG功能类别. Note: a: KOG function category. |
||
无论是聚集态还是分散态菌体,大部分独有表达基因RPKM值均比较低(低于RPKM平均值),特别是分散态菌体,低表达量表明这些独有表达基因可能在菌体形态分化中作用不很突出。而且根据基因功能注释,这些独有表达基因大部分(均为14个)均为功能未知,特别是一些表达量相对最高的独有基因。
参照基因功能注释,在聚集态华根霉中有2个高表达独有基因,A02897和A00080,可能为研究形态差异机制提供了一些线索。在“代谢”类别中,A02897作为G蛋白α亚基,可能参与了调控酶代谢、离子通道、转运蛋白等诸多细胞活动[40]。磷酸氨基葡萄糖N乙酰转移酶基因A00080,可能参与了与细胞壁相关的氨基糖代谢,与细胞的特定形态形成或许相关。此外,两个不同的转录因子基因A05800和A04197分别在聚集和分散菌体中独有表达。总体而言,聚集态菌体独有表达基因在基因种类和表达量上都要明显高于分散态菌体,暗示聚集态菌体中可能存在更为复杂的生理活动,这可能是细胞分化的结果,也可能与菌体聚集造成细胞微环境的改变,从而对菌体产生影响有关。
2.4 不同形态华根霉脂肪酶基因表达分析针对华根霉脂肪酶,分析两种形态菌体中对应的两个脂肪酶基因A06672与A07506[41]的表达量,结果如表 8所示。数据表明,两个脂肪酶基因在聚集态菌体中的表达量都远高于分散态菌体,差异表达倍数均上百倍。前期华根霉脂肪酶蛋白质分析也表明,聚集态菌体脂肪酶表达量远高于分散态菌体[24]。在米根霉脂肪酶(R. oryzae lipase)的研究中,有报道认为菌体形态主要影响脂肪酶在细胞中的定位[42]。本研究结果表明,华根霉脂肪酶在聚集菌体中较高的生产水平可能与脂肪酶基因的高水平转录有关,与米根霉脂肪酶的分泌表达存在差异。
| 脂肪酶基因 Lipase gene |
聚集态BM值 Base mean of the aggregated mycelia |
分散态BM值 Base mean of the dispersed mycelia |
差异倍数b Fold changeb |
| A06672 | 4 270.21 | 16.78 | 254.48 |
| A07506 | 11 398.33 | 70.87 | 160.83 |
| 注:b:差异倍数指聚集态与分散态菌体BM值之比. Note: b: Fold change is the ratio of BM value between the aggregated and dispersed of R. chinensis. |
|||
本研究对华根霉液态培养生产脂肪酶过程中形成的聚集态和分散态菌体进行了RNA-Seq转录组分析,以考察不同形态菌体及其细胞代谢在转录组水平的内在差异。测序结果显示,两种形态菌体基因转录模式存在明显的差异。分别对不同形态菌体转录表达量最高的基因和差异表达最显著的基因分析表明,不仅不同菌体形态的基因转录有明显不同,而且功能相同的蛋白在不同菌体形态下也多是由不同基因表达,可能承担着不同的作用。聚集态菌体高表达基因主要为不同类别的核糖体蛋白,这些基因除了参与蛋白质的翻译合成,也可能执行了其他一些与菌体形态分化相关的功能。而分散态菌体高表达基因中还有可能与细胞形态相关的几丁质酶及与信号传导相关的基因。在两种形态菌体显著差异表达的基因分析中,除了涉及代谢的基因有明显差异外,分散态菌体中还有一些涉及“细胞过程与信号”的显著差异表达基因。进一步对这两种形态菌体独有表达基因进行分析,独有表达基因总体表达量均较低,但聚集态菌体独有表达基因在基因种类和表达量上都要明显高于分散态菌体。针对华根霉脂肪酶基因转录分析表明,聚集菌体较高的脂肪酶生产水平可能首先与脂肪酶基因的高水平转录有关。综合以上结果,华根霉聚集态菌体中可能存在更为复杂的生理过程,这或许是细胞分化的结果,也可能是由于菌体聚集造成细胞微环境的改变,华根霉细胞针对环境的变化产生的一种响应。而分散菌体由于散布于环境中,缺少菌体聚集的保护,对于环境影响更为敏感,信号传导可能更为复杂。本研究结果为了解丝状真菌形态分化的内在机制及其影响提供了一些线索。此外,研究中还有相当一部分高表达基因和显著差异表达基因的功能并不明确,表明与菌体形态分化相关的一些重要基因及其功能信息还相当缺乏,值得进一步研究。
| [1] |
Pérez-Rodríguez N, Oliveira F, Pérez-Bibbins B, et al. Optimization of xylanase production by filamentous fungi in solid-state fermentation and scale-up to horizontal tube bioreactor[J]. Applied Biochemistry and Biotechnology, 2014, 173(3): 803-825. DOI:10.1007/s12010-014-0895-1 |
| [2] |
Hevekerl A, Kuenz A, Vorlop KD. Filamentous fungi in microtiter plates-an easy way to optimize itaconic acid production with Aspergillus terreus[J]. Applied Microbiology and Biotechnology, 2014, 98(16): 6983-6989. DOI:10.1007/s00253-014-5743-2 |
| [3] |
Dufossé L, Fouillaud M, Caro Y, et al. Filamentous fungi are large-scale producers of pigments and colorants for the food industry[J]. Current Opinion in Biotechnology, 2014, 26: 56-61. DOI:10.1016/j.copbio.2013.09.007 |
| [4] |
Gibbs PA, Seviour RJ, Schmid F. Growth of filamentous fungi in submerged culture: problems and possible solutions[J]. Critical Reviews in Biotechnology, 2000, 20(1): 17-48. |
| [5] |
Papagianni M. Fungal morphology and metabolite production in submerged mycelial processes[J]. Biotechnology Advances, 2004, 22(3): 189-259. DOI:10.1016/j.biotechadv.2003.09.005 |
| [6] |
Papagianni M, Mattey M, Kristiansen B. The influence of glucose concentration on citric acid production and morphology of Aspergillus niger in batch and culture[J]. Enzyme and Microbial Technology, 1999, 25(8/9): 710-717. |
| [7] |
Papagianni M. Fungal morphology[A]//Kristiansen B, Mattey M, Linden J. Citric Acid Biotechnology[M]. London: Taylor and Francis, 1999: 69-84
|
| [8] |
Papagianni M. Quantification of the fractal nature of mycelial aggregation in Aspergillus niger submerged cultures[J]. Microbial Cell Factories, 2006, 5: 5. DOI:10.1186/1475-2859-5-5 |
| [9] |
Shi XZ, Sha Y, Kaminskyj S. Aspergillus nidulans hypA regulates morphogenesis through the secretion pathway[J]. Fungal Genetics and Biology, 2004, 41(1): 75-88. DOI:10.1016/j.fgb.2003.09.004 |
| [10] |
Liao W, Liu Y, Chen SL. Studying pellet formation of a filamentous fungus Rhizopus oryzae to enhance organic acid production[A]//Mielenz JR, Klasson KT, Adney WS, et al. Applied Biochemistry and Biotechnology[M]. Totowa, NJ: Humana Press Inc., 2007: 689-701
|
| [11] |
Papagianni M, Moo-Young M. Protease secretion in glucoamylase producer Aspergillus niger cultures: fungal morphology and inoculum effects[J]. Process Biochemistry, 2002, 37(11): 1271-1278. DOI:10.1016/S0032-9592(02)00002-X |
| [12] |
Grimm LH, Kelly S, Krull R, et al. Morphology and productivity of filamentous fungi[J]. Applied Microbiology and Biotechnology, 2005, 69(4): 375-384. DOI:10.1007/s00253-005-0213-5 |
| [13] |
Machida M, Asai K, Sano M, et al. Genome sequencing and analysis of Aspergillus oryzae[J]. Nature, 2005, 438(7071): 1157-1161. DOI:10.1038/nature04300 |
| [14] |
Pel HJ, de Winde JH, Archer DB, et al. Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88[J]. Nature Biotechnology, 2007, 25(2): 221-231. DOI:10.1038/nbt1282 |
| [15] |
Meyer V. Genetic engineering of filamentous fungi-progress, obstacles and future trends[J]. Biotechnology Advances, 2008, 26(2): 177-185. DOI:10.1016/j.biotechadv.2007.12.001 |
| [16] |
Conesa A, Madrigal P, Tarazona S, et al. A survey of best practices for RNA-Seq data analysis[J]. Genome Biology, 2016, 17: 13. DOI:10.1186/s13059-016-0881-8 |
| [17] |
Meijueiro ML, Santoyo F, Ramírez L, et al. Transcriptome characteristics of filamentous fungi deduced using high-throughput analytical technologies[J]. Briefings in Functional Genomics, 2014, 13(6): 440-450. DOI:10.1093/bfgp/elu033 |
| [18] |
Levin JZ, Yassour M, Adiconis X, et al. Comprehensive comparative analysis of strand-specific RNA sequencing methods[J]. Nature Methods, 2010, 7(9): 709-715. DOI:10.1038/nmeth.1491 |
| [19] |
Kröber M, Verwaaijen B, Wibberg D, et al. Comparative transcriptome analysis of the biocontrol strain Bacillus amyloliquefaciens fzb42 as response to biofilm formation analyzed by RNA sequencing[J]. Journal of Biotechnology, 2016, 231: 212-223. DOI:10.1016/j.jbiotec.2016.06.013 |
| [20] |
Xu Y, Wang D, Mu XQ, et al. Efficient esterification of sorbitan oleate by lipase in a solvent-free system[J]. Journal of the American Oil Chemists' Society, 2003, 80(7): 647-651. DOI:10.1007/s11746-003-0753-6 |
| [21] |
Shan TY, Wang D, Xu Y, et al. Kinetic resolution of 2-octanol by esterification with mycelium-bound lipase from Rhizopus chinensis[J]. Chinese Journal of Catalysis, 2008, 29(4): 403-408. 单天宇, 王栋, 徐岩, 等. 华根霉菌丝体结合脂肪酶催化脂合成动力学拆分2-辛醇[J]. 催化学报, 2008, 29(4): 403-408. |
| [22] |
He Q, Xu Y, Teng Y, et al. Biodiesel production catalyzed by whole-cell lipase from Rhizopus chinensis[J]. Chinese Journal of Catalysis, 2008, 29(1): 41-46. 贺芹, 徐岩, 滕云, 等. 华根霉全细胞脂肪酶催化合成生物柴油[J]. 催化学报, 2008, 29(1): 41-46. |
| [23] |
Wang D, Wu R, Xu Y, et al. Draft genome sequence of Rhizopus chinensis CCTCC M201021, used for brewing traditional chinese alcoholic beverages[J]. Genome Announcements, 2013, 1(2): e00195-12. |
| [24] |
Teng Y, Xu Y, Wang D. Changes in morphology of Rhizopus chinensis in submerged fermentation and their effect on production of mycelium-bound lipase[J]. Bioprocess and Biosystems Engineering, 2009, 32(3): 397-405. DOI:10.1007/s00449-008-0259-8 |
| [25] |
Xu Y, Wang D, Mu XQ, et al. Biosynthesis of ethyl esters of short-chain fatty acids using whole-cell lipase from Rhizopus chinesis CCTCC M201021 in non-aqueous phase[J]. Journal of Molecular Catalysis B: Enzymatic, 2002, 18(1/3): 29-37. |
| [26] |
Wang D, Xu Y, Teng Y. Synthetic activity enhancement of membrane-bound lipase from Rhizopus chinensis by pretreatment with isooctane[J]. Bioprocess and Biosystems Engineering, 2007, 30(3): 147-155. DOI:10.1007/s00449-006-0097-5 |
| [27] |
Xiong Y, Guo YC, Zhang Z. Study on methods for RNA extraction from Auricularia auricular[J]. Acta Agriculturae Zhejiangensis, 2014, 26(2): 456-460. 熊毅, 郭彦萃, 张志. 木耳总RNA提取方法研究[J]. 浙江农业学报, 2014, 26(2): 456-460. |
| [28] |
Dang YF, Xu XY, Shen YB, et al. Transcriptome analysis of the innate immunity-related complement system in spleen tissue of Ctenopharyngodon idella infected with Aeromonas hydrophila[J]. PLoS One, 2016, 11(7): e0157413. DOI:10.1371/journal.pone.0157413 |
| [29] |
Tatusov RL, Fedorova ND, Jackson JD, et al. The COG database: an updated version includes eukaryotes[J]. BMC Bioinformatics, 2003, 4: 41. DOI:10.1186/1471-2105-4-41 |
| [30] |
Wagner GP, Kin K, Lynch VJ. Measurement of mRNA abundance using RNA-Seq data: RPKM measure is inconsistent among samples[J]. Theory in Biosciences, 2012, 131(4): 281-285. DOI:10.1007/s12064-012-0162-3 |
| [31] |
Anders S, Huber W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10): R106. DOI:10.1186/gb-2010-11-10-r106 |
| [32] |
Bullard JH, Purdom E, Hansen KD, et al. Evaluation of statistical methods for normalization and differential expression in mRNA-Seq experiments[J]. BMC Bioinformatics, 2010, 11: 94. DOI:10.1186/1471-2105-11-94 |
| [33] |
Wang D, Zhu ZL, Wang XQ, et al. Improving mycelium-bound lipase production by aggregating Rhizopus chinensis on a draft tube in a modified stirred tank fermentor[J]. Process Biochemistry, 2015, 50(12): 2019-2028. DOI:10.1016/j.procbio.2015.10.004 |
| [34] |
Chen G, Li RY, Shi LM, et al. Revealing the missing expressed genes beyond the human reference genome by RNA-Seq[J]. BMC Genomics, 2011, 12: 590. DOI:10.1186/1471-2164-12-590 |
| [35] |
Cherepneva GN, Schmidt KH, Kulaeva ON, et al. Expression of the ribosomal proteins S14, S16, L13a and L30 is regulated by cytokinin and abscisic acid: implication of the involvement of phytohormones in translational processes[J]. Plant Science, 2003, 165(5): 925-932. DOI:10.1016/S0168-9452(03)00204-8 |
| [36] |
Ramakrishnan V, White SW. Ribosomal protein structures: insights into the architecture, machinery and evolution of the ribosome[J]. Trends in Biochemical Sciences, 1998, 23(6): 208-212. DOI:10.1016/S0968-0004(98)01214-6 |
| [37] |
Tarze A, Deniaud A, Le Bras M, et al. GAPDH, a novel regulator of the pro-apoptotic mitochondrial membrane permeabilization[J]. Oncogene, 2007, 26(18): 2606-2620. DOI:10.1038/sj.onc.1210074 |
| [38] |
Sámi L, Pusztahelyi T, Emri T, et al. Autolysis and aging of Penicillium chrysogenum cultures under carbon starvation: chitinase production and antifungal effect of allosamidin[J]. The Journal of General and Applied Microbiology, 2001, 47(4): 201-211. DOI:10.2323/jgam.47.201 |
| [39] |
Gunning PW, Ghoshdastider U, Whitaker S, et al. The evolution of compositionally and functionally distinct actin filaments[J]. Journal of Cell Science, 2015, 128(11): 2009-2019. DOI:10.1242/jcs.165563 |
| [40] |
Neves SR, Ram PT, Iyengar R. G protein pathways[J]. Science, 2002, 296(5573): 1636-1639. DOI:10.1126/science.1071550 |
| [41] |
Wu R. The analysis of Rhizopus chinensis genome sequence and its preliminary application[D]. Wuxi: Master's Thesis of Jiangnan University, 2013 (in Chinese) 吴荣. 华根霉全基因组序列分析及其初步应用[D]. 无锡: 江南大学硕士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10295-1013309501.htm |
| [42] |
Hama S, Tamalampudi S, Fukumizu T, et al. Lipase localization in Rhizopus oryzae cells immobilized within biomass support particles for use as whole-cell biocatalysts in biodiesel-fuel production[J]. Journal of Bioscience and Bioengineering, 2006, 101(4): 328-333. DOI:10.1263/jbb.101.328 |
 2018, Vol. 45
2018, Vol. 45




