扩展功能
文章信息
- 梁沪莲, 郭小雅, 刘洋, 刘传丽, 宋志文, 徐爱玲
- LIANG Hu-Lian, GUO Xiao-Ya, LIU Yang, LIU Chuan-Li, SONG Zhi-Wen, XU Ai-Ling
- 基于高通量测序的4种硝化细菌富集培养物微生物群落结构分析
- Microbial community of four nitrobacteria enrichments by high-throughput sequencing
- 微生物学通报, 2017, 44(9): 2112-2119
- Microbiology China, 2017, 44(9): 2112-2119
- DOI: 10.13344/j.microbiol.china.160961
-
文章历史
- 收稿日期: 2016-12-28
- 接受日期: 2017-05-04
- 优先数字出版日期(www.cnki.net): 2017-05-09
2. 山东省胶东调水局 山东 济南 250100
2. Jiaodong Water Transfer Bureau of Shandong Province, Jinan, Shandong 250100, China
硝化细菌是一类广泛存在于自然界且在氮循环过程中起到重要作用的微生物,包括氨氧化菌和亚硝酸盐氧化菌两个生理菌群[1-2],二者能通过硝化作用把氨转化为亚硝酸盐,再进一步转化为硝酸盐。硝化细菌具有好氧、附着生长、代时长、生长繁殖速度慢、对环境因素敏感等特点[3-4],导致其在污水处理系统和水产养殖系统中含量较低,从而直接影响系统的硝化效果和脱氮效率。由于硝化细菌浓度与硝化速率成正比[5],因此建立高效硝化细菌富集培养方法,在污水处理、水产养殖和水体富营养化治理等领域具有重要的意义。
传统的硝化细菌生态学研究主要是通过富集、纯化培养,借助显微镜观察,根据细胞形态构造和生理生化特点来进行,使得对硝化细菌生态学的研究基本停留在总体计数水平,很难为其可靠的种群结构研究和系统发育分析提供证据,并且往往不能正确反映自然环境样品的真实情况[6-7]。随着现代分子生物学技术的发展,一些学者采用高通量测序方法[8-9],通过控制温度、pH、盐度和载体等条件富集培养硝化细菌,并对微生物群落结构进行分析,可最大限度地保留原有微生物群落结构组成和分布特征。
不同应用领域对硝化细菌富集培养物的要求不同,如在污水处理领域主要考虑去除氨氮,水产养殖领域则要重点解决去除亚硝酸盐的问题。另外,由于硝化细菌对环境条件敏感,在淡水、常温条件下富集得到的培养物通常不适于低温、海水等环境。本研究利用前期建立的一种硝化细菌富集培养方法[10-11],分别制备以铵盐为氮源的淡水富集物A、以亚硝酸盐为氮源的淡水富集物B、以铵盐为氮源的低温淡水富集物C和以亚硝酸盐为氮源的海水富集物D,这些富集培养物在常温、低温、高盐等环境条件下表现出较高的氨氧化或亚硝酸盐氧化活性,同时采用高通量测序方法比较分析4种富集培养物的微生物群落结构组成。
1 材料与方法 1.1 主要试剂和仪器土壤DNA提取试剂盒,美国Omega Bio-Tek公司。台式离心机,德国Sigma公司;超微量分光光度计,德国Implen公司。
1.2 硝化细菌富集培养方法硝化细菌的富集培养方法参考钱生财等[10-11],污泥来源及培养条件见表 1。
| 样品 Sample |
污泥来源 Sludge source |
能源物质 Energy substance |
温度 Temperature (℃) |
盐度 Salinity (%) |
周期 Period (d) |
| 富集培养物A Enrichment culture A |
夏季污水处理厂 曝气池污泥 |
NH4Cl 初始浓度200 mg/L |
28 | 0.3 | 20 |
| 富集培养物B Enrichment culture B |
夏季污水处理厂 曝气池污泥 |
NaNO2 初始浓度200 mg/L |
28 | 0.3 | 20 |
| 富集培养物C Enrichment culture C |
冬季污水处理厂 曝气池污泥 |
NH4Cl 初始浓度200 mg/L |
15 | 0.3 | 40 |
| 富集培养物 D Enrichment culture D |
海岸泥沙 | NaNO2 初始浓度200 mg/L |
28 | 3.0 | 40 |
分别取4种硝化细菌富集培养物15 mL,用10%盐酸预处理后置于100 mL离心管13 000×g离心20 min,弃去上清液收集沉淀于2 mL离心管中,采用上述土壤DNA提取试剂盒提取DNA,提取得到的DNA在-20 ℃冻存备用。DNA浓度和纯度通过超微量分光光度计测定。
1.4 高通量测序委托上海派森诺生物科技股份有限公司,利用Illumina MiSeq平台对16S rRNA基因V4区进行高通量测序,采用滑动窗口法对双端的FASTQ序列做质量过滤,利用软件FLASH (version 1.2.7,http://ccb.jhu.edu/software/FLASH/)[12]对通过质量过滤的序列进行连接,对连接上的序列进行过滤和去除嵌合体,得到优质序列。基于OTU聚类和注释的分析结果,绘制稀释曲线,进行多样性指数分析,并在各分类水平上进行群落结构的统计分析和物种丰度差异分析。
2 结果与分析 2.1 样品取样验证及多样性分析在原核生物分子生态学研究中,基于16S rRNA基因的菌群多样性分析会引起一定程度的高估,通常用来进行焦磷酸测序区域中的V4-V5区域显示了最低的高估程度(约为3.0%),而V6区域的高估程度最高(约13%)[13-14]。本研究4个样品细菌16S rRNA基因的V4区域更适合作为焦磷酸测序的目的片段,也是细菌多样性研究中常用的区域。通过测序总计测得原始序列155 250条,经去杂分析得到优质序列147 851条。在97%相似度下将其聚类为用于物种分类的OTU,共产生2 634个OTU,各样品测序信息结果见表 2。采用Alpha多样性指标中的香农指数(Shannon index)和Chao1指数估算样品群落的异质性和物种总数[15]。由表 2可以看出,不同培养物Simpson、Shannon差别较小,但培养物C的OTU数、Chao1、ACE指数明显高于其它3个样品,具有较高的微生物多样性和总体丰度,说明低温条件有助于保护微生物多样性,与邹雨璇等[16]的研究结果一致。
| 样品 Sample |
原始序列数 Original sequence number |
优质序列数 High quality sequence number |
OTUs | Chao1 | ACE | Simpson | Shannon |
| A | 37 038 | 33 372 | 965 | 344.0 | 371.76 | 0.93 | 5.36 |
| B | 47 127 | 45 555 | 929 | 321.0 | 411.53 | 0.96 | 5.44 |
| C | 30 318 | 29 630 | 1 194 | 466.0 | 513.34 | 0.94 | 5.83 |
| D | 40 767 | 39 294 | 773 | 309.0 | 352.81 | 0.95 | 5.43 |
4个样品中的细菌总共分布在24个门,具体情况见图 1。变形菌门(Proteobacteria)占绝对优势,占比分别为73.64%、55.06%、60.89%和74.71%,与顾锦钊[17]对海洋低温硝化细菌富集培养物的研究结果一致;样品B和C中的拟杆菌门(Bacteroidetes)和异常球菌-栖热菌门(Deinococcus-Thermus),样品A中的放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes),以及样品D中的厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)也占一定优势;其它占比较大的门还包括绿弯菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)、芽单胞菌门(Gemmatimonadetes)、浮霉菌门(Planctomycetes)等。在24个门中,14个门为4个样品所共有,Ignavibacteriae、厚壁菌门(Firmicutes)和Caldithrix为样品B所特有,热脱硫杆菌门(Thermodesulfobacteria)和黏胶球形菌门(Lentisphaerae)为样品C所特有,样品A、D没有特有门。
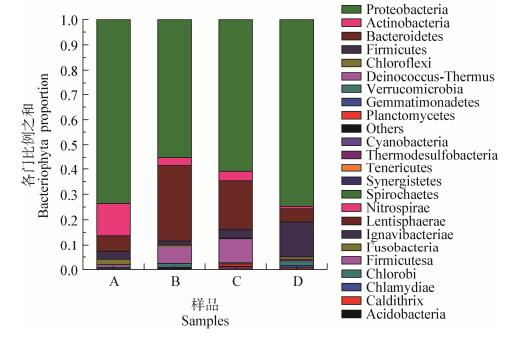
|
| 图 1 4种硝化细菌富集培养物的细菌门水平组成 Figure 1 The composition of 4 kinds of nitrobacteria enrichments at phyla level |
|
|
4个样品中细菌总共分布在47个纲,具体情况见图 2。由图 2可以看出,样品A、B的优势菌纲均为β-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)和α-变形菌纲(Alphaproteobacteria);样品C的优势菌纲为β-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)和黄杆菌纲(Flavobacteriia);样品D的优势菌纲为γ-变形菌纲(Gammaproteobacteria)、δ-变形菌纲(Deltaproteobacteria)和芽孢杆菌纲(Bacilli)。样品D与其它样品差别较大的原因可能与其取自海洋环境样品有关。4个样品中其它占比较大的有放线菌纲(Actinobacteria)、热微菌纲(Thermomicrobia)、梭菌纲(Clostridia)、异常球菌纲(Deinococci)等。在47个纲中,有24个纲为4个样品所共有,全噬菌纲(Holophagae)为样品A所特有,球杆菌亚纲(Sphaerobacteridae)、Dehalococcoidetes和Caldithrixae为样品B所特有,热脱硫杆菌纲(Thermodesulfobacteria)、集球藻亚纲(Synechococcophycideae)、颤藻亚纲(Oscillatoriophycideae)、纤线杆菌纲(Ktedonobacteria)、厚壁菌纲(Firmicutes)、产芽孢菌纲(Erysipelotrichi)、红蝽菌纲(Coriobacteriia)、绿弯菌纲(Chloroflexi)为样品C所特有,样品D没有特有纲类。
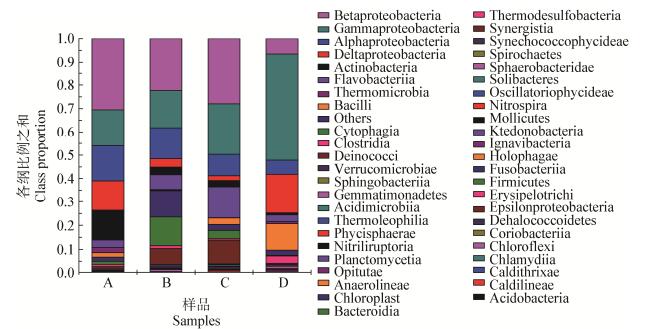
|
| 图 2 4种硝化细菌富集培养物的细菌纲水平组成 Figure 2 The composition of 4 kinds of nitrobacteria enrichments at classes level |
|
|
4个样品中细菌总共分布在129个属,不同样品优势菌属均不同,其中样品A为亚硝化单胞菌属(Nitrosomonas,24.56%)、Halobacteriovorax litoralis (7.97%)和大洋单胞菌属(Oceanimonas doudoroffii,4.47%);样品B为链霉菌属(Streptomyces lushanensis,7.15%)、新疆盐地杆菌属(Salinibacterium xinjiangense,6.67%)和Tistrella bauzanensis (6.34%);样品C为噬菌弧菌属(Bacteriovorax stolpii,19.36%)、类诺卡氏菌属(Nocardioides daecheongensis,19.35%)和Polycyclovorans algicola TG408 (8.26%);样品D为嗜酸菌属(Acidovorax anthurii,13.6%)、柄杆菌属(Caulobacter segnis,11.5%)和蛭弧菌属(Bdellovibrio bacteriovorus str. Tiberius,6.03%)。
2.3 样品中功能菌群分析4个样品中共检测出7种具有硝化功能的类群,样品A、B、C、D分别含5、3、4、4种,具体情况见表 3。其中亚硝化单胞菌属菌株N. eutropha C91为4个样品所共有;Nitrospirillum amazonense为3个样品所共有;N. europaea ATCC 19718为2个样品所共有;硝化螺旋菌属菌株N. japonica为样品C所特有。此外,样品A、C和D中还检测到蜡样芽胞杆菌(Bacillus cereus ATCC 14579),占比分别为0.21%、0.18%和0.44%;样品C中检测到红育菌属(Rhodoferax antarcticus ANT. BR),占比为1.39%,有研究显示它们均可以通过同化作用利用氨[18-21]。
| 硝化细菌 Nitrifying bacteria |
属名 Genus |
百分比 Percentage (%) |
|||
| A | B | C | D | ||
| Nitrosococcus watsonii | 亚硝化球菌属 | - | - | 0.55 | 0.38 |
| Nitrosomonas europaea ATCC 19718 | 亚硝化单胞菌属 | 0.65 | - | - | 0.16 |
| Nitrosomonas eutropha C91 | 2.48 | 4.64 | 0.26 | 0.47 | |
| Nitrosomonas halophila | 1.24 | 0.18 | - | - | |
| Nitrosomonas sp. AL212 | 20.19 | 0.12 | - | - | |
| Nitrospirillum amazonense | Nitrospirillum | 0.18 | - | 0.69 | 0.42 |
| Nitrospira japonica | 硝化螺旋菌属 | - | - | 0.69 | - |
| 总计Total | 24.74 | 4.94 | 2.19 | 1.43 | |
| 注:-:样品中细菌占比为0. Note: -: the proportion of bacteria in the samples was zero. |
|||||
4个样品中还检测出其它与环境物质循环相关或具有特殊生理特性的菌群,如样品D中的红灯食烷菌(Alcanivorax hongdengensis A-11-3) 甲基菌属(Methylophaga nitratireducenticrescens)等为石油等降解菌或潜在降解菌,羽扇豆根瘤菌(Bradyrhizobium lupini)具有固氮作用,特吕珀菌属(Truepera radiovictrix DSM 17093) 具有较强耐辐射能力等[22]。
样品中也检测出部分致病菌或条件致病菌,比如:弧菌属(Vibrio)某些菌株可能导致霍乱样疾病或轻度腹泻;伯克霍尔德菌属(Burkholderia)某些菌株可能导致惠特莫尔氏病;费克蓝姆菌属(Facklamia)菌株可引起抵抗力低下宿主的机会感染。
3 结论与讨论硝化细菌包括氨氧化菌和亚硝酸盐氧化菌两个生理菌群,尽管二者生理特性相似,但系统发育具有高度的多样性,其中氨氧化菌分为两个不同的系统发育组:一组为氨氧化菌主要成员,属于β-变形菌亚纲(β-Proteobacteria),包括亚硝化单胞菌属(Nitrosomonas)、亚硝化叶菌属(Nitrosolobus)、亚硝化螺菌属(Nitrosospira)和亚硝化弧菌属(Nitrosovibrio);另外一组为亚硝化球菌属(Nitrosococcus),属γ-变形菌亚纲着色菌目(Chromatiales)着色菌科(Chromatiaceae)。亚硝酸盐氧化菌则由进化上截然不同的四类菌群构成:(1) α-变形菌亚纲(α-Proteobacteria)的根瘤菌目(Rhizobiales)慢生根瘤菌科(Bradyrhizobiaceae)硝酸菌属(Nitrobacter);(2) γ-变形菌亚纲(γ-Proteobacteria)的着色菌目(Chromatiales)外硫红螺旋菌科(Ectothiorhodospiraceae)硝化球菌属(Nitrococcus);(3) δ-变形菌亚纲(δ-Proteobacteria)的脱硫杆菌目(Desulfobacterales)脱硫盒菌科(Desulfoarculaceae)硝化刺菌属(Nitrospina);(4) 硝化螺旋菌门(Nitrospira)硝化螺旋菌纲(Nitrospira)硝化螺旋菌科(Nitrospiraceae)硝化螺旋菌属(Nitrospira)[23-24]。
在本研究中,由于不同富集培养物的污泥来源及富集条件不同,所以其微生物多样性组成存在差异。不同培养物优势菌门均为变形菌门,样品A、B、C的优势菌纲为β-变形菌纲和γ-变形菌纲,样品D优势菌纲为γ-变形菌纲和δ-变形菌纲,在属水平上差异较大,主要细菌类群分别为亚硝化单胞菌属、链霉菌属、噬菌弧菌属和嗜酸菌属。硝化细菌对光照、温度、盐度等环境因子敏感,多数硝化细菌的生长温度在15-35 ℃之间,当温度降至15 ℃以下时,硝化细菌的生长和硝化速率均会显著降低;不同硝化细菌种类对盐度需求也存在较大差异,可分为需盐种、非需盐种和嗜盐种,导致其在不同生态系统中分布不同[25-27]。海洋环境盐度高,硝化菌群贫乏[16],使得样品D经过40 d的富集培养,具有硝化功能的类群占比仅为1.43%。Yao等[28]通过逐级增加溶解氧浓度,富集低温异氧硝化-好氧反硝化细菌,分析表明优势菌纲为变形菌纲,与本研究结果一致;但是在富集培养过程中细菌群落多样性明显降低,其中α-变形菌纲、δ-变形菌纲和拟杆菌门占比减少,而γ-变形菌纲中的假单胞菌属和β-变形菌纲中的红育菌属成为优势菌属,与本研究结果不同。分析原因可能是他们富集的是异养硝化菌群,在富集过程中加入了有机碳源,而本研究富集的是自养硝化细菌,培养过程中无有机碳源加入。
本研究样品中起主要作用的亚硝酸盐氧化菌(NOB)为硝化螺旋菌属(Nitrospira)和Nitrospirillum,前者在污水处理系统和反应器中经常被检出。样品A中有硝化功能菌群的比例最高,占比24.74%,且主要为亚硝化单胞菌属细菌,与其富集培养过程中的能源物质及适宜的温度、盐度和pH条件有关。Yao等[29]利用活性污泥,通过昼夜交替间歇运行、连续进氨方式富集培养硝化细菌,经过25 d富集培养,硝化细菌群落多样性得到提高,亚硝化单胞菌属(Nitrosomonas)和硝化螺旋菌属(Nitrospira)占比也从2.94%提高到了60.66%。Young等[30]研究硝化MBBR反应器不同氨负荷下细菌群落结构,表明亚硝化单胞菌属占优势地位。侯爱月等[31]利用PCR-DGGE分析好氧颗粒污泥中微生物群落结构,表明氨氧化菌(AOB)均为β-变形菌纲的亚硝化单胞菌属(Nitrosomonas)。在本研究中,N. eutropha C91为非需盐种,但可耐受3% NaCl[32-33],在4个样品中均被检测到;Nitrosomonas sp. AL212[34]常见于淡水环境,在样品A中占比达20.19%。亚硝化球菌属菌株N. watsonii是一种新型海洋厌氧氨氧化菌,为需盐种[35-36],在样品C和D中被检测到。此外,本研究还检测到具有脱氮作用的异养菌,如对温度和盐度等具有较强适应能力的芽孢杆菌属[37],在样品A、C、D中均被检测到;红育菌属(R. antarcticus ANT. BR)属耐寒物种,低温有利于其生长[38],在样品C中被检测到,与Yao等[28]研究结果一致。样品中检测到的红灯食烷菌是海洋环境中重要的烷烃降解菌,Alcanivorax hongdengensis A-11-3分离自马六甲海峡表层海水,是一种石油降解新种[39];胚芽乳酸杆菌(Lactococcus plantarum)[40]可分解养殖塘底的有机物;Aequorivita sublithincola则是潜在的有机物降解菌。
本研究仅针对4种富集培养物中细菌多样性进行了分析,近年来随着对氨氧化古菌(AOA)研究的深入,发现其广泛存在于土壤、自然水体、污水处理厂、垃圾渗滤液等环境中,甚至在某些环境中占主导地位[41-43]。因此,对于4种培养物富集过程中微生物多样性的变化,特别是古菌的群落结构变化将是我们后续研究的重点。
综上所述,本研究富集了4种不同条件下的培养物,采用Illumina MiSeq高通量测序方法比较分析了4种硝化细菌富集培养物微生物群落结构及多样性差异,确定了在不同培养物中起主要作用的硝化细菌类群以及部分有益菌和致病菌类群,研究结果为硝化细菌富集培养物的实际应用奠定了基础。
| [1] |
Qu JN, Jin ZG, He QB, et al. Research on enrichment and cultivation technology of high-efficient nitrifying bacteria[J]. Journal of Tongji University, 1996, 27(3): 351-354. (in Chinese) 屈计宁, 金志刚, 何群彪, 等. 高效硝化细菌的富集技术研究[J]. 同济大学学报, 1996, 27(3): 351-354. |
| [2] |
Ma Y, Qian LM, Wang YS, et al. Progress in molecular ecology of nitrifying bacteria[J]. Journal of Fishery Sciences of China, 2007, 14(5): 872-879. (in Chinese) 马英, 钱鲁闽, 王永胜, 等. 硝化细菌分子生态学研究进展[J]. 中国水产科学, 2007, 14(5): 872-879. |
| [3] |
Song ZW, Xu M, Wen SP, et al. Effect of nitrobacteria preparation on water purification of freshwater aquarium[J]. Hebei Fisheries, 2007(11): 29-31, 60. (in Chinese) 宋志文, 徐敏, 温少鹏, 等. 硝化细菌制剂对淡水水族箱水质的净化效果[J]. 河北渔业, 2007(11): 29-31, 60. DOI:10.3969/j.issn.1004-6755.2007.11.008 |
| [4] |
Luo XX, Gao JZ, Chen ZZ. The research and application of nitrifying bacteria enrichment culture[J]. Fisheries Science & Technology Information, 2013, 40(6): 320-323. (in Chinese) 罗小溪, 高建忠, 陈再忠. 硝化细菌富集培养及应用研究进展[J]. 水产科技情报, 2013, 40(6): 320-323. |
| [5] |
Wang XJ, He CP, Wang Z, et al. Screening and characteristics of an efficient nitrobacterium[J]. China Environmental Science, 2013, 33(2): 286-292. (in Chinese) 王小菊, 何春平, 王震, 等. 高效硝化细菌的筛选及特性研究[J]. 中国环境科学, 2013, 33(2): 286-292. |
| [6] |
Cho KH, Kim JO, Kang S, et al. Achieving enhanced nitrification in communities of nitrifying bacteria in full-scale wastewater treatment plants via optimal temperature and pH[J]. Separation and Purification Technology, 2014, 132: 697-703. DOI:10.1016/j.seppur.2014.06.027 |
| [7] |
Peng L, Ni BJ, Erler D, et al. The effect of dissolved oxygen on N2O production by ammonia-oxidizing bacteria in an enriched nitrifying sludge[J]. Water Research, 2014, 66: 12-21. DOI:10.1016/j.watres.2014.08.009 |
| [8] |
Zhang ZD, Gu MY, Wang W, et al. Analysis of bacterial community in radiation polluted soils by high-throughput sequencing[J]. Microbiology China, 2016, 43(6): 1218-1226. (in Chinese) 张志东, 顾美英, 王玮, 等. 基于高通量测序的辐射污染区细菌群落特征分析[J]. 微生物学通报, 2016, 43(6): 1218-1226. |
| [9] |
Qin N, Li DF, Yang RF. Next-generation sequencing technologies and the application in microbiology--a review[J]. Acta Microbiologica Sinica, 2011, 51(4): 445-457. (in Chinese) 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报, 2011, 51(4): 445-457. |
| [10] |
Qian SC, Xu M, Wen SP, et al. The influence of different carriers on nitrifying bacteria enrichment process[J]. Hebei Fisheries, 2013(2): 1-4, 16. (in Chinese) 钱生财, 徐敏, 温少鹏, 等. 载体对硝化细菌富集培养过程的影响[J]. 河北渔业, 2013(2): 1-4, 16. |
| [11] |
Wang W, Tan XY, Sun XF, et al. A study on marine nitrifying bacteria enrichment cultivation process[J]. Journal of Qingdao Technological University, 2006, 27(3): 67-70. (in Chinese) 王玮, 谭潇也, 孙贤风, 等. 海洋硝化细菌富集培养过程研究[J]. 青岛理工大学学报, 2006, 27(3): 67-70. |
| [12] |
Magoč T, Salzberg SL. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [13] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [14] |
Jin L, Gao XM, Du JH, et al. Peat bacterial diversity and community structure in Gahai Lake wetlandin Gan'nan[J]. Microbiology China, 2016, 43(11): 2396-2404. (in Chinese) 靳亮, 高学梅, 杜建华, 等. 尕海湖湿地泥炭细菌多样性分析[J]. 微生物学通报, 2016, 43(11): 2396-2404. |
| [15] |
Amato KR, Yeoman CJ, Kent A, et al. Habitat degradation impacts black howler monkey (Alouatta pigra) gastrointestinal microbiomes[J]. The ISME Journal, 2013, 7(7): 1344-1353. DOI:10.1038/ismej.2013.16 |
| [16] |
Zou YX. Enhance ammonia removal effect in constructed wetland at low temperature[D]. Changchun: Master's Thesis of Jilin University, 2014(in Chinese) 邹雨璇. 低温条件下人工湿地氨氮强化脱氮机理的研究[D]. 长春: 吉林大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/article/cdmd-10183-1014281527.htm |
| [17] |
Gu JZ. Study on enrichment process and influence factors analysis of marine low temperatuer nitrifying bacteria[D]. Qingdao: Master's Thesis of Qingdao University of Technology, 2014(in Chinese) 顾锦钊. 海洋低温硝化细菌的富集培养过程及影响因素分析[D]. 青岛: 青岛理工大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/article/cdmd-10429-1015536559.htm |
| [18] |
Zhou YQ, Xin X, Yao L, et al. Isolation and identification of a heterotrophic nitrification-aerobic denitrification bateria with high efficiency and its denitrification characteristics[J]. Chinese Journal of Environmental Engineering, 2013, 7(10): 4127-4132. (in Chinese) 周迎芹, 信欣, 姚力, 等. 一株高效异养硝化-好氧反硝化菌的分离鉴定及脱氮性能[J]. 环境工程学报, 2013, 7(10): 4127-4132. |
| [19] |
Yang L, Ren YX, Liang X, et al. Nitrogen removal characteristics of a heterotrophic nitrifier Acinetobacter junii YB and its potential application for the treatment of high-strength nitrogenous wastewater[J]. Bioresource Technology, 2015, 193: 227-233. DOI:10.1016/j.biortech.2015.05.075 |
| [20] |
Mols M, de Been M, Zwietering MH, et al. Metabolic capacity of Bacillus cereus strains ATCC 14579 and ATCC 10987 interlinked with comparative genomics[J]. Environmental Microbiology, 2007, 9(12): 2933-2944. DOI:10.1111/emi.2007.9.issue-12 |
| [21] |
Madigan MT, Jung DO, Woese CR, et al. Rhodoferax antarcticus sp. nov., a moderately psychrophilic purple nonsulfur bacterium isolated from an Antarctic microbial ma[J]. Archives of Microbiology, 2000, 173(4): 269-277. DOI:10.1007/s002030000140 |
| [22] |
Katipoglu-Yazan T, Merlin C, Pons MN, et al. Chronic impact of sulfamethoxazole on the metabolic activity and composition of enriched nitrifying microbial culture[J]. Water Research, 2016, 100: 546-555. DOI:10.1016/j.watres.2016.05.043 |
| [23] |
Garrity GM, Bell JA, Lilburn TG, et al. Bergey's Manual of Systematic Bacteriology[M]. 2nd Edition. USA: Springer-Verlag, 2005, 137-153.
|
| [24] |
Liu ZP, Liu SJ. Advances in the molecular biology of nitrifying microorganisms[J]. Chinese Journal of Applied & Environmental Biology, 2004, 10(4): 521-525. (in Chinese) 刘志培, 刘双江. 硝化作用微生物的分子生物学研究进展[J]. 应用与环境生物学报, 2004, 10(4): 521-525. |
| [25] |
Kuhn DD, Drahos DD, Marsh L, et al. Evaluation of nitrifying bacteria product to improve nitrification efficacy in recirculating aquaculture systems[J]. Aquacultural Engineering, 2010, 43(2): 78-82. DOI:10.1016/j.aquaeng.2010.07.001 |
| [26] |
Ward BB, Arp DJ, Klotz MG. Nitrification[M]. Washington, DC, USA: ASM Press, 2011.
|
| [27] |
John RC, Okpokwasili GC. Crude oil-degradation and plasmid profile of nitrifying bacteria isolated from oil-impacted mangrove sediment in the Niger Delta of Nigeria[J]. Bulletin of Environmental Contamination and Toxicology, 2012, 88(6): 1020-1026. DOI:10.1007/s00128-012-0609-8 |
| [28] |
Yao S, Ni JR, Chen Q, et al. Enrichment and characterization of a bacteria consortium capable of heterotrophic nitrification and aerobic denitrification at low temperature[J]. Bioresource Technology, 2013, 127: 151-157. DOI:10.1016/j.biortech.2012.09.098 |
| [29] |
Yao RD, Yang H, Yu MY, et al. Enrichment of nitrifying bacteria and microbial diversity analysis by high-throughput sequencing[J]. RSC Advances, 2016, 3(15): 113959-113966. |
| [30] |
Young B, Delatolla R, Kennedy K, et al. Low temperature MBBR nitrification: microbiome analysise[J]. Water Research, 2017, 111: 224-233. DOI:10.1016/j.watres.2016.12.050 |
| [31] |
Hou AY, Li J, Wang CW, et al. Characteristics of microbial community structure in different aerobic granular sludge[J]. China Environmental Science, 2016, 36(4): 1136-1144. (in Chinese) 侯爱月, 李军, 王昌稳, 等. 不同好氧颗粒污泥中微生物群落结构特点[J]. 中国环境科学, 2016, 36(4): 1136-1144. |
| [32] |
Kjeldal H, Pell L, Pommerening-Röser A, et al. Influence of p-cresol on the proteome of the autotrophic nitrifying bacterium Nitrosomonas eutropha C91[J]. Archives of Microbiology, 2014, 196(7): 497-511. DOI:10.1007/s00203-014-0985-z |
| [33] |
Stein LY, Arp DJ, Berube PM, et al. Whole-genome analysis of the ammonia-oxidizing bacterium, Nitrosomonas eutropha C91: implications for niche adaptation[J]. Environmental Microbiology, 2007, 9(12): 2993-3007. DOI:10.1111/emi.2007.9.issue-12 |
| [34] |
Yuichi S, Norton JM, Bollmann A, et al. Genome sequence of Nitrosomonas sp. strain AL212, an ammonia-oxidizing bacterium sensitive to high levels of ammonia[J]. Journal of Bacteriology, 2011, 193(18): 5047-5048. DOI:10.1128/JB.05521-11 |
| [35] |
Fečkaninová A, Koščová J, Mudroňová D, et al. The use of probiotic bacteria against Aeromonas infections in salmonid aquaculture[J]. Aquaculture, 2017, 469: 1-8. DOI:10.1016/j.aquaculture.2016.11.042 |
| [36] |
Campbell MA, Chain PSG, Dang HY, et al. Nitrosococcus watsonii sp. nov., a new species of marine obligate ammonia-oxidizing bacteria that is not omnipresent in the world's oceans: calls to validate the names 'Nitrosococcus halophilus' and 'Nitrosomonas mobilis'[J]. Microbiology Ecology, 2011, 76(1): 39-48. DOI:10.1111/fem.2011.76.issue-1 |
| [37] |
Huang X, Bai J, Li KR, et al. Characteristics of two novel cold-and salt-tolerant ammonia-oxidizing bacteria from Liaohe Estuarine Wetland[J]. Marine Pollution Bulletin, 2017, 114(1): 192-200. DOI:10.1016/j.marpolbul.2016.08.077 |
| [38] |
Song ZW, Li T, Wang QX, et al. Influence of microbial community structure of seed sludge on the properties of aerobic nitrifying granules[J]. Journal of Environmental Sciences, 2015, 35: 144-150. DOI:10.1016/j.jes.2015.01.033 |
| [39] |
Wang WP, Shao ZZ. Genes involved in alkane degradation in the Alcanivorax hongdengensis strain A-11-3[J]. Applied Microbiology and Biotechnology, 2012, 94(2): 437-448. DOI:10.1007/s00253-011-3818-x |
| [40] |
Beck BR, Kim D, Jeon J, et al. The effects of combined dietary probiotics Lactococcus lactis BFE920 and Lactobacillus plantarum FGL0001 on innate immunity and disease resistance in olive flounder (Paralichthys olivaceus)[J]. Fish & Shellfish Immunology, 2015, 42(1): 177-183. |
| [41] |
Spang A, Hatzenpichler R, Brochier-Armanet C, et al. Distinct gene set in two different lineages of ammonia-oxidizing archaea supports the phylum Thaumarchaeota[J]. Trends in Microbiology, 2010, 18(8): 331-340. DOI:10.1016/j.tim.2010.06.003 |
| [42] |
Liu ZH, Li DH. Ammonia-oxidizing archaea and their contribution to global nitrogen cycling: a review[J]. Microbiology China, 2015, 42(4): 774-782. (in Chinese) 刘正辉, 李德豪. 氨氧化古菌及其对氮循环贡献的研究进展[J]. 微生物学通报, 2015, 42(4): 774-782. |
| [43] |
Zhou LL, Zhu GB, Wang SY, et al. Abundance, biodiversity and contribution to ammonia oxidization of ammonia-oxidizing archaea in littoral sediments of Dongting Lake[J]. Acta Scientiae Circumstantiae, 2013, 33(6): 1741-1747. (in Chinese) 周磊榴, 祝贵兵, 王衫允, 等. 洞庭湖岸边带沉积物氨氧化古菌的丰度、多样性及对氨氧化的贡献[J]. 环境科学学报, 2013, 33(6): 1741-1747. |
 2017, Vol. 44
2017, Vol. 44




